2. 兰州理工大学土木工程学院, 兰州 730050;
3. 中国科学院生态环境研究中心水污染控制实验室, 北京 100085;
4. 中国科学院大学, 北京 100049
2. School of Civil Engineering, Lanzhou University of Technology, Lanzhou 730050, China;
3. Department of Water Pollution Control, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China;
4. University of Chinese Academy of Sciences, Beijing 100049, China
污水处理厂是抗生素抗性基因(antibiotic resistance genes, ARG)的重要储存库, 也是控制抗性污染传播的研究重点[1~3].然而抗生素抗性基因在污水处理过程中变化的影响机制非常复杂, 目前仍处于黑箱状态[4].已有研究表明, 污水处理过程中水质特征对菌群结构有重要影响[5], 并且菌群结构[6, 7]和水质特征[3, 8]对ARG的丰度变化均有重要影响, 因此有必要对污水处理过程中三者之间的关系进行深入分析.抗生素制药废水是处理难度较大的工业废水之一, 具有污染物浓度高、成分复杂、生物毒性大和难降解等特点[9], 常含有高浓度COD、SO42-、NH4+-N、残余抗生素以及高丰度的ARG和耐药菌[6, 10], 也是向下游受纳水体传播ARG的重要源头.抗生素制药废水中的ARG丰度往往高于城市污水中的ARG丰度, 引起抗性污染传播的潜在风险更高[11~13], 更应引起人们的重视.螺旋霉素是一种广泛使用的大环内酯类抗生素, 具有16元环结构, 常用于耳、鼻、喉和呼吸道感染、尿路感染以及脑膜炎等病症的治疗.目前人们对螺旋霉素制药废水处理过程中的菌群结构变化及ARG分布特征研究较少, 尚有待于进一步地考察与研究.
本课题组前期考察了某实际螺旋霉素制药废水处理厂A2O处理工艺全流程中, 大环内酯类抗性基因ereA、ermB、ermF、mefA、mphB和ermX, 以及可移动遗传元件(mobile genetic elements, MGE)Tn916/1545、intI1和ISCR1的分布特征[14].在此基础上, 本研究重点考察螺旋霉素制药废水各处理单元的菌群结构的演替、水质特征及其对ARG转归的影响, 探讨三者之间的影响关系, 以期为抗生素废水处理过程中抗性污染控制提供科学参考.
1 材料与方法 1.1 样品采集本研究的样品同文献[14], 简述如下:样品取自于江苏省某螺旋霉素生产废水处理站的A2O工艺, 于2014年4月(春季)和2014年9月(秋季)在调节池、厌氧池、缺氧池、好氧池和二沉池取样, 分别标记为S1~S5(春季)和A1~A5(秋季).前期研究已对废水处理过程的水质和ARG分布变化进行了分析与讨论[14], 本研究重点考察菌群结构在废水处理过程中的演替, 探讨其对水质特征和ARG的影响.
1.2 菌群结构分析DNA样品与文献[14]一致, 本研究选择引物515F/806R, 使用巢式PCR对DNA样品16S的V4区进行扩增, 通过Illumina MiSeq进行高通量测序, 将所得序列通过OTUs(operational taxonomic units)以97%的相似度进行聚类, 并使用RDP(ribosomal database project)分类器对样品中的序列进行分类, 从而分析各样品中菌群结构的演变情况.
1.3 数据分析取每个样品丰度排前10的优势菌属, 通过Heml 1.0软件制作菌群结构的热图, 并对其基于欧氏距离的相似度进行聚类分析; 采用Gephi 0.9.1软件, 基于Spearman相关性分析, 对水/泥相中ARG和菌群结构进行网络分析, 考察螺旋霉素制药废水处理过程中二者的相关性; 采用Canoco 5.0(Microcomputer Power, US)软件, 通过主成分分析(principal component analysis, PCA)对各样品微生物群落、ARG和MGE的分布进行聚类, 并通过普鲁克分析(procrustes analysis, PA)考察ARG和菌群结构的关系, 通过冗余分析(redundancy analysis, RDA)考察废水特性对ARG、MGE和菌群结构的影响.
2 结果与讨论 2.1 污水处理过程中水质特征变化前期的研究仅描述了螺旋霉素制药废水在A2O工艺过程中常规理化指标的去除效果[14], 但未对处理过程中理化指标变化规律进行详细探讨.本研究为了考察菌群结构演替与各污水处理单元水质特征的关系, 对理化指标在废水处理过程中的变化进行了进一步分析(图 1).由图 1可知, 螺旋霉素制药废水具有高COD、NH4+-N、NO3--N以及低PO43--P的水质特征, 春季进水COD比秋季进水高800 mg·L-1, 分别为2 280 mg·L-1和1 480 mg·L-1, 但春、秋季进水的NH4+-N浓度相似, 分别为223.52 mg·L-1和218.61 mg·L-1.秋季进水中NO3--N浓度为49.42 mg·L-1, 比春季进水高6.59 mg·L-1.春、秋季污水处理过程中, 由于反硝化作用, NO3--N在厌氧后显著下降, 并在好氧段由于硝化作用显著增加, 导致二沉池内NO3--N甚至高于进水的NO3--N.值得注意的是, 在缺氧阶段后NO3--N均略有升高(增加了1.72~4.99 mg·L-1), 这可能与缺氧池内因好氧池混合液的回流而引入大量的硝酸盐但未能及时进行反硝化作用有关.春、秋两季进水中均未检出NO2--N, 但由于硝化反硝化作用, 秋季出水中NO2--N增加到1.64 mg·L-1.在春季各处理单元样品中均未检出PO43--P, 在秋季进水中检出浓度为1.86 mg·L-1, 出水仅为0.05 mg·L-1.总体而言, A2O工艺对COD和NH4+-N的削减效果较好; 春、秋季COD的削减率分别为94.08%和89.86%, NH4+-N的削减率分别为89.55%和86.90%; 但出水中仍含有高浓度的NO3--N(66.07~68.08 mg·L-1).
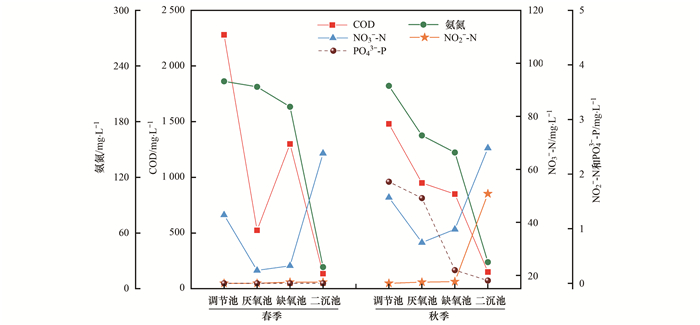
|
图 1 废水处理过程中水质特征的变化 Fig. 1 Variations of wastewater characteristics during wastewater treatment |
通过对春、秋两季螺旋霉素制药废水在生物处理过程中属水平菌群结构进行主成分分析(PCA, 图 2), 发现各处理单元的样品中菌群结构大体可分为3大类:春、秋季的调节池、厌氧池和缺氧池为一类; 春季好氧池、二沉池和秋季好氧池为一类; 秋季二沉池单独为一类.这个结果表明:①虽然春季和秋季进水的COD相差800 mg·L-1, 但并未引起进水(调节池)的微生物菌群结构产生显著差异; ②进水中的菌群结构与厌氧和缺氧阶段的菌群结构相似, 这可能与进水储存于调节池中, 使其处于厌氧或缺氧状态有关; ③春、秋季的好氧池内菌群结构相似, 也未受到进水水质不同的影响, 但均与厌氧、缺氧池的菌群结构有较大差异; ④只有秋季二沉池的菌群结构与其他样品差异较大, 这可能跟秋季二沉池内的NO2--N(1.64 mg·L-1)高于其他工艺单元有关.但污水处理过程中菌群结构的变化受到的影响因素较多, 影响机制也较复杂, 目前仍需进一步探讨影响微生物菌群结构的各种驱动因素.
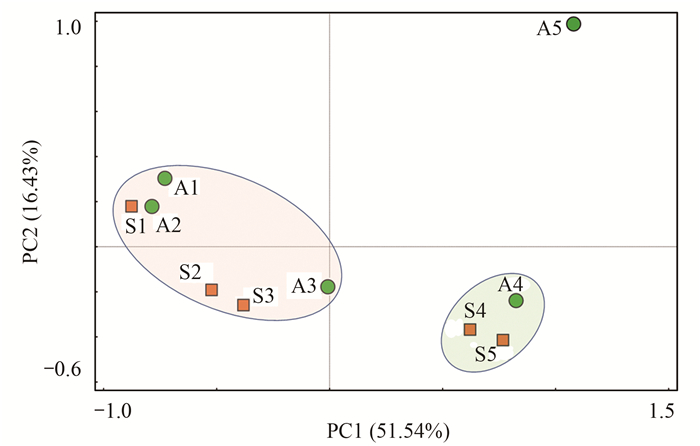
|
S1~S5表示春季调节池、厌氧池、缺氧池、好氧池和二沉池样品; A1~A5表示秋季调节池、厌氧池、缺氧池、好氧池和二沉池样品, 下同 图 2 基于属水平菌群结构的PCA分析 Fig. 2 PCA analysis of microbial community at the genus level |
通过热图进一步分析发现(图 3), 春季调节池中的优势菌属为Desulfomicrobium(14.93%)、Parabacteroides(10.18%)、Sulfurospirillum(7.37%)和Desulfovibrio(5.93%)等, 秋季调节池的优势菌属为Parabacteroides(14.75%)、Proteiniclasticum(9.97%)、Trichococcus(8.22%)和Desulfovibrio(7.57%)等.Desulfomicrobium是一种在SO42-废水厌氧处理单元中经常检出的硫酸盐还原菌(sulfate reducing bacteria, SRB), 可以以乳酸、丙酮酸、甘油、乙酸盐和乙醇为电子供体还原硫酸盐[15]; Desulfovibrio也是一种专性厌氧的SRB, 并可代谢多种碳水化合物, 少数菌株还有生物固氮能力[16]; Parabacteroides是有糖酵解作用的专性厌氧菌[17]; Sulfurospirillum菌属中的大多细菌可在有毒底物条件下生存, 可吸收有机卤代物, 也可以以硝酸盐和硫化合物为电子受体削减污染物, 还可通过降解高氯酸盐促进自身生长[18, 19]; Proteiniclasticum是严格厌氧的蛋白水解细菌, 也常在反硝化系统出现[20]; Trichococcus厌氧时可氧化糖类产酸, 也可利用不同碳源在低温和高盐度条件下生存[21, 22].
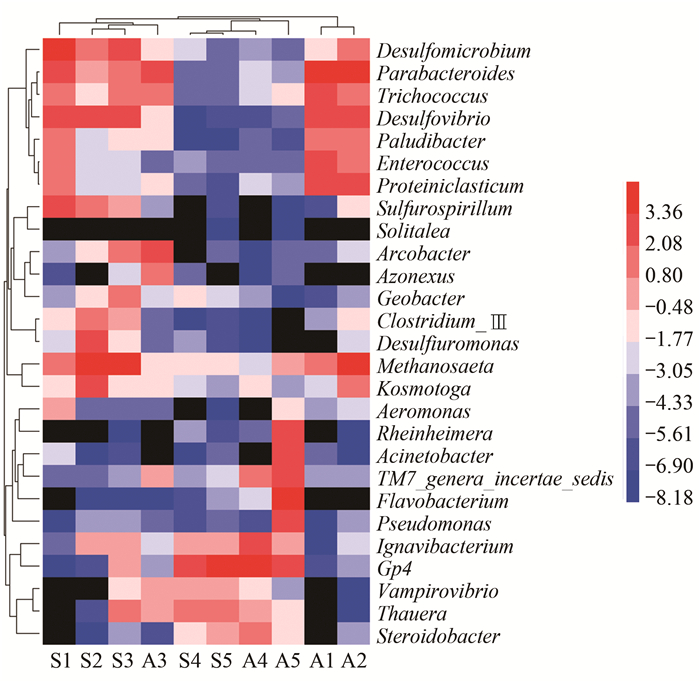
|
Desulfomicrobium为脱硫微菌属, Parabacteroides为副杆状菌属, Trichococcus为毛球菌属, Desulfovibrio为脱硫弧菌属, Paludibacter为沼杆菌属, Enterococcus为肠球菌, Sulfurospirillum为硫磺单胞菌属, Arcobacter为弓形杆菌属, Azonexus为固氮捲菌属, Geobacter为地杆菌属, Clostridium_Ⅲ为梭菌属Ⅲ, Desulfuromonas为脱硫单孢菌属, Methanosaeta为甲烷八叠球菌, Aeromonas为气单胞菌属, Acinetobacter为不动杆菌属, Flavobacterium为黄杆菌属, Pseudomonas为假单胞菌属, Ignavibacterium为懒小杆菌属, Vampirovibrio为吸血弧菌属, Thauera为陶厄氏菌属, Steroidobacter为甾类杆菌属; 每个样品取前10个菌属, 微生物丰度以2为底的对数处理 图 3 属水平菌群结构热图 Fig. 3 Heatmap of the microbial community at the genus level |
春季厌氧池的优势菌属为Methanosaeta(25.06%)、Kosmotoga(6.24%)、Desulfovibrio(5.15%)和Desulfuromonas(4.81%)等; 秋季厌氧池的优势菌属与春季相似, 为Parabacteroides(15.36%)、Methanosaeta(12.43%)、Desulfovibrio(6.48%)和Proteiniclasticum(4.42%)等, 说明厌氧池内由产甲烷菌Methanosaeta, 水解菌如Proteiniclasticum和产酸菌如Parabacteroides主导.Desulfuromonas可通过氧化乙酸盐实现自身生长, 也可降解有机酸[23]; 而Ignavibacterium可利用多种糖和胺产生有机酸, 提高有机污染物的降解效率, Ignavibacterium也是阿莫西林废水、林可霉素废水厌氧处理单元的优势菌[24, 25].虽然厌氧池中Thauera(0%~0.01%)和Arcobacter(0.22%~0.47%)丰度不高, 但这些菌属在厌氧条件下会以NO3-为电子受体进行呼吸作用而生长[26], 属于反硝化功能菌, 对厌氧过程NO3--N的去除起到促进作用.此外, 适量的硝酸盐可使产甲烷和反硝化耦合, 且当COD/NO3--N为121.43~15.18时耦合效果最佳[27], 本研究中厌氧池内COD/NO3--N比值为23.95~29.29, 厌氧阶段NO3--N降低了16.99~20.91 mg·L-1, 由此推断, 本研究厌氧过程中硝氮的削减除了反硝化作用外, 可能还与产甲烷和反硝化耦合作用有关.
春季缺氧池的菌群结构与厌氧池相似, 优势菌仍以产甲烷功能菌和脱硫功能菌为主, 如Methanosaeta(11.38%)、Desulfomicrobium(6.69%)和Desulfovibrio(4.44%)等, 并且缺氧池内兼具降解有机物和反硝化的功能菌Arcobacter、Geobacter和Thauera等菌属丰度升高; 秋季缺氧池优势菌属为反硝化功能菌Arcobacter(5.60%)和Azonexus(2.86%)以及发酵功能菌Parabacteroides(4.64%)和Trichococcus(1.82%)等.此外, 秋季缺氧池中反硝化菌的丰度高于春季缺氧池, 这与秋季缺氧池中NO3--N浓度较春季更高有关.此外, 厌氧池中反硝化菌主要为Thauera和Arcobacter, 缺氧池为Thauera、Arcobacter和Azonexus, 可见Thauera对氮的去除发挥了重要作用.已有研究也发现抗生素废水处理过程中Thauera是重要的反硝化功能菌[28, 29].
春季好氧池和二沉池的优势菌属均为Gp4和Thauera.秋季好氧池中的优势菌属和春季好氧池相似, 但二沉池的优势菌群变为Flavobacterium(23.60%)、Pseudomonas(9.18%)、Gp4(9.02%)和Acinetobacter(7.72%)等.春、秋季二沉池中Flavobacterium丰度均升高(增幅为0.08%和23.60%), 好氧池中氨氧化菌(ammonia oxidizing bacteria, AOB)Nitrosomonas和亚硝酸盐氧化菌(nitrite oxidizing bacteria, NOB)Nitrospira的丰度分别为0.02%~0.10%和0.09%~0.48%, 对好氧阶段氨氮去除起到重要贡献.有研究通过功能基因定量分析认为, 螺旋霉素废水处理过程中氨氧化古菌(ammonia-oxidizing archaea, AOA)在脱氮过程中可能比AOB起到更重要的作用[30], 但在本研究中未检测到AOA的丰度.Nitrospira在春季二沉池中的丰度高于秋季二沉池(分别为0.43%和0.17%), 说明春季二沉池中亚硝酸盐NO2--N被NOB氧化成NO3--N的几率更高, 不易产生NO2--N累积现象.此外, Flavobacterium在秋季二沉池的丰度高达23.60%, 该菌属可还原NO3--N为NO2--N, 说明二沉池内可能继续发生反硝化作用, 并可能导致NO2--N累积.因此, 秋季二沉池内相对低丰度的Nitrospira和高丰度的Flavobacterium的存在, 是该池内NO2--N高于其他处理单元的主要原因.
2.3 潜在致病菌的变化由于潜在致病菌在抗生素的选择压力下可能产生耐药性, 从而兼具致病性和耐药性, 因此, 本研究根据VFDB(http://www.mgc.ac.cn/VFs/)数据库中确定的致病菌菌属类别, 考察了螺旋霉素制药废水处理过程中潜在致病菌的丰度变化情况.这些致病菌菌属中包含了致病菌株, 可以作为污水处理过程中细菌潜在致病性变化的参考.
如图 4所示, 进水中存在丰度较高的潜在致病菌(总丰度为11.73%~13.78%), 包括Desulfovibrio、Enterococcus、Aeromonas、Clostridium、Acinetobacter和Pseudomonas, 厌氧过程对部分潜在致病菌的削减较为明显, 例如Enterococcus削减了3.18%~4.04%, 但也会使部分潜在致病菌如Clostridium的丰度升高; 秋季缺氧过程对Desulfovibrio和Enterococcus的削减显著优于春季.此外, 几乎所有潜在致病菌的丰度在好氧池均显著下降, 说明好氧处理工艺是削减潜在致病菌的主要贡献单元.而二沉池中潜在致病菌丰度增加(春季和秋季分别升高了0.04%和17.38%), 尤其是秋季二沉池中Acinetobacter和Pseudomonas的丰度大幅增加, 这可能是由于秋季二沉池NO2--N的积累, 促进了这两种具有反硝化功能的潜在致病菌的增殖.总体来说, 春季二沉池出水中潜在致病菌的总丰度低于进水中总丰度, 而秋季二沉池出水中潜在致病菌的总丰度则高于进水, 可见螺旋霉素废水处理过程对潜在致病菌总量的削减效果并不稳定.
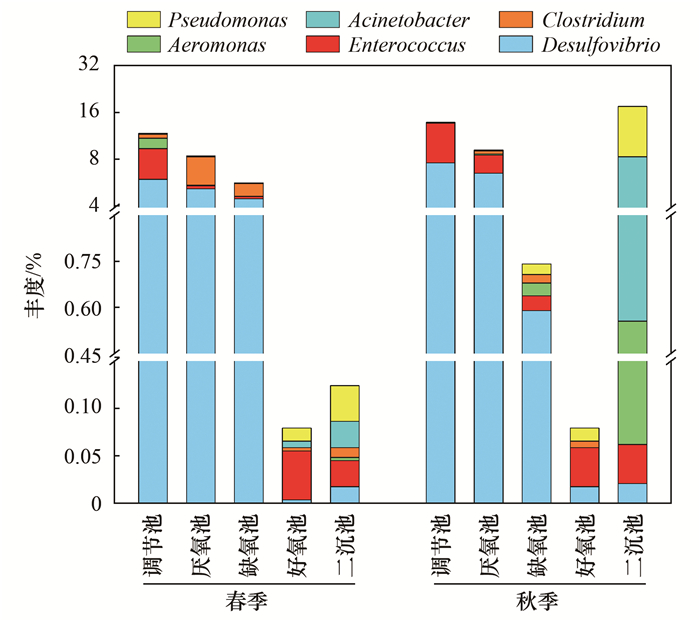
|
图 4 废水处理过程中潜在致病菌的丰度变化 Fig. 4 Variation of potentially pathogenic bacteria during wastewater treatment |
本课题组前期研究结果表明, 螺旋霉素制药废水经过A2O工艺处理, ereA、ermB、ermF、mefA、mphB和Tn916/1545的绝对丰度减少, 而ermX、intI1和ISCR1的绝对丰度则有所增加, 并且ereA、mefA和Tn916/1545在春季的削减效果优于秋季[14].在此基础上, 本研究通过RDA分析进一步考察废水处理过程中水质特性对ARG和MGE的分布以及对菌群结构演变的影响(图 5).由图 5(a)可知, RDA分析结果对ARG和MGE变化的解释度为92.20%, 其中轴1的解释度为70.68%, 轴2的解释度为17.76%. 16S rRNA、NH4+-N和COD对ARG和MGE的分布有重要影响, 其中16S rRNA代表系统中的生物量[31], 其与ereA和ermB的分布密切相关, 而COD与Tn916/1545、mphB和ermF的分布密切相关.另外, intI1通常被看作ARG水平转移的重要标志物[32]; 前期研究结果表明[14], 春季、秋季出水中ermX绝对和相对丰度均呈增加趋势, 本研究RDA分析发现ermX与intI1显著相关, 说明ermX极有可能发生了水平转移, 因而导致出水中ermX的升高.

|
S表示春季样品, A表示秋季样品; 红色箭头代表环境变量, 蓝色箭头代表ARG、MGE和物种变量(属水平):1. Desulfomicrobium; 2. Parabacteroides; 3. Sulfurospirillum; 4. Desulfovibrio; 5. Methanosaeta; 6. Paludibacter; 7. Enterococcus; 8. Trichococcus; 9. Proteiniclasticum; 10.Aeromonas; 11. Clostridium_Ⅲ; 12. Kosmotoga; 13. Acinetobacter; 14. Desulfuromonas; 15. Arcobacter; 16. Geobacter; 17. Ignavibacterium; 18. TM7_genera_incertae_sedis; 19. Azonexus; 20.Pseudomonas; 21. Gp4; 22. Vampirovibrio; 23. Thauera; 24. Steroidobacter; 25. Solitalea; 26. Rheinheimera; 27. Flavobacterium 图 5 水质特征对ARG、MGE分布以及菌群结构的影响 Fig. 5 Influence of wastewater characteristics on the distribution of ARG/MGE and microbial community structure |
如图 5(b)所示, 水质特征对螺旋霉素制药废水处理过程中菌群结构变化的解释度为94.40%, 其中轴1的解释度为48.29%, 轴2的解释度为17.99%.COD、NO2--N和PO43--P与部分微生物密切正相关:COD与Parabacteroides、Paludibacter和Trichococcus显著正相关, 其中Parabacteroides通过糖酵解作用产生乙酸、琥珀酸和少量其他酸[17], Paludibacter会发酵各种糖类产生乙酸盐、丙酸盐和琥珀酸盐, 并可参与生物缺氧系统高浓度COD的生物降解[33, 34], Trichococcus会发酵糖类, 可见这3种细菌均对有机物的产酸发酵过程起作用, 因此其丰度变化与系统中与COD的变化密切相关; NO2--N与Acinetobacter、TM7_genera_incertae_sedis、Pseudomonas、Rheinheimera和Flavobacterium正相关, 其中Pseudomonas和Flavobacterium具有反硝化功能, 尤其在秋季伴随二沉池中NO2--N的升高, Pseudomonas和Flavobacterium的丰度也大幅升高(增幅为9.17%~23.37%), 从而促进反硝化作用的进行.因此, NO2--N对秋季二沉池的菌群结构变化有重要影响.此外, PO43--P对Enterococcus和Proteiniclasticum的正向影响较大, 16S rRNA和NH4+-N与Desulfovibrio密切正相关.
2.5 ARG、MGE和菌群结构之间的关系对春、秋季螺旋霉素制药废水在生物处理过程中ARG和MGE的分布进行PCA分析(图 6).春秋两季各处理单元的ARG和MGE分布特征可分为3类:春、秋季的调节池、厌氧池和缺氧池为一类, 春、秋季的好氧池为一类, 春、秋季的二沉池为一类.对比菌群结构PCA分析结果可知(图 2), 除春、秋季的二沉池的菌群结构差异较大、但ARG和MGE的分布相似之外, 其他样品的菌群结构的聚类、ARG和MGE分布的聚类结果一致.进一步通过Procrustes分析考察微生物群落与ARG和MGE之间的关联度(图 7).结果表明, 轴1和轴2之间相关系数R分别为0.93和0.80, 59.00%的ARG和MGE分布变化可以由菌群结构演替来解释, 表明菌群结构对ARG和MGE的分布有重要影响.

|
图 6 污水处理过程中ARG和MGE分布PCA分析 Fig. 6 PCA analysis of the distribution of ARG and MGE during wastewater treatment |
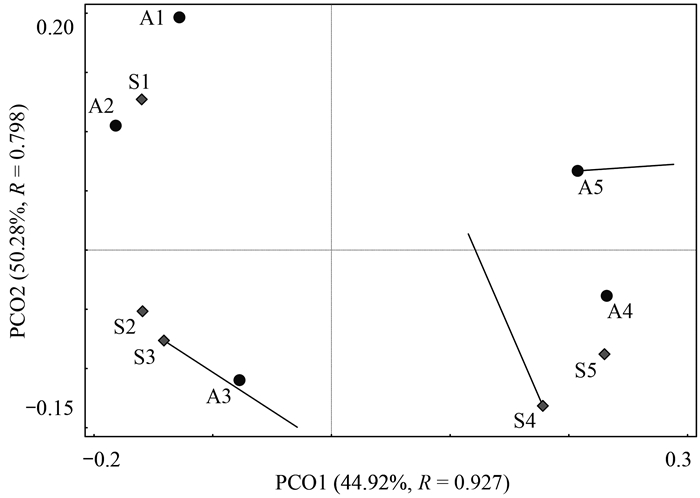
|
图 7 ARG和MGE与菌群结构的Procrustes分析 Fig. 7 Procrustes analysis of the relationship between ARG, MGE, and microbial community structure |
基于Spearman相关性对水相(调节池、二沉池)及泥相(厌氧池、缺氧池和好氧池)的ARG、MGE和菌群结构分别进行网络分析(图 8), 结果表明无论在水相或泥相中, 与mefA显著相关的微生物种类最多, 并且这些微生物(如Desulfovibrio和Pseudomonas)是部分处理单元中的优势菌, 且大多数菌属的丰度在整个处理过程中呈下降趋势, 说明本研究mefA潜在宿主微生物丰度的削减对mefA的去除有重要意义.同时, intI1在水相和泥相中均与ermX显著正相关, ermX在水相中与Ignavibacterium显著正相关, 在泥相中与TM7_genera_incertae_sedis显著正相关, 并且这两种细菌的丰度在整个抗生素废水生物处理过程中呈上升趋势, 这也可以解释前期研究所发现的ermX和intI1的丰度在出水中升高的现象[14].
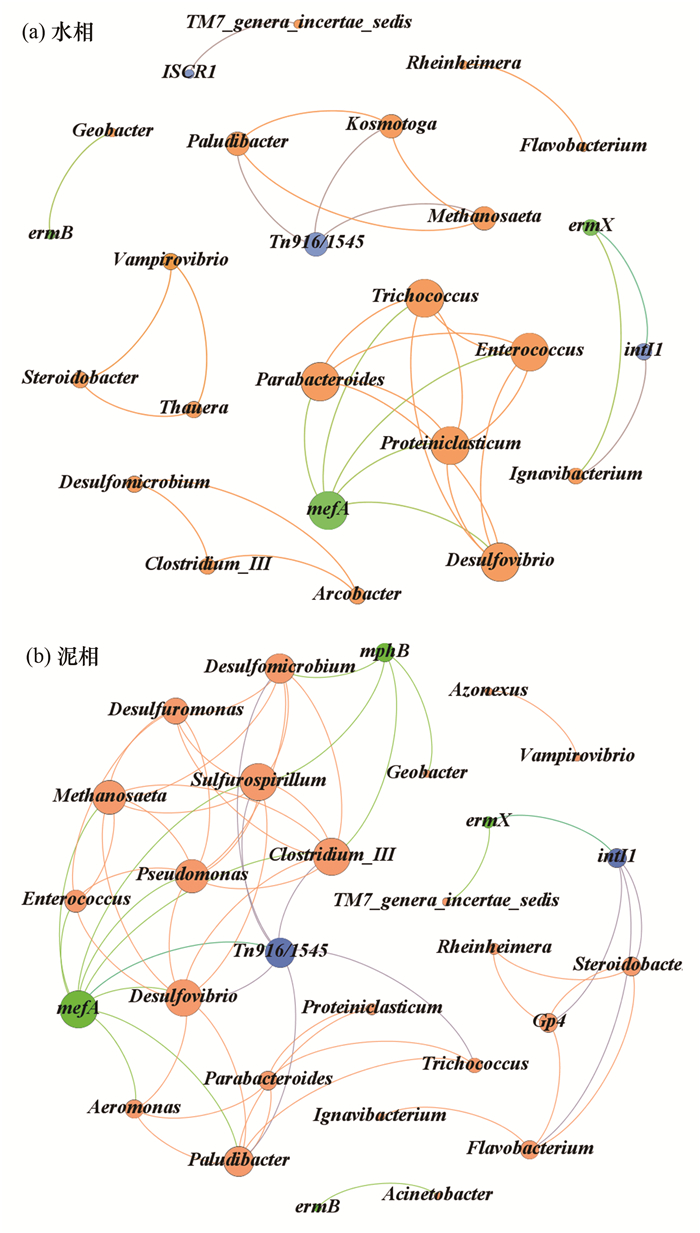
|
橙色节点代表微生物(前10菌属), 绿色节点代表ARG,蓝色节点代表MGE 图 8 水相和泥相中ARG、MGE和菌群结构的关系 Fig. 8 Relationship between ARG, MGE, and microbial community structure in the water phase and sludge phase |
此外, ARG在水相和泥相中分别与不同的菌属显著正相关, 如ermX在水相中与Ignavibacterium显著相关, 而在泥相中与TM7_genera_incertae_sedis显著相关; ermB在水相中与Geobacter显著相关, 而在泥相中与Acinetobacter显著相关; mphB在水相中不与任何微生物显著正相关, 而在泥相中与Desulfomicrobium、Sulfurospirillum、Clostridium_Ⅲ和Geobacter显著相关等.由此可见, 泥相中比水相中存在更多与ARG显著相关的潜在宿主菌类型.此外, 可移动遗传元件Tn916/1545与微生物的显著相关性在泥相中也比水相中更突出.由于Tn916/1545可以接合在染色体和质粒上, 并且可通过细胞间的接触在微生物之间转移[35], 因此, 以上结果说明泥相中细菌传播抗性污染几率更大.
另外, 水相和泥相中ARG与潜在致病菌的相关性也明显不同, 水相中仅mefA与Enterococcus显著正相关, 而在泥相中, 所有目标ARG均至少与一种潜在致病菌显著正相关, 并且泥相中潜在致病菌与其他微生物的显著相关性也比水相中更加明显, 尤其是Pseudomonas在泥相中与Desulfomicrobium、Sulfurospirillum和Methanosaeta等7种微生物显著相关, 而在水相中不与任何微生物显著相关; Clostridium在泥相中与Desulfomicrobium、Sulfurospirillum和Desulfovibrio等6种微生物显著相关, 而在水相中仅与Desulfomicrobium和Arcobacter显著正相关.由此可见, 泥相中ARG与潜在致病菌的显著相关性较水相中更为复杂, 表明污泥中微生物同时获得耐药性和致病性的可能性比水相中的微生物更大.
3 结论(1) 螺旋霉素制药废水A2O工艺处理过程中, 春季和秋季各单元菌群结构受进水水质影响较小; 其中厌氧和缺氧池中以产甲烷菌与硫酸盐还原菌为优势菌, 好氧池和二沉池中优势菌功能则相对较为复杂; 整个废水处理系统对潜在致病菌的削减效果不稳定.
(2) 水质特征和菌群结构对ARG和MGE分布均有重要影响, 且水质特征和菌群结构之间也存在显著的相互影响关系.生物量、NH4+-N和COD对ARG和MGE的分布密切相关, 而在水相和泥相中对ARG分布有显著影响的菌属不同.
(3) 螺旋霉素制药废水在A2O工艺处理过程中, 泥相中ARG与潜在致病菌的显著相关性较水相中更为复杂, 污泥中微生物同时获得耐药性和致病性的可能性比水相中的可能性高, 是今后抗性污染控制的重点关注对象.
| [1] | Rizzo L, Manaia C, Merlin C, et al. Urban wastewater treatment plants as hotspots for antibiotic resistant bacteria and genes spread into the environment: a review[J]. Science of the Total Environment, 2013, 447: 345-360. DOI:10.1016/j.scitotenv.2013.01.032 |
| [2] | Rodríguez-Molina D, Mang P, Schmitt H, et al. Do wastewater treatment plants increase antibiotic resistant bacteria or genes in the environment? Protocol for a systematic review[J]. Systematic Reviews, 2019, 8(1). DOI:10.1186/s13643-019-1236-9 |
| [3] | Guo J H, Li J, Chen H, et al. Metagenomic analysis reveals wastewater treatment plants as hotspots of antibiotic resistance genes and mobile genetic elements[J]. Water Research, 2017, 123: 468-478. DOI:10.1016/j.watres.2017.07.002 |
| [4] | Manaia C M, Rocha J, Scaccia N, et al. Antibiotic resistance in wastewater treatment plants: tackling the black box[J]. Environment International, 2018, 115: 312-324. DOI:10.1016/j.envint.2018.03.044 |
| [5] | Ouyang E M, Liu Y, Ouyang J T, et al. Effects of different wastewater characteristics and treatment techniques on the bacterial community structure in three pharmaceutical wastewater treatment systems[J]. Environmental Technology, 2019, 40(3): 329-341. DOI:10.1080/09593330.2017.1393010 |
| [6] | Aydin S, Ince B, Ince O. Assessment of anaerobic bacterial diversity and its effects on anaerobic system stability and the occurrence of antibiotic resistance genes[J]. Bioresource Technology, 2016, 207: 332-338. DOI:10.1016/j.biortech.2016.01.080 |
| [7] | Jia S Y, Bian K Q, Shi P, et al. Metagenomic profiling of antibiotic resistance genes and their associations with bacterial community during multiple disinfection regimes in a full-scale drinking water treatment plant[J]. Water Research, 2020, 176. DOI:10.1016/j.watres.2020.115721 |
| [8] | Narciso-da-Rocha C, Rocha J, Vaz-Moreira I, et al. Bacterial lineages putatively associated with the dissemination of antibiotic resistance genes in a full-scale urban wastewater treatment plant[J]. Environment International, 2018, 118: 179-188. DOI:10.1016/j.envint.2018.05.040 |
| [9] | 魏健, 何锦垚, 宋永会, 等. 臭氧催化氧化-BAF深度处理抗生素废水效能及微生物群落结构分析[J]. 环境科学学报, 2020, 40(6): 2090-2100. |
| [10] | Wang M M, Liu H L, Cheng X M, et al. Hydrothermal treatment of lincomycin mycelial residues: antibiotic resistance genes reduction and heavy metals immobilization[J]. Bioresource Technology, 2019, 271: 143-149. DOI:10.1016/j.biortech.2018.09.106 |
| [11] | 刘苗苗, 张昱, 李栋, 等. 制药废水受纳河流中四环素抗药基因及微生物群落结构变化研究[J]. 环境科学学报, 2010, 30(8): 1551-1557. |
| [12] | Guardabassi L, Petersen A, Olsen J E, et al. Antibiotic resistance in Acinetobacter spp. isolated from sewers receiving waste effluent from a hospital and a pharmaceutical plant[J]. Applied and Environmental Microbiology, 1998, 64(9): 3499-3502. DOI:10.1128/AEM.64.9.3499-3502.1998 |
| [13] | Schwartz T, Kohnen W, Jansen B, et al. Detection of antibiotic-resistant bacteria and their resistance genes in wastewater, surface water, and drinking water biofilms[J]. FEMS Microbiology Ecology, 2003, 43(3): 325-335. DOI:10.1111/j.1574-6941.2003.tb01073.x |
| [14] | 覃彩霞, 佟娟, 申佩弘, 等. 螺旋霉素制药废水处理过程中耐药菌和抗性基因的转归特征[J]. 环境科学, 2015, 36(9): 3311-3318. |
| [15] | Huang H, Biswal B K, Chen G H, et al. Sulfidogenic anaerobic digestion of sulfate-laden waste activated sludge: evaluation on reactor performance and dynamics of microbial community[J]. Bioresource Technology, 2020, 297. DOI:10.1016/j.biortech.2019.122396 |
| [16] | Wang Y B, Feng M Q, Liu Y H, et al. Comparison of three types of anaerobic granular sludge for treating pharmaceutical wastewater[J]. Journal of Water Reuse and Desalination, 2018, 8(4): 532-543. DOI:10.2166/wrd.2017.040 |
| [17] | Sakamoto M, Benno Y. Reclassification of Bacteroides distasonis, Bacteroides goldsteinii and Bacteroides merdae as Parabacteroides distasonis gen. nov., comb. nov., Parabacteroides goldsteinii comb. nov. and Parabacteroides merdae comb. nov[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(7): 1599-1605. DOI:10.1099/ijs.0.64192-0 |
| [18] | Hubert C, Voordouw G. Oil field souring control by nitrate-reducing Sulfurospirillum spp. that outcompete sulfate-reducing bacteria for organic electron donors[J]. Applied and Environmental Microbiology, 2007, 73(8): 2644-2652. DOI:10.1128/AEM.02332-06 |
| [19] | Yin P N, Guo J B, Xiao S M, et al. Rapid of cultivation dissimilatory perchlorate reducing granular sludge and characterization of the granulation process[J]. Bioresource Technology, 2019, 276: 260-268. DOI:10.1016/j.biortech.2018.12.070 |
| [20] | Pan H P, Yang X N, Zhong Y M, et al. Response of environmental variables and microbial community to sodium percarbonate addition to contaminated sediment[J]. Chemosphere, 2018, 211: 500-509. DOI:10.1016/j.chemosphere.2018.07.120 |
| [21] | Liu J R, Tanner R S, Schumann P, et al. Emended description of the genus Trichococcus, description of Trichococcus collinsii sp. nov., and reclassification of Lactosphaera pasteurii as Trichococcus pasteurii comb. nov. and of Ruminococcus palustris as Trichococcus palustris comb. nov. in the low-G+C gram-positive bacteria[J]. International Journal of Systematic and Evolutionary Microbiology, 2002, 52(4): 1113-1126. |
| [22] | Strepis N, Naranjo H D, Meier-Kolthoff J, et al. Genome-guided analysis allows the identification of novel physiological traits in Trichococcus species[J]. BMC Genomics, 2020, 21(1). DOI:10.1186/s12864-019-6410-x |
| [23] | Xin X D, Chen B Y, Hong J M. Unraveling interactive characteristics of microbial community associated with bioelectric energy production in sludge fermentation fluid-fed microbial fuel cells[J]. Bioresource Technology, 2019, 289. DOI:10.1016/j.biortech.2019.121652 |
| [24] | Meng L W, Li X K, Wang X R, et al. Amoxicillin effects on functional microbial community and spread of antibiotic resistance genes in amoxicillin manufacture wastewater treatment system[J]. Journal of Environmental Sciences, 2017, 61: 110-117. DOI:10.1016/j.jes.2017.09.020 |
| [25] | Li Y C, Zhou J, Gong B Z, et al. Cometabolic degradation of lincomycin in a Sequencing Batch Biofilm Reactor (SBBR) and its microbial community[J]. Bioresource Technology, 2016, 214: 589-595. DOI:10.1016/j.biortech.2016.04.085 |
| [26] | Zheng W S, Liang Q Y, Du Z J. Thauera lacus sp. nov., isolated from a saline lake in Inner Mongolia[J]. International Journal of Systematic and Evolutionary Microbiology, 2019, 69(12): 3786-3791. DOI:10.1099/ijsem.0.003679 |
| [27] | Yi X H, Wu R R, Han D H, et al. Adaptation of methane recovery, sludge characteristics and evolution of microbial community response to elevated nitrate under the methanogenic condition[J]. Journal of Cleaner Production, 2020, 258. DOI:10.1016/j.jclepro.2020.120713 |
| [28] | Zhang M Q, Yuan L, Li Z H, et al. Tetracycline exposure shifted microbial communities and enriched antibiotic resistance genes in the aerobic granular sludge[J]. Environment International, 2019, 130. DOI:10.1016/j.envint.2019.06.012 |
| [29] | Guo N, Ma X F, Ren S J, et al. Mechanisms of metabolic performance enhancement during electrically assisted anaerobic treatment of chloramphenicol wastewater[J]. Water Research, 2019, 156: 199-207. DOI:10.1016/j.watres.2019.03.032 |
| [30] | Zhang Y, Tian Z, Liu M M, et al. High concentrations of the antibiotic spiramycin in wastewater lead to high abundance of ammonia-oxidizing archaea in nitrifying populations[J]. Environmental Science & Technology, 2015, 49(15): 9124-9132. |
| [31] | Tong J, Lu X T, Zhang J Y, et al. Factors influencing the fate of antibiotic resistance genes during thermochemical pretreatment and anaerobic digestion of pharmaceutical waste sludge[J]. Environmental Pollution, 2018, 243: 1403-1413. DOI:10.1016/j.envpol.2018.09.096 |
| [32] | Gillings M R, Gaze W H, Pruden A, et al. Using the class 1 integron-integrase gene as a proxy for anthropogenic pollution[J]. The ISME Journal, 2015, 9(6): 1269-1279. DOI:10.1038/ismej.2014.226 |
| [33] | Ueki A, Akasaka H, Suzuki D, et al. Paludibacter propionicigenes gen. nov., sp. nov., a novel strictly anaerobic, Gram-negative, propionate-producing bacterium isolated from plant residue in irrigated rice-field soil in Japan[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(1): 39-44. DOI:10.1099/ijs.0.63896-0 |
| [34] | Si Z H, Song X S, Wang Y H, et al. Intensified heterotrophic denitrification in constructed wetlands using four solid carbon sources: denitrification efficiency and bacterial community structure[J]. Bioresource Technology, 2018, 267: 416-425. DOI:10.1016/j.biortech.2018.07.029 |
| [35] | Clewell D B, Flannagan S E, Jaworski D D, et al. Unconstrained bacterial promiscuity: the Tn916-Tn1545 family of conjugative transposons[J]. Trends in Microbiology, 1995, 3(6): 229-236. DOI:10.1016/S0966-842X(00)88930-1 |
 2021, Vol. 42
2021, Vol. 42


