2. 南阳师范学院生命科学与农业工程学院, 南阳 473061;
3. 德国基尔大学水文与水资源管理系, 基尔 24098
2. School of Life Science and Agricultural Engineering, Nanyang Normal University, Nanyang 473061, China;
3. Department of Hydrology and Water Resources Management, University of Kiel, Kiel 24098, Germany
随着工农业的飞速发展, 我国面临较为严重的土壤重金属污染, 其中镉(Cd)污染占比最高, 超标率达7.0%, 土壤重金属污染治理与修复迫在眉睫[1, 2].植物修复是目前重金属污染修复研究中的热点.之前的修复植物主要集中于超积累植物和高生物量植物等, 近年来因能源植物自身独特的优势, 在重金属污染土壤修复中得到越来越多的关注[3~6].选用能源植物作为修复植物既可以修复重金属污染土壤, 同时又能发展能源农业, 缓解发展能源产业所面临的耕地压力, 为治理和合理利用重金属污染土壤开辟了一条新的途径[7, 8].芒草(Miscanthus)作为第二代能源植物的代表, 研究表明其对重金属具有良好的耐受能力, 已用于重金属污染土壤修复研究[9~13].
土壤中具有种类丰富、功能多样的微生物, 这些微生物对修复植物的生长起到重要作用[14].目前关于芒草修复土壤重金属污染过程中土壤微生物群落组成和功能研究开展较少. Pham等[15]的研究表明重金属污染能影响芒草根际细菌群落组成. Bourgeois等[16]的研究表明污水灌溉农田中种植芒草能富集Rhizobiales、Nistrospira、Glomeromycota和Mortierella等细菌和真菌群落.Zhao等[11]的研究表明汞污染土壤种植中国芒(M. sinensis)能提高微生物多样性和丰度.这些研究结果为探究芒草修复土壤重金属污染过程中微生物组成提供依据, 但其主要关注于微生物群落结构及其影响因素, 对微生物之间的相互作用和生态功能研究尚未开展.基于高通量测序技术的分子生态网络分析和PICRUSt功能预测分析已应用于土壤、植物和水体等多种生境, 在微生物之间相互作用、关键种群的识别、微生物生态功能等分析中得到良好应用[17~24], 其在芒草修复土壤重金属污染中应用, 将为探究该过程中微生物生态功能提供重要帮助.
我国是芒草集中分布区, 具有丰富的芒草自然资源, 为重金属污染土壤修复提供了良好的材料[25].本研究测定芒草品种南荻(M. saccariflorus)对土壤Cd的耐受和富集能力, 通过高通量测序和分子生态网络分析Cd胁迫对芒草根际细菌群落组成的影响, 通过PICRUSt分析细菌功能, 以期为探明芒草根际细菌群落组成及其在修复Cd污染中的作用提供帮助.
1 材料与方法 1.1 芒草种植及Cd含量测定芒草品种南荻(M. saccariflorus)繁殖材料取自河南南阳国家农业科技园区能源植物栽培示范基地(32°56′45.34″N, 112°24′56.28″E), 选取芒草根状茎进行栽培, 在温室完成盆栽实验.本实验设置高浓度100 mg ·kg-1Cd污染土壤水平和对照.将采集的无污染农田土壤风干, 过4 mm筛充分混匀后分为2份, 其中一份加入分析纯CdSO4 · 8H2 O使土壤中的Cd离子含量为100 mg ·kg-1, 搅拌均匀装入盆钵中, 每盆2 kg, 放置30 d使土壤充分吸附和固定Cd离子.挑选长势相同且生长旺盛的根状茎种入盆钵中, 保持土壤含水量, 控制湿度、温度以满足芒草生长.盆栽种植60 d后, 将芒草植株连根拔起, 采用抖落法收集根际土壤.每组样品设置9个重复(编号分别为Cd0- 1~Cd0- 9和Cd100- 1~Cd100- 9).将芒草植株分为根、茎、叶3部分, 于105℃下杀青20 min, 然后在70℃下烘干至恒重, 测定干重.烘干样品研磨后采用HNO3-HClO4法(1 ∶3, 体积比)消煮, 使用1% HNO3定容后用电感耦合等离子体发射光谱仪(ICP-OES)测定Cd含量.
1.2 根际土壤细菌总DNA提取根际土壤细菌总DNA提取采用FastDNA® Spin Kit for Soil试剂盒(MP Biomedicals, USA), 按照说明书的提取步骤进行, 将提取得到的土壤总DNA通过微量紫外分光光度计(NanoDrop® ND- 1000, Wilmington, DE, USA)测定DNA浓度和纯度.
1.3 高通量测序和PCR扩增采用通用引物338F(5′-ACTCCTACGGGAGG CAGCA- 3′)和806R(5′-GGACTACHVGGGTWTC TAAT- 3′)对细菌V3-V4区进行扩增, 以获得目的基因片段. PCR反应体系为20 μL, 包含5×FastPfu缓冲液4 μL、2.5 mmol ·L-1 dNTPs 2 μL、Forward Primer(5 μmol ·L-1) 0.8 μL、Reverse Primer (5 μmol ·L-1) 0.8 μL、FastPfu Polymerase 0.4 μL、BSA 0.2 μL和DNA模板10 ng, 补充dH2 O至20 μL. PCR反应条件为95℃预变性3 min, 27×(95℃变性30 s; 55℃复性30 s; 72℃延伸45 s); 72℃延伸10 min, 10℃直到停止.将不同样品的PCR扩增物均一化后寄送上海美吉生物医药科技有限公司, 使用MiSeq PE300测序仪(Illumina Inc, San Diego, CA, USA)完成序列测定.
1.4 高通量数据分析将Illumina MiSeq测序得到的下机数据(raw data)经处理后得到有效数据(effective tags).采用QIIME软件进行生物信息学分析, 根据序列的相似度, 将序列归为多个OTU(operational taxonomic unit)[26].采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析, 并在各个分类水平下统计每个样品的群落组成.基于OTU计算稀释曲线、群落丰富度指数(Chao1、ACE)和群落多样性指数(Shannon、Simpson)等[27].采用LEfSe在线工具寻找组与组之间有统计学差异的生物标志物[28].分子生态网络分析选取随机矩阵理论(random matrix theory, RMT), 在Molecular Ecological Network Analyses Pipeline (MENAP)(http://ieg2.ou.edu/MENA/main.cgi)上完成[29, 30].利用软件Cytoscape 3.1.1对网络进行可视化处理, 得到可视网络结构图及相关信息.功能和代谢途径预测采用PICRUSt2软件进行分析, 利用QIIME获得的closed OTU-table与COG、KEGG数据库进行比对, 获得不同的数据库功能预测信息, 具体分析步骤基于在线分析平台https://github.com/picrust/picrust2)[31].
2 结果与分析 2.1 芒草生物量及其Cd积累从表 1可以看出, 100 mg ·kg-1 Cd的添加会抑制芒草的生长, 不同组织生物量与无污染对照相比均有所降低, 幅度为根部26.72%、茎部57.24%、叶部32.41%, 均显著低于对照(P < 0.05).不同组织Cd含量测定结果表明, 根部Cd含量远高于茎部和叶部含量(表 1). Cd积累量与之类似, 根部远高于茎部和叶部积累量.
|
|
表 1 芒草不同组织生物量、Cd含量和积累量1) Table 1 Biomass, Cd content, and total Cd uptake in the tissues of Miscanthus grown in uncontaminated and Cd-contaminated soils |
2.2 群落组成与多样性指数分析
高通量测序结果表明, 无污染芒草根际土壤样品细菌由32个门和373个属组成, Cd污染组细菌由28个门和365个属组成.其中变形菌门(Proteobacteria, 占比24.20% ~32.99%)、酸杆菌门(Acidobacteria, 12.58% ~15.00%)、拟杆菌门(Bacteroidetes, 7.93% ~15.84%)、绿弯菌门(Chloroflexi, 9.23% ~16.14%)、放线菌门(Actinobacteria, 5.89% ~16.87%)为其优势种群(图 1).对属水平的丰度占比前30种群进行分析, 其主要由鞘氨醇单胞菌属(Sphingomonas)、RB 41 、玫瑰弯菌(Roseiflexus)、Haliangium、Ohtaekwangia和链霉菌属(Streptomyces)等组成, 总丰度占比分别为20.36%和21.80%.其中鞘氨醇单胞菌属、芽孢杆菌属(Bacillus)、芽单胞菌属(Gemmatimonas)、链霉菌属和德沃斯氏菌属是报道的具有植物促生特性的种群(图 2).
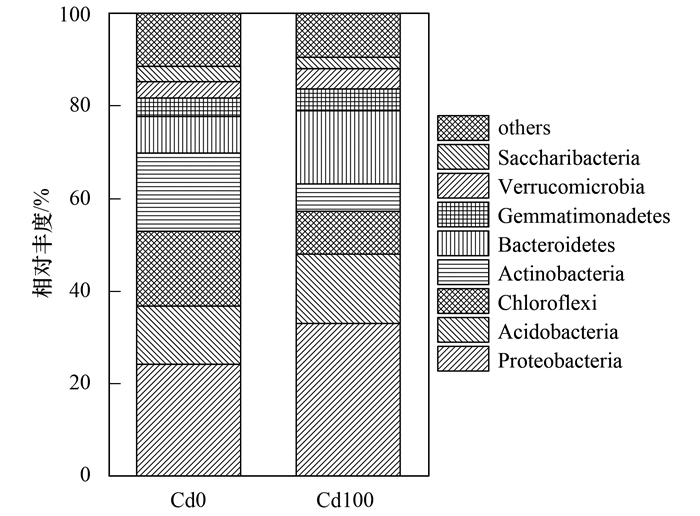
|
图 1 门分类水平细菌群落相对丰度分布 Fig. 1 Relative abundance of sequences at the phylum level of the bacterial communities in the different samples |
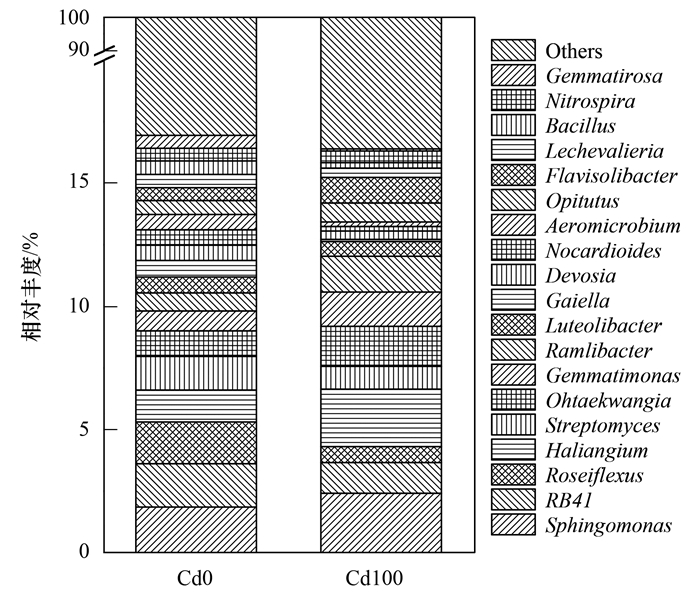
|
图 2 属分类水平细菌群落相对丰度分布 Fig. 2 Relative abundance of sequences at the genera level of the bacterial communities in the different samples |
采用基于UniFrac的PCoA分析和UPGMA分析不同芒草样品细菌群落差异, 无污染组聚集于图 3(a), 添加Cd污染组分布于图 3(b).UPGMA聚类结果与PCoA分析类似, 在0.2相似性水平上分为无污染组和添加Cd污染组两大组, 以上结果表明100 mg ·kg-1 Cd的添加影响了芒草根际细菌群落组成(图 4).非参数检验方法ADONIS和ANOSIM分析结果表明, Cd胁迫导致细菌群落结构存在极显著差异(P < 0.01).多样性指数分析表明, 无污染组芒草根际细菌Sobs数、Shannon指数、ACE指数、Chao1指数均显著高于相对应添加Cd污染组, Simpson指数显著低于相对应添加Cd污染组(P < 0.05), 表明Cd的添加能影响芒草根际细菌群落, 降低其群落多样性(表 2).

|
图 3 不同样品细菌群落的PCoA分析 Fig. 3 Principal coordinates analysis (PCoA) of the bacterial communities in the different samples |
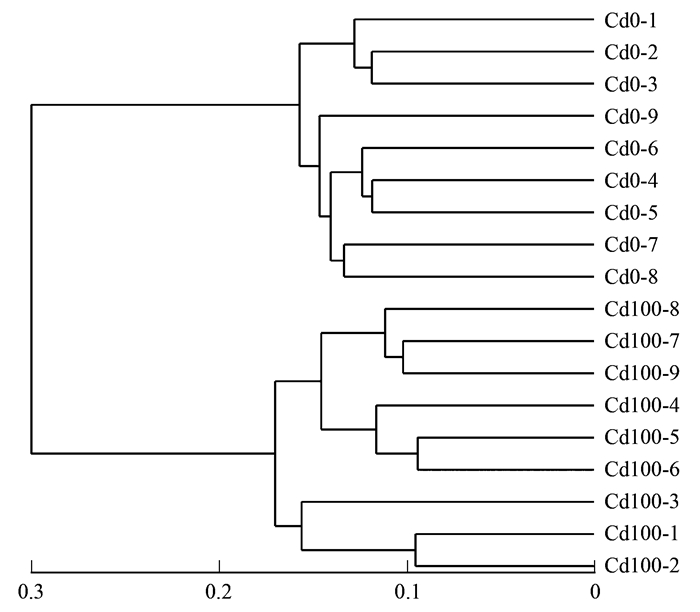
|
图 4 不同样品UPGMA聚类 Fig. 4 UPGMA tree of the bacterial communities in the different samples |
|
|
表 2 不同样品细菌群落多样性评估1) Table 2 Estimation of bacterial community diversity in different samples |
2.3 差异分析
采用LEFSE软件寻找芒草根际细菌群落具有统计学差异的生物标志物(biomarker), 结果表明在LDA≥3.0的水平上共有24个属细菌存在差异, 主要分布于变形菌门(9属)、放线菌门(6属)和拟杆菌门(3属)[图 5(a)].在无污染土壤中存在显著差异的为酸杆菌门的RB 41 , 放线菌门的游动放线菌属(Actinoplanes)、气微菌属(Aeromicrobium)、芽生球菌属(Blastococcus)、Gaiella、类诺卡氏属(Nocardioides)和壤红杆菌属(Solirubrobacter), 绿弯菌门的玫瑰弯菌属(Roseiflexus), 厚壁菌门(Firmicutes)的芽孢杆菌属, 芽单胞菌门(Gemmatimonadetes)的Gemmatirosa, 变形菌门的德沃斯氏菌属、红游动菌属(Rhodoplanes)和Roseateles.添加Cd污染组中显著差异的为拟杆菌门的黄色土源菌属(Flavisolibacter)、Ohtaekwangia和Terrimonas, 芽单胞菌门的芽单胞菌属, 变形菌门的Azohydromonas、Haliangium、苯基杆菌属(Phenylobacterium)、沙壤土杆菌属(Ramlibacter)、堆囊菌属(Sorangium)和鞘氨醇单胞菌属, 疣微菌门(Verrucomicrobia)的丰佑菌属(Opitutus).添加Cd污染组中显著差异细菌丰度均显著高于无污染组[图 5(b)], 其中芽单胞菌属、苯基杆菌属、鞘氨醇单胞菌属和Azohydromonas具有植物促生特性, 鞘氨醇单胞菌属被用于植物-PGPR联合修复重金属污染土壤研究[图 5(c)].
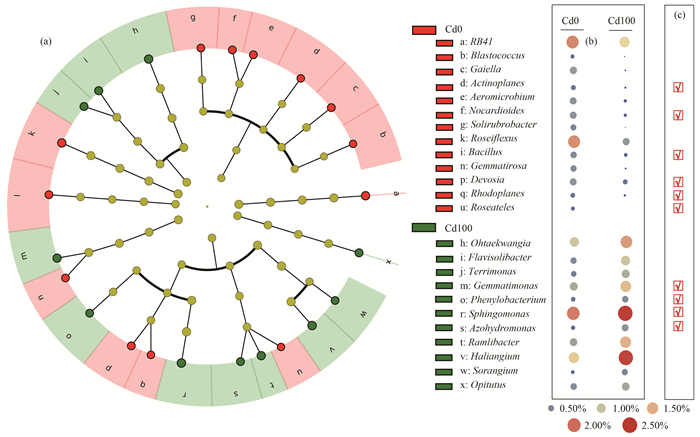
|
(a)差异细菌分布; (b)相对丰度; (c) 植物促生细菌分布 图 5 不同样品差异细菌与潜在功能 Fig. 5 LEfSe analysis identifying the most differentially abundant taxa and their potential function |
采用PICRUSt软件对芒草根际细菌功能进行预测分析, 共获得代谢(metabolism)、遗传信息处理(genetic information processing)和环境信息处理(environmental information processing)为主要组成的6类一级功能层生物代谢通路, 占比分别为69.06% ~69.68%、10.73% ~10.76%和7.09% ~7.15%(图 6).除人类疾病(human diseases)外, 其他5个一级功能层无污染组预测基因拷贝数均高于添加Cd污染组.对二级功能层生物代谢通路进行分析, 发现其由碳水化合物代谢(carbohydrate metabolism)、氨基酸代谢(amino acid metabolism)和能量代谢(energy metabolism)等46个子功能组成.其中环境适应(environmental adaptation)、转运与分解代谢(transport and catabolism)、细胞生长和死亡(cell growth and death)、信号转导(signal transduction)、细胞运动(cell motility)和其他氨基酸代谢(metabolism of other amino acids)等20个二级功能层预测基因拷贝数均为添加Cd污染组高于无污染组.
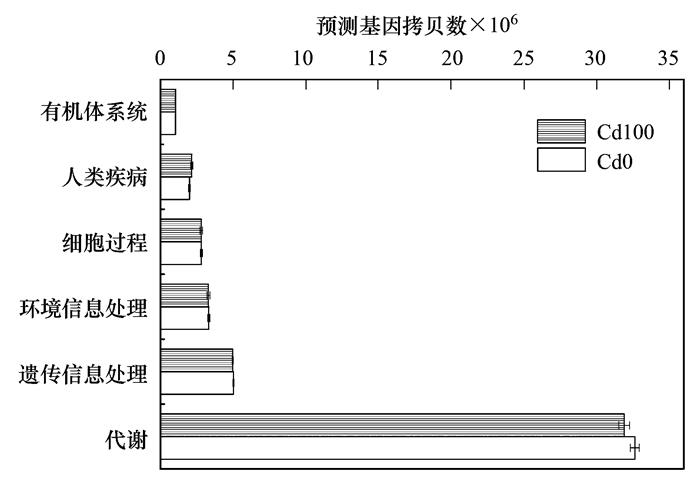
|
图 6 不同样品细菌PICRUSt功能预测 Fig. 6 PICRUSt metabolic predictions of the bacterial communities from different samples |
利用细菌相对丰度基于RMT方法构建无污染组和污染组芒草根际细菌分子生态网络, 在可视化网络结构图中, 每个节点代表一个OTU, 节点之间的红色或者蓝色连线代表OTU之间正负相关关系[图 7(a)和7(b)].描述分子生态网络性质的指标主要有总节点数(total nodes)、总连接数(total links)、相关性阈值(similarity threshold)、平均路径长度(geodesic distance)、平均聚集系数(average clustering coefficient, avgCC)、平均连通度(average degree, avgK)和正相关连线数比例(positive link percentage, %)等.所构建的2个网络相关性阈值(similarity threshold)均高于0.90, 可以用于后续微生物相互关系的研究.与无污染组芒草根际样品相比, 污染组网络的总节点数(468 vs. 631)、总连接数(902 vs. 1217)均有所降低.同时无污染组网络每个节点的平均连通度相近(3.857 vs. 3.855)、平均路径长度更大(6.705 vs. 6.046)、聚集能力(0.182 vs. 0.217)和模块性(0.665 vs. 0.719)稍弱、模块数(96 vs. 55)更高.无污染和污染组网络中负相关连线数分别为472和394, 分别占相应总连线数的38.78%和43.68%.

|
图 7 芒草根际细菌生态网络 Fig. 7 Overview of the bacterial networks in the Miscanthus rhizosphere soil samples |
参考Deng等[29]的研究, 将Zi≥2.5或是Pi≥0.62的所有节点(OTU)定为关键细菌(key bacteria).无污染组样品关键细菌数(OTU数)为13个, 其中鉴定到属水平的分别为酸杆菌门的Haliangium、拟杆菌门的玫瑰弯菌、绿弯菌门的Ohtaekwangia和拟杆菌门的黄色土源菌属.污染组样品关键细菌数(OTU数)同为13个, 其中鉴定到属水平的分别为酸杆菌门的RB 41、放线菌门的 CL500- 29 _marine_group、壤红杆菌属和变形菌门的沙壤土杆菌属, 关键细菌组成与无污染组有所差异.
3 讨论 3.1 芒草根际细菌组成及其潜在功能土壤微生物能影响重金属污染土壤中植物的生长和修复效率, 目前关于芒草修复重金属污染过程中微生物群落组成开展较少[32, 33]. Pham等[15]采用MiSeq测序分析发现芒草根际细菌主要由变形菌门、拟杆菌门、酸杆菌门和芽单胞菌门等组成, 重金属污染能影响细菌群落结构.本实验MiSeq测序结果表明, 芒草无污染和Cd污染条件下根际细菌由变形菌门、酸杆菌门、拟杆菌门、绿弯菌门、放线菌门等32个门和425个属组成(图 1).有研究表明植物根际土壤存在大量具有植物促生特性的细菌, 其在植物生长、植物耐受逆境胁迫等方面起到重要的作用[34]. Pham等[15]的研究发现芒草根际含有假单胞菌属(Pseudomonas)、草螺菌属(Herbaspirillum)、固氮螺菌属(Azospirillum)和梭菌属(Clostridium)等植物促生细菌种群.Zadel等[35]的研究发现, 芒草根际Luteolibacter和小单孢菌属(Micromonospora)在重金属胁迫条件下到显著地富集, 是后续芒草-植物促生细菌(PGPR)联合修复的良好菌株材料.与之类似, 本研究对芒草属水平上丰度占比前30种群(图 2)以及种群差异分析[图 5(c)]发现, 鞘氨醇单胞菌属、芽孢杆菌属、芽单胞菌属和链霉菌属等优势种群或差异种群为已报告的PGPR, 其中芽孢杆菌属[36]、鞘氨醇单胞菌属[37]和链霉菌属[38]已用于重金属污染土壤植物-PGPR联合修复研究中.目前芒草生长过程中PGPR的有效利用开展较少, 对芒草根际PGPR分离、纯化并开展联合修复是后续研究的重要内容之一.
3.2 Cd的胁迫改变芒草根际细菌网络和功能重金属污染对土壤微生物具有毒害作用, 其对芒草根际微生物群落组成影响研究已有开展. Pham等[15]的研究表明重金属能改变芒草根际细菌群落和组成.与之类似, 本实验中Cd的添加能显著影响芒草根际细菌群落组成, 降低其群落多样性(表 2、图 3和图 4).基于高通量测序的PICRUSt分析能预测细菌群落的代谢功能, 在植物修复土壤HM污染研究中已有应用.Jiang等[20]采用PICRUSt预测了海州香薷(Elsholtzia splendens)根际细菌功能, 发现其主要由氨基酸代谢、碳水化合物代谢等41个子功能组成.本实验芒草根际细菌预测分析表明, 其由碳水化合物代谢、氨基酸代谢和能量代谢等子功能组成.高浓度的重金属对土壤微生物产生明显的毒害作用, 降低微生物活性和群落结构[39].本实验多样性指数分析表明, Cd的添加能降低芒草根际细菌群落的Sobs数、Shannon指数、ACE指数和Chao1指数(表 2), 对预测基因拷贝数进行分析发现, 无污染样品均高于Cd污染样品(图 6), 结果表明Cd的胁迫同时降低了芒草根际土壤细菌功能.
在生态系统中, 微生物以复杂的网络结构共存和相互作用[40].分子生态网络(phylogenentic molecular ecology networks, pMEN)是基于随机矩阵理论来研究生态系统内作用关系的一种方法, 对理解微生物群落内潜在的相互作用关系提供了解决途径[29, 30].本实验构建了无污染和Cd污染两组芒草根际细菌分子生态网络, 结果发现Cd的胁迫降低了芒草根际细菌之间的相互作用, 导致其网络结构更为简单, 表现为更低的总节点数和总连接数.同时污染组网络平均路径长度和聚集能力等网络特性表明当外界环境发生扰动时, 该网络能在很短的时间内将环境扰动传递到整个网络, 导致网络结构不稳定, 表明Cd的胁迫降低了网络缓冲环境变化的能力[29, 41].网络中物种间的相互关系可描述为正相关和负相关, 正相关表示物种的生态位相同或具有共生关系, 负相关代表着竞争或捕食关系[42].污染组网络负相关连线数高于无污染组, 表明Cd的胁迫使芒草根际细菌物种之间的竞争关系更强.同时pMENs在关键细菌识别得到广泛应用, 这些关键细菌在生态系统维持稳定和发挥功能中起到重要作用[42].网络节点的拓扑学角色分析表明Cd的胁迫使关键细菌组成发生了改变, 这些关键细菌可能在芒草耐受和富集Cd过程中起到作用.
4 结论(1) MiSeq测序表明芒草根际细菌群落由变形菌门、酸杆菌门、拟杆菌门、绿弯菌门和放线菌门等32个门组成, 包含鞘氨醇单胞菌属、芽孢杆菌属、芽单胞菌属和链霉菌属等植物促生细菌(PGPR)种群.
(2) PCoA分析和UPGMA分析表明Cd的添加能显著影响芒草根际细菌群落组成, 多样性指数分析Cd的添加降低其群落多样性.LEFSE分析表明Cd的添加能显著提高黄色土源菌属、苯基杆菌属、芽单胞菌属、鞘氨醇单胞菌属等11个属的相对丰度, 这些细菌可能在芒草耐受和富集重金属中起到作用.
(3) 分子生态网络分析表明, Cd的添加改变了芒草根际细菌间的相互作用, 导致其网络结构更为简单、降低了网络缓冲环境变化的能力、提高了负相关连线数并改变关键细菌组成. PICRUSt功能预测分析表明, Cd的胁迫降低了芒草根际土壤细菌功能.
| [1] | Zhao F J, Ma Y B, Zhu Y G, et al. Soil contamination in China: current status and mitigation strategies[J]. Environmental Science & Technology, 2015, 49(2): 750-759. |
| [2] |
骆永明, 章海波, 涂晨, 等. 中国土壤环境与污染修复发展现状与展望[J]. 中国科学院院刊, 2015, 30(S1): 257-266. Luo Y M, Zhang H B, Tu C, et al. Soil environment and pollution remediation in China: current status and prospectives research development[J]. Bulletin of Chinese Academy of Sciences, 2015, 30(S1): 257-266. |
| [3] | Tripathi V, Edrisi S A, Abhilash P C. Towards the coupling of phytoremediation with bioenergy production[J]. Renewable and Sustainable Energy Reviews, 2016, 57: 1386-1389. DOI:10.1016/j.rser.2015.12.116 |
| [4] | Gomes H I. Phytoremediation for bioenergy: challenges and opportunities[J]. Environmental Technology Reviews, 2012, 1(1): 59-66. DOI:10.1080/09593330.2012.696715 |
| [5] |
余海波, 宋静, 骆永明, 等. 典型重金属污染农田能源植物示范种植研究[J]. 环境监测管理与技术, 2011, 22(3): 71-76. Yu H B, Song J, Luo Y M, et al. Field demonstration of energy plants production on heavy metal contaminated farmland[J]. The Administration and Technique of Environmental Monitoring, 2011, 22(3): 71-76. DOI:10.3969/j.issn.1006-2009.2011.03.014 |
| [6] |
贾伟涛, 吕素莲, 冯娟娟, 等. 利用能源植物治理土壤重金属污染[J]. 中国生物工程杂志, 2015, 35(1): 88-95. Jia W T, Lv S L, Feng J J, et al. Restore heavy metal contaminated soil with energy plants[J]. China Biotechnology, 2015, 35(1): 88-95. |
| [7] | Weyens N, van der Lelie D, Taghavi S, et al. Exploiting plant-microbe partnerships to improve biomass production and remediation[J]. Trends in Biotechnology, 2009, 27(10): 591-598. DOI:10.1016/j.tibtech.2009.07.006 |
| [8] |
丁传雨, 郑远, 任学敏, 等. 能源植物修复土壤镉污染过程中细菌群落分析[J]. 环境科学学报, 2016, 36(8): 3009-3016. Ding C Y, Zheng Y, Ren X M, et al. Changes in bacterial community composition during the remediation of Cd-contaminated soils of bioenergy crops[J]. Acta Scientiae Circumstantiae, 2016, 36(8): 3009-3016. |
| [9] | Rusinowski S, Krzy Dz·ak J, Sitko K, et al. Cultivation of C4 perennial energy grasses on heavy metal contaminated arable land: impact on soil, biomass, and photosynthetic traits[J]. Environmental Pollution, 2019, 250: 300-311. DOI:10.1016/j.envpol.2019.04.048 |
| [10] | Nurzhanova A, Pidlisnyuk V, Abit K, et al. Comparative assessment of using Miscanthus×giganteus for remediation of soils contaminated by heavy metals: a case of military and mining sites[J]. Environmental Science and Pollution Research, 2019, 26(13): 13320-13333. DOI:10.1007/s11356-019-04707-z |
| [11] | Zhao A Q, Gao L Y, Chen B Q, et al. Phytoremediation potential of Miscanthus sinensis for mercury-polluted sites and its impacts on soil microbial community[J]. Environmental Science and Pollution Research, 2019, 26(34): 34818-34829. |
| [12] | Zhang J, Yang S Y, Huang Y J, et al. The tolerance and accumulation of Miscanthus Sacchariflorus (maxim.) benth., an energy plant species, to cadmium[J]. International Journal of Phytoremediation, 2015, 17(6): 538-545. DOI:10.1080/15226514.2014.922925 |
| [13] |
吴道铭, 陈晓阳, 曾曙才. 芒属植物重金属耐性及其在矿山废弃地植被恢复中的应用潜力[J]. 应用生态学报, 2017, 28(4): 1397-1406. Wu D M, Chen X Y, Zeng S C. Heavy metal tolerance of Miscanthus plants and their phytoremediation potential in abandoned mine land[J]. Chinese Journal of Applied Ecology, 2017, 28(4): 1397-1406. |
| [14] | Wood J L, Tang C X, Franks A E. Microbial associated plant growth and heavy metal accumulation to improve phytoextraction of contaminated soils[J]. Soil Biology and Biochemistry, 2016, 103: 131-137. DOI:10.1016/j.soilbio.2016.08.021 |
| [15] | Pham H N, Pham P A, Nguyen T T H, et al. Influence of metal contamination in soil on metabolic profiles of Miscanthus x giganteus belowground parts and associated bacterial communities[J]. Applied Soil Ecology, 2018, 125: 240-249. DOI:10.1016/j.apsoil.2018.01.004 |
| [16] | Bourgeois E, Dequiedt S, Lelièvre M, et al. Miscanthus bioenergy crop stimulates nutrient-cycler bacteria and fungi in wastewater-contaminated agricultural soil[J]. Environmental Chemistry Letters, 2015, 13(4): 503-511. |
| [17] | Chen Z J, Zheng Y, Ding C Y, et al. Integrated metagenomics and molecular ecological network analysis of bacterial community composition during the phytoremediation of cadmium-contaminated soils by bioenergy crops[J]. Ecotoxicology and Environmental Safety, 2017, 145: 111-118. DOI:10.1016/j.ecoenv.2017.07.019 |
| [18] | Chen Z J, Xu G, Ding C Y, et al. Illumina MiSeq sequencing and network analysis the distribution and co-occurrence of bacterioplankton in Danjiangkou Reservoir, China[J]. Archives of Microbiology, 2020, 202(4): 859-873. DOI:10.1007/s00203-019-01798-7 |
| [19] | Wang M, Chen S B, Chen L, et al. Responses of soil microbial communities and their network interactions to saline-alkaline stress in Cd-contaminated soils[J]. Environmental Pollution, 2019, 252: 1609-1621. |
| [20] | Jiang L F, Song M K, Yang L, et al. Exploring the influence of environmental factors on bacterial communities within the rhizosphere of the Cu-tolerant plant, Elsholtzia splendens[J]. Scientific Reports, 2016, 6(1). DOI:10.1038/srep36302 |
| [21] |
胡晓婧, 刘俊杰, 魏丹, 等. 东北黑土区不同纬度农田土壤真菌分子生态网络比较[J]. 应用生态学报, 2018, 29(11): 3802-3810. Hu X J, Liu J J, Wei D, et al. Comparison on fungal molecular ecological networks of agricultural soils with different latitudes in the black soil region of Northeast China[J]. Chinese Journal of Applied Ecology, 2018, 29(11): 3802-3810. |
| [22] |
李冰, 李玉双, 魏建兵, 等. 不同土地利用方式对土壤细菌分子生态网络的影响[J]. 环境科学, 2020, 41(3): 1456-1465. Li B, Li Y S, Wei J B, et al. Effects of different land use typess on the molecular ecological network of soil bacteria[J]. Environmental Science, 2020, 41(3): 1456-1465. |
| [23] |
周石磊, 孙悦, 张艺冉, 等. 基于网络分析解析水源水库春季水体反硝化群落演变特征及驱动因素[J]. 环境科学, 2020, 41(4): 1666-1675. Zhou S L, Sun Y, Zhang Y R, et al. Evolution characteristics and driving factors of denitrification community based on network analysis in the process of spring thermal layer formation in Zhoucun reservoir[J]. Environmental Science, 2020, 41(4): 1666-1675. |
| [24] |
孙峰, 田伟, 张菲, 等. 丹江口库区库滨带植被土壤细菌群落多样性及PICRUSt功能预测分析[J]. 环境科学, 2019, 40(1): 421-429. Sun F, Tian W, Zhang F, et al. Composition and predictive functional analysis of rhizosphere bacterial communities in riparian buffer strips in the Danjiangkou Reservoir, China[J]. Environmental Science, 2019, 40(1): 421-429. |
| [25] |
易自力. 芒属能源植物资源的开发与利用[J]. 湖南农业大学学报(自然科学版), 2012, 38(5): 455-463. Yi Z L. Exploitation and utilization of Miscanthus as energy plant[J]. Journal of Hunan Agricultural University (Natural Sciences), 2012, 38(5): 455-463. |
| [26] | Caporaso J G, Kuczynski J, Stombaugh J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. |
| [27] | Schloss P D, Westcott S L, Ryabin T, et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. |
| [28] | Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biology, 2011, 12(6). DOI:10.1186/gb-2011-12-6-r60 |
| [29] | Deng Y, Jiang Y H, Yang Y, et al. Molecular ecological network analyses[J]. BMC Bioinformatics, 2012, 13. DOI:10.1186/1471-2105-13-113 |
| [30] | Zhou J Z, Deng Y, Luo F, et al. Phylogenetic molecular ecological network of soil microbial communities in response to elevated CO2[J]. mBio, 2011, 2(4). DOI:10.1128/mBio.00122-11 |
| [31] | Douglas G M, Maffei V J, Zaneveld J, et al. PICRUSt2:an improved and extensible approach for metagenome inference[J]. bioRxiv, 2019. DOI:10.1101/672295 |
| [32] | Chen Y, Tian W, Shao Y, et al. Miscanthus cultivation shapes rhizosphere microbial community structure and function as assessed by Illumina MiSeq sequencing combined with PICRUSt and FUNGUIld analyses[J]. Archives of Microbiology, 2020, 202(5): 1157-1171. |
| [33] | Li D F, Voigt T B, Kent A D. Plant and soil effects on bacterial communities associated with Miscanthus×giganteus rhizosphere and rhizomes[J]. GCB Bioenergy, 2016, 8(1): 183-193. |
| [34] | Timmusk S, Paalme V, Pavlicek T, et al. Bacterial distribution in the rhizosphere of wild barley under contrasting microclimates[J]. PLoS One, 2011, 6(3). DOI:10.1371/journal.pone.0017968 |
| [35] | Zadel U, Nesme J, Michalke B, et al. Changes induced by heavy metals in the plant-associated microbiome of Miscanthus x giganteus[J]. Science of the Total Environment, 2020, 711. DOI:10.1016/j.scitotenv.2019.134433 |
| [36] | Babu A G, Kim J D, Oh B T. Enhancement of heavy metal phytoremediation by Alnus firma with endophytic Bacillus thuringiensis GDB-1[J]. Journal of Hazardous Materials, 2013, 250-251: 477-483. |
| [37] | Chen B, Zhang Y B, Rafiq M T, et al. Improvement of cadmium uptake and accumulation in Sedum alfredii by endophytic bacteria Sphingomonas SaMR12: effects on plant growth and root exudates[J]. Chemosphere, 2014, 117: 367-373. |
| [38] | Ali A, Guo D, Mahar A, et al. Role of Streptomyces pactum in phytoremediation of trace elements by Brassica juncea in mine polluted soils[J]. Ecotoxicology and Environmental Safety, 2017, 144: 387-395. |
| [39] | Wang F, Yao J, Si Y, et al. Short-time effect of heavy metals upon microbial community activity[J]. Journal of Hazardous Materials, 2010, 173(1-3): 510-516. |
| [40] | Coyte K Z, Schluter J, Foster K R. The ecology of the microbiome: networks, competition, and stability[J]. Science, 2015, 350(6261): 663-666. |
| [41] | Wang Y, Zhang R, Zheng Q, et al. Bacterioplankton community resilience to ocean acidification: evidence from microbial network analysis[J]. ICES Journal of Marine Science, 2016, 73(3): 865-875. |
| [42] | Layeghifard M, Hwang D M, Guttman D S. Disentangling interactions in the microbiome: a network perspective[J]. Trends in Microbiology, 2017, 25(3): 217-228. |
 2021, Vol. 42
2021, Vol. 42


