Effects of Three Commonly Used Herbicides on Bacterial Antibiotic Resistance
自抗生素使用以来, 人类医疗和畜禽养殖行业得到快速的改善和发展, 但与此同时也带来了严重的公共卫生问题——抗生素耐药性的产生与传播.在全球范围内, 每年约有70万人死于耐药感染性疾病, 预计到2050年, 每年因此而死亡的人数将达到1 000万[1].由于抗生素在人体和牲畜体内代谢率低, 导致大量的抗生素及其代谢产物残留于水体和土壤环境中[2~4].在外界环境的选择压力下, 细菌可通过基因突变、垂直基因转移和水平基因转移产生抗生素耐药性.通常认为, 抗生素滥用是导致耐药性出现和传播的主要驱动力.近年来, 研究者们在原始森林、永久冻土等没有任何抗生素污染的环境中也检测到大量的抗生素抗性基因存在[5], 表明抗生素不是唯一驱动耐药基因扩散传播的动力, 可能存在其他因素加速其快速传播.
最近研究发现, 非抗生素物质也可以驱动耐药基因的传播.例如, 消毒剂和消毒副产物[6]、汽油和柴油废气颗粒[7]、纳米材料[8, 9]、离子液体[10]、非抗生素药物(三氯生、氟西汀和卡马西平)[11~13]、CO2[14]和游离亚硝酸[15]等物质可以促进多重耐药质粒在细菌之间的水平转移, 进而加速耐药基因在环境中的传播.这些物质通过促进细胞膜通透性增加, 导致耐药质粒更容易进出细胞膜, 从而加速抗性基因在细菌之间的转移频率[8].此外, 还可以促进胞内活性氧(reactive oxygen species, ROS)含量升高, 具有强氧化能力的ROS可直接诱导染色体DNA损伤进而增加复制过程中的突变频率[14, 16].尽管已有多种非抗生素物质被证明通过增加耐药质粒(譬如RP4质粒)转移频率从而加速耐药基因的传播.但是, 环境中依然有大量的非抗生素物质在抗生素耐药性方面的贡献被忽视.
除草剂作为一种有效控制杂草的农药在全球农业生产中被广泛使用[17].我国使用除草剂的农田面积高达6 000万hm2, 未来可能以每年200万hm2的速度增加[18].除草剂的广泛使用导致其大量残留于自然环境中[19], 从而使除草剂在环境中的检测浓度从μg·L-1上升至mg·L-1[20], 尤其在土壤环境中浓度高达10 mg·kg-1[21].因此, 除草剂在环境中, 特别是在土壤中的风险不容忽视.已有报道证明除草剂和抗生素耐药性之间存在潜在的联系, Kurenbach等[22]的研究发现草甘膦、麦草畏和2, 4-D的暴露会引起细菌改变对抗生素的敏感性.Xing等[23]的研究结果显示, 大肠杆菌连续暴露于环境浓度的农药和抗生素复合污染的环境中, 能够诱导耐药相关基因发生突变, 从而加速剧耐药性的传播.尽管已有农药等化学物质影响细菌耐药性的报道, 然而, 这些研究存在化学处理成分复杂、选择的化学物质浓度过高和暴露时间过短等局限性.在土壤环境浓度胁迫下, 不同种类除草剂(草甘膦、草铵膦和麦草畏)对细菌抗生素耐药性的影响鲜见报道, 特别是影响细菌耐药性的分子机制依然未知.
因此, 本文选取农业上广泛使用的3种除草剂(草甘膦、草铵膦和麦草畏)和E. coli DH5α模式菌株为研究对象, 探究除草剂的持续胁迫对细菌耐药性的影响.利用液体连续传代培养实验和全基因组测序解析除草剂诱导抗生素耐药性产生的作用机制.本文发现除草剂等农业化学制剂可促进抗生素耐药性的产生和传播, 以期为评估除草剂在环境中的风险评价提供了新的视角.
1 材料与方法
1.1 实验材料
草甘膦(glyphosate, Gly)、草铵膦(glufosinate, Glu)和麦草畏(dicamba, Dic)纯度为99%, 其分子结构式如图 1所示; 本文选用临床上使用广泛且常见的6种抗生素, 用于表征抗生素耐药性[24, 25], 如氯霉素(chloramphenicol, Chl)、庆大霉素(gentamycin, Gen)、卡那霉素(kanamycin, Kan)、利福平(rifampicin, Rif)、硫酸链霉素(streptomycin sulfate, Str)和四环素(tetracycline, Tet), 具体的抗生素质量浓度见表 1, 以上除草剂和抗生素均购自上海阿拉丁生化科技股份有限公司; Luria-Bertani(LB)液体和固体培养基(5 g·L-1酵母粉, 10 g·L-1胰蛋白胨, 10 g·L-1 NaCl, 2%琼脂)购自北京索莱宝科技有限公司; 本文选用无上述6种抗生素抗性的大肠杆菌(E. coli DH5α)作为研究对象, 由本课题组菌种库保藏.
表 1
(Table 1)
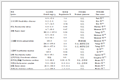
表 1 抗生素质量浓度
Table 1 Antibiotic concentrations
| 抗生素 |
缩写 |
类别 |
质量浓度/μg·mL-1 |
| 氯霉素 |
Chl |
氯霉素类 |
16 |
| 庆大霉素 |
Gen |
氨基糖苷类 |
10 |
| 卡那霉素 |
Kan |
氨基糖苷类 |
50 |
| 利福平 |
Rif |
利福平类 |
100 |
| 硫酸链霉素 |
Str |
氨基糖苷类 |
30 |
| 四环素 |
Tet |
四环素类 |
4 |
|
表 1 抗生素质量浓度
Table 1 Antibiotic concentrations
|
1.2 除草剂暴露实验
E. coli DH5α经过12 h活化后, 取50 μL菌液接种至含有除草剂的4.95 mL LB培养基中, 随后在37℃、150 r·min-1条件下培养, 每隔24 h重复上述步骤, 连续传代培养30 d.除草剂浓度根据土壤中残留的除草剂水平(10 mg·kg-1)[20, 21], 草甘膦和草铵膦暴露浓度为:5、25和50 mg·L-1, 麦草畏暴露浓度为:2、10和20 mg·L-1.通过添加相同体积的无菌水作为对照处理(0 mg·L-1).实验过程中, 在第0、10、15、20、25和30 d取100 μL菌液并选择合适的稀释梯度, 分别涂布至含有10 mg·L-1Gen和无任何抗生素的LB平板上, 以统计除草剂暴露后耐药菌株数量和总细菌数量, 每个菌液梯度3个重复, 所有平板在37℃培养24 h后进行菌落计数.抗生素耐药性突变频率等于耐药菌株的数量除以细菌总数.
1.3 最小抑制浓度和生长曲线的测定
最小抑制浓度(minimum inhibitory concentration, MIC)是反映细菌抗生素耐药能力的重要指标, 采用二倍稀释法测定细菌MIC[11].具体步骤为:经过除草剂暴露30 d后, 在抗生素平板上筛选出突变的抗性菌株, 活化12 h后, 用PBS调整细胞密度为106 CFU·mL-1, 分别测定突变菌株在6种抗生素下的MIC.本实验体系为:5 μL上述调整后的菌液、15 μL连续2倍稀释的抗生素溶液(根据抗生素质量浓度稀释)和130 μL新鲜LB.在37℃培养24 h后, 使用酶标仪(Biotek 800TS, 美国)检测D600来表征菌株的耐药能力.MIC90是指能够抑制90%菌落生长的抗生素浓度.
生长曲线测定的具体步骤为:E. coli DH5α活化12 h后, 稀释100倍至含有除草剂的LB培养基中.草甘膦和草铵膦暴露浓度为5 mg·L-1和50 mg·L-1, 麦草畏暴露浓度为2 mg·L-1和20 mg·L-1.随后在37℃、150 r·min-1条件下培养, 采样间隔为1 h, 总共取样12次, 最后置于酶标仪检测600 nm处的吸光度以表征细菌的生长情况.
1.4 大肠杆菌突变体DNA的提取及全基因组测序
经过除草剂诱变的大肠杆菌突变菌[草甘膦突变菌(30d-Gly)、草铵膦突变菌(30d-Str)和麦草畏突变菌(30d-Dic)]活化12 h后, 使用TIAN Bacteria DNA Kit(TIANGEN, 美国)试剂盒提取细菌DNA, 采用NanoDrop分光光度计(Thermo Fisher Scientific, 美国)和电泳凝胶定量分析DNA浓度.合格的DNA样品提交给广东美格基因科技有限公司, 以HiSeq平台(Illumina)进行双端测序.使用Trimmomatic 0.36对Illumina双端测序的原始数据进行质量修剪, 保留正确配对的数据用于后续分析.将检测的突变位点与E. coli K-12基因组(NCBI登录号NC000913.3)进行比对, 以分析突变位点所在基因的变异.例如单核苷酸多态性(SNP)、插入和缺失.
1.5 活性氧的检测
测定·OH、H2O2和O2-·这3种主要活性氧的浓度. 3种活性氧的检测均在除草剂浓度为10 mg·L-1时测定.·OH采用对苯二甲酸法测定[26], 对苯二甲酸(终浓度0.5 mmol·L-1)添加到含有大肠杆菌的LB中, 通过添加除草剂处理6 h, 取其上清液于荧光光度计检测荧光强度[λ(Ex)=320 nm, λ(Em)=425 nm]; H2O2的测定需提前制备缓冲液(10 mg对羟基苯甲酸和2 mg过氧化物催化酶溶于50 mL 0.1 mmol·L-1的Tris溶液, pH=8.8), 取5 mL制备的缓冲液于20 mL含有大肠杆菌的LB中, 添加除草剂处理6 h后, 取其上清液于λ(Ex)=316.5 nm, λ(Em)=408.5 nm处检测荧光值[27]; O2-·采用氮蓝四唑法测定[28](氮蓝四唑与O2-·反应生成蓝色物质甲腙), 氮蓝四唑(终浓度50 μmol·L-1)添加到经过除草剂处理6 h的菌液中反应20 min, 检测560 nm处的吸光度.
1.6 数据分析
本实验数据均用Microsoft Excel 2010整理, 采用Origin 9.0软件作图, 采用SPSS 25.0软件进行统计学分析, 采用独立样本t检验进行显著性检验.若P < 0.05则认为具有显著性的差异, P < 0.01则认为具有极显著性的差异.
2 结果与讨论
2.1 除草剂对大肠杆菌单一抗生素耐药性的影响
为了研究除草剂暴露对细菌耐药性的影响, 本文选择无抗生素抗性的E. coli DH5α作为研究对象, 在实验室条件下分别暴露于不同浓度的草甘膦(5、25和50 mg·L-1)、草铵膦(5、25和50 mg·L-1)和麦草畏(2、10和20 mg·L-1)中, 进行30 d的液体传代培养实验.因为氨基糖苷类抗生素是临床上使用最广泛的抗生素之一, 所以暴露期间重点观察E. coli DH5α耐受庆大霉素的突变频率变化.
首先验证工作浓度的草甘膦、草铵膦和麦草畏对E. coli DH5α的生长没有显著影响[图 2(a)、2(c)和2(e)], 排除了除草剂对实验菌株的直接毒性作用.图 2(b)、2(d)和2(f)结果显示, 与对照处理相比, 不同类型除草剂均显著(P < 0.05)增加庆大霉素耐药的突变频率, 其中草甘膦诱导突变的频率最高, 麦草畏次之, 草铵膦最低.可能的原因是细菌在面对外源性化合物的选择性压力时, 造成细菌细胞膜通透性的增加和强氧化性物质的产生, 从而诱导细菌突变产生抗生素抗性[12, 13].其次, 3种除草剂实验组的突变频率均随时间逐渐增加, 在第30 d突变频率达到最高.如草甘膦实验组为(6.39±0.93)×10-4、草铵膦实验组为(1.90±0.18)×10-4和麦草畏实验组为(2.92±0.98)×10-4.这表明除草剂诱导的抗生素抗性具有时间依赖性.由于土壤中的除草剂都以相对较高的浓度存在且残留时间较长[19], 除草剂的累积作用会加剧自然环境中抗生素抗性的产生和传播.然而, 在无除草剂暴露的对照组, 笔者发现其自发突变频率始终维持在较低的水平(10-6~10-8), 这与前人的研究结果一致[12, 29].此外, 3种除草剂实验组的突变频率并不遵循剂量依赖性效应, 如草甘膦实验组突变频率变化趋势表现为5 mg·L-1>25 mg·L-1>50 mg·L-1; 草铵膦实验组变化趋势为5 mg·L-1>50 mg·L-1>25 mg·L-1; 麦草畏实验组变化趋势为2 mg·L-1>10 mg·L-1>20 mg·L-1.这是由于除草剂的工作浓度较低, 大肠杆菌对其敏感性较差[30], 导致不同质量浓度的除草剂诱导的突变频率差异较小.以上结果表明, 3种除草剂对细菌突变频率的诱导效果略有差异, 但总体变化趋势类似, 表明3种常用除草剂可以促进E. coli DH5α耐药性的产生.
2.2 除草剂对大肠杆菌多重耐药性的影响
在验证了除草剂能够促进大肠杆菌单一耐药性产生的基础上, 进一步探究除草剂是否诱导多重耐药性的产生.分别从草甘膦(标记为30d-Gly)、草铵膦(标记为30d-Glu)和麦草畏(标记为30d-Dic)暴露处理30 d的菌株中, 随机挑选9株(每种除草剂3株)耐受庆大霉素的突变菌株并测试其多重耐药能力.结果如图 3(a)所示, 经过除草剂诱变的突变菌株对Chl、Gen、Str和Tet抗生素均表现出耐药性, 而无除草剂暴露的对照组菌株对所测试的6种抗生素表现出敏感性.同时, MIC结果显示[图 3(b)], 由除草剂诱变得到的突变菌株显著提高了四环素、氯霉素和氨基糖苷类抗生素的MIC90(P < 0.05), 表明这3种突变菌株对抗生素表现出更强的耐药能力, 并且具有多重耐药性, 进一步证实了除草剂诱导大肠杆菌产生多重耐药性.例如, 30d-Gly突变菌对Gen、Kan和Str抗性分别提高了8.9、9.3和4倍; 30d-Glu突变菌对Chl、Gen、Kan、Str和Tet的MIC90均有显著性提高(P < 0.05), 其中30d-Glu突变菌对Str抗性高达19.8倍; 30d-Dic突变菌对Chl、Kan、Rif和Str的抗性提高了1.7~6.7倍(P < 0.05).据报道, 在外源性化合物的胁迫下, E. coli通过启动AcrAB-TolC和MdtEF-TolC等外排泵将阿莫西林、氯霉素和四环素排出胞外而获得多重耐药性[12].本文中, 除草剂诱导E. coli同时对多种抗生素(Gen、Kan和Str)耐药性的提高, 可能是由于除草剂暴露导致E. coli外排泵作用增强所致. Xing等[23]的研究也发现多种农药混合物可以诱导大肠杆菌产生多种抗生素耐药性.
由于抗生素耐药性的遗传能力对其在环境中传播具有重要意义, 因此本文进一步探讨除草剂诱导的多重耐药性是否具有稳定的遗传能力.突变菌在无除草剂与抗生素选择压力下连续传代培养5 d(大约360代, 每20 min一代), 大部分抗生素的MIC90在培养前后没有发生显著的差异[图 3(c)~3(e)], 表明除草剂诱导的多重耐药性具有较稳定的遗传性, 也表明耐药的机制可能是改变遗传物质导致.因此, 多重耐药性的稳定遗传性会导致耐药基因在环境中长期存在, 进而加剧了耐药性在环境中传播的风险.
2.3 除草剂诱导大肠杆菌耐药性的机制初探
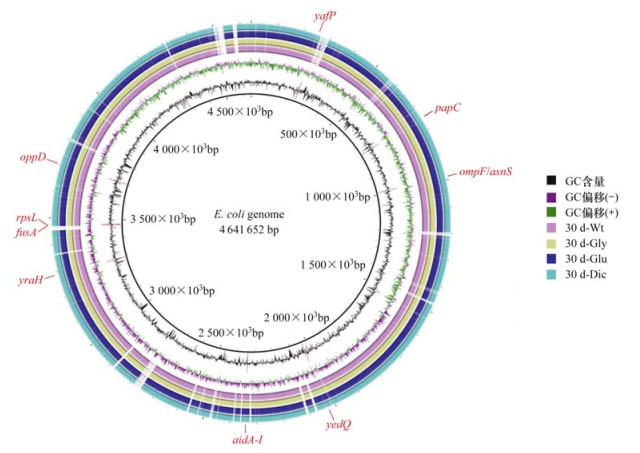
为了揭示除草剂胁迫下大肠杆菌多重耐药性产生的潜在机制, 对9株突变菌株(每种除草剂3株)及3株对照组的敏感菌株进行全基因组测序研究.与对照组的敏感菌相比, 突变菌株中总共鉴定出400多个突变(包括缺失、插入和取代), 并且大部分突变与核酸代谢、氮代谢、生物合成途径和氧化还原过程有关.根据以前的研究报道, 发现其中存在许多与抗生素抗性相关基因的突变(表 2).例如, 3株突变菌株均在外膜蛋白F(ompF)和天冬酰胺-tRNA合成酶(asnS)基因间产生了8 bp的移码突变(图 4), 这些基因突变可能涉及碳青霉烯类和头孢吡肟抗生素耐药[31]. Kurenbach等[22]的研究发现ompF基因的缺失增加了大肠杆菌对环丙沙星和四环素类抗生素的耐药性.除了共有的突变, 不同除草剂诱导的突变菌存在某些与耐药相关的特异性基因突变.例如, 核糖体蛋白质S12(rpsL)、延长因子G(fusA)和自主转运蛋白(aidA-I)基因的取代突变仅在30d-Gly突变菌出现.有研究表明, rpsL、fusA和aidA-I的基因突变与链霉素、黄霉素和氨苄青霉素耐药性相关[32~34]; 在30d-Glu突变菌中检测到菌毛蛋白(yraH), 双鸟苷酸环化酶(yedQ)和寡肽转运蛋白(oppD)的基因SNP突变, 这与庆大霉素、多黏菌素、氨基糖苷类和大环内酯类抗生素的耐药性相关[35~37]; 30d-Dic突变菌在N-乙酰基转移酶(yafP)
和外膜蛋白(papC)基因上发生点突变, 已有研究证明这些基因的yafP和papC突变有助于提高氨基糖苷类和β-内酰胺类抗生素耐药性[38, 39].总之, 这些突变可能在破坏细胞膜通透性、生物膜形成、毒力特性、代谢和应激反应等方面发挥重要作用[40~42], 以上结果证实了多重耐药性是除草剂胁迫大肠杆菌产生基因突变所导致的.
表 2
(Table 2)

表 2 突变菌株基因组中与耐药性相关的突变1)
Table 2 Genetic mutations associated with antibiotic resistome in the genome of mutant strains
| 基因 |
突变 |
注释 |
功能 |
30d-Gly |
30d-Glu |
30d-Dic |
耐药性 |
文献 |
| ompF/asnS |
Δ8 bp |
Intergenic (TAAAAAAA) |
外膜蛋白F/天冬酰胺- tRNA合成酶 |
+ |
+ |
+ |
碳青霉烯类头孢吡肟 |
[31] |
| rpsL |
G→T |
R85S(ACG→ACT) |
核糖体蛋白质S12 |
+ |
- |
- |
链霉素 |
[32] |
| fusA |
G→A |
A607V(CGC→CAC) |
延伸因子G |
+ |
- |
- |
黄霉素 |
[33] |
| aidA-I |
A→T |
F1053Y(AAA→ATA) |
自主转运蛋白 |
+ |
- |
- |
氨苄青霉素头孢菌素 |
[34] |
| yraH |
A→C |
I137L(ATT→CTT) |
菌毛蛋白 |
- |
+ |
- |
庆大霉素 |
[35] |
| yedQ |
C→T |
D99N(ATC→ATT) |
双鸟苷酸环化酶 |
- |
+ |
- |
多黏菌素诺氟沙星 |
[36] |
| oppD |
T→A |
E68V(CTC→CAC) |
寡肽转运蛋白 |
- |
+ |
- |
大环内酯类氨基糖苷类 |
[37] |
| yafP |
C→T |
Y151H(CAT→TAT) |
N-乙酰基转移酶 |
- |
- |
+ |
氨基糖苷类 |
[38] |
| papC |
C→T |
M1185I(CAT→TAT) |
外膜蛋白 |
- |
- |
+ |
β-内酰胺类 |
[39] |
| 1)+表示存在相应的突变, -表示无相应突变 |
|
表 2 突变菌株基因组中与耐药性相关的突变1)
Table 2 Genetic mutations associated with antibiotic resistome in the genome of mutant strains
|
为了进一步探究除草剂胁迫大肠杆菌产生基因突变的原因, 通过查阅前人的研究发现, 抗生素通过激活ROS介导的氧化应激反应和SOS反应, 从而诱导基因突变产生抗生素耐药性[11].因此本文检测了除草剂是否诱导大肠杆菌产生强氧化性的ROS(·OH、H2O2和O2-·).结果表明, 在10 mg·L-1的暴露浓度下3种除草剂不会显著影响·OH、H2O2和O2-·的浓度(数据未显示), 这表明除草剂诱导的基因突变不是由ROS驱动, 其潜在的突变机制需进一步研究.
综上所述, 除草剂诱导大肠杆菌产生多重耐药性的可能机制如图 5所示.当大肠杆菌暴露于环境浓度的除草剂时, 会对其造成一定的选择性压力, 大肠杆菌为了维持自身生存而产生适应性进化, 从而导致耐药相关的基因(如ompF、yraH、asnS和rspL等)发生突变, 进而影响转运蛋白、外膜孔蛋白和多药外排泵编码基因的调控, 最终增强了对抗生素的耐受能力.
3 结论
本研究结果表明, 草甘膦、草铵膦和麦草畏常用除草剂暴露显著增加细菌耐受抗生素的突变频率, 并且不同除草剂之间作用效果相似, 最终诱导大肠杆菌产生可遗传的多重耐药性(氨基糖苷类、四环素类和氯霉素类抗生素), 抗生素耐药性最高提高19.8倍.全基因组测序分析表明, 除草剂诱导的多重耐药性可能由膜蛋白(ompF、papC)、菌毛蛋白(yraH)和核糖体(rpsL)等染色体上的基因突变导致.本文发现环境除草剂的残留对细菌造成一定的选择压力, 增加耐药相关基因的突变频率, 从而加速环境中耐药菌的出现和传播.因此, 需要重新审视与评估环境中除草剂等非抗生素类化学物质在加速抗生素耐药传播的潜在风险.
 2021, Vol. 42
2021, Vol. 42