2. 华东师范大学生态与环境科学学院, 上海市城市化生态过程与生态恢复重点实验室, 上海 200241;
3. 上海污染控制与生态安全研究院, 上海 200092
2. Shanghai Key Laboratory for Urban Ecological Processes and Eco-Restoration, School of Ecological and Environmental Sciences, East China Normal University, Shanghai 200241, China;
3. Shanghai Institute of Pollution Control and Ecological Security, Shanghai 200092, China
在过去的几十年间, 抗生素广泛应用于人类医疗和畜禽养殖领域.然而, 抗生素的不规范使用和不完全代谢导致其被释放并进入环境中, 从而诱导抗生素抗性基因(antibiotic resistance genes, ARGs)的产生和传播扩散[1].近年来, ARGs已在各种有机固体废弃物如污泥和畜禽粪便中广泛赋存迁移[2, 3].目前, 作为有机固废的重要组成部分, 餐厨垃圾等城市有机固废已成为环境中ARGs的来源和储存库之一[4, 5].已有研究报道, ARGs广泛存在于如萝卜、西红柿和生菜等蔬菜与肉类和海产品中, 餐厨垃圾的生物处理过程中也检出高丰度的ARGs(例如四环素类抗性基因:tetM、tetQ和tetW以及磺胺类抗性基因:sul1和sul2)[4, 6].此外, ARGs可能在生物处理系统中通过水平基因转移(horizontal gene transfer, HGT)方式在耐药菌甚至人类致病菌间传播, 不仅提高了ARGs的传播风险, 还对环境和人体健康造成了潜在威胁[7, 8].
厌氧消化不仅能够实现有机固废的减量化, 还可实现可持续能源回收和减少有机固废潜在污染风险, 是目前城市有机固废处理的重要技术[9].此外, 已有研究表明厌氧消化过程对城市污泥中部分ARGs具有削减效果, 可抑制ARGs在环境中传播扩散[10, 11], 但仍缺乏规律性认识.如Ma等[11]的研究发现厌氧消化过程对污泥中几种磺胺类和四环素类抗性基因具有削减作用, 但红霉素类抗性基因反而得到富集.目前, 餐厨垃圾等城市有机固废厌氧消化过程中ARGs和抗性细菌的行为特征和传播机制研究尚不明确.
活性炭具有比表面积大、孔隙结构发达和导电性质优良等特点[9], 已被证实对厌氧生物处理产甲烷效率和系统稳定性具有良好的提升效果[12].活性炭能够为微生物提供定殖“位点”, 从而影响厌氧生物处理中微生物生长与群落演替, 且活性炭可促进微生物间直接电子传递[13, 14].ARGs的赋存与迁移转化与微生物群落组成与多样性有关[15].因此, 活性炭可能通过影响微生物生长或微生物间HGT影响厌氧消化过程中ARGs的增殖和迁移转化.Zhang等[16, 17]的研究发现活性炭添加促进有机固废厌氧消化过程几种四环素类和磺胺类抗性基因削减, 但是其促进效果与厌氧消化的底物有关.此外, 并非所有类型ARGs均受活性炭影响[18].因此, 活性炭添加对城市有机固废厌氧消化过程中不同类型ARGs行为特征的影响及其潜在机制仍不清楚, 需进一步研究.
基于此, 本研究通过在城市有机固废厌氧消化体系中添加两种不同粒径的活性炭, 采用定量PCR(qPCR)方法和高通量测序技术分别对目标基因和细菌群落进行分析, 以探究多种类型ARGs和整合子基因在城市有机固废厌氧消化过程中的变化特征, 解析活性炭添加对ARGs行为特征的影响以及ARGs变化的潜在微生物机制, 以期为了解城市有机固废厌氧消化过程中ARGs的转归特征以及外源添加活性炭对ARGs的影响机制提供理论基础.
1 材料与方法 1.1 厌氧消化基质、接种物与活性炭本实验采用的城市有机固废底物为餐厨垃圾和果蔬垃圾的混合基质, 餐厨垃圾主要由20%米饭、10%面条、20%卷心菜、15%土豆、6%豆腐、5.8%胡萝卜、8%猪肉、7%鸡肉和7%鸡蛋组成, 果蔬垃圾主要由30%生卷心菜、12%生土豆、8%胡萝卜、20%苹果、20%香蕉皮和10%橘子皮组成.使用电动搅拌器(Yalixi, XP07, China)将餐厨垃圾和果蔬垃圾分别打碎均化, 并过10目筛以去除大颗粒物质.接种物取自上海黎明有机质固废处理厂稳定运行的中温厌氧消化反应器, 在实验前将接种物置于(35±0.2)℃环境中驯化14 d.在加入反应器前, 颗粒活性炭(granular activated carbon, GAC, 0.84~1 mm)和粉末活性炭(powered activated carbon, PAC, 61~75 μm)经去离子水多次清洗后, 烘干备用.
1.2 厌氧反应器构建与运行厌氧反应器为120 mL的玻璃血清瓶, 工作体积为90 mL.餐厨垃圾和果蔬垃圾根据挥发性固体(volatile solid, VS)比值为1∶2加入反应器中, 反应器内基质的总VS量固定为1.9 g, 接种物的总VS量为1.9 g, 总固体(total solid, TS)量为5.4 g.设置R0为对照组, R0包含接种物和混合基质, 但不添加GAC和PAC, 设置6组处理组, 分别添加不同剂量和粒径的活性炭材料, 设为R1(5 g·L-1 GAC)、R2(10 g·L-1 GAC)、R3(15 g·L-1 GAC)、R4(5 g·L-1 PAC)、R5(10 g·L-1 PAC)和R6(15 g·L-1 PAC), 对照组和6个处理组均设置3个平行.采用缓冲液(5 mol·L-1 NaOH和5 mol·L-1 HCl)将混合体系的初始pH值调至约7.0.用橡胶塞和铝盖将反应器密封, 并用高纯氮气冲洗顶空1~2 min, 以保证反应器内厌氧条件.最后将反应器置于恒温振荡培养箱(35±0.2)℃, 振荡频率为130 r·min-1.当生物气产量几乎停滞时停止实验, 共85 d.
1.3 定量PCR与细菌群落分析分别取厌氧消化初始和各组最终体系的混合基质用于DNA提取和微生物分析.使用DNA提取试剂盒(MoBio Laboratories)提取样品的DNA, 微量紫外分光光度计(Thermo Fisher Scientific, USA)用于检测DNA浓度和纯度.本实验采用qPCR方法对ARGs、整合子和细菌16S rRNA进行定量分析, 共选取8种ARGs和2种整合子基因作为目标基因, 包括氨基糖苷类抗性基因(aadA1)、大环内酯类抗性基因(ermB和mefA)、β-内酰胺类抗性基因(blaOXA)、四环素类抗性基因(tetM和tetQ)、磺胺类抗性基因(sul1和sul2)和整合子基因(intl1和intl2).这10种目标基因是文献[4, 5]报道的餐厨垃圾中检出的几种典型ARGs和可移动遗传元件(mobile genetic elements, MGEs).目标基因的扩增引物序列信息和反应条件如表 1所示.目标基因和细菌16S rRNA基因的qPCR反应体系分别为10 μL(2×SYBR Green qPCR mix为5.0 μL, 引物F为0.5 μL, 引物R为0.5 μL, DNA模板为1.0 μL和ddH2O为3.0 μL)和20 μL(2×SYBR Green qPCR mix为10.0 μL, 引物F为1.0 μL, 引物R为1.0 μL, DNA模板为1.0 μL和ddH2O为7.0 μL).qPCR的反应程序为:预变性期:95℃, 5 min; 变性:94℃, 30 s; 退火:30 s; 再延伸:72℃, 30 s(读板); 最后延伸:72℃, 7 min; 溶解曲线:65~95℃, 每5 s升温0.5℃.为保证实验数据的准确性和可靠性, 实验样品设置3个平行.
|
|
表 1 qPCR的引物序列和反应程序 Table 1 Primer sequences and procedures for qPCR |
对DNA样品进行PCR扩增(引物为338F和806R), 通过Illumina MiSeq-PE250平台对16S rRNA的V3~V4区进行高通量测序, 该过程由上海派森诺生物科技股份有限公司完成.
1.4 数据分析采用SPSS 23(IBM, USA)进行单因素方差分析和皮尔逊相关性分析, 采用Origin 9.3(Origin Lab Corporation, USA)绘制柱形图, 采用R3.4.2绘制热图和相关性图, 采用Gephi 0.9.2进行网络分析.
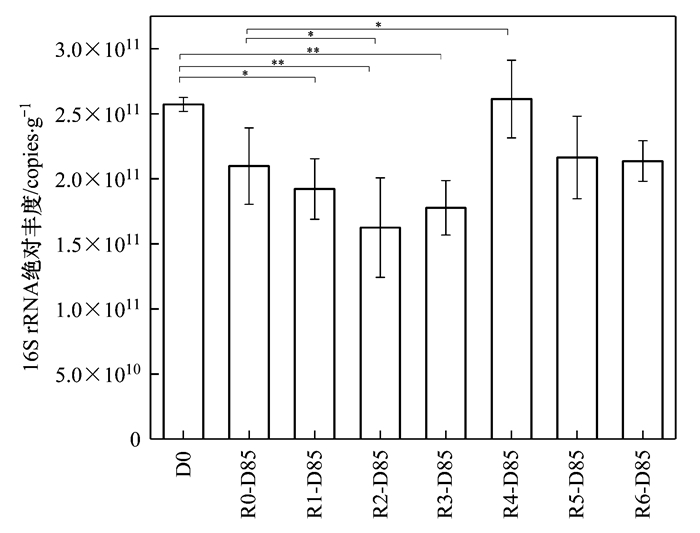
2 结果与讨论 2.1 活性炭对厌氧消化过程生物量变化的影响16S rRNA拷贝数可用于衡量厌氧消化基质中细菌生物量[19].厌氧消化过程中16S rRNA绝对丰度的变化以及活性炭对16S rRNA变化的影响如图 1所示.在厌氧消化初始体系(0 d)中, 16S rRNA基因的绝对丰度(以干物质计, 下同)为(2.57×1011±5.33×109)copies·g-1.相比于初始, 除R4外, 最终体系(85 d)中R0(对照组)与其它处理组的16S rRNA绝对丰度均降低, 特别地, R1、R2和R3这3组GAC添加组中16S rRNA绝对丰度显著降低(P < 0.05).此外, 厌氧消化最终体系中, 与对照组相比, 3组GAC添加组中16S rRNA绝对丰度降低, 相反地, R4、R5和R6这3组PAC添加组中16S rRNA绝对丰度升高.这表明厌氧消化过程能够降低系统中的细菌生物量, 且GAC添加促进了生物量的降低.
|
D0表示各组中厌氧消化初始体系(0 d), D85表示厌氧消化最终体系(85 d); *表示P < 0.05, **表示P < 0.01, ***表示P < 0.001, 下同 图 1 厌氧消化过程中16S rRNA绝对丰度的变化 Fig. 1 Variations in the absolute abundances of 16S rRNA during anaerobic digestion |
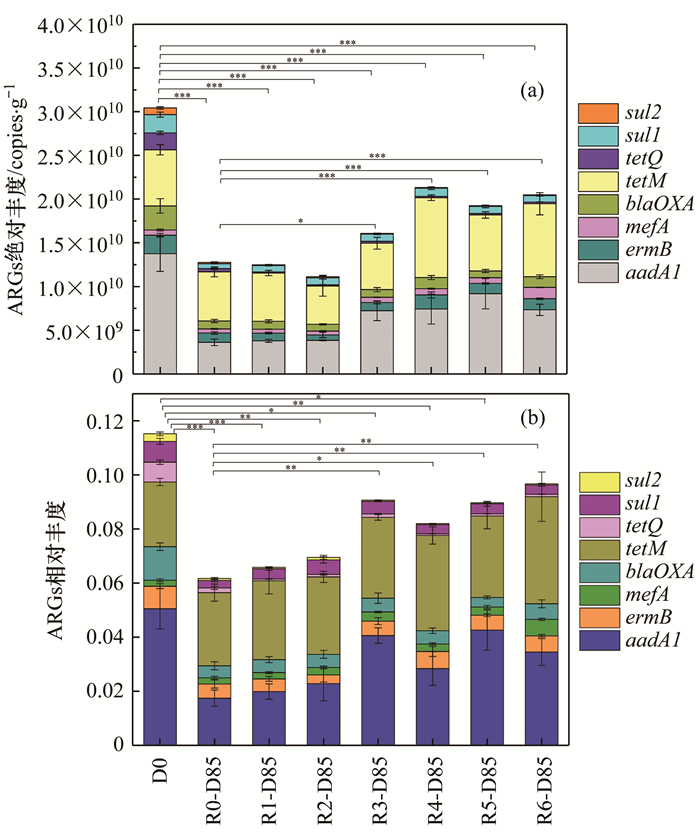
厌氧消化过程中ARGs绝对丰度的变化如图 2(a)所示.在厌氧消化初始时, 总ARGs绝对丰度最高, 为3.04×1010 copies·g-1.无论是否添加活性炭, 厌氧消化最终体系中总ARGs绝对丰度均极显著降低(P < 0.001), 总ARGs的削减率为29.95%~63.40%.表明厌氧消化过程对初始体系中总ARGs具有良好的削减效果, 与已有研究的结果一致[16, 20].与对照组相比, R4、R5和R6这3组PAC添加组中总ARGs绝对丰度均极显著升高(P < 0.001), 总ARGs削减率显著下降, 为29.95%~36.77%.这表明, PAC添加不利于城市有机固废厌氧消化过程中总ARGs的削减, 甚至可能抑制厌氧消化过程对总ARGs的去除作用.除R3以外, GAC添加对总ARGs的削减效果无显著影响.此外, 与对照组相比, 除tetQ和sul2外, 3组PAC添加组中其它6种ARGs绝对丰度均升高, 表明PAC添加有利于多种类型ARGs增殖.这可能是由于PAC添加促使厌氧消化体系中生物量增加, 微生物活性增强, 有助于多种类型ARGs增殖和传播.
|
图 2 厌氧消化过程中目标ARGs绝对丰度和相对丰度的变化 Fig. 2 Variations in the absolute abundances and relative abundances of the target ARGs during anaerobic digestion |
图 2(b)显示了ARGs相对丰度的变化, 与初始体系相比, 各组的最终体系中总ARGs的相对丰度均显著降低(P < 0.05), 表明经过厌氧消化过程各组中总ARGs得到削减.此外, 相比于对照组, GAC和PAC添加组的总ARGs相对丰度均升高, 表明不同粒径活性炭添加均可能不利于ARGs削减, 其中PAC的抑制效果更显著.在厌氧消化初始时, aadA1和tetM占比最高, 分别占总ARGs的43.85%和20.73%, 其次是blaOXA和ermB.厌氧消化后, 对照组和活性炭添加组中, aadA1、blaOXA和ermB相对丰度均降低, 而tetM相对丰度均升高.因此, 与其它目标基因相比, tetM更难以被去除, 趋于在城市有机固废厌氧消化系统及后续产品中赋存.tetM已广泛存在于各种有机固废中, 且研究表明tetM在餐厨垃圾厌氧消化处理过程中丰度变化较小, 较难被去除[4], 这与本研究的结果一致.
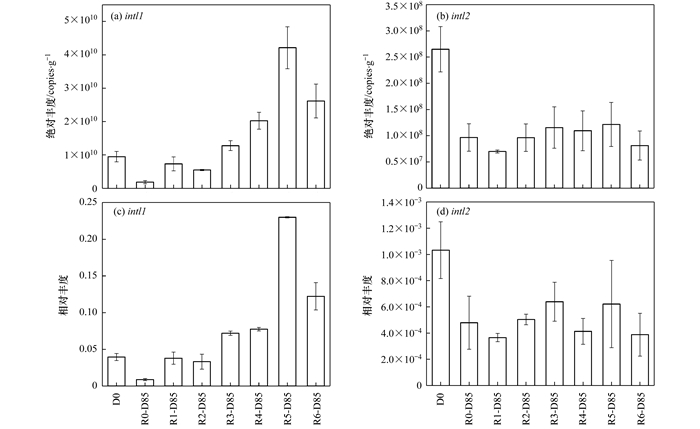
2.3 活性炭对厌氧消化过程遗传元件变化的影响HGT能够通过整合子、转座子和质粒等MGEs介导在微生物间发生, 从而促使ARGs传播扩散[21].可移动整合子在细菌间HGT中起重要作用, 本研究选取一类整合子(intl1)和二类整合子(intl2)探究活性炭添加对厌氧消化过程HGT的影响.厌氧消化过程中intl1和intl2绝对丰度的变化如图 3(a)和图 3(b)所示, intl1绝对丰度远高于intl2.厌氧消化初始体系中intl1绝对丰度为(9.49×109±1.57×109)copies·g-1.相较于初始体系, 在最终体系中, 对照组中intl1绝对丰度降低, 削减率为80.43%.然而GAC和PAC添加的6组中intl1绝对丰度均高于对照组, 表明活性炭添加可能不利于intl1削减.在3组PAC添加组中, 与初始体系相比, intl1绝对丰度升高, intl1反而得到富集.与对照组相比, GAC和PAC添加对intl2绝对丰度变化的影响均较不明显.相似地, 如图 3(c)和图 3(d)所示, intl1相对丰度在3组PAC添加组中均升高, GAC和PAC添加后intl2相对丰度变化不明显.因此, PAC添加组中intl1显著富集, 可能促使ARGs传播, 从而影响厌氧消化过程对城市有机固废中ARGs的削减效果.
|
图 3 厌氧消化过程中intl1和intl2绝对丰度与相对丰度的变化 Fig. 3 Variations in the absolute abundances and relative abundances of intl1 and intl2 during anaerobic digestion |
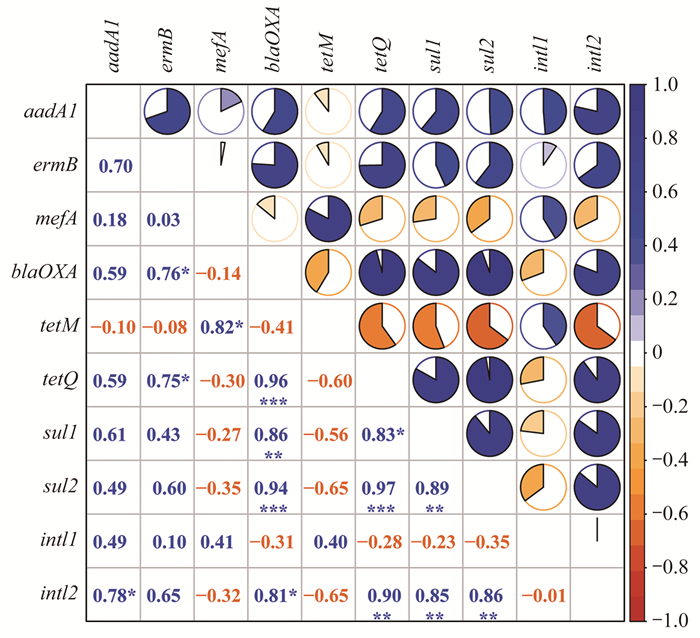
ARGs在环境中传播扩散往往由MGEs介导, 而整合子基因能够参与ARGs在微生物间的水平转移过程[22].ARGs和整合子基因间的皮尔逊相关关系如图 4所示.intl1与各种ARGs间均不存在显著相关性.intl2相对丰度显著低于intl1, 但是intl2与多种类型ARGs均显著正相关(P < 0.05), 特别是与tetQ、sul1和sul2极显著正相关(P < 0.01), 表明intl2可能介导ARGs特别是tetQ、sul1和sul2的跨细菌水平转移过程.已有研究报道sul1和sul2这2种磺胺类抗性基因在环境中的传播扩散常由整合子介导[23].值得注意的是, blaOXA、tetQ、sul1和sul2这4种ARGs两两之间均存在显著正相关关系(P < 0.05), 这表明多种ARGs可能同时存在于同一宿主微生物或同一基因盒中, 且水平转移可能促进ARGs传播.
|
图 4 ARGs和整合子基因间的皮尔逊相关关系 Fig. 4 Pearson's correlations of ARGs and integron genes |
16S rRNA基因测序分析了城市有机固废厌氧消化过程中细菌群落演替和活性炭对细菌群落变化的影响.如图 5(a)所示, 厌氧消化初始体系中的优势细菌主要为梭菌纲(Clostridia, 57.06%)、拟杆菌纲(Bacteroidia, 21.14%)和厌氧绳菌纲(Anaerolineae, 2.42%).经过厌氧消化过程后, 细菌群落发生了变化, Clostridia和互营养菌纲(Synergistia)细菌相对丰度升高, Bacteroidia相对丰度降低.此外, 与对照组相比, 3组PAC添加组中Clostridia相对丰度均明显升高, 从71.84%升至82.90%~83.81%, 不同添加剂量组间无明显差异.Clostridia属于厚壁菌门(Firmicutes), 是污泥和餐厨垃圾等有机固废厌氧消化系统中的主要优势细菌, 具有高效降解有机物的能力[24, 25].Clostridia主要参与有机固废厌氧消化过程中蛋白质、脂质和碳水化合物等大分子有机物向乙酸和丙酸等挥发性脂肪酸(volatile fatty acids, VFAs)的转化过程[24, 26].由此可见, PAC添加促使Clostridia富集, 可能利于有机物转化和VFAs产生.
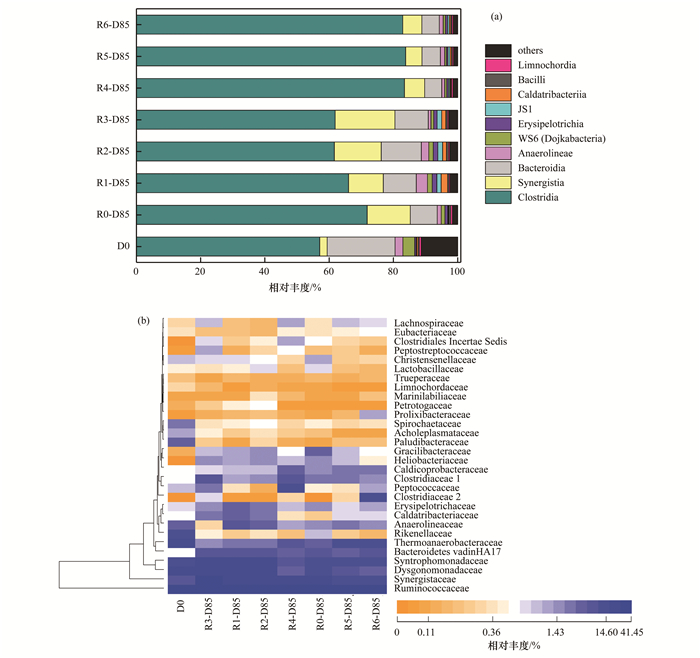
|
图 5 厌氧消化过程中细菌纲水平和科水平(丰度前30科)的相对丰度 Fig. 5 Relative abundances of bacteria at the class level and family level (top 30) during anaerobic digestion |
为了进一步了解活性炭对细菌群落组成的影响, 图 5(b)展示了细菌科水平的组成(丰度前30).厌氧消化初始体系中主要优势科细菌为瘤胃菌科(Ruminococcaceae, 22.34%)、Dysgonomonadaceae(9.26%)、热厌氧杆菌科(Thermoanaerobacteraceae, 6.41%)、互营单胞菌科(Syntrophomonadaceae, 5.37%)、理研菌科(Rikenellaceae, 4.15%)和互养菌科(Synergistaceae, 2.32%).经过厌氧消化过程后, 对照组和各处理组中Ruminococcaceae(29.27%~41.45%)和Synergistaceae(5.08%~13.41%)相对丰度均明显升高.Ruminococcaceae属于Clostridia, 参与纤维素等复杂有机物的水解过程且利于VFAs产生[27, 28], Synergistaceae具有促进VFAs转化为乙酸的能力[28, 29].由此可见, 厌氧消化过程富集了参与有机物降解和产酸过程的细菌.在最终体系中, 与对照组相比, GAC添加组中Syntrophomonadaceae的相对丰度明显升高, 从4.26%升至6.49%~10.46%, PAC添加组中Caldicoprobacteraceae的相对丰度明显升高, 从0.81%升至1.19%~1.45%.Syntrophomonadaceae具有协同降解VFAs和产甲烷能力[30], Caldicoprobacteraceae能够在厌氧消化前期降解蛋白质和半纤维素等大分子有机物, 促进有机物水解[31].GAC和PAC添加的6组中, 梭菌科(Clostridiaceae)细菌均明显富集, 有助于降解碳水化合物产生乙酸和丁酸[25].因此, GAC和PAC添加可能利于参与有机物降解和VFAs产生与转化过程的功能细菌富集, 加速厌氧消化过程.
2.6 抗性基因与细菌群落的相关关系为了分析ARGs的潜在宿主微生物和探究ARGs与细菌群落的内在联系, 对ARGs、整合子和细菌群落进行了网络分析.如图 6所示, 厌氧消化过程中, ARGs的潜在宿主细菌主要属于Clostridia、Bacteroidia和Synergistia.部分细菌与多种ARGs显著正相关(P < 0.05), 例如Gelria与aadA1、ermB和blaOXA均存在显著正相关关系, Rikenellaceae RC9 gut group和Acholeplasma均与blaOXA、tetQ、sul1、sul2和intl2显著正相关, Tissierella与aadA1和ermB显著正相关, Ruminiclostridium 1与aadA1和intl1显著正相关(P < 0.05).这表明在城市有机固废厌氧消化过程中, 某些细菌可能成为多种ARGs的潜在宿主, 携带多种类型ARGs, 可促进ARGs传播扩散.如图 2和图 3所示, aadA1、ermB、blaOXA和intl1在3组PAC添加组中均得到富集.这几种目标基因的潜在宿主细菌如Gelria、Tissierella和Ruminiclostridium 1均属于Clostridia, 且Clostridia相对丰度在PAC添加组中均明显升高.因此, 在PAC添加组中, aadA1、ermB、blaOXA和intl1的增加可能与Clostridia的富集有关.表明PAC添加时, 潜在宿主细菌的富集是目标基因增殖的重要原因, 从而影响总ARGs的变化特征, 且Clostridia可能是厌氧消化过程中ARGs消长和传播的主要驱动因子.厌氧消化系统中, PAC添加可能促使某些功能细菌生长, 从而加速ARGs增殖.此外, PAC可能促进微生物间能量和电子传递[32], 其对微生物间生物电连接的强化作用可能导致微生物间发生遗传物质的传递, 从而促使ARGs通过MGEs在微生物间水平转移, 提升ARGs的传播风险.
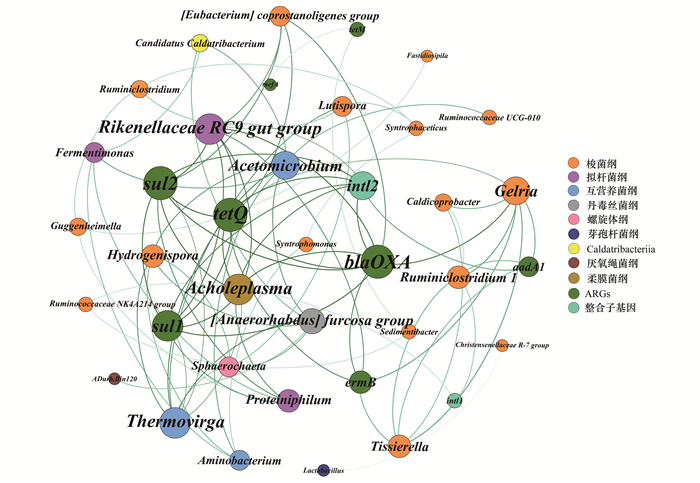
|
网络分析基于Pearson相关性分析(P < 0.05), 边连接代表节点间正相关关系, 节点大小代表与该节点相关的节点数多少 图 6 厌氧消化过程中ARGs、MGEs和细菌群落(丰度前30属)的关系 Fig. 6 Connections between ARGs, MGEs, and bacterial communities (top 30 genera) during anaerobic digestion |
此外, 网络分析揭示了HGT在ARGs传播过程中的作用.intl1的潜在宿主细菌为Ruminiclostridium 1和Ruminococcaceae NK4A214 group, intl2的潜在宿主为Rikenellaceae RC9 gut group和Acholeplasma.由ARGs和整合子间相关性分析可知, intl2与blaOXA、tetQ、sul1和sul2均显著正相关, 且blaOXA、tetQ、sul1和sul2相互之间显著正相关(P < 0.05).值得注意的是, blaOXA、tetQ、sul1、sul2和intl2拥有相同的宿主Rikenellaceae RC9 gut group和Acholeplasma.因此, 整合子对ARGs的传播具有促进作用, 且blaOXA、tetQ、sul1和sul2可能由intl2介导在这些细菌间水平转移扩散.已有研究报道Acholeplasma中包含典型致病菌[1], 在城市有机固废厌氧消化过程中具有携带多种类型ARGs的能力, 且ARGs在微生物间的水平转移会加剧致病菌的潜在风险, 从而对厌氧消化后续产品利用的安全性产生威胁.
3 结论(1) 无论是否添加活性炭, 厌氧消化过程对城市有机固废厌氧消化初始体系中的总ARGs均具有削减作用.
(2) 厌氧消化最终体系中, 相较于对照组, PAC添加促使ARGs增殖, 即PAC添加削弱了厌氧消化过程对ARGs的削减效果; GAC添加对ARGs变化无显著影响.
(3) 城市有机固废厌氧消化过程中, ARGs的潜在宿主细菌主要归属于Clostridia、Bacteroidia和Synergistia; 某些宿主细菌与多种ARGs和整合子基因显著正相关, 可促进ARGs增殖与传播扩散.
(4) PAC添加时, 宿主细菌的富集是目标基因增殖的重要原因, 且Clostridia可能是厌氧消化过程中ARGs消长的主要驱动因子.
| [1] | Su Y L, Wang J X, Xia H P, et al. Comparative network analysis revealing the mechanisms of antibiotic resistance genes removal by leachate recirculation under different hydraulic loadings[J]. Science of the Total Environment, 2019, 649: 318-326. DOI:10.1016/j.scitotenv.2018.08.361 |
| [2] |
姜明吉, 薛罡, 陈红, 等. pH值对污泥热水解/厌氧消化过程抗性基因的影响[J]. 中国给水排水, 2019, 35(13): 16-22. Jiang M J, Xue G, Chen H, et al. Effect of pH value on the decline of resistance genes in sludge during the thermal hydrolysis and anaerobic digestion combined process[J]. China Water & Wastewater, 2019, 35(13): 16-22. |
| [3] | Guo H G, Chen Q L, Hu H W, et al. Fate of antibiotic resistance genes during high-solid anaerobic co-digestion of pig manure with lignite[J]. Bioresource Technology, 2020, 303. DOI:10.1016/j.biortech.2020.122906 |
| [4] | He P J, Yu Z F, Shao L M, et al. Fate of antibiotics and antibiotic resistance genes in a full-scale restaurant food waste treatment plant: implications of the roles beyond heavy metals and mobile genetic elements[J]. Journal of Environmental Sciences, 2019, 85: 17-34. DOI:10.1016/j.jes.2019.04.004 |
| [5] | Wang P L, Qiao Z R, Li X N, et al. Fate of integrons, antibiotic resistance genes and associated microbial community in food waste and its large-scale biotreatment systems[J]. Environment International, 2020, 144. DOI:10.1016/j.envint.2020.106013 |
| [6] | Wang P, Chen X T, Liang X F, et al. Effects of nanoscale zero-valent iron on the performance and the fate of antibiotic resistance genes during thermophilic and mesophilic anaerobic digestion of food waste[J]. Bioresource Technology, 2019, 293. DOI:10.1016/j.biortech.2019.122092 |
| [7] |
黄福义, 周曙仡聃, 颜一军, 等. 生活垃圾渗滤液处理过程中抗生素抗性基因的变化特征[J]. 环境科学, 2019, 40(10): 4685-4690. Huang F Y, Zhou S Y D, Yan Y J, et al. Dynamics of antibiotic resistance genes during the municipal solid waste leachate treatment[J]. Environmental Science, 2019, 40(10): 4685-4690. |
| [8] | Zhao R X, Feng J, Liu J, et al. Deciphering of microbial community and antibiotic resistance genes in activated sludge reactors under high selective pressure of different antibiotics[J]. Water Research, 2019, 151: 388-402. DOI:10.1016/j.watres.2018.12.034 |
| [9] |
马佳莹, 汪冰寒, 乔子茹, 等. 碳基材料对餐厨垃圾厌氧消化效率和微生物群落的影响研究进展[J]. 应用与环境生物学报, 2020, 26(3): 730-738. Ma J Y, Wang B H, Qiao Z R, et al. Effects of carbon-based materials on anaerobic digestion efficiency and microbial community of food waste[J]. Chinese Journal of Applied & Environmental Biology, 2020, 26(3): 730-738. |
| [10] | Wu Y, Cui E P, Zuo Y R, et al. Influence of two-phase anaerobic digestion on fate of selected antibiotic resistance genes and class I integrons in municipal wastewater sludge[J]. Bioresource Technology, 2016, 211: 414-421. DOI:10.1016/j.biortech.2016.03.086 |
| [11] | Ma Y J, Wilson C A, Novak J T, et al. Effect of various sludge digestion conditions on sulfonamide, macrolide, and tetracycline resistance genes and class I integrons[J]. Environmental Science & Technology, 2011, 45(18): 7855-7861. |
| [12] | Zhang J S, Zhao W Q, Zhang H W, et al. Recent achievements in enhancing anaerobic digestion with carbon-based functional materials[J]. Bioresource Technology, 2018, 266: 555-567. DOI:10.1016/j.biortech.2018.07.076 |
| [13] | Yin Q D, Yang S, Wang Z Z, et al. Clarifying electron transfer and metagenomic analysis of microbial community in the methane production process with the addition of ferroferric oxide[J]. Chemical Engineering Journal, 2018, 333: 216-225. DOI:10.1016/j.cej.2017.09.160 |
| [14] |
杨波, 贾丽娟, 徐辉, 等. 投加颗粒活性炭和二氧化锰对剩余污泥厌氧消化的影响[J]. 环境科学, 2020, 41(4): 1816-1824. Yang B, Jia L J, Xu H, et al. Effect of adding granular activated carbon (GAC)/manganese dioxide (MnO2) for the anaerobic digestion of waste activated sludge[J]. Environmental Science, 2020, 41(4): 1816-1824. |
| [15] | Sentchilo V, Mayer A P, Guy L, et al. Community-wide plasmid gene mobilization and selection[J]. The ISME Journal, 2013, 7(6): 1173-1186. DOI:10.1038/ismej.2013.13 |
| [16] | Zhang J X, Chua Q W, Mao F J, et al. Effects of activated carbon on anaerobic digestion-methanogenic metabolism, mechanisms of antibiotics and antibiotic resistance genes removal[J]. Bioresource Technology Reports, 2019, 5: 113-120. DOI:10.1016/j.biteb.2019.01.002 |
| [17] | Zhang J X, Mao F J, Loh K C, et al. Evaluating the effects of activated carbon on methane generation and the fate of antibiotic resistant genes and class I integrons during anaerobic digestion of solid organic wastes[J]. Bioresource Technology, 2018, 249: 729-736. DOI:10.1016/j.biortech.2017.10.082 |
| [18] | Zhang J X, Zhang L, Loh K C, et al. Enhanced anaerobic digestion of food waste by adding activated carbon: fate of bacterial pathogens and antibiotic resistance genes[J]. Biochemical Engineering Journal, 2017, 128: 19-25. DOI:10.1016/j.bej.2017.09.004 |
| [19] |
李慧莉, 武彩云, 唐安平, 等. 不同污泥在微波预处理-厌氧消化过程中抗性基因分布及菌群结构演替[J]. 环境科学, 2021, 42(1): 323-332. Li H L, Wu C Y, Tang A P, et al. Occurrence of antibiotic resistance genes and bacterial community structure of different sludge during microwave pretreatment-anaerobic digestion[J]. Environmental Science, 2021, 42(1): 323-332. |
| [20] |
杨帆, 徐雯丽, 钱雅洁, 等. 零价铁对污泥厌氧消化过程中四环素抗性基因水平转移的作用影响[J]. 环境科学, 2018, 39(4): 1748-1755. Yang F, Xu W L, Qian Y J, et al. Effect of zero valent iron on the horizontal gene transfer of tetracycline resistance genes during anaerobic sludge digestion process[J]. Environmental Science, 2018, 39(4): 1748-1755. |
| [21] | Soucy S M, Huang J L, Gogarten J P. Horizontal gene transfer: building the web of life[J]. Nature Reviews Genetics, 2015, 16(8): 472-482. DOI:10.1038/nrg3962 |
| [22] | Ma L P, Li A D, Yin X L, et al. The prevalence of integrons as the carrier of antibiotic resistance genes in natural and man-made environments[J]. Environmental Science & Technology, 2017, 51(10): 5721-5728. |
| [23] | Hsu J T, Chen C Y, Young C W, et al. Prevalence of sulfonamide-resistant bacteria, resistance genes and integron-associated horizontal gene transfer in natural water bodies and soils adjacent to a swine feedlot in northern Taiwan[J]. Journal of Hazardous Materials, 2014, 277: 34-43. DOI:10.1016/j.jhazmat.2014.02.016 |
| [24] |
苏宇傲, 刘宏波, 毛秋燕, 等. 死菌DNA对厌氧消化污泥中抗生素抗性基因及微生物群落分析的干扰[J]. 环境科学, 2020, 41(6): 2812-2821. Su Y A, Liu H B, Mao Q Y, et al. Interference of dead cell DNA on the analysis of antibiotic resistance genes and microbial community in anaerobic digestion sludge[J]. Environmental Science, 2020, 41(6): 2812-2821. |
| [25] |
潘婧冉, 高苏, 赵国柱, 等. 餐厨垃圾厌氧消化处理主要过程的微生物群落结构分析[J]. 微生物学通报, 2019, 46(11): 2886-2899. Pan J R, Gao S, Zhu G Z, et al. Microbial community structure in the main processes of anaerobic digestion of restaurant food waste[J]. Microbiology China, 2019, 46(11): 2886-2899. |
| [26] | Su C Y, Lin X M, Zheng P, et al. Effect of cephalexin after heterogeneous Fenton-like pretreatment on the performance of anaerobic granular sludge and activated sludge[J]. Chemosphere, 2019, 235: 84-95. DOI:10.1016/j.chemosphere.2019.06.136 |
| [27] |
唐涛涛, 李江, 杨爱江, 等. 秸秆类型及配比变化对污泥厌氧消化中微生物群落的影响[J]. 化工进展, 2020, 39(2): 667-678. Tang T T, Li J, Yang A J, et al. Effects of straw type and ratio change on microbial community in anaerobic digestion of sludge[J]. Chemical Industry and Engineering Progress, 2020, 39(2): 667-678. |
| [28] | Liu Y, Wachemo A C, Yuan H R, et al. Anaerobic digestion performance and microbial community structure of corn stover in three-stage continuously stirred tank reactors[J]. Bioresource Technology, 2019, 287. DOI:10.1016/j.biortech.2019.121339 |
| [29] | Suksong W, Tukanghan W, Promnuan K, et al. Biogas production from palm oil mill effluent and empty fruit bunches by coupled liquid and solid-state anaerobic digestion[J]. Bioresource Technology, 2020, 296. DOI:10.1016/j.biortech.2019.122304 |
| [30] | Capson-Tojo G, Moscoviz R, Ruiz D, et al. Addition of granular activated carbon and trace elements to favor volatile fatty acid consumption during anaerobic digestion of food waste[J]. Bioresource Technology, 2018, 260: 157-168. DOI:10.1016/j.biortech.2018.03.097 |
| [31] | Wang Y Y, Zhang J X, Sun Y B, et al. Effects of intermittent mixing mode on solid state anaerobic digestion of agricultural wastes[J]. Chemosphere, 2020, 248. DOI:10.1016/j.chemosphere.2020.126055 |
| [32] | Xu S Y, He C Q, Luo L W, et al. Comparing activated carbon of different particle sizes on enhancing methane generation in upflow anaerobic digester[J]. Bioresource Technology, 2015, 196: 606-612. DOI:10.1016/j.biortech.2015.08.018 |
 2021, Vol. 42
2021, Vol. 42


