2. 泰安市岱岳区畜牧局, 泰安 271000;
3. 泰安市中心医院, 泰安 271000
2. Animal Husbandry Bureau of Daiyue Region, Tai'an 271000, China;
3. Center Hospital of Tai'an City, Tai'an 271000, China
动物粪便在堆肥发酵处理后, 由于其含有多种营养物质和有机物质, 常作为“农家肥/有机肥”来改良农业土壤[1].然而, 动物粪便中也含有大量耐药菌和耐药基因(antibiotic resistant genes, ARG), 虽然堆肥过程可以显著降低其耐药水平但并不能完全消除[2].因此, 动物粪肥的施用是大量耐药性进入土壤生态系统的主要途径之一, 其中的ARG会通过水平转移至土壤的固有微生物, 甚至人类致病菌中[3, 4], 同时粪肥中还存在的大量可移动遗传元件(mobile genetic elements, MGE), 如质粒、整合子和转座子等, 会进一步促进耐药性的转移和扩散[5, 6].耐药基因已然成为土壤环境的一种新型污染物.
土壤不仅是植物赖以生存的物质基础, 也是植物内生菌的主要来源, 植物内生菌是指定殖于植物组织或细胞内部的微生物群落, 其多样性一定程度上取决于根部土壤微生物菌群的多样性[7, 8].在动物粪肥改良过的土壤中, 植物根系会从中获取耐药菌, 进而可以定植于植物组织内成为内生细菌; 植物内生菌也可以作为ARG的载体, 通过水平基因转移将ARG从土壤转移到植物中[9].因此, 日常生活中直接用于生食的蔬菜, 如苦菊、生菜和甘蓝等, 在生长过程中会从粪肥改良土壤中获得耐药性的内生菌, 并且用水清洗蔬菜表面并不能去除蔬菜内部的耐药菌和耐药基因, 食用后很可能通过水平转移向人体内微生物扩散, 具有潜在的健康威胁[10~12].但是, 目前关于田间动物粪肥施用对生食蔬菜内生菌中ARG的影响以及ARG在粪便-土壤-蔬菜系统的转移路径却很少被关注.
因此, 本文以常出现于人们餐桌上凉拌菜或蔬菜沙拉的苦菊为研究对象, 采用高通量定量PCR平台, 在蔬菜基地的大田土壤中施用鸡粪肥, 分析其对土壤、苦菊根和叶片内生菌中ARG的影响, 并深入探讨ARG在粪便-土壤-蔬菜系统的潜在传播路径, 以期为在农业生态系统中动物粪肥的无害化施用提供理论依据.
1 材料与方法 1.1 样品采集2020年6月在山东省泰安市良庄蔬菜种植基地选择两个苦菊大棚.在种植苦菊前, 将备好的沙土壤(无有机肥料施用历史)分为两部分:一部分土壤与已堆肥发酵处理的鸡粪肥均匀混合(poultry manure, PM组); 另一部分土壤不施加任何粪肥(对照组).将两种土壤分别铺于两个大棚内提前挖好的畦沟中, 然后在沟内均匀撒播苦菊种子, 再覆盖一层细土.之后两个大棚内苦菊的管理方式和生长条件完全相同, 包括温度、湿度、浇灌和光照等.土壤和鸡粪肥理化性质见表 1.
|
|
表 1 土壤和鸡粪肥的理化性质 Table 1 Physicochemical properties of soil and chicken manure |
苦菊在4~5 d出苗, 大约长到150 g(约15 d)采收.每个大棚内随机选择3颗苦菊植株.用乙醇消毒剪刀将苦菊地上叶片部分和地下根分离, 并分别放入取样袋中; 以苦菊根上附着的土壤为土壤样品.共收集18份样品(PM组:3个地上叶片部分、3个地下根和3份土壤; 对照组:3个地上叶片部分、3个地下根和3份土壤).
1.2 DNA提取首先去除驻留在根和叶表面的细菌和污染物, 将叶片和地下根样品浸泡在30%双氧水(H2O2) 中30 min, 用无菌水清洗3次; 再经70%乙醇处理1 min, 无菌水清洗3次.然后, 每个土壤、叶片、地下根样品分别取0.5 g, 根据FastDNA Spin Kit for Soil DNA(MP Biomedicals, USA)试剂盒说明书提取土壤细菌DNA、苦菊组织内生细菌DNA.由NanoDrop One仪器(Thermo Fisher Scientific, USA)测定DNA浓度和纯度.
1.3 高通量定量PCR分析采用Wafergen SmartChip Real-time PCR系统(Fremont, USA) 进行高通量定量PCR(High-throughput quantitative PCR, HT-qPCR)分析, 以检测土壤和苦菊样品中ARG和MGE的多样性和丰度.Wafergen系统具有5 184个反应孔, 每个反应为100 nL体系.本研究采用了384对引物组中的296对, 主要涉及到8个主要ARG型(285对引物组)、8个转座酶、1个Ⅰ类整合子、1个临床Ⅰ类整合子和16S rRNA基因[12, 13].
将SmartChip放至Wafergen SmartChip Real-time PCR Cycler中, PCR程序设定为95℃ 10 min, 随后40个循环包括95℃ 30 s和60℃ 30 s.每个反应孔仅保留单一有效融解峰且扩增效率在1.7~2.3范围内的数据.设置循环阈值(threshold cycle, Ct)为31[14].每个样品设置3次重复, 取3次重复的平均值进行后续分析.
1.4 细菌16S rRNA基因的定量PCR分析构建qPCR分析的标准曲线, 首先对细菌16S rRNA基因进行PCR扩增并将其连接到pGEM-T Easy载体上, 随后将连接产物转化到JM109感受态细胞中, 再选择含有16S rRNA基因片段的克隆, 提取质粒DNA, 进行10倍连续稀释制备标准品.采用Bio-Rad CFX384 Real-Time PCR检测系统(Bio-Rad, USA)检测16S rRNA基因的丰度, 引物和反应程序与HT-qPCR相同.
1.5 MiSeq测序和数据处理PCR扩增16S rRNA基因的V5-V7区域, 使用排除叶绿体的引物799F/1193R, 799F(5′-AACMG GATTAGATACCCKG-3′)和1193R(5′-ACGTCATC CCCACCTTCC-3′), PCR程序参考文献[15].PCR产物经纯化回收后构建DNA文库, 随后在Illumina MiSeq平台(上海美优生物公司)上进行双末端测序.
使用QIIME管道处理原始序列数据, 将末端可配对的序列合并为约350 bp的单个序列, 去掉具有3个或多个模糊核苷酸的序列、片段平均质量分数 < 20且 < 100 nt的低质量序列.剩余序列根据97%的相似性归为操作分类单元(operational taxonomic unit, OTU), 使用BLAST算法和Greengenes数据库对每个OTU的分类进行确认, 在最终的OTU数据集中剔除嵌合体、线粒体OTU、细胞质OTU和单一序列(singletons)[16].所有的序列上传至NCBI的SRA(Sequence Read Archive)数据库, 登录号为PRJNA663721.
1.6 统计分析使用SPSS v19.0 (IBM, Armonk, NY, USA)中的单因素方差分析(ANOVA)检验两组之间ARG多样性和丰度的差异、细菌菌群相对丰度的差异.使用在线软件生成Circos图(http://circos.ca/), 显示两个处理组中ARG的分类.基于Bray-Curtis距离的非度量多维标尺(Non-metric multidimensional scaling, NMDS)分析ARGs和细菌菌群的总体分布模式.去趋势对应分析(detrended correspondence analysis, DCA)的梯度长度 < 3, 线性模型-冗余分析(redundancy analysis, RDA)作为多变量回归分析用于检测ARG与细菌菌群结构之间的相关性.Mantel检验分析ARG和MGE之间的相关性.利用Gephi(https://gephi.org/.)中Yifan-Hu layout, 示出PM组的鸡粪肥施用土壤、地下根内生菌和叶片内生菌之间共有和独有ARG和MGE.
在R语言中用vegan计算细菌菌群与ARG亚型之间(在所有样本中≥50%)所有可能的配对Spearman相关系数(ρ)来构建相关性矩阵, 将细菌菌群与ARG之间的潜在关系可视化, 仅正向(ρ≥0.8)且显著(P≤0.01)相关的两个节点被用于网络分析.
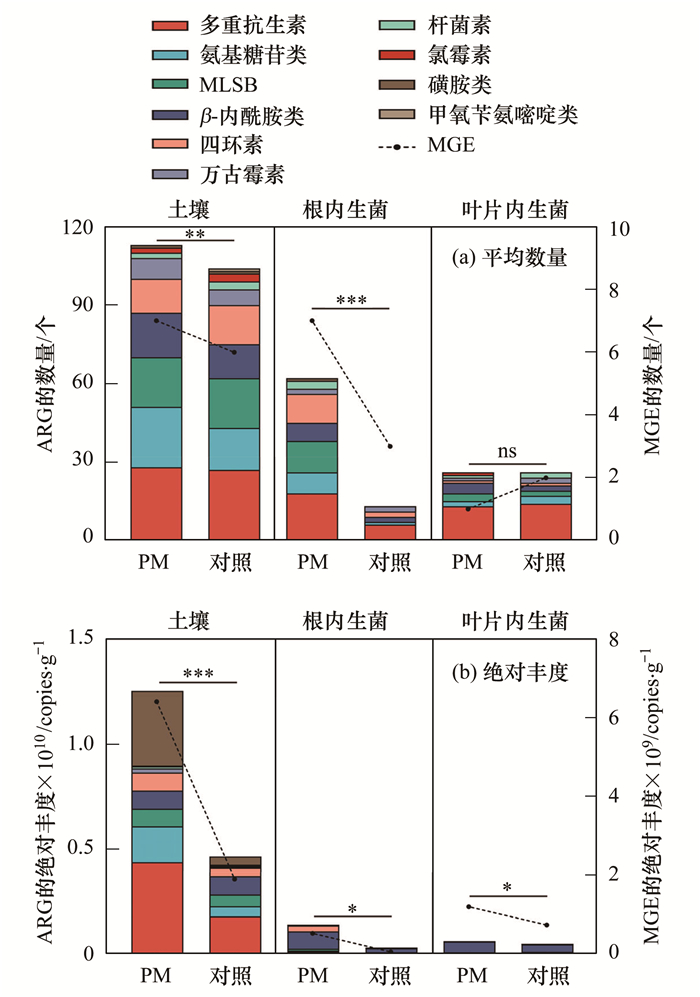
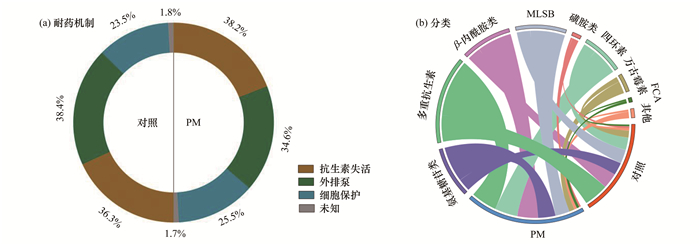
2 结果与讨论 2.1 土壤和苦菊内生菌中ARG的组成分布分析了土壤和苦菊内生菌中ARG多样性[图 1(a)].总体上, 土壤和苦菊内生菌中共检测到149个ARG亚型.每个样品中检测到的ARG数量在16~120之间, 其中土壤中ARG(110~120)的多样性最大, 其次是苦菊的根(16~69)和叶片(27~28).同时, 所有样品中ARG均可归为3种主要的耐药机制[图 2(a)]:抗生素失活、外排泵和细胞保护, 其中抗生素失活和外排泵是最主要的两种, 所有样品中多重抗生素耐药基因检测频率最高,其次是β-内酰胺类、氨基糖苷类、大环内酯类-林可酰胺-链霉菌素B类(macrolides-lincosamide-streptogramines B, MLSB)和四环素类耐药基因[图 2(b)].
|
*表示P < 0.05; **表示P < 0.01; ***表示P < 0.001; ns表示无统计学意义(no significance), 下同 图 1 土壤、苦菊根内生菌和叶片内生菌中的ARG和MGE Fig. 1 ARG and MGE detected in soil, root endophytes, and leaf endophytes of Cichorium endivia L. |
|
图 2 两个处理组中ARG所属的耐药机制和基因型分类 Fig. 2 Resistance mechanisms and classification of ARG in two treatment groups |
分析了土壤和苦菊内生菌中ARG的绝对丰度[图 1(b)].土壤中ARG的总丰度最高, 为3.36×106~4.37×109 copies·g-1; 蔬菜内生菌中ARG的总丰度为1.89×106~8.36×108 copies·g-1.土壤样品中以多重抗生素耐药基因的丰度最高, 而苦菊的根和叶片内生菌中以β-内酰胺类耐药基因丰度最高.本结果与之前报道一致, 土壤一直是耐药菌增殖和ARG水平转移的研究热点, 并且由于蔬菜扎根于土壤, 从中摄取营养或者污染物, 也已经成为潜在的ARG转移途径[17~19].
2.2 鸡粪肥施用对土壤和苦菊内生菌中ARG的影响鸡粪肥施用增加了土壤和苦菊内生菌中ARG的多样性和丰度.与对照组相比, PM组中鸡粪肥的施用增加了土壤(P < 0.01)和苦菊根内生菌(P < 0.001)中的ARG数量[图 1(a)], 其中在PM组根内生菌的多重抗生素、氨基糖苷类、MLSB、β-内酰胺类和四环素耐药基因数量均显著增加, 叶片内生菌中ARG数量无明显增加.与对照组相比, PM组中鸡粪肥的施用显著提高了土壤(P < 0.001)、苦菊根内生菌(P < 0.05)和叶片内生菌中(P < 0.05)的ARG丰度[图 1(b)].PM组土壤和苦菊根内生菌中ARG丰度均比对照组上升了1个数量级, 苦菊叶片内生菌中ARG丰度也比对照组高出1.3倍.这说明施加鸡粪肥的土壤中ARG的多样性和丰度显著高于原来的土壤, 导致苦菊在生长过程中有更多的ARG通过转移或者通过细菌的携带进入蔬菜的根和叶片[20, 21].
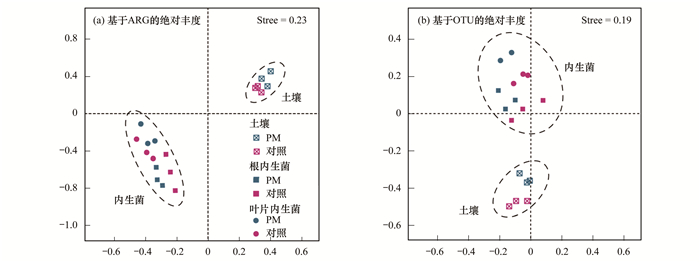
基于Bray-Curtis距离的NMDS分析显示[图 3(a)], ARG组成的分布是按照不同处理组(PM组和对照组)和不同样品类型(土壤、根内生菌、叶片内生菌)聚集的.可以看出ARG组成结构明显受到处理方式和样品类型的影响.
|
图 3 不同处理组和不同样本类型之间ARG和OTU的NMDS分析 Fig. 3 NMDS analysis of ARG and OTU between different sample types and different treatments |
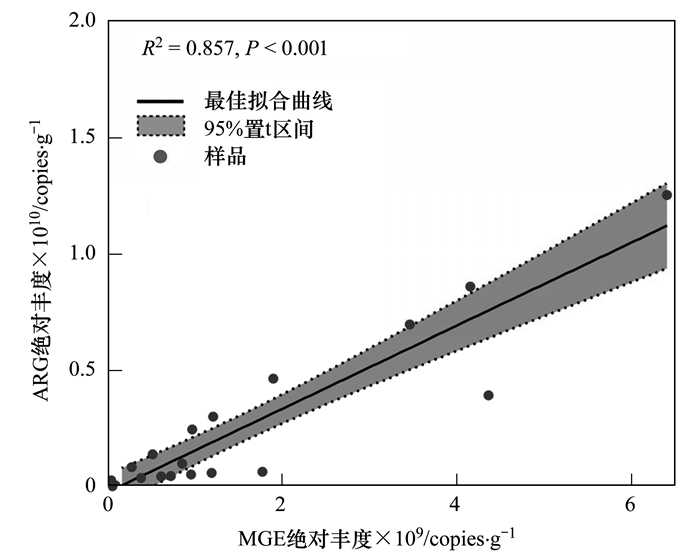
总体上, 鸡粪肥施用增加了土壤和苦菊内生菌中MGE的多样性和丰度.在所有样品中共检测到10个MGE[图 1(a)].土壤中MGE的绝对丰度为2.12×106~3.66×109 copies·g-1, 苦菊内生菌中MGE的绝对丰度为1.66×106~1.20×109 copies·g-1[图 1(b)].与对照组相比, 施用鸡粪肥后土壤和苦菊内生菌中MGE的数量和丰度均增高.经回归分析(图 4), MGE丰度与ARG总丰度之间属于线性正相关关系(R2=0.857, P < 0.001).Mantel检验(表 2)进一步显示MGE丰度(转座酶和整合子)与多个ARG型具有强的正相关关系, 如:多重抗生素、氨基糖苷类、MLSB、四环素、万古霉素、杆菌素和磺胺类耐药基因.MGE对ARG水平基因转移起着关键作用, 因此这些结果说明鸡粪肥的施用可能会进一步促进ARG在土壤和蔬菜内生菌中的增加和散播.
|
图 4 ARG和MGE绝对丰度之间相关性的的回归分析 Fig. 4 Regression analysis of relationship between the absolute abundance of ARG and MGE |
|
|
表 2 ARG和MGE绝对丰度之间相关性的Mantel检验1) Table 2 Mantel test of correlations between the absolute abundance of ARG and MGE |
2.3 土壤和苦菊中细菌菌群及其与ARG的共存模式
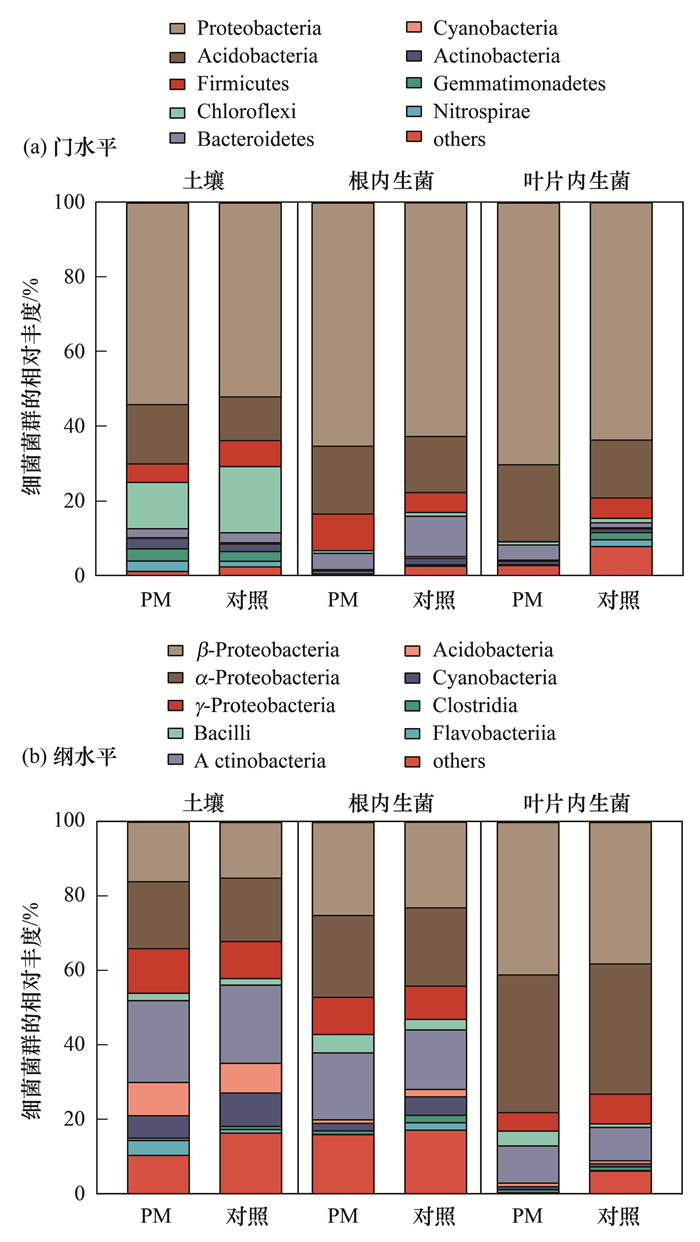
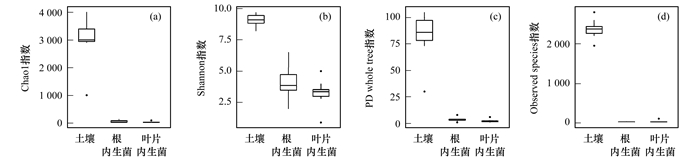
分析了土壤细菌菌群和苦菊根和叶片内生菌群的结构.本研究中共检测到47个门和94个纲的OTU.在门水平[图 5(a)], 所有样品中变形菌门(Proteobacteria)的丰度最高(51.96%~70.60%); 与对照组相比, 在PM组的土壤、苦菊根和叶片中变形菌门丰度均有增加.在纲水平上[图 5(b)], β-变形菌纲(β-Proteobacteria, 15.0%~41.0%)、α-变形菌纲(α-Proteobacteria, 17.0%~37.0%)、放线菌纲(Actinobacteria, 9.0%~22.0%)是土壤、苦菊根内生菌和叶片内生菌的优势菌群.经Chao1、Shannon、PD whole tree和Observed species多样性分析(图 6), 土壤中细菌菌群的多样性最高, 其次是根内生菌和叶片内生菌.基于Bray-Curtis距离的NMDS分析[图 3(b)], 显示细菌菌群结构分布同样会受到样品类型(土壤、苦菊根和叶片)的影响.
|
图 5 两种处理组土壤、根内生菌和叶片内生菌OTU的分布 Fig. 5 Distributions of the OTU in the soil, root endophytes, and leaf endophytes among the two treatments |
|
图 6 不同样本类型中细菌菌群α多样性分析 Fig. 6 Bacterial α-diversity in different sample types |
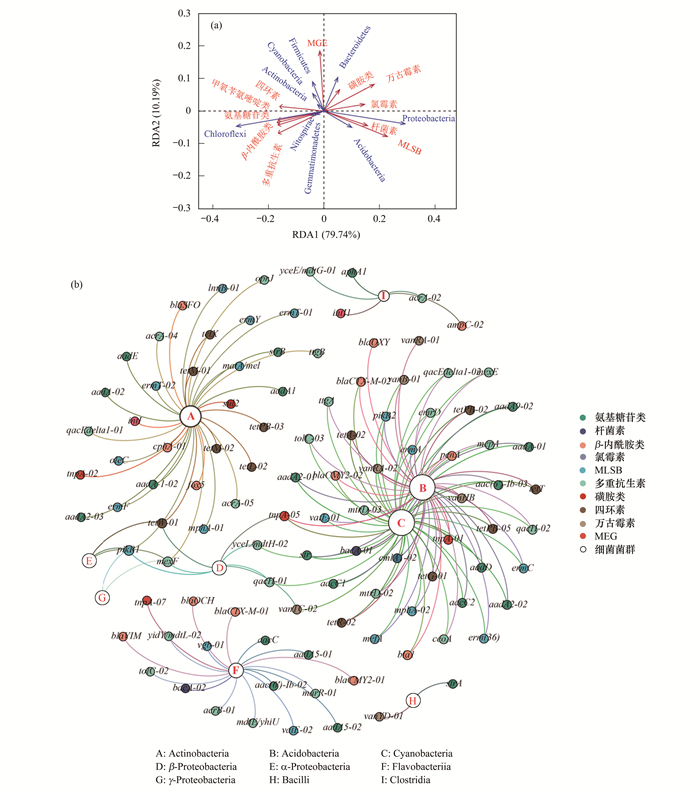
进一步分析发现ARG与细菌菌群具有显著相关性.经RDA分析了门水平细菌菌群/MGE与ARG型之间的关系[图 7(a)], ARG中89.93%的变量可以由所选的前两轴变量解释.变形菌门、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、放线菌门(Actinobacteria)、芽单胞菌门(Gemmatimonadetes)与第一轴显著相关(解释了79.47%的变量), 涉及到氯霉素、杆菌素、MLSB、氨基糖苷类、β-内酰胺类、甲氧苄氨嘧啶和多重抗生素等耐药基因; 厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)与第二轴显著相关(解释10.19%的变量), 涉及到MGE和磺胺类耐药基因等.
|
(a)冗余分析(门水平):以细菌菌群作为解释变量对ARG谱进行分析.蓝色箭头代表细菌菌群, 红色箭头代表ARG和MGE; (b)网络分析(纲水平):每个节点的大小与其连线数成正比, 一个连接代表一个强相关 图 7 ARG与细菌菌群/MGE之间的共存关系 Fig. 7 Co-occurrence relationship between ARG and bacterial community/MGE |
网络分析显示了纲水平细菌菌群与ARG亚型之间的共存模式[图 7(b)], 共有9个细菌菌群与105个ARG亚型(属于9个ARG型)具有显著正相关关系(ρ>0.8, P < 0.01).在细菌菌群中, 蓝藻细菌纲(Cyanobacteria)、酸杆菌纲、放线菌纲、黄杆菌纲(Flavobacteriia)和变形杆菌纲与多种ARG型高度相关.在ARG型中, 多重抗生素(40个ARG亚型)、氨基糖苷类(30)、MLSB(28)、四环素(20)和β-内酰胺类(17)耐药基因同样与多个细菌菌群具有强的相关性.此结果说明这9个纲水平细菌菌群可能是ARG的主要宿主.有研究表明, 不同的环境中, 包括土壤、河流、地下水和污泥等, 细菌菌群结构都是影响ARG谱动态变化的重要因素[22~25].本研究中土壤中以多重抗生素耐药基因丰度最高, 而苦菊根和叶片中β-内酰胺类耐药基因丰度最高, 这种差异可能是由于土壤中特定的细菌菌群可能携带某些特定ARG型, 从而转移至苦菊组织中[26].
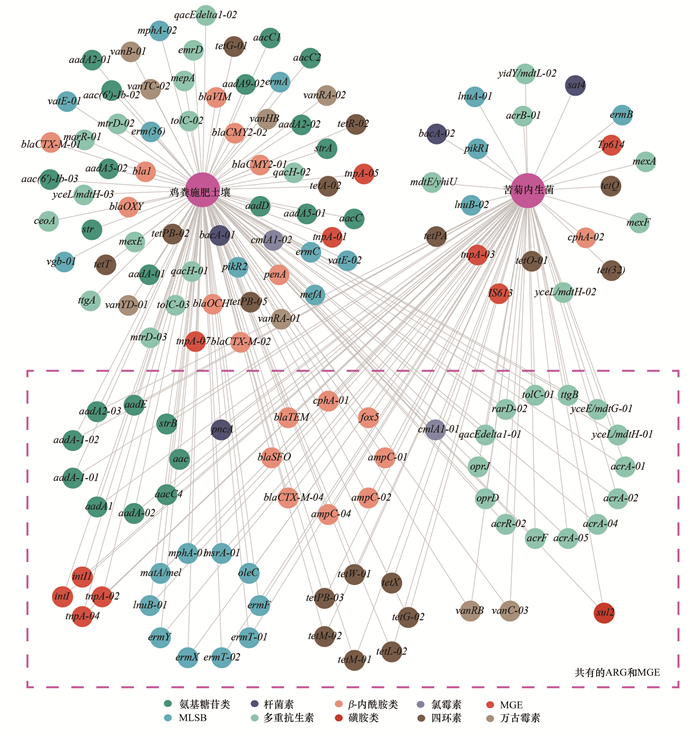
2.4 施肥土壤与苦菊内生菌中共有的细菌菌群和ARG及其传播路径通过分析施肥土壤与苦菊(根内生菌和叶片内生菌)之间的共有ARG, 来预测苦菊组织中ARG的来源.从图 8可以看出, 在鸡粪肥施用土壤与苦菊内生菌之间共有52个ARG亚型和4个MGE, 涉及到多重抗生素、MLSB、氨基糖苷类、β-内酰胺类、四环素、万古霉素、杆菌素、氯霉素和磺胺类等9个ARG型, MGE包括intI、intI1、tnpA-02和tnp-04整合子/转座酶基因.结果表明这些耐药基因很可能从土壤和鸡粪肥中转移至苦菊的根和叶片内生菌中.本研究结果与之前研究的一致, 即土壤和粪肥中的ARG是蔬菜中ARG的重要来源, 同时由于粪肥中有大量的MGE存在, 会进一步促进ARG在粪肥-土壤-蔬菜系统之间的水平传播[12, 27, 28].
|
图 8 鸡粪施肥土壤和苦菊内生菌之间共有的施用ARG和MGE Fig. 8 Shared ARG and MGE between poultry manure-amended soil and endophytes of Cichorium endivia L. |
进一步分析了鸡粪肥施用土壤中ARG向苦菊不同组织的传播, 来探索ARG在土壤-苦菊中潜在的传播路径(图 9).鸡粪肥施用土壤和苦菊根内生菌之间共有49个相同的ARG(Group A1), 这些基因可能来自鸡粪肥改良后的土壤, 随后可能被苦菊根内生菌获得.此外, 土壤、根内生菌和叶片内生菌之间共有11个ARG(Group A2), 包括多重抗生素(acrA-01、acrF、oprD、rarD-02、yceE/mdtG-01)、β-内酰胺类(ampC-02、blaTEM)、氨基糖苷类(aac)、MLSB(ermX)、四环素类(tetG-02)和万古霉素类(vanRB), 都是对人和动物临床中最常用抗生素表现抗性, 说明这些基因可能通过苦菊组织内部从根内生菌向上转移到叶片内生菌.ARG从土壤通过植物组织向植物的传播途径被称为内部路径[12].那么, 本研究中苦菊是通过根吸收从鸡粪肥施用土壤中获取ARG, 并通过内部路径转移到根和叶片内生菌中.
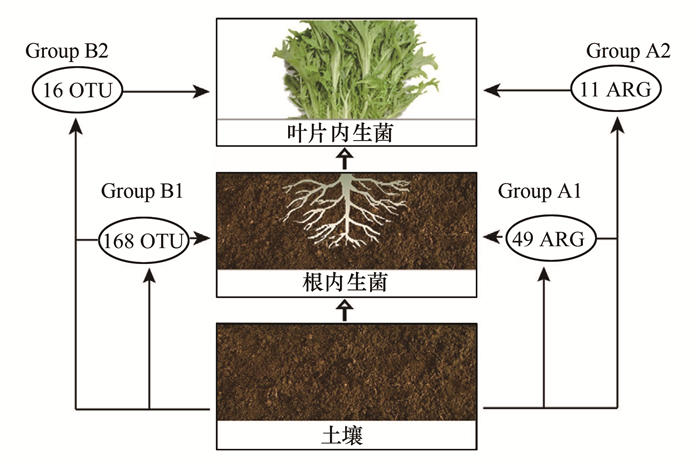
|
图 9 鸡粪施肥土壤中细菌(纲水平)和ARG向苦菊不同组织传播的潜在路径 Fig. 9 Potential transmission pathways of soil and manure-derived resistome and microbiome to different tissues |
由于细菌菌群对ARG分布起重要作用, 本研究通过分析鸡粪肥施用土壤与苦菊组织之间共有的细菌OTU, 来进一步说明施肥土壤中细菌菌群是否向蔬菜内生系统传播(图 9).在鸡粪肥施用土壤和苦菊根内生菌之间共有168个细菌OTU(Group B1).这与之前研究一致, 土壤可能是根内生菌群的种子库, 即植物的大部分内生菌是从生长环境中水平获得, 而不是通过种子或花粉从亲本植株垂直传播而来[29, 30].但是, 仅有16个OTU(Group B2) 可能被进一步传递到叶片内生菌, 这表明根内生菌和叶片内生菌中的细菌菌群不同.值得注意的是, 共有的16个OTU包括α-变形菌纲(6)、β-变形菌纲(5)、γ-变形菌纲(3)和放线菌纲(2), 这些是土壤样品中丰度最高的前4个细菌纲(图 5).此外, 变形菌门和放线菌门与多重抗生素、β-内酰胺类、氨基糖苷类等ARG的存在成正相关(图 7).有研究表示甘蓝和有机生菜叶片中都与土壤之间存在相当大的共有的细菌菌群, 而本研究中苦菊叶片和土壤之间共有的细菌菌群相对较少, 这说明土壤环境中外源细菌对蔬菜内生菌的影响程度可能与蔬菜种类有关[27, 31, 32].以上研究结果表明, 共有的细菌OTU可能是ARG的宿主, 进入苦菊的内生系统, 最终形成了苦菊的根内生菌、叶片内生菌的ARG组成.
动物粪便是耐药菌的重要存储库, 而粪肥的施用很可能将大量的ARG带入到农业土壤-蔬菜系统中[2, 22].本研究中苦菊内生系统ARG的定量检测结果证实, 在大田土壤中添加鸡粪肥会促进蔬菜内生菌中ARG数量和丰度的增加, 甚至蔬菜中会出现了携带多种耐药基因的细菌[7].施用粪肥的土壤环境中, 造成这种结果的原因主要有两个:①蔬菜根会从土壤中吸收耐药菌, 定植于组织内成为内生细菌, 和/或在抗生素选择压下诱导蔬菜内生耐药菌比例增加, 进而导致蔬菜内生系统中ARG丰度增高; ②在MGE作用下, 外源ARG通过土壤-蔬菜互作, 水平转移进入蔬菜内生系统, 促进蔬菜内生菌耐药性的增加[9].以往的研究已证实施肥蔬菜中存在具有耐药性的人类致病菌, 而这很可能通过食物链向周围环境和人类传播, 尤其是日常频繁食用或直接生食的蔬菜, 这对公共健康造成巨大威胁.
3 结论本研究发现鸡粪肥施用可显著提高苦菊根和叶片内生菌中ARG的数量和丰度, 且ARG的富集与变形菌门和放线菌门等细菌菌群高度相关; 分析了ARG和细菌菌群从鸡粪肥施用土壤向苦菊不同部位传播的潜在内部路径.动物粪肥的施用可能导致动物源病原菌和/或ARG在粪便-土壤-蔬菜-人类这个大环境系统之间散播.因此, 动物粪肥的处理和利用形式应该得到有效地指导和严格地控制.
| [1] |
张宁, 李淼, 刘翔. 土壤中抗生素抗性基因的分布及迁移转化[J]. 中国环境科学, 2018, 38(7): 2609-2617. Zhang N, Li M, Liu X. Distribution and transformation of antibiotic resistance genes in soil[J]. China Environmental Science, 2018, 38(7): 2609-2617. DOI:10.3969/j.issn.1000-6923.2018.07.029 |
| [2] | Gou M, Hu H W, Zhang Y J, et al. Aerobic composting reduces antibiotic resistance genes in cattle manure and the resistome dissemination in agricultural soils[J]. Science of the Total Environment, 2018, 612: 1300-1310. DOI:10.1016/j.scitotenv.2017.09.028 |
| [3] | Fang H, Wang H F, Cai L, et al. Prevalence of antibiotic resistance genes and bacterial pathogens in long-term manured greenhouse soils as revealed by metagenomic survey[J]. Environmental Science & Technology, 2015, 49(2): 1095-1140. |
| [4] |
张昊. 抗生素及其耐药性在畜禽粪便-土壤-蔬菜中的传播和转移[D]. 新乡: 河南师范大学, 2018. Zhang H. Spread and transfer of antibiotics and antibiotic resistance in the livestock manure-soil-vegetable endophytic system[D]. Xinxiang: Henan Normal University, 2018. |
| [5] | Han X M, Hu H W, Chen Q L, et al. Antibiotic resistance genes and associated bacterial communities in agricultural soils amended with different sources of animal manures[J]. Soil Biology and Biochemistry, 2018, 126: 91-102. DOI:10.1016/j.soilbio.2018.08.018 |
| [6] | Zhang H P, Zhang Q K, Song J J, et al. Tracking resistomes, virulence genes, and bacterial pathogens in long-term manure-amended greenhouse soils[J]. Journal of Hazardous Materials, 2020, 396. DOI:10.1016/j.jhazmat.2020.122618 |
| [7] | Yang Q X, Zhang H, Guo Y H, et al. Influence of chicken manure fertilization on antibiotic-resistant bacteria in soil and the endophytic bacteria of pakchoi[J]. International Journal of Environmental Research and Public Health, 2016, 13(7). DOI:10.3390/ijerph13070662 |
| [8] | Berg G, Krechel A, Ditz M, et al. Endophytic and ectophytic potato-associated bacterial communities differ in structure and antagonistic function against plant pathogenic fungi[J]. FEMS Microbiology Ecology, 2005, 51(2): 215-229. DOI:10.1016/j.femsec.2004.08.006 |
| [9] | Carpentieri-Pipolo V, De Almeida Lopes K B, Degrassi G. Phenotypic and genotypic characterization of endophytic bacteria associated with transgenic and non-transgenic soybean plants[J]. Archives of Microbiology, 2019, 201(8): 1029-1045. DOI:10.1007/s00203-019-01672-6 |
| [10] | Verraes C, Van Boxstael S, Van Meervenne E, et al. Antimicrobial resistance in the food chain: a review[J]. International Journal of Environmental Research and Public Health, 2013, 10(7): 2643-2669. DOI:10.3390/ijerph10072643 |
| [11] | Chen Q L, Cui H L, Su J Q, et al. Antibiotic resistomes in plant microbiomes[J]. Trends in Plant Science, 2019, 24(6): 530-541. DOI:10.1016/j.tplants.2019.02.010 |
| [12] | Zhang Y J, Hu H W, Chen Q L, et al. Transfer of antibiotic resistance from manure-amended soils to vegetable microbiomes[J]. Environment International, 2019, 130. DOI:10.1016/j.envint.2019.104912 |
| [13] | Hu H W, Wang J T, Singh B K, et al. Diversity of herbaceous plants and bacterial communities regulates soil resistome across forest biomes[J]. Environmental Microbiology, 2018, 20(9): 3186-3220. DOI:10.1111/1462-2920.14248 |
| [14] | Su J Q, Wei B, Ouyang W Y, et al. Antibiotic resistome and its association with bacterial communities during sewage sludge composting[J]. Environmental Science & Technology, 2015, 49(12): 7356-7363. |
| [15] | Beckers B, De Beeck M O, Thijs S, et al. Performance of 16s rDNA primer pairs in the study of rhizosphere and endosphere bacterial microbiomes in metabarcoding studies[J]. Frontiers in Microbiology, 2016, 7. DOI:10.3389/fmicb.2016.00650 |
| [16] | Edgar R C. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [17] | Pan M, Chu L M. Phytotoxicity of veterinary antibiotics to seed germination and root elongation of crops[J]. Ecotoxicology and Environmental Safety, 2016, 126: 228-237. DOI:10.1016/j.ecoenv.2015.12.027 |
| [18] | Heuer H, Smalla K. Plasmids foster diversification and adaptation of bacterial populations in soil[J]. FEMS Microbiology Reviews, 2012, 36(6): 1083-1104. DOI:10.1111/j.1574-6976.2012.00337.x |
| [19] | Wang F H, Qiao M, Chen Z, et al. Antibiotic resistance genes in manure-amended soil and vegetables at harvest[J]. Journal of Hazardous Materials, 2015, 299: 215-221. DOI:10.1016/j.jhazmat.2015.05.028 |
| [20] | Zhu B K, Chen Q L, Chen S C, et al. Does organically produced lettuce harbor higher abundance of antibiotic resistance genes than conventionally produced?[J]. Environment International, 2017, 98: 152-159. DOI:10.1016/j.envint.2016.11.001 |
| [21] | Cerqueira F, Christou A, Fatta-Kassinos D, et al. Effects of prescription antibiotics on soil-and root-associated microbiomes and resistomes in an agricultural context[J]. Journal of Hazardous Materials, 2020, 400. DOI:10.1016/j.jhazmat.2020.123208 |
| [22] | Forsberg K J, Patel S, Gibson M K, et al. Bacterial phylogeny structures soil resistomes across habitats[J]. Nature, 2014, 509(7502): 612-616. DOI:10.1038/nature13377 |
| [23] |
张红娜, 崔娜, 申红妙. 基于宏基因组学探讨东平湖水库的菌群结构、耐药基因谱及其公共健康风险[J]. 环境科学, 2020, 42(1): 211-220. Zhang H N, Cui N, Shen H M. Metagenomic analysis provides insights into bacterial community, antibiotic resistome and public health risk in Dongping Lake reservoir[J]. Environmental Science, 2020, 42(1): 211-220. |
| [24] |
毛秋燕, 赵栩宁, 苏宇傲, 等. 不同预处理方式对剩余污泥中活菌菌群及ARGs的影响[J]. 中国环境科学, 2020, 40(6): 2537-2545. Mao Q Y, Zhao X N, Su Y A, et al. Impact of different pretreatments on ARGs and live microbial communities in excess sludge[J]. China Environmental Science, 2020, 40(6): 2537-2545. DOI:10.3969/j.issn.1000-6923.2020.06.024 |
| [25] | Jia S Y, Shi P, Hu Q, et al. Bacterial community shift drives antibiotic resistance promotion during drinking water chlorination[J]. Environmental Science & Technology, 2015, 49(20): 12271-12279. |
| [26] | Li B, Yang Y, Ma L P, et al. Metagenomic and network analysis reveal wide distribution and co-occurrence of environmental antibiotic resistance genes[J]. The ISME Journal, 2015, 9(11): 2490-2502. DOI:10.1038/ismej.2015.59 |
| [27] | Chen Q L, An X L, Zhu Y G, et al. Application of struvite alters the antibiotic resistome in soil, rhizosphere, and phyllosphere[J]. Environmental Science & Technology, 2017, 51(14): 8149-8157. |
| [28] |
张汝凤, 宋渊, 高浩泽, 等. 北京蔬菜地土壤中抗生素抗性基因与可移动元件的分布特征[J]. 环境科学, 2020, 41(1): 385-393. Zhang R F, Song Y, Gao H Z, et al. Distribution characteristics of antibiotic resistance genes and mobile genetic elements in Beijing vegetable base soils[J]. Environmental Science, 2020, 41(1): 385-393. |
| [29] | Frank A C, Saldierna Guzmán J P, Shay J E. Transmission of bacterial endophytes[J]. Microorganisms, 2017, 5(4). DOI:10.3390/microorganisms5040070 |
| [30] | Vandenkoornhuyse P, Quaiser A, Duhamel M, et al. The importance of the microbiome of the plant holobiont[J]. New Phytologist, 2015, 206(4): 1196-1206. DOI:10.1111/nph.13312 |
| [31] | Durán P, Thiergart T, Garrido-Oter R, et al. Microbial interkingdom interactions in roots promote Arabidopsis survival[J]. Cell, 2018, 175(4): 973-983. DOI:10.1016/j.cell.2018.10.020 |
| [32] | Liao J L, Xu Q C, Xu H L, et al. Natural farming improves soil quality and alters microbial diversity in a cabbage field in Japan[J]. Sustainability, 2019, 11(11). DOI:10.3390/su11113131 |
 2021, Vol. 42
2021, Vol. 42


