超级耐药细菌(super antibiotic resistant bacteria, SARB)是一类细菌的总称, 对几乎所有抗生素耐药是其最大的特点[1].2010年8月, 世界卫生组织指出人类将面临SARB的威胁[2], 甚至有人提出SARB如NDM-1细菌10年内将无药可救.SARB产生抗生素耐药性的遗传基础是超级耐药基因(super antibiotic resistance genes, SARGs[3]), 它兼具“可复制传播”的生物特性和“不易消亡或环境持久”的物理化学特性, 可在生物体内持续传播[4].SARGs在自然界中有两种存在形式:细胞内超级耐药基因(intracellular super antibiotic resistance genes, iSARGs)和细胞外超级耐药基因(extracellular super resistance genes, eSARGs[5]), 其中eSARGs是菌体活体分泌或死亡后释放的, 可在水中持久存在[6].污水处理厂(wastewater treatment plants, WWTP)主要是处理居民生活、医疗和畜禽养殖等方面废水, 被认为是ARGs的重要储存库, 且在整个污水处理过程中各个单元均有ARGs被检测出, 同时也是细菌之间发生水平基因转移的高发区[7].目前, ARGs已经被定义为一种新型环境污染物[8], 因此深入研究污水处理过程中SARGs的污染特征, 对今后有效控制水中SARGs污染有重要意义.
消毒处理对提高WWTP出水的生物安全性具有重要意义, 它能够高效杀灭水中多种病菌和病毒.然而, 有研究表明消毒过程并不能有效去除iARGs[9, 10], 而且更为严重的是, 笔者发现WWTP出水经二氧化氯(chlorine dioxide, ClO2)消毒后, 16种iARGs的相对浓度升高[11].考虑到ClO2杀菌机制主要与细菌蛋白质变性和渗漏有关, 且已有研究表明, 只有当消毒剂使用浓度远远高于杀菌浓度时, 才会破坏消毒致死细菌释放的质粒及其ARGs[12].因此, 消毒过程有可能帮助死亡SARB释放其DNA, 成为水体游离的eSARGs.目前, 关于ClO2消毒对水中iSARGs和eSARGs污染的影响, 还未见报道.
本研究对天津市某污水处理厂出水消毒前后的水样分别进行iSARGs和eSARGs的富集和定量检测, 通过分析ClO2消毒对水中SARGs的影响, 探讨ClO2消毒对水中SARGs的去除效果, 以期为以后WWTP消毒工艺的改进提供参考.
1 材料与方法 1.1 主要仪器与试剂正压过滤器(海宁市能大过滤设备有限公司)、蠕动泵(上海安亭科学仪器厂)、ViiA 7Dx荧光定量PCR仪(美国ABI公司)和台式冷冻高速离心机(美国Sigma公司)等.
水相微孔过滤膜(北京北化黎明膜分离技术有限责任公司)、硅胶(中国昆海公司)、SYBR Green荧光定量PCR试剂盒(瑞士Roche公司)、普通DNA产物纯化试剂盒(北京天根生化科技有限公司)和Fast DNA Spin Kit for Soil(美国MP Bio公司)等.
1.2 水样采集水样采自天津市某WWTP出水池, 采集ClO2消毒前(采样点1)和消毒后(采样点2)的水样, 具体如图 1所示, 消毒池中ClO2使用浓度为8~9mg·L-1, 消毒后水样中ClO2残留量为0.07~0.63 mg·L-1.采样时间为2016年1月至2016年12月, 每月采集水样3次(因7月天津持续强暴雨影响, 污水厂水位过高, 未能采样).采集的水样冷藏条件下迅速送至实验室开展实验.
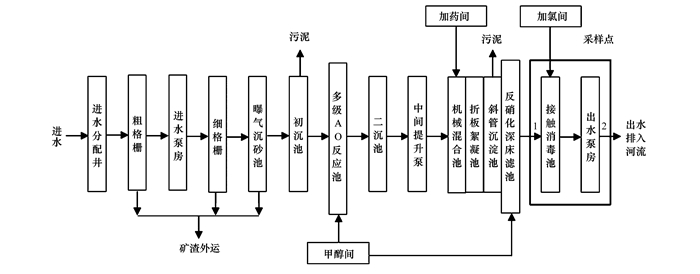
|
图 1 污水处理厂工艺示意 Fig. 1 Schematic diagram of wastewater treatment in the WWTP |
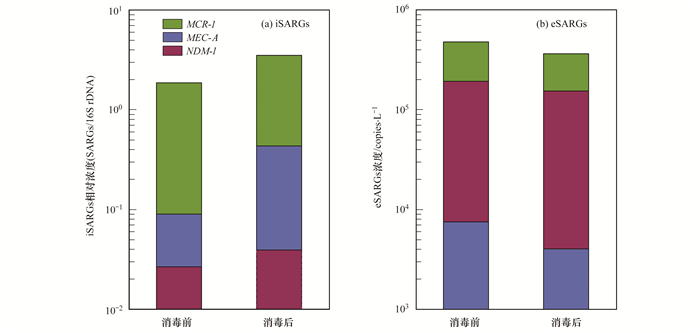
|
图 2 全年污水处理厂出水中超级耐药基因浓度变化(n=33) Fig. 2 Change in the concentration of SARGs in the WWTP throughout the year |
将无菌微孔滤膜(孔径0.22 μm)装入正压过滤器, 并通过蠕动泵, 使20 L水样匀速(50 mL·min-1)通过滤膜, 然后将滤膜剪碎并收集到装有100 mL 3%牛肉膏洗液的玻璃瓶中, 置于磁力搅拌器上搅拌30 min(4℃).将细菌悬液收集到50 mL的离心管中, 离心(4℃, 8 000 r·min-1, 10 min)弃上清后, 将细菌沉淀物悬浮于10 mL PBS缓冲液中, 置于-80℃保存, 待检测.细菌DNA采用Fast DNA Spin Kit for Soil进行提取.
1.4 胞外游离核酸的富集文献[13]将高压灭菌的Al(OH)3溶液与60~100目的硅胶充分混合, 制备核酸吸附颗粒.以质粒PBR322作为内参, 加入到20 L水样中, 充分混匀, 使其最终浓度为20 copies·mL-1.利用蠕动泵, 使水样以一定流速(50 mL·min-1)通过装有80~90 g核酸吸附颗粒的过滤柱(直径5 cm), 过滤结束后用300 mL洗脱液(15 g·L-1氯化钠, 30 g·L-1胰蛋白胨, 15 g·L-1牛肉粉, 3.75 g·L-1甘氨酸, 0.28 g·L-1氢氧化钠, pH 9.3±0.2)洗脱滤柱, 收集洗脱液并加入等体积异丙醇, 室温放置16 h后离心(4℃, 10 000 r·min-1, 10 min)弃上清; 75%乙醇重悬并洗涤沉淀, 离心(4℃, 10 000 r·min-1)5 min后放置1~2 min, 使乙醇充分挥发, 加入1 mL无菌Tris-EDTA缓冲液(pH 8.0)进行重悬后, 利用普通DNA产物纯化试剂盒纯化胞外核酸, 置于-80℃保存, 待检测.
1.5 SARGs的定量检测利用荧光定量PCR技术(quantitative real-time polymerase chain reaction, qPCR)检测9种SARGs.其中, 耐碳青霉烯类基因包括NDM-1、KPC、IMP、VIM和OXA-48; 耐万古霉素类基因包括VAN-B; 耐多粘菌素类基因包括MCR-1; 耐甲氧西林类基因包括MEC-A和FEM-A.SARGs标准曲线的建立方法参照文献[14].各种SARGs的引物及标准品均由上海生工生物工程有限公司合成(表 1).
|
|
表 1 本文所用的检测引物1) Table 1 Primers used in this study |
qPCR反应体系包括FastStart Universal SYBR Green Master(Rox)10 μL, 基因上下游引物(10 μmol·L-1)各0.4 μL, 模板2 μL, 无酶水补至20 μL.反应条件:50℃ 2 min; 95℃ 10 min; 95℃ 15 s, 60℃ 60 s, 40个循环.每批次反应均设置阳性对照(SARGs阳性标准品为模板)和阴性对照(以无酶水为模板), 每个基因检测3次.
1.6 水中eSARGs浓度的计算为保证水样eSARGs浓度的准确计算, 将PBR322作为内参, 加入到待测水样中, 利用qPCR检测其富集前后的浓度, 确定其回收率[11].胞外核酸回收率和eSARGs实际浓度的计算参照公式(1)和(2):
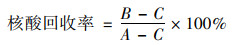
|
(1) |
式中, A为水样里PBR322的实际加标总拷贝数(copies), B为浓集液中PBR322的拷贝数(copies), C是水样中PBR322的背景拷贝数(copies).
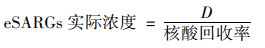
|
(2) |
式中, D为利用qPCR测得的水样eSARGs浓度(copies·L-1).
1.7 数据统计与分析用SPSS 18.0(SPSS Inc., Chicago, Illinois)软件进行统计学分析, 利用Origin 9.6(Microcal Software, USA)进行作图.数据满足正态性要求的(比较消毒前后变化)用Paired-Sample t检验, 数据不满足正态性要求的(比较消毒前后变化)用Wilcoxon检验, 比较多个独立样本的分布差异用Kruskal-Wallis H检验, P<0.05为差异有统计学意义.
2 结果与讨论 2.1 ClO2消毒对水中SARGs检出频率的影响将WWTP出水ClO2消毒前后的水样同时进行iSARGs和eSARGs检测, 结果显示(表 2), 仅有NDM-1、MCR-1和MEC-A这3种SARGs被不同程度检出, 而其它6种SARGs均未被检出.对于iSARGs, 消毒后水样的MCR-1和MEC-A检出率较消毒前均发生了下降; 在eSARGs中, 可发现MEC-A的检出率在消毒后出现了上升, 由消毒前的45%变为消毒后的55%.曹振华等在南京地区污水厂的出水中, 也检出MCR-1和NDM-1污染[14], 以上结果表明目前的污水处理工艺无法有效去除MCR-1、NDM-1和MEC-A等SARGs.
|
|
表 2 污水处理厂超级耐药基因的检出率(n=33)/% Table 2 Detection rate of SARGs in the WWTP/% |
2.2 ClO2消毒对水中iSARGs的组成及其浓度的影响
将全年收集的所有水样进行统计分析, 观察ClO2消毒对水中iSARGs的影响[图 2(a)].结果表明, 未经ClO2消毒时, 水中NDM-1、MCR-1和MEC-A的平均相对总浓度为1.86, 经过ClO2消毒后, 胞内平均相对总浓度发生明显上升(P<0.05), 达到3.52, 较消毒前提高1.89倍[图 2(a)].进一步观察可发现, 经过ClO2消毒后的3种基因中, 以MEC-A上升最明显(P<0.05), 相对浓度达到0.4, 较消毒前提高7倍, NDM-1和MCR-1相对浓度在消毒后具有上升的趋势, 但没有显著性差异(P>0.05).以上结果说明ClO2消毒具有促进水中iSARGs污染的作用.Liu等[11]的研究也发现ClO2消毒会增加iARGs的丰度, 相对浓度最高可增加7.8倍, 而Shi等[25]根据宏基因组测序, 发现氯化过程会提高ampC和tetA丰度.该现象可能与细菌消毒剂抗性/抗生素耐药性的共选择有关[12], 也可能与消毒导致细菌死亡释放ARGs, 而ARGs可转化存活损伤细菌导致iSARGs浓度升高有关, 另外, 氯剂量不足下的耐药质粒结合转移提升也可能与之有关[26].因此, 为了控制水环境中SARB的传播和扩散, 目前的污水处理厂ClO2消毒工艺有待进一步提高.
结果还显示, 对于消毒前NDM-1、MCR-1和MEC-A呈阳性的水样, ClO2消毒后的相对浓度发生显著性变化(P<0.05), 其中既有相对浓度增加的现象, 也有相对浓度减少的个例[图 3(a)], 对于MEC-A, 100%阳性水样发生增加(P<0.05), 最高提高倍数为8倍(8月); 对于MCR-1和NDM-1, 分别为86%和66%阳性水样发生增加, 但均无显著性差异(P>0.05).值得关注的是, MEC-A在9和11月的消毒前水样中未测出, 但在消毒后其浓度分别达到0.04和0.1.
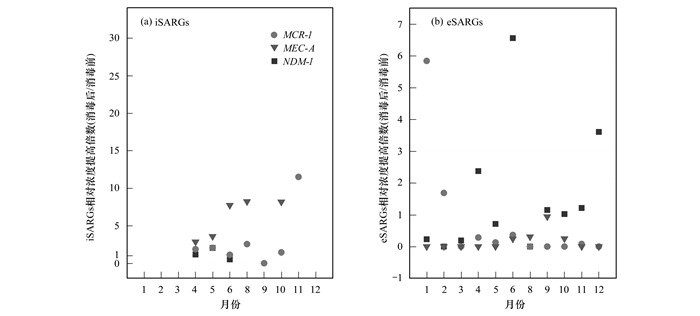
|
图 3 ClO2消毒后水中超级耐药基因浓度的提高倍数(n=3) Fig. 3 Change in the concentration of SARGs in the water before and after ClO2 disinfection |
进一步研究不同时间段ClO2消毒对水中iSARG组成的影响[图 4(a)], 结果表明, iSARGs的检出时间分布于4~11月, 而其它时间点均无检出.同时, NDM-1只出现在4~6月, 平均相对浓度由消毒前的0.008, 变为消毒后的0.013, MEC-A和MCR-1均出现在4~11月, 平均相对浓度分别由消毒前的0.009和0.25, 变为消毒后的0.057和0.44.因此, 对于污水处理厂消毒后的水样, 除12月和1~3月未被检测出, 4~6月的iSARGs种类最丰富, 包含NDM-1、MCR-1和MEC-A, 而其他月份只含有2种上述iSARGs.
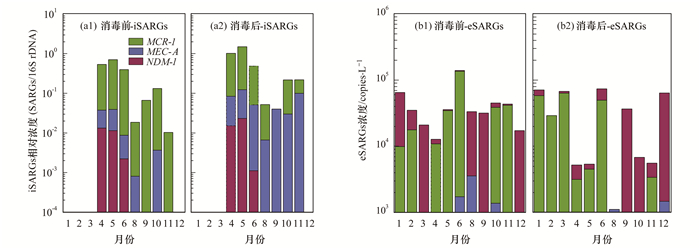
|
图 4 每月污水处理厂出水中超级耐药基因的组成及其浓度(n=3) Fig. 4 Compositions and concentrations of SARGs in the WWTP by month |
将全年收集的所有水样进行统计分析, 观察ClO2消毒对水中eSARGs的影响[图 2(b)].结果表明, 未经ClO2消毒时, 水中胞外NDM-1、MCR-1和MEC-A总浓度平均为4.80×105 copies·L-1, 其中MCR-1浓度最高, 为2.86×105 copies·L-1, MEC-A最低, 仅有7.55×103 copies·L-1.经ClO2消毒后, 水中胞外NDM-1、MCR-1和MEC-A总浓度平均与消毒前无显著性差异(P>0.05), 为3.65×105 copies·L-1.结果还显示[图 3(b)], 对于消毒前呈NDM-1阳性的水样, 有55%发生显著增加(P<0.05), 提高倍数最高为6.5倍.对于消毒前呈MCR-1阳性的水样, 有28%发生增加, 但无显著性差异(P>0.05).同时还发现, MCR-1在3月未消毒水样中没有被检测出, 但消毒后浓度增加至6.34×104 copies·L-1, 而MEC-A, 在12月未消毒水样中没有被检测出, 消毒后水样中其浓度为1.46×103 copies·L-1.水中eSARGs的污染与细菌死亡释放的核酸有关, 据报道, 即使ARB通过消毒完全失活, 其释放的质粒仍可在水中持久存在且保持转化功能, 在一定条件下, 它们可以转化自然感受态细菌而进入细菌, 导致抗生素耐药性的水平转移和扩散[12].另外, 不同eSARGs对消毒剂的抵抗能力不同, 可能是它们在水中污染种类和丰度呈多态性的主要原因.
进一步研究不同时间段ClO2消毒对水中eSARGs组成的影响[图 4(b)], 结果显示, 经过ClO2消毒后, NDM-1在1、3、4、5、6、9、10、11和12月被检测出, 平均浓度由消毒前的1.53×104 copies·L-1, 变为消毒后的1.67×104 copies·L-1, 而MEC-A出现在1、6、8、9、10和12月[1、6、9和10月因浓度较低, 图 4(b)未显示], 平均浓度由消毒前的1.18×103 copies·L-1, 变为消毒后的6.74×102 copies·L-1, MCR-1出现在1、2、3、4、5、6和11月, 平均浓度由消毒前的3.55×104 copies·L-1, 变为消毒后的3.01×104 copies·L-1.由上可观察到, 水样经消毒后, 全年所有月份均检出eSARGs, 其中1月和6月的eARGs最丰富, 可检出NDM-1、MCR-1和MEC-A, 而其它时间, 只能检出1~2种.
2.4 不同季节ClO2消毒对水中SARGs浓度的影响根据消毒时间所处季节, 观察ClO2消毒对水中iSARGs的影响[图 5(a)].结果显示, 未经ClO2消毒时, iSARGs平均相对总浓度在春季最高, 为1.23, 其次是夏季和秋季, 分别为0.42和0.21.经ClO2消毒后, 春季、夏季和秋季的iSARGs平均相对总浓度均发生上升, 其中春季达到一年中的最高值, 为2.46, 与消毒前相比, 春季iSARGs平均相对总浓度升高2倍(P<0.05).值得关注的是, 冬季的水样不管消毒与否均未检出iSARGs, 暗示SARB可能在冬季流行相对较弱, Yuan等[27]的研究也指出季节对于污水处理厂的ARB释放负荷有相当大影响, 其在冬季释放负荷极低.进一步研究不同季节ClO2消毒对各个基因相对浓度的影响[图 5(a)], 可观察到, 在春季, 以NDM-1相对浓度增加倍数最大, 由消毒前的0.01, 变为消毒后的0.038, 提高3.8倍(P>0.05); 在夏季, 以MEC-A相对浓度增加倍数最大, 由消毒前的0.007, 变为消毒后的0.013, 提高1.86倍(P>0.05); 在秋季, 以MEC-A相对浓度增加倍数最大, 由消毒前的0.004, 变为消毒后的0.037, 提高9.25倍(P>0.05).贺赟等[28]的研究也发现不同季节污水处理厂处理效果不同, 其优势菌属也存在差异.因此, 污水处理厂出水中iSARGs的污染种类和浓度与季节密切有关, 且与ClO2消毒过程息息相关.
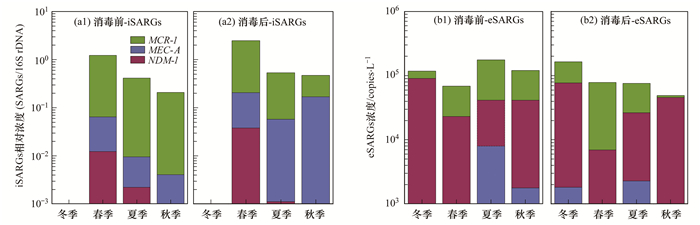
|
图 5 不同季节污水处理厂出水中超级耐药基因浓度(n=9) Fig. 5 Concentrations of SARGs in the WWTP in different seasons |
对全年收集的所有水样进行统计分析, 观察不同季节ClO2消毒对水中eSARGs的影响[图 5(b)].结果显示, 未经ClO2消毒时, eSARGs总浓度平均在夏季达到最高, 为1.76×105 copies·L-1, 其次是秋季和冬季, 最低是春季, 其总浓度平均为6.85×104 copies·L-1.经ClO2消毒后, 总浓度平均在冬季达到最高, 为1.63×105 copies·L-1, 其次是春季和夏季, 最低是秋季, 其总浓度平均为4.88×104 copies·L-1.但是, 经统计学分析, 各季节的eSARGs总浓度在消毒前后并无显著性差异(P>0.05).进一步研究各个基因在不同季节的变化[图 5(b)], 可观察到各个基因总浓度在消毒前后均无显著性差异(P>0.05).以上结果表明, ClO2消毒对水中eSARGs的影响与温度无关.
3 结论(1) WWTP出水消毒后, 仍可检出胞内和胞外NDM-1、MCR-1和MEC-A等SARGs, 其中, iSARGs在4~6月, 而eSRAGs在1月和6月可同时检出上述3种SARGs.
(2) 经ClO2消毒后, WWTP出水的胞内NDM-1、MCR-1和MEC-A平均相对总浓度显著提升, 且MEC-A提高倍数达7倍; ClO2消毒对iSARGs相对浓度的影响与季节有关, iSARGs相对总浓度在春季、夏季和秋季均发生上升, 且春季升高最为明显, 达2倍, 而冬季出水在消毒前后均未测出iSARGs.
(3) 经ClO2消毒后, WWTP出水的胞外NDM-1、MCR-1和MEC-A总浓度平均未明显变化.
| [1] | 张锦红, 江涛, 曾昭智. 预防"超级细菌"的滋长-合理规范使用抗生素[J]. 国外医药(抗生素分册), 2011, 32(1): 46-48. |
| [2] | 赵晖. 面对"超级耐药基因", 重视头孢菌素临床运用价值[J]. 中国社区医师, 2010, 26(41): 15. |
| [3] |
文汉卿, 史俊, 寻昊, 等. 抗生素抗性基因在水环境中的分布、传播扩散与去除研究进展[J]. 应用生态学报, 2015, 26(2): 625-635. Wen H Q, Shi J, Xun H, et al. Distribution, dissemination and removal of antibiotic resistant genes (ARGs) in the aquatic environment[J]. Chinese Journal of Applied Ecology, 2015, 26(2): 625-635. |
| [4] |
徐冰洁, 罗义, 周启星, 等. 抗生素抗性基因在环境中的来源、传播扩散及生态风险[J]. 环境化学, 2010, 29(2): 169-178. Xu B J, Luo Y, Zhou Q X, et al. Sources, dissemination, and ecological risks of antibiotic resistances genes (ARGs) in the environment[J]. Environmental Chemistry, 2010, 29(2): 169-178. |
| [5] | Zhang Y P, Snow D D, Parker D, et al. Intracellular and extracellular antimicrobial resistance genes in the sludge of livestock waste management structures[J]. Environmental Science & Technology, 2013, 47(18): 10206-10213. |
| [6] | Mao D Q, Luo Y, Mathieu J, et al. Persistence of extracellular DNA in river sediment facilitates antibiotic resistance gene propagation[J]. Environmental Science & Technology, 2014, 48(1): 71-78. |
| [7] | 苗雨. 抗生素抗性基因在污水处理系统和消毒工艺中的行为特性[D]. 天津: 天津大学, 2018. |
| [8] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants: studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [9] | Yuan Q B, Guo M T, Yang J. Fate of antibiotic resistant bacteria and genes during wastewater chlorination: implication for antibiotic resistance control[J]. PLoS One, 2015, 10(3). DOI:10.1371/journal.pone.0119403 |
| [10] | Hu Y R, Zhang T Y, Jiang L, et al. Occurrence and reduction of antibiotic resistance genes in conventional and advanced drinking water treatment processes[J]. Science of the Total Environment, 2019, 669: 777-784. DOI:10.1016/j.scitotenv.2019.03.143 |
| [11] | Liu S S, Qu H M, Yang D, et al. Chlorine disinfection increases both intracellular and extracellular antibiotic resistance genes in a full-scale wastewater treatment plant[J]. Water Research, 2018, 136: 131-136. DOI:10.1016/j.watres.2018.02.036 |
| [12] | Jin M, Liu L, Wang D N, et al. Chlorine disinfection promotes the exchange of antibiotic resistance genes across bacterial genera by natural transformation[J]. The ISME Journal, 2020, 14(7): 1847-1856. DOI:10.1038/s41396-020-0656-9 |
| [13] | Wang D N, Liu L, Qiu Z G, et al. A new adsorption-elution technique for the concentration of aquatic extracellular antibiotic resistance genes from large volumes of water[J]. Water Research, 2016, 92: 188-198. DOI:10.1016/j.watres.2016.01.035 |
| [14] |
曹振华, 张媛, 马奔, 等. 南京地区污水厂、自来水厂及长江中抗性基因MCR-1和NDM-1的污染特征[J]. 环境科学研究, 2019, 32(3): 406-414. Cao Z H, Zhang Y, Ma B, et al. Pollution characteristics of MCR-1 and NDM-1 in wastewater treatment plants, waterworks and Yangtze river of Nanjing section[J]. Research of Environmental Sciences, 2019, 32(3): 406-414. |
| [15] | McGann P, Hang J, Clifford R J, et al. Complete sequence of a novel 178-kilobase plasmid carrying blaNDM-1 in a Providencia stuartii strain isolated in Afghanistan[J]. Antimicrobial Agents and Chemotherapy, 2012, 56(4): 1673-1679. DOI:10.1128/AAC.05604-11 |
| [16] | Zheng F, Sun J J, Cheng C C, et al. The establishment of a duplex real-time PCR assay for rapid and simultaneous detection of blaNDM and blaKPC genes in bacteria[J]. Annals of Clinical Microbiology and Antimicrobials, 2013, 12(1): 30. DOI:10.1186/1476-0711-12-30 |
| [17] | van der Zee A, Roorda L, Bosman G, et al. Multi-centre evaluation of real-time multiplex PCR for detection of carbapenemase genes OXA-48, VIM, IMP, NDM and KPC[J]. BMC Infectious Diseases, 2014, 14(1): 27. DOI:10.1186/1471-2334-14-27 |
| [18] | Bordin A, Trembizki E, Windsor M, et al. Evaluation of the SpeeDx Carba (beta) multiplex real-time PCR assay for detection of NDM, KPC, OXA-48-like, IMP-4-like and VIM carbapenemase genes[J]. BMC Infectious Diseases, 2019, 19(1): 571. DOI:10.1186/s12879-019-4176-z |
| [19] | Naas T, Cotellon G, Ergani A, et al. Real-time PCR for detection of blaOXA-48 genes from stools[J]. Journal of Antimicrobial Chemotherapy, 2013, 68(1): 101-104. DOI:10.1093/jac/dks340 |
| [20] | Adams D N. Shortcut detection of the vanB gene cluster in enterococci by a duplex real-time PCR assay[J]. Pathology, 2006, 38(4): 349-352. DOI:10.1080/00313020600820807 |
| [21] | Daniels J B, Campbell D, Boyd S, et al. Development and validation of a clinical laboratory improvement amendments-compliant multiplex real-time PCR assay for detection of mcr genes[J]. Microbial Drug Resistance, 2019, 25(7): 991-996. DOI:10.1089/mdr.2018.0417 |
| [22] | Johnson E J, Zemanick E T, Accurso F J, et al. Molecular identification of Staphylococcus aureus in airway samples from children with cystic fibrosis[J]. PLoS One, 2016, 11(1). DOI:10.1371/journal.pone.0147643 |
| [23] |
董莲华, 隋志伟, 王晶, 等. 数字PCR方法准确测量质粒DNA拷贝浓度[J]. 计量学报, 2017, 38(2): 247-251. Dong L H, Sui Z W, Wang J, et al. Accurate plasmid DNA copy concentration quantification by digital PCR[J]. Acta Metrologica Sinica, 2017, 38(2): 247-251. DOI:10.3969/j.issn.1000-1158.2017.02.27 |
| [24] | Crisafi F, Denaro R, Genovese M, et al. Comparison of 16SrDNA and toxR genes as targets for detection of Vibrio anguillarum in Dicentrarchus labrax kidney and liver[J]. Research in Microbiology, 2011, 162(3): 223-230. DOI:10.1016/j.resmic.2010.11.004 |
| [25] | Shi P, Jia S Y, Zhang X X, et al. Metagenomic insights into chlorination effects on microbial antibiotic resistance in drinking water[J]. Water Research, 2013, 47(1): 111-120. DOI:10.1016/j.watres.2012.09.046 |
| [26] | Guo M T, Yuan Q B, Yang J. Distinguishing effects of ultraviolet exposure and chlorination on the horizontal transfer of antibiotic resistance genes in municipal wastewater[J]. Environmental Science & Technology, 2015, 49(9): 5771-5778. |
| [27] | Yuan Q B, Guo M T, Yang J. Monitoring and assessing the impact of wastewater treatment on release of both antibiotic-resistant bacteria and their typical genes in a Chinese municipal wastewater treatment plant[J]. Environmental Science: Processes & Impacts, 2014, 16(8): 1930-1937. |
| [28] |
贺赟, 李魁晓, 王佳伟, 等. 不同季节城市污水处理厂微生物群落特性[J]. 环境科学, 2021, 42(3): 1488-1495. He Y, Li K X, Wang J W, et al. Microbial community structure of waste water treatment plants in different seasons[J]. Environmental Science, 2021, 42(3): 1488-1495. |
 2021, Vol. 42
2021, Vol. 42


