2. 上海浦东威立雅自来水有限公司, 上海 200127
2. Shanghai Pudong Veolia Water Corporation Limited, Shanghai 200127, China
水中对公共健康风险最大的是微生物[1], 可通过污染饮用水传播疾病的病原微生物多种多样.与细菌和原生动物相比, 病毒是一种没有细胞结构的特殊生物体, 尺寸更小并具有高抗性[2], 几乎可以感染所有具有细胞结构的生命体(动物、植物和微生物等)[3].病毒不仅存在于宿主细胞内, 也可独立存在于生态环境中[4], 饮用水是病毒传播的主要介质之一.在饮用水消毒处理过程中, 致病性病毒通常比细菌更具抗性[5].我国武汉以长江为水源的3座水厂出厂水中检测到了腺病毒、轮状病毒和肠道病毒[6].病毒借饮用水传播的风险不容忽视, 加强供水系统中不同消毒技术对病毒的影响特性研究十分重要.
氯胺是饮用水处理中常用消毒剂, 稳定性高且持续时间长, 但当水中氯胺低于0.45 mg·L-1时, 微生物可能会产生适应性, 供水输配系统中的生物膜也可以保护微生物以减轻消毒剂的影响[7].紫外线(ultraviolet, UV)消毒可以有效灭活对氯/氯胺有抵抗力的水生病原体, 如隐孢子虫[8], 而且水中病原体可以在短时间内被有效灭活, 但因UV不具持久消毒能力, 往往需要与其他消毒方式联合使用.经UV处理后补加氯/氯胺的联合处理工艺[UV/chlor(am)ines]既能有效灭活水中微生物, 降低氯化消毒剂投加量, 还能高效去除微量有机污染物, 如紫外/氯胺处理可部分降低两种吡唑啉酮药物[异丙酚菲(PRP)和氨基比林(AMP)]的遗传毒性[9].但目前该联合工艺主要针对两虫和细菌有相关研究, 对病毒微生物的影响及其在输配系统中的迁移变化还知之甚少.
研究供水系统中病毒微生物的净化效能难点在于病毒微生物的检测.现阶段水环境中病毒的检测方法主要有:细胞培养法和基于病毒核酸扩增方法, 如聚合酶链式反应(PCR)、逆转录PCR和实时荧光定量PCR等.细胞培养法是检测病毒感染性的最可靠方法, 但该方法需要特定培养细胞, 大多数病毒根本无法培养.基于病毒核酸扩增方法已成为病毒检测的普遍方法, 目前已用于在地表水、河水和自来水中检测病毒[10].但该方法仅能检测病毒的核酸, 但却不可以表明病毒的感染性, 细胞培养与PCR法相结合的检测方法, 其灵敏度高, 可以检测到具有感染性的病毒[11].但是PCR检测要求在诊断前已知目标病毒的基因组序列[12], 没有用于PCR检测的特异引物, 给序列未知的新病毒的识别带来了困难.病毒宏基因组分析是一种与培养无关的方法, 不需要事先知道病毒的基因组序列[13], 能够在同一时间在一个样本中识别多种病毒, 可为供水系统中病毒多样性的检测提供全面快速的方法.
本文以生产规模紫外+氯胺组合消毒供水系统为研究对象, 通过对水样病毒宏基因组分析, 全面探究了供水系统中病毒微生物的分布规律和紫外氯胺组合消毒对供水系统中病毒群落和病毒宿主的影响特性.
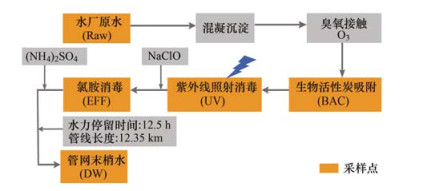
1 材料与方法 1.1 样品采集水样来自上海市某水厂及其管网末梢(如图 1), 主要包括:原水(Raw)、臭氧生物活性炭深度处理出水(BAC)、紫外消毒处理出水(UV)、氯胺消毒处理出水(EFF)和管网末梢水样(DW).紫外照射剂量为:15 mJ·cm-2, 氯胺消毒总氯质量浓度为1.07 mg·L-1, CT值为34.24 mg·min·L-1.管网采样点距水厂12.35 km, 水力停留时间(HRT)为12.5 h, 管径为DN300.样品采集于2019年10月.其它水质参数如表 1.
|
图 1 供水系统中的采样点分布示意 Fig. 1 Sampling sites in the drinking water supply system |
|
|
表 1 采样点水质特性 Table 1 Water quality characteristics for different sampling sites |
1.2 DNA提取和宏基因组测序
采用CTAB法对样本基因组DNA进行提取, 所有DNA样品送至上海美吉生物公司进行宏基因组建库和测序.插入片段长度约为400 bp, 构建PE文库:使用NEXTFLEXTM Rapid DNA-Seq Kit试剂盒, 首先进行接头连接, 使用磁珠筛选去除接头自连片段, 利用PCR扩增进行文库模板的富集, 磁珠回收PCR产物得到最终的文库.桥式PCR和测序:使用NovaSeq Reagent Kits/HiSeq X Reagent Kits试剂盒, 首先文库分子一端与引物碱基互补, 经过一轮扩增, 将模板信息固定在芯片上, 固定在芯片上的分子另一端随机与附近的另外一个引物互补, 也被固定住, 形成“桥(bridge)”, PCR扩增, 产生DNA簇, DNA扩增子线性化成为单链, 加入改造过的DNA聚合酶和带有4种荧光标记的dNTP, 每次循环只合成一个碱基, 用激光扫描反应板表面, 读取每条模板序列第一轮反应所聚合上去的核苷酸种类, 将“荧光基团”和“终止基团”化学切割, 恢复3′端粘性, 继续聚合第二个核苷酸, 统计每轮收集到的荧光信号结果, 获知模板DNA片段的序列.
1.3 宏基因组数据分析序列的质量控制:首先使用软件fastp剪切序列3′端和5′端的adapter序列; 使用软件fastp去除质量剪切后长度小于50 bp、平均质量值低于20以及含N碱基的reads, 保留高质量的pair-end reads和single-end reads; 如果在进行数据分析之前, 确认需要对样本进行宿主剔除(比如人或动物的粪便样本), 而且该宿主本身的基因组已被发表, 则通过软件BWA将reads比对宿主DNA序列, 并去除比对相似性高的污染reads.
序列组装:通过Multiple Megahit拼接软件, 选择最短contig长度≥300 bp的序列, 对结果进行ORF预测.选择核酸长度≥100 bp的基因, 并将其翻译为氨基酸序列.
基因集构建与丰度计算:通过CD-HIT软件对样本预测出来的基因序列进行聚类, 构建非冗余基因集, 得到非冗余基因集基因的碱基序列.用TPM[14](Transcripts Per Million):[(Reads Number/Gene Length)_Relative]×1 000 000表示各样本的基因丰度, 即基因丰度以该基因通过基因长度标准化后的基因丰度在该样本中所有基因长度标准化后的基因丰度总和中占比的百万倍来表示:
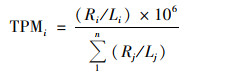
|
式中, Ri表示Genei在某一样本中的丰度值, 即该样本中比对到Genei上的Reads数; Li表示Genei的核苷酸长度; 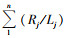
物种注释:使用BLASTP(BLAST Version 2.2.28+, http://blast.ncbi.nlm.nih.gov/Blast.cgi)将非冗余基因集与NR数据库进行比对(BLAST比对参数设置期望值e-value为1e-5), 并通过NR库对应的分类学信息数据库获得物种注释结果, 然后使用物种对应的基因丰度总和计算该物种的丰度, 并在Domain(域)、Kingdom(界)、Phylum(门)、Class(纲)、Order(目)、Family(科)、Genus(属)和Species(种)各个分类学水平上统计物种在各个样品中的丰度, 从而构建相应分类学水平上的丰度表.
1.4 数据处理图形绘制在R包“ circlize”完成.
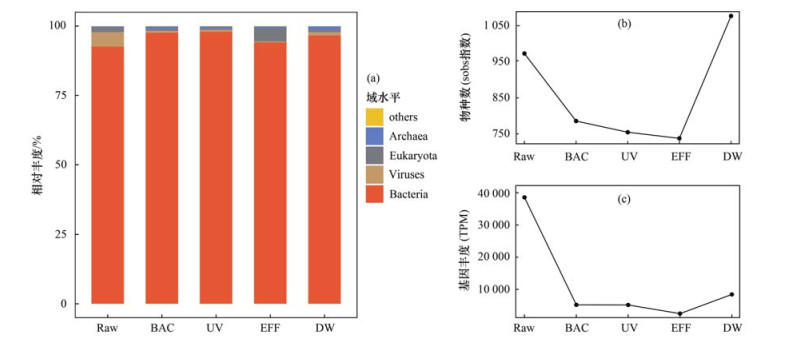
2 结果与讨论 2.1 病毒群落在供水系统中的迁移变化本研究中宏基因组测序共有25 828个基因, 总长度为15 197 245 bp, 平均基因长度为588.4 bp.在该供水系统中, 检测到细菌(92.59%~97.97%)、病毒(0.35%~5.19%)、真菌(0.49%~4.86%)、古菌(0.38%~1.30%)和其它(0.10%~0.12%).细菌群落在整个供水系统中基因相对丰度最高[图 2(a)].
|
图 2 病毒群落基因丰度和物种数变化规律 Fig. 2 Variations in gene abundance and species number in the virus community |
本文用Sobs指数代表病毒物种数, 用基因丰度(TPM)计算病毒灭活率.在该供水系统中, 病毒物种数(sobs指数)和基因丰度(TPM)呈现相同的变化趋势[图 2(b)和图 2(c)], 经过混凝沉淀+臭氧生物活性炭深度处理, 病毒种类数减少19.28%, 基因丰度降低86.38%.据报道, 臭氧氧化可将病毒的数量减少到无法检测的水平, 可以减少许多致病性人类病毒的传播[15]; 相比较快速砂滤和生物活性炭吸附氧化, 混凝沉淀和臭氧处理对饮用水处理系统PMMoV病毒的减少贡献最大[16].本研究中, 在经过紫外消毒和紫外氯胺组合消毒后, 病毒种类数依次减少3.96%和6.13%, 基因丰度依次降低0.79%和51.97%.培养法观察150 mJ·cm-2 UV剂量对浓缩血小板中的埃博拉病毒和中东呼吸综合征冠状病毒的灭活率超过4.5个对数单位和3.7个对数单位[17]. UV消毒对MS2、Φ174、Qβ和T7等噬菌体也具有良好的灭活能力[18].有研究报道了培养法检测氯胺消毒对多种病毒的灭活效果[19], 达到3个对数单位灭活率时, 埃可病毒1型、诺如病毒和柯萨奇病毒B3型所需的CT值为:18、78和330 mg·min·L-1.由于管网微生物的附着生长[20], 长时间的管道输送和复杂的管网生长环境可使病毒丰度升高.本研究中, 到达管网末梢时物种数增加46.12%, 基因丰度增加235.85%.
美国环保署(USEPA)认为[21], 在滤池滤后水的浊度低于0.3 NTU时, 培养法检测病毒的去除率为2 lg(99%), 即常规处理工艺在去除浊度的同时可同步取得99%的病毒去除率.氯胺消毒的CT值为1 243 mg·min·L-1时, 对水温为1℃下的肠道病毒的去除率为2 lg (99%).本研究中, 原水经过混凝沉淀+臭氧生物活性炭处理后, 浊度由5 NTU降至0.34 NTU, 宏基因组数据检测到的病毒去除率为86.38%;经过紫外消毒后, 浊度由0.34 NTU降至0.17 NTU, 病毒去除率仅为0.79%;氯胺的CT值为34.24 mg·min·L-1, 病毒去除率为51.59%, 经过水厂净水工艺后病毒去除率为93.46%.对比USEPA培养法检测的单个病毒在水处理中的去除率, 利用宏基因组测得的总病毒去除率要低, 这也侧面反映了培养法检测水中病毒的局限性.
2.2 供水系统中病毒群落组成特征病毒寄生在细胞内, 其基因组包含在蛋白质衣壳内.根据它们的基因组类型, 可以分为:双链DNA (dsDNA)病毒、单链DNA (ssDNA)病毒、双链RNA(dsRNA)病毒和单链RNA(ssRNA)病毒[22].在本研究供水系统中, dsDNA病毒占有99.99%, ssDNA病毒占有0.006 9%, ssRNA病毒占有0.002 7%, dsDNA病毒占绝对优势.
图 3(a)为科水平紫外氯胺组合消毒工艺下的病毒群落组成.其中, 基因丰度最高的是unclassified_d_Viruses(26.05%~56.89%), 其次是Siphoviridae(长尾噬菌体科)(8.92%~24.78%)、Myoviridae(肌尾噬菌体科)(9.41%~22.95%)和Podoviridae(短尾噬菌体科)(4.87%~31.56%).这3个科均属于Caudovirales(有尾噬菌体目), 是dsDNA病毒.在目水平下, 有尾噬菌体目基因丰度最丰富, 在整个供水系统中占比37.34%~66.49%.如图 3(b), 由原水经过混凝沉淀+臭氧生物活性炭深度处理, 有尾噬菌体目基因丰度降低了76.34%;经过UV和氯胺消毒后, 有尾噬菌体目基因丰度先升高1.73%, 再降低59.70%.有尾噬菌体目对氯胺消毒处理较为敏感, 其中只有短尾噬菌体科对UV照射具有抗性, 基因丰度增加10.48%.管网中有尾噬菌体目基因丰度整体升高262.41%.
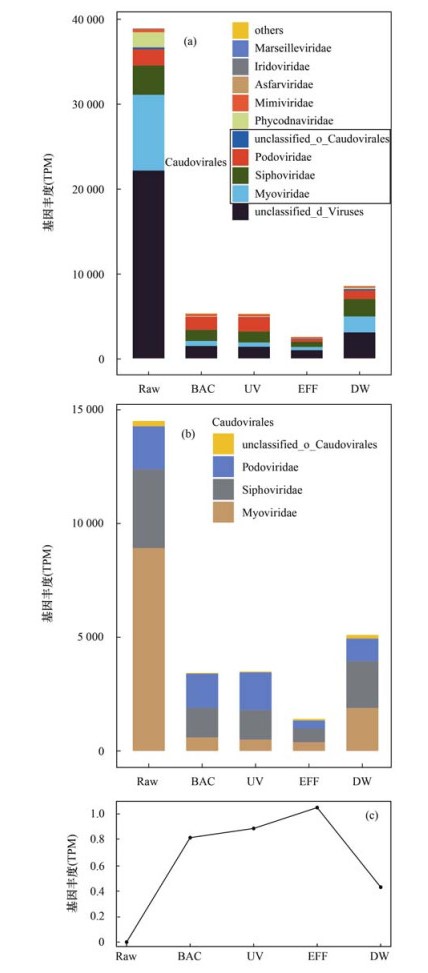
|
(a)病毒物种基因丰度分布规律(科水平); (b)有尾噬菌体目基因丰度分布规律; (c)慢病毒属(Lentivirus) 图 3 科水平和属水平下病毒基因丰度分布规律 Fig. 3 Distribution of viral gene abundance at the family and genus level |
水介质中常见病毒[23]:adenovirus(腺病毒)、coronavirus(冠状病毒)、enterovirus(肠道病毒)、norovirus(诺如病毒)、hepatitis A virus(HAV, 甲型肝炎病毒)和rotavirus(轮状病毒)在该供水系统的宏基因组数据中均没有检出, 其中enterovirus(肠道病毒)包括poliovirus(脊髓灰质炎病毒)、coxsackievirus(柯萨奇病毒)和echovirus(埃可病毒)等.
已检出的6个科的病毒中均含有致病性病毒[24], 包括鱼贝类致病病毒[Alloherpesviridae(0.002 5%)]、猪致病病毒[Asfarviridae(0.31%)]、无脊椎动物病毒[Iridoviridae(0.12%)、Baculoviridae(0.058%)]、人及动物病毒[Retroviridae(0.001 7%)]和类病毒[Phycodnaviridae(2.10%)]等.其中感染人类的Retroviridae(逆转录病毒科)在该系统中检测到两个属:Lentivirus(慢病毒属)(0.002 4%)和Gammaretrovirus(γ反转录病毒属)(0.000 3%).慢病毒属是ssRNA病毒, 包括8种能够感染人和脊椎动物的病毒、例如人类免疫缺陷病毒(HIV)、猴免疫缺陷病毒(SIV)、马传染性贫血(EIA)和猫免疫缺陷病毒(FIV)都是慢病毒属[25].如图 3(c), 慢病毒属在经过混凝沉淀+臭氧生物活性炭深度处理后被检出, 紫外照射和氯胺消毒后基因丰度依次升高8.81%、18.36%, 慢病毒属对紫外照射和氯胺消毒都具有强抵抗力.进入管网后, 基因丰度降低59.17%.
紫外氯胺组合消毒可以有效降低病毒物种数和基因丰度, 但该组合消毒方式并不能完全去除经过其它水处理工艺后的病毒.在供水系统中, dsDNA病毒占绝对优势.有研究发现[26], 病毒基因结构会影响UV消毒效率, dsDNA病毒比ssDNA病毒对UV灭活的抗性更强, 可能是由于当dsDNA病毒在UV光照时只有一条链受到损伤, 剩余未受损的链可作为宿主细胞酶修复的模板.
2.3 病毒宿主群落组成病毒是一种必须在活细胞内寄生并以复制方式增殖的非细胞型生物, 研究宿主在供水系统中的变化规律有助于了解病毒在供水系统的迁移机制和控制途径.除了驱动宿主种群动态和基因水平转移外, 病毒还通过宿主细胞裂解将生物质转化为溶解和微粒有机物来影响微生物群落结构和功能[27].
除unclassified、unidentified、uncultured和unknown宿主外, 本系统中病毒的宿主共有259种, 包括动物、植物、微生物以及虫藻共生系统.
本供水系统中, 最丰富的病毒是uncultured Mediterranean phage uvMED(31.94%).根据文献[28]将uncultured Mediterranean phage uvMED的宿主分为3类:细菌(24%)、真核生物(1%)和unclassified(76%). uncultured Mediterranean phage uvMED的细菌宿主又分为:变形杆菌(59%)、蓝藻细菌(26%)、拟杆菌(7%)、厚壁菌门(2%)和unclassified细菌(5%).
本供水系统中最大宿主是细菌(61.50%), 主要寄生在Synechococcus(聚球藻属)中.列出基因丰度除unclassified Host之外的以宿主分类的前15种病毒[图 4(a)和图 4(b)], 其中有13种病毒宿主属于细菌, 1种属于病毒, 1种属于真核生物.聚球藻属和uncultured Mediterranean phage中的Proteobacteria(变形菌门)在原水中作为优势病毒宿主, 基因丰度分别为16.20%和6.34%.经过混凝沉淀和臭氧生物活性炭深度处理后, 病毒宿主基因丰度明显降低(86.38%)[图 4(c)], 优势病毒宿主发生了变化, 主要是:Pseudomonas(假单胞菌属)和Caulobacter(新月柄杆菌), 基因丰度分别为:18.55%、10.54%.之前有文献[29]用16S rRNA基因高通量测序得出活性炭表层生物膜中细菌种群多样性十分丰富, 且检出了致病菌, 因此生物活性炭表面微生物附着生长对后续悬浮水中微生物群落结构可产生明显影响.紫外消毒前后, 宿主基因丰度变化不大, 降低0.79%, 优势病毒宿主依旧是假单胞菌属(16.79%)和新月柄杆菌属(8.57%).有研究表明病毒聚集物可能通过紫外线辐射干扰病毒失活, 因为它们可能增强宿主细胞内受损病毒之间的基因组重组, 从而部分减少病毒失活[30].氯胺消毒后, 病毒宿主基因丰度大幅度降低51.59%, 优势病毒宿主为假单胞菌属(7.40%), 紫外氯胺组合消毒后宿主丰度整体降低51.97%.紫外氯胺组合消毒比单紫外消毒更有利于病毒宿主的去除(51.97%与0.79%).水厂处理后进入管网系统, 病毒宿主基因丰度增加76.42%, 优势病毒宿主依然是假单胞菌(11.21%), 没有变化.
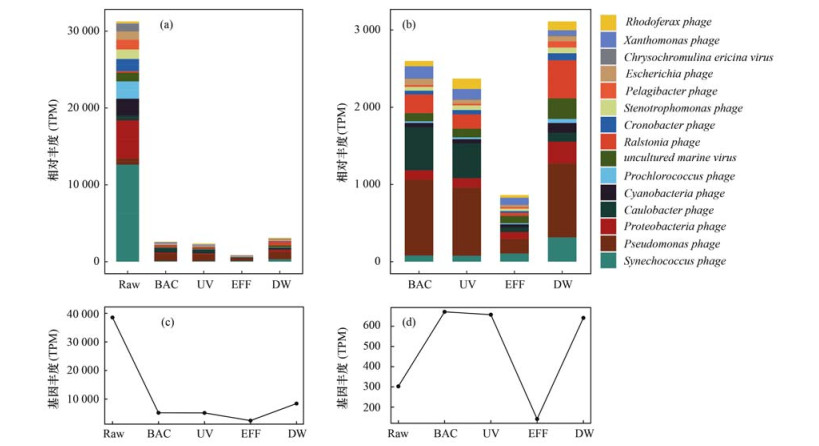
|
图 4 病毒宿主群落组成和基因丰度变化规律 Fig. 4 Variations in the community composition and gene abundance of virus hosts |
该供水系统中共检出100种寄生在假单胞菌中的病毒, 其中有59种病毒寄生在Pseudomonas aeruginosa(铜绿假单胞菌)中.铜绿假单胞菌是一种致病性细菌, 可在植物和动物(包括人)中引起疾病, 是患者院内获得性感染(HAIs)的主要原因[31].供水系统中寄生在铜绿假单胞菌中的病毒基因丰度变化如图 4(d), 经过混凝沉淀和臭氧生物活性炭深度处理后, 铜绿假单胞菌宿主病毒基因丰度增加119.9%;经紫外和氯胺消毒后, 基因丰度依次降低2.09%和77.95%;水厂处理后进入管网系统, 基因丰度升高342.62%, 应加强关注输配管网中病毒微生物风险变化.
3 结论(1) 利用宏基因组技术分析, 紫外氯胺组合消毒工艺能降低病毒物种数(6.13%)和基因丰度(51.97%), 该组合消毒方式在目前工艺条件下不能去除所有水中病毒微生物.
(2) 对比USEPA培养法检测的单个病毒在水处理中的去除率(99%~99.99%), 利用宏基因组测得的总病毒去除率要低(93.46%), 以培养法评价不能保证净水工艺对病毒微生物的完全去除.
(3) 在该供水系统中, dsDNA病毒占绝对优势. Caudovirales(有尾噬菌体目)在整个供水系统中基因丰度最大, 对氯胺消毒处理有一定的敏感性. Lentivirus(慢病毒属)作为能够感染人和脊椎动物的病毒, 对紫外照射和氯胺消毒都具有较强抵抗力.
(4) 本供水系统中最大的病毒宿主是细菌(61.50%).原水中病毒主要寄生在Synechococcus(聚球藻属)中, 经过水厂净水工艺以及到达管网后, 优势病毒宿主均为Pseudomonas(假单胞菌属).其中Pseudomonas aeruginosa(铜绿假单胞菌)宿主病毒在进入管网系统后, 基因丰度升高342.62%, 应加强关注管网系统病毒微生物风险.
(5) 在去除病毒宿主方面, 氯胺消毒效率(51.59%)明显高于紫外照射(0.79%), 组合消毒工艺更有利于病毒宿主的去除(51.97%).
| [1] | Bergion V, Lindhe A, Sokolova E, et al. Risk-based cost-benefit analysis for evaluating microbial risk mitigation in a drinking water system[J]. Water Research, 2018, 132: 111-123. DOI:10.1016/j.watres.2017.12.054 |
| [2] | Xagoraraki I, Yin Z Q, Svambayev Z. Fate of viruses in water systems[J]. Journal of Environmental Engineering, 2014, 140(7). DOI:10.1061/(ASCE)EE.1943-7870.0000827 |
| [3] | Fuhrman J A. Microbial community structure and its functional implications[J]. Nature, 2009, 459(7244): 193-199. DOI:10.1038/nature08058 |
| [4] | Zhou Y F, Fernandez S, Yoon I K, et al. Metagenomics study of viral pathogens in undiagnosed respiratory specimens and identification of human enteroviruses at a Thailand hospital[J]. The American Journal of Tropical Medicine and Hygiene, 2016, 95(3): 663-669. DOI:10.4269/ajtmh.16-0062 |
| [5] | Fujioka R S, Yoneyama B S. Sunlight inactivation of human enteric viruses and fecal bacteria[J]. Water Science and Technology, 2002, 46(11-12): 291-295. DOI:10.2166/wst.2002.0752 |
| [6] | Ye X Y, Ming X, Zhang Y L, et al. Real-time PCR detection of enteric viruses in source water and treated drinking water in Wuhan, China[J]. Current Microbiology, 2012, 65(3): 244-253. DOI:10.1007/s00284-012-0152-1 |
| [7] | Herath B S, Sathasivan A. The chloramine stress induces the production of chloramine decaying proteins by microbes in biomass (biofilm)[J]. Chemosphere, 2020, 238. DOI:10.1016/j.chemosphere.2019.124526 |
| [8] | Adeyemo F E, Singh G, Reddy P, et al. Efficiency of chlorine and UV in the inactivation of Cryptosporidium and Giardia in wastewater[J]. PLoS One, 2019, 14(5). DOI:10.1371/journal.pone.0216040 |
| [9] | Jiang B Q, Tian Y J, Zhang Z C, et al. Degradation behaviors of Isopropylphenazone and Aminopyrine and their genetic toxicity variations during UV/chloramine treatment[J]. Water Research, 2020, 170. DOI:10.1016/j.watres.2019.115339 |
| [10] | Shaheen M N F, Elmahdy E M, Chawla-Sarkar M. Quantitative PCR-based identification of enteric viruses contaminating fresh produce and surface water used for irrigation in Egypt[J]. Environmental Science and Pollution Research, 2019, 26(21): 21619-21628. DOI:10.1007/s11356-019-05435-0 |
| [11] | Ryu H, Schrantz K A, Brinkman N E, et al. Applicability of integrated cell culture reverse transcriptase quantitative PCR (ICC-RTqPCR) for the simultaneous detection of the four human enteric enterovirus species in disinfection studies[J]. Journal of Virological Methods, 2018, 258: 35-40. DOI:10.1016/j.jviromet.2018.05.008 |
| [12] | Bibby K. Metagenomic identification of viral pathogens[J]. Trends in Biotechnology, 2013, 31(5): 275-279. DOI:10.1016/j.tibtech.2013.01.016 |
| [13] | Martínez-Porchas M, Vargas-Albores F. Microbial metagenomics in aquaculture: a potential tool for a deeper insight into the activity[J]. Reviews in Aquaculture, 2017, 9(1): 42-56. DOI:10.1111/raq.12102 |
| [14] | Wagner G P, Kin K, Lynch V J. Measurement of mRNA abundance using RNA-seq data: RPKM measure is inconsistent among samples[J]. Theory in Biosciences, 2012, 131(4): 281-285. DOI:10.1007/s12064-012-0162-3 |
| [15] | Wang H, Sikora P, Rutgersson C, et al. Differential removal of human pathogenic viruses from sewage by conventional and ozone treatments[J]. International Journal of Hygiene and Environmental Health, 2018, 221(3): 479-488. DOI:10.1016/j.ijheh.2018.01.012 |
| [16] | Kato R, Asami T, Utagawa E, et al. Pepper mild mottle virus as a process indicator at drinking water treatment plants employing coagulation-sedimentation, rapid sand filtration, ozonation, and biological activated carbon treatments in Japan[J]. Water Research, 2018, 132: 61-70. DOI:10.1016/j.watres.2017.12.068 |
| [17] | Eickmann M, Gravemann U, Handke W, et al. Inactivation of Ebola virus and Middle East respiratory syndrome coronavirus in platelet concentrates and plasma by ultraviolet C light and methylene blue plus visible light, respectively[J]. Transfusion, 2018, 58(9): 2202-2207. DOI:10.1111/trf.14652 |
| [18] | Hijnen W A M, Beerendonk E F, Medema G J. Inactivation credit of UV radiation for viruses, bacteria and protozoan(oo)cysts in water: a review[J]. Water Research, 2006, 40(1): 3-22. DOI:10.1016/j.watres.2005.10.030 |
| [19] | Cromeans T L, Kahler A M, Hill V R. Inactivation of adenoviruses, enteroviruses, and murine norovirus in water by free chlorine and monochloramine[J]. Applied and Environmental Microbiology, 2010, 76(4): 1028-1033. DOI:10.1128/AEM.01342-09 |
| [20] |
白晓慧, 蔡云龙, 支兴华, 等. 供水管网不同管材内壁微生物分布的显微观察[J]. 环境科学, 2009, 30(9): 2555-2559. Bai X H, Cai Y L, Zhi X H, et al. Microscope observation of the microbial distribution on the wall of different pipe material in water supply system[J]. Environmental Science, 2009, 30(9): 2555-2559. |
| [21] |
解跃峰, 马军. 饮用水厂病毒去除与控制[J]. 给水排水, 2020, 46(3): 1-3. Xie Y F, Ma J. Virus removal and control in drinking water plant[J]. Water & Wastewater Engineering, 2020, 46(3): 1-3. |
| [22] | Flint J, Mackay T F C. Genetic architecture of quantitative traits in mice, flies, and humans[J]. Genome Research, 2009, 19(5): 723-733. DOI:10.1101/gr.086660.108 |
| [23] |
董慧峪, 李凌菲, 刘沛峰, 等. 饮用水消毒工艺对病毒的灭活[J]. 环境工程学报, 2020, 7(14): 1718-1727. Dong H Y, Li Y F, Liu P F, et al. Inactivation of virus by drinking water disinfection process[J]. Chinese Journal of Environmental Engineering, 2020, 7(14): 1718-1727. |
| [24] |
葛英亮, 于水利, 时文歆. 应用Illumina Miseq测序分析饮用水源水中病毒多样性[J]. 食品科学, 2018, 39(2): 287-292. Ge Y L, Yu S L, Shi W X. Analysis of virus diversity in drinking source water by using Illumina MiSeq sequencing technology[J]. Food Science, 2018, 39(2): 287-292. |
| [25] | Bukong T N, Hall W W, Jacque J M. Lentivirus-associated MAPK/ERK2 phosphorylates EMD and regulates infectivity[J]. Journal of General Virology, 2010, 91(9): 2381-2392. DOI:10.1099/vir.0.019604-0 |
| [26] | Calgua B, Carratalà A, Guerrero-Latorre L, et al. UVC inactivation of dsDNA and ssRNA viruses in water: UV fluences and a qPCR-based approach to evaluate decay on viral infectivity[J]. Food and Environmental Virology, 2014, 6(4): 260-268. DOI:10.1007/s12560-014-9157-1 |
| [27] | Suttle C A. Marine viruses—major players in the global ecosystem[J]. Nature Reviews Microbiology, 2007, 5(10): 801-812. DOI:10.1038/nrmicro1750 |
| [28] | Ghai R, Martin-Cuadrado A B, Molto A G, et al. Metagenome of the mediterranean deep chlorophyll maximum studied by direct and fosmid library 454 pyrosequencing[J]. The ISME Journal, 2010, 4(9): 1154-1166. DOI:10.1038/ismej.2010.44 |
| [29] |
王敏, 尚海涛, 郝春博, 等. 饮用水深度处理活性炭池中微生物群落分布研究[J]. 环境科学, 2011, 32(5): 1497-1504. Wang M, Shang M T, Hao C B, et al. Diversity and bacteria community structure of activated carbon used in advanced drinking water treatment[J]. Environmental Science, 2011, 32(5): 1497-1504. |
| [30] | Mattle M J, Kohn T. Inactivation and tailing during UV254 disinfection of viruses: contributions of viral aggregation, light shielding within viral aggregates, and recombination[J]. Environmental Science & Technology, 2012, 46(18): 10022-10030. |
| [31] | Azam M W, Khan A U. Updates on the pathogenicity status of Pseudomonas aeruginosa[J]. Drug Discovery Today, 2019, 24(1): 350-359. DOI:10.1016/j.drudis.2018.07.003 |
 2021, Vol. 42
2021, Vol. 42


