2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
抗生素抗性基因被认为是一种新型环境污染物[1], 与传统化学污染物不同, 抗生素抗性基因具有能够随着微生物繁殖而自我复制(污染倍增)和微生物物种间横向迁移(污染扩散)等独特的生物学特性[2~4].目前, 在城市河流[5]、城市污水[6]、饮用水源地的水库水[7]和湖泊水[8]等水体环境中都有抗生素抗性基因的检出报道, 这些水体环境检测出抗性基因具有多样性高和丰度水平高的特点, 甚至与微塑料和重金属元素等形成了复合环境污染的特征[9, 10].这些水体环境所在区域往往直接或间接受到了各种人类活动的影响, 同时也深刻影响了环境微生物抗性基因的种类和分布[11].不仅仅是水体环境, 在农田土壤[12]、植物叶际[13]和空气微生物[14]等也发现抗生素抗性基因的普遍存在, 并受到多种环境因子和人类活动的影响, 引发了人类干扰下的环境微生物全球大迁徙[15].
尾矿库一般是指筑坝拦截山谷谷口或者围地构成的, 用以堆存金属或非金属矿山进行矿石采选后排出的尾矿或废渣的场地.尾矿库是一个具有较高势能的人造泥石流危险源, 存在溃坝危险, 容易发生泥石流等地质灾害, 可能造成生态环境严重污染[16].尾矿库的库区内收集了矿石采选后的随尾矿排放的废水, 并涵盖一定范围的雨水汇水区, 形成了库区内的独特水体环境.此外, 尾矿坝一般设有尾矿排渗水收集系统, 用以排出库区尾矿堆积体的水分, 保障尾矿库的总体安全.尾矿库水体环境, 直接受到尾矿处理等矿业活动的影响, 是一个特殊水体环境, 对地表水和地下水具有潜在影响.目前针对尾矿库水体环境抗生素抗性基因的相关研究还比较少, 尾矿库水体环境抗生素抗性基因污染特征和驱动机制还不甚清楚.本研究采用超高通量PCR技术, 从抗生素抗性基因谱集合的角度, 深入研究了尾矿库水体环境抗生素抗性基因的多样性、丰度和影响因素, 阐释抗性基因分布和驱动机制, 以期为尾矿库生态环境风险评估提供科学支撑和决策依据.
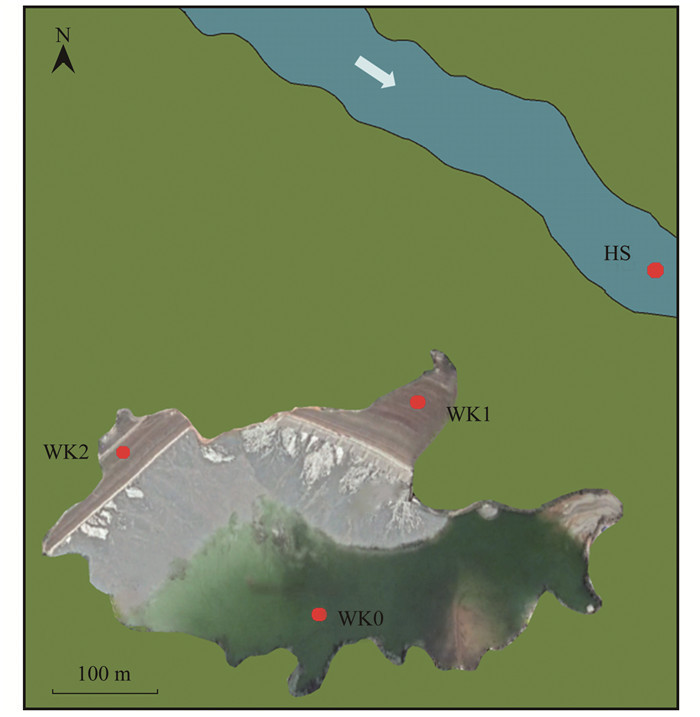
1 材料与方法 1.1 尾矿库水体环境本研究选定的尾矿库位于福建省龙岩市境内, 为山谷型尾矿库, 采用上游式筑坝法, 初期坝类型为透水堆石坝(有排渗管导出尾矿堆体内部的滞水, 即排渗水), 设计总库容为627.98万m3, 总坝高为57 m, 初期坝坝高13.5 m, 堆积坝坝高43.5 m, 库区汇水面积为0.662 km2, 属四等库.该尾矿库的初期坝具体由主坝和副坝组成, 尾矿库的东北方向有河流(汀江)经过(图 1).
|
图 1 尾矿库水体环境及采样点示意 Fig. 1 Water environment and sampling points of the tailings reservoir |
针对该尾矿库的水环境地理特征, 采集了尾矿库主坝(WK1)和尾矿库副坝(WK2)的排渗水, 同时采集了尾矿库库区水体(WK0)和附近的河流水(HS), 这4个不同点位的水样能够较好地代表该尾矿库的总体水体环境(图 1).采集的水样, 用低温保存箱运回实验室, 用六联排抽滤装置进行水样负压抽滤, 获得含有微生物的滤膜, 抽滤膜选用的是0.22 μm无菌滤膜(ADVANTEC Co., 日本).
用无菌的不锈钢手术剪将得到的滤膜剪碎成约1 mm×1 mm的形态, 选用FastDNAⓇ Spin Kit(MP, 美国)试剂盒.确保滤膜上微生物充分震荡裂解, 进而保证DNA提取效率.根据试剂盒说明书的DNA提取步骤, 获得水样中微生物的总DNA样品(洗脱液DES的体积为100 μL).随后用Qubit 3荧光计(ThermoFisher, 美国)来测定DNA样品初始浓度.然后根据浓度计算每个DNA样品的原液所需体积, 配置成浓度为30 ng ·μL-1, 体积为45 μL的样品, 放在-80℃冰箱保存, 用于后续的超高通量qPCR上机实验.
1.3 金属元素测定与总有机碳测定采集的尾矿库水体环境样品采用0.45 μm水系滤膜过滤后, 每个样品得到35 mL的过滤水样.取10 mL过滤水样, 采用ICP-MS方法(Agilent 7500cx)测定水体的Ni、Cu、As和Cd等元素的浓度; 另取25 mL过滤水样, 采用燃烧法(Shimadzu TOC VCPH)测定总有机碳(TOC)的含量.
1.4 超高通量qPCR本研究采用的超高通量qPCR反应平台是TaKaRa公司旗下的SmartChip Real-Time PCR系统, 定量的目标基因是课题组研究选定的296种基因引物[14, 17], 涵盖了283种抗性基因.另外, 引物组合中还包含了12种可移动遗传元件(MGEs, 其中转座子8种, 整合子4种)和1种核糖体16S rRNA基因.这些抗生素抗性基因/可移动遗传元件对应的抗性机制具有典型的代表性, 而且类别全面.该qPCR平台单次理论上最大PCR通量为5 184个, 单个独立PCR反应体系为100 nL, 具有组合灵活、成本低和通量高的特点.芯片SmartChip中的各试剂都是由超微量自动加样器(MSND)进行设定后自动精确加样.超高通量qPCR反应采用“两步法”, 具体PCR反应程序与条件参考Ding等[14]的方法. qPCR反应结束后, Cycler会自动进行熔解曲线分析, 并可以对Ct值进行初步筛选, 保留扩增效率介于1.85~2.15的有效数据(Ct值).
此外, 还根据标准质粒外标法, 采用荧光定量PCR仪(Roche 480Ⅱ)对尾矿库水体环境微生物的16S rRNA基因进行了常规荧光定量PCR.
1.5 数据处理与分析方法将超高通量qPCR定量得到的各个样品的16S rRNA基因Ct值, 计算相应的相对拷贝数数值[5], Pearson线性回归拟合结果显示, 其与常规荧光定量PCR得到的16S rRNA基因绝对丰度呈极显著正相关的关系(P<0.01), 说明两者的线性关系极为显著.参照相关方法[5, 18], 最终可以得到尾矿库水体环境各种抗生素抗性基因的绝对丰度.超高通量qPCR的数据通过Microsoft公司的Excel 2019进行综合计算整理, 采用IBM SPSS 22.0进行数据的显著性和相关性分析, 采用OriginPro 9.1进行直方图等的作图, 采用R 4.0.1版本软件进行冗余分析(RDA)和热图分析.
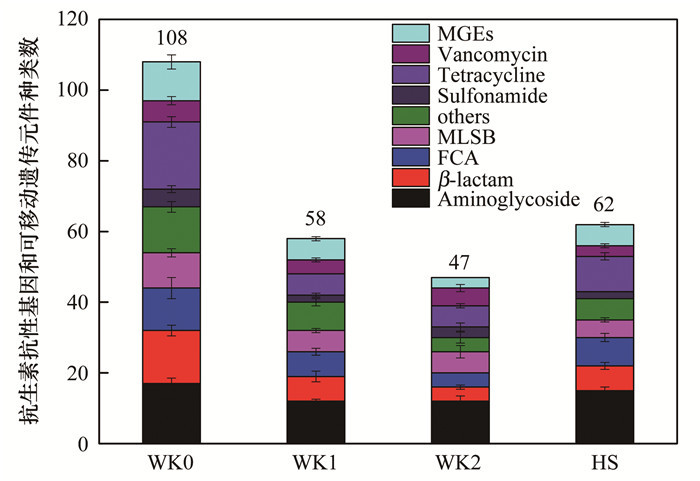
2 结果与分析 2.1 尾矿库水体环境抗生素抗性基因的种类分析尾矿库水体环境总共检测到了106种抗生素抗性基因(ARGs)和11种可移动遗传元件(MGEs), 水体中各类抗性基因种类数目具体如图 2所示.这些检测到的抗生素抗性基因涵盖了不同类型的抗性基因.具体包含有:万古霉素类(Vancomycin)、四环素类(Tetracycline)、磺胺类(Sulfonamide)、大环内酯类/林肯酰胺类/链阳性菌素B类(MLSB)、氟喹诺酮类/氯霉素类/胺酰醇类(FCA)、β-内酰胺大类(β-Lactam)和氨基糖苷大类(Aminoglycoside)等.
|
图 2 抗生素抗性基因和可移动遗传元件种类数 Fig. 2 Detected number of ARGs and MGEs |
从图 2可以看出, 尾矿库水体环境具有类型多样的抗生素抗性基因, 库区水体(WK0)的抗生素抗性基因和可移动遗传元件的种类数都是最高的, 分别为97和11种.主坝排渗水(WK1)和副坝排渗水(WK2)分别检测出抗生素抗性基因52和44种, 可移动遗传元件分别检测到6和3种, 主坝排渗水(WK1)的抗性基因种类数高于副坝排渗水(WK2), 但是都显著低于库区水体(WK0).尾矿库附近河水(HS)则检测到了抗生素抗性基因56种和可移动遗传元件6种, 总体上高于主坝和副坝的排渗水(WK1和WK2).从抗生素抗性基因种类数上分析, 库区水体(WK0)的抗性基因拥有最多的种类, 可能通过水体下渗过程, 进而影响排渗水和河水的抗性组成和分布.
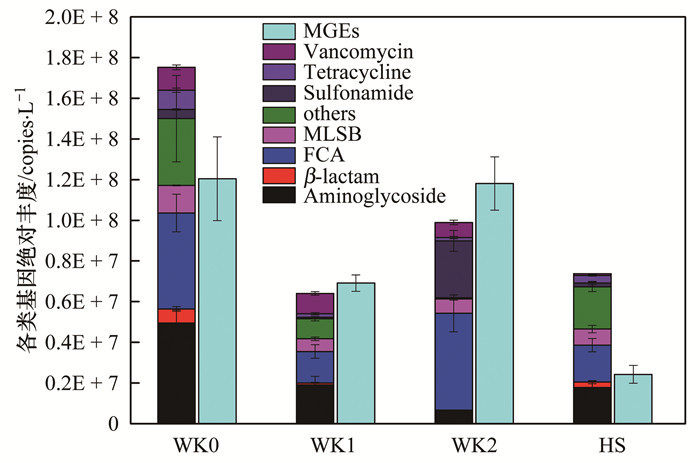
2.2 尾矿库水体环境抗生素抗性基因的丰度分析尾矿库水体环境中抗生素抗性基因的绝对丰度水平如图 3所示.可以看出, 库区水体(WK0)的抗生素抗性基因的绝对丰度最高, 达到了1.75×108 copies ·L-1, 可移动遗传元件丰度为1.21×108 copies ·L-1.主坝排渗水(WK1)和副坝排渗水(WK2)抗生素抗性基因的丰度分别为6.39×107 copies ·L-1和9.90×107 copies ·L-1, 而可移动遗传元件丰度分别为6.93×107 copies ·L-1和1.18×108 copies ·L-1, 都高于抗生素抗性基因的丰度水平, 因此排渗水可能存在较高频次的水平基因转移情况.此外, 尾矿库附近河水(HS)的抗生素抗性基因水平为7.37×107 copies ·L-1, 可移动遗传元件的丰度为2.44×107 copies ·L-1.在丰度水平上, 尾矿库附近河水(HS)的抗生素抗性基因和可移动遗传元件都显著低于库区水体(WK0)和副坝排渗水(WK2), 暗示着库区水环境可能是一个抗生素抗性基因的存储库, 并可能直接或间接影响河水抗性基因的分布和组成.
|
图 3 抗生素抗性基因和可移动遗传元件的丰度 Fig. 3 Abundance of ARGs and MGEs |
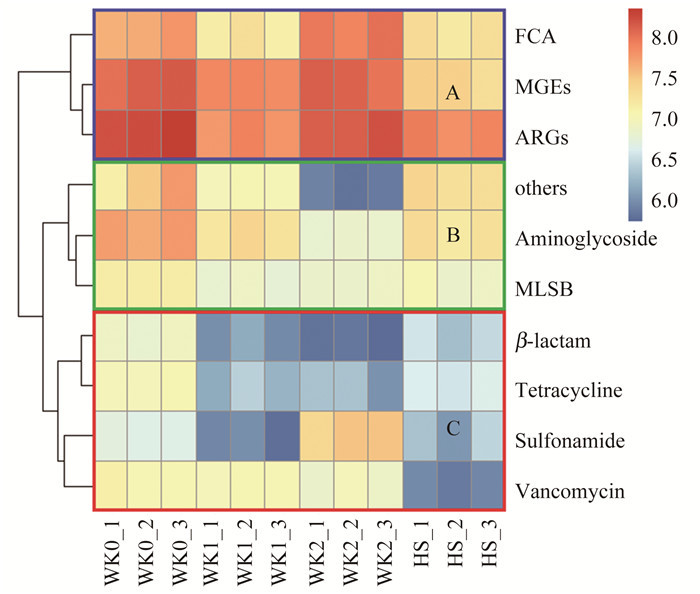
尾矿库水体环境中抗生素抗性基因的种类和丰度差异较大, 将各类抗生素抗性基因和可移动遗传元件进行综合热图分析(图 4), 可以比较直观地展示尾矿库水体环境抗性基因的差异和分布格局.
|
图 4 河水抗生素抗性基因聚类分布热图 Fig. 4 Heatmap of ARGs in river water |
图 4可以看出, 各类抗性基因与可移动遗传元件形成了A、B和C这3大簇的分布格局. A簇包括总抗性基因ARGs、可移动遗传元件MGEs和氟喹诺酮类/氯霉素类/胺酰醇类(FCA)抗性基因, 暗示着ARGs和MGEs可能存在相互影响的关系, 存在较高频次水平基因转移情况, 进而改变抗性基因的分布格局. B簇则包含了氨基糖苷类(Aminoglycoside)、大环内酯类/林肯酰胺类/链阳性菌素B类(MLSB)和其它未知机制(others)的抗性基因, B簇的抗性基因丰度水平总体略低于第一簇的基因丰度, 但总体高于C簇, 相对B簇的其它点位, 副坝排渗水的这几类抗性基因丰度相对较低. C簇则含有β-内酰胺类(β-lactam)、四环素类(Tetracycline)、磺胺类(Sulfonamide)和万古霉素类(Vancomycin)抗性基因, C簇的抗性基因丰度低于A簇和B簇, 同时内部不同点位的各类抗性基因丰度水平差异较大, 特别是主坝排渗水与副坝排渗水差异大, 河水(HS)中这些类别抗性基因也低于其它点位的丰度水平, C簇分布格局比较复杂, 表明这几类抗性基因可能与所在的具体水环境状况有紧密的关系.
3 讨论环境抗生素抗性基因的影响因素和驱动机制国内外科学家都开展了一些相关研究.有研究认为As和Cu等元素, 可能会增加抗生素抗性的选择压力, 进而增加抗性基因丰度[19, 20]. Chen等[7]关于河流-水库系统的研究认为, 可移动遗传元件(MGEs)是水环境抗生素抗性基因变化的主要因素. Bakkeren等[21]认为微生物的进化演变是细菌抗性基因存在和传播的主要原因.因此, 在各种环境因素和微生物耦合作用下, 抗生素抗性基因在不同的水体环境中的赋存和演变机制可能有不同的具体特征, 但是人类活动是流域环境微生物污染的主要因素[22]. 图 2可以看出, 库区水体的抗生素抗性基因和可移动遗传元件种类最多, 库区水体主要来自于周围自然降水汇集而成, 并含有填埋尾矿带入的废水, 可能形成了一个独特的水体环境(营养元素高、金属元素、成分复杂的洗选矿工艺废液等), 进而影响了水体环境微生物的分布和结构.而在丰度水平上(图 3), 虽然库区水体环境抗生素抗性基因丰度总体低于城市河水[5, 23], 但是主坝和副坝排渗水中可移动遗传元件丰度高于抗生素抗性基因丰度, 这与Chen等[24]关于地下水抗生素抗性基因的研究一致, 暗示着在尾矿堆体水环境中可能存在高频次和显著的抗性基因水平转移现象, 尾矿库排渗水的微生物演变及其驱动机制还需结合高通量测序等手段进一步深入研究.
本研究显示, 铜Cu、总有机碳TOC、可移动遗传元件(MGEs)与抗生素抗性基因呈显著正相关的关系(P<0.05). RDA分析表明(图 5), 尾矿库水体环境抗生素抗性基因主要受到铜Cu、总有机碳TOC和可移动遗传元件(MGEs)影响和驱动, 这3种影响因子能够解释抗生素抗性基因61.64%(前两轴)的变化特征. 图 5还可以看出, 铜Cu和可移动遗传元件(MGEs)对于库区水体(WK0)和副坝排渗水(WK2)抗生素抗性基因的影响比较大, 而总有机碳TOC与河水(HS)的抗生素抗性基因关系更为密切.
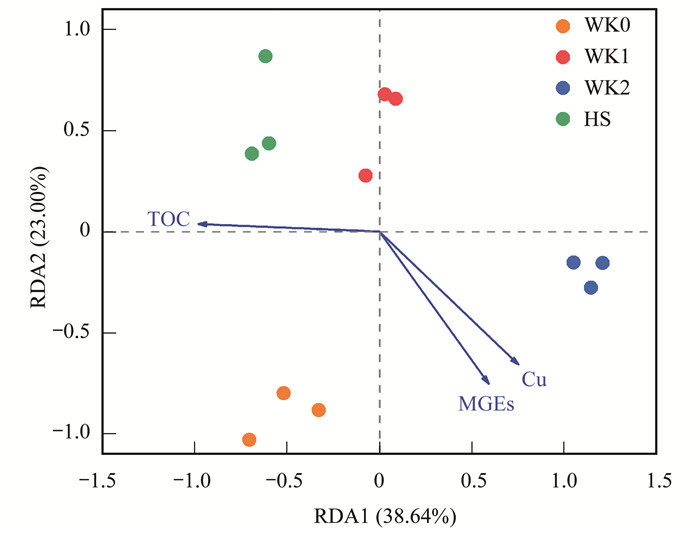
|
图 5 影响因素与抗生素抗性基因的冗余分析 Fig. 5 Redundancy analysis (RDA) of the relationships between TOC, Cu, MGEs, and ARGs |
本研究中, 尾矿库水体环境各金属元素浓度水平见表 1.可以看出, 水中的铜Cu和砷As的浓度水平都没有超过《地表水环境质量标准》(GB 3838-2002)规定的Ⅰ类水标准.镉Cd虽然在河水中未检出, 但是在尾矿库水体中浓度则属于上述标准的Ⅱ类水标准.因此, 该尾矿库水体总体金属元素含量满足Ⅱ类水体标准.但是, 低浓度金属元素仍可能对水中微生物群落结构和功能产生了影响, 并进而影响抗生素抗性基因的组成和结构[25].库区内镍Ni等含量较高元素对排渗水和河流水环境也可能构成了直接或间接的生态风险, 虽然统计显示Ni与本研究中的抗生素抗性基因无显著相关关系, 需采取更多研究手段对此进行综合分析与评估可能存在的潜在影响.
|
|
表 1 金属元素和总有机碳含量1) Table 1 Metal analysis and TOC |
4 结论
(1) 尾矿库水体环境存在着种类较丰富和丰度水平较高的抗生素抗性基因, 库区水体(WK0)抗生素抗性基因的种类和丰度都是最高的, 可能通过水体下渗直接影响了主坝和副坝排渗水的抗性基因组成, 并可能间接影响河水抗生素抗性基因.
(2) 铜Cu、总有机碳TOC和可移动遗传元件(MGEs)与抗生素抗性基因(ARGs)呈显著相关的关系, 总共解释了抗生素抗性基因61.64%(前两轴)的变化特征, 表明尾矿库水体环境抗生素抗性基因的分布主要受到了这3个因素的影响和驱动.
| [1] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants: studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [2] |
苏建强, 黄福义, 朱永官. 环境抗生素抗性基因研究进展[J]. 生物多样性, 2013, 21(4): 481-487. Su J Q, Huang F Y, Zhu Y G. Antibiotic resistance genes in the environment[J]. Biodiversity Science, 2013, 21(4): 481-487. |
| [3] | McInnes R S, McCallum G E, Lamberte L E, et al. Horizontal transfer of antibiotic resistance genes in the human gut microbiome[J]. Current Opinion in Microbiology, 2020, 53: 35-43. DOI:10.1016/j.mib.2020.02.002 |
| [4] | Fan X T, Li H, Chen Q L, et al. Fate of antibiotic resistant Pseudomonas putida and broad host range plasmid in natural soil microcosms[J]. Frontiers in Microbiology, 2019, 10: 194. DOI:10.3389/fmicb.2019.00194 |
| [5] | Ouyang W Y, Huang F Y, Zhao Y, et al. Increased levels of antibiotic resistance in urban stream of Jiulongjiang River, China[J]. Applied Microbiology and Biotechnology, 2015, 99(13): 5697-5707. DOI:10.1007/s00253-015-6416-5 |
| [6] | Su J Q, An X L, Li B, et al. Metagenomics of urban sewage identifies an extensively shared antibiotic resistome in China[J]. Microbiome, 2017, 5: 84. DOI:10.1186/s40168-017-0298-y |
| [7] | Chen Y H, Su J Q, Zhang J Y, et al. High-throughput profiling of antibiotic resistance gene dynamic in a drinking water river-reservoir system[J]. Water Research, 2019, 149: 179-189. DOI:10.1016/j.watres.2018.11.007 |
| [8] | Yang Y Y, Song W J, Lin H, et al. Antibiotics and antibiotic resistance genes in global lakes: a review and meta-analysis[J]. Environment International, 2018, 116: 60-73. DOI:10.1016/j.envint.2018.04.011 |
| [9] | Moore R E, Millar B C, Moore J E. Antimicrobial resistance (AMR) and marine plastics: can food packaging litter act as a dispersal mechanism for AMR in oceanic environments?[J]. Marine Pollution Bulletin, 2020, 150. DOI:10.1016/j.marpolbul.2019.110702 |
| [10] | Yang Y Y, Liu G H, Song W J, et al. Plastics in the marine environment are reservoirs for antibiotic and metal resistance genes[J]. Environment International, 2019, 123: 79-86. DOI:10.1016/j.envint.2018.11.061 |
| [11] | Flandroy L, Poutahidis T, Berg G, et al. The impact of human activities and lifestyles on the interlinked microbiota and health of humans and of ecosystems[J]. Science of the Total Environment, 2018, 627: 1018-1038. DOI:10.1016/j.scitotenv.2018.01.288 |
| [12] | Chen Q L, An X L, Li H, et al. Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil[J]. Environment International, 2016, 92: 1-10. |
| [13] | Chen Q L, An X L, Zheng B X, et al. Long-term organic fertilization increased antibiotic resistome in phyllosphere of maize[J]. Science of the Total Environment, 2018, 645: 1230-1237. DOI:10.1016/j.scitotenv.2018.07.260 |
| [14] | Ding L J, Zhou X Y, Zhu Y G. Microbiome and antibiotic resistome in household dust from Beijing, China[J]. Environment International, 2020, 139. DOI:10.1016/j.envint.2020.105702 |
| [15] | Zhu Y G, Gillings M, Simonet P, et al. Microbial mass movements[J]. Science, 2017, 357(6356): 1099-1100. DOI:10.1126/science.aao3007 |
| [16] |
刘玉林, 刘长淼, 刘红召, 等. 我国矿山尾矿利用技术及开发利用建议[J]. 矿产保护与利用, 2018(6): 140-144, 150. Liu Y L, Liu C M, Liu H Z, et al. Utilization technology of mine tailings in China and exploitation suggestions[J]. Conservation and Utilization of Mineral Resources, 2018(6): 140-144, 150. |
| [17] |
黄福义, 杨凯, 张子兴, 等. 微塑料对河口沉积物抗生素抗性基因的影响[J]. 环境科学, 2019, 40(5): 2234-2239. Huang F Y, Yang K, Zhang Z X, et al. Effects of microplastics on antibiotic resistance genes in estuarine sediments[J]. Environmental Science, 2019, 40(5): 2234-2239. |
| [18] | Su J Q, Wei B, Ouyang W Y, et al. Antibiotic resistome and its association with bacterial communities during sewage sludge composting[J]. Environmental Science & Technology, 2015, 49(12): 7356-7363. |
| [19] | Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440. DOI:10.1073/pnas.1222743110 |
| [20] | Poole K. At the nexus of antibiotics and metals: the impact of Cu and Zn on antibiotic activity and resistance[J]. Trends in Microbiology, 2017, 25(10): 820-832. DOI:10.1016/j.tim.2017.04.010 |
| [21] | Bakkeren E, Diard M, Hardt W D. Evolutionary causes and consequences of bacterial antibiotic persistence[J]. Nature Reviews Microbiology, 2020, 18(9): 479-490. DOI:10.1038/s41579-020-0378-z |
| [22] |
庄芳芳, 苏建强, 陈辉煌, 等. 基于高通量定量PCR研究城市化小流域微生物污染特征[J]. 生态毒理学报, 2017, 12(5): 141-152. Zhuang F F, Su J Q, Chen H H, et al. Characterization of microbial contaminants using high-throughput quantitative PCR in a small urbanizing catchment[J]. Asian Journal of Ecotoxicology, 2017, 12(5): 141-152. |
| [23] | Xu L K, Ouyang W Y, Qian Y Y, et al. High-throughput profiling of antibiotic resistance genes in drinking water treatment plants and distribution systems[J]. Environmental Pollution, 2016, 213: 119-126. DOI:10.1016/j.envpol.2016.02.013 |
| [24] | Chen Q L, Li H, Zhou X Y, et al. An underappreciated hotspot of antibiotic resistance: The groundwater near the municipal solid waste landfill[J]. Science of the Total Environment, 2017, 609: 966-973. DOI:10.1016/j.scitotenv.2017.07.164 |
| [25] | Pal C, Asiani K, Arya S, et al. Metal resistance and its association with antibiotic resistance[J]. Advances in Microbial Physiology, 2017, 70: 261-313. |
 2021, Vol. 42
2021, Vol. 42


