外来入侵植物成功定殖后, 通过种群扩张可迅速形成单优势种群落, 造成入侵地生境群落生物多样性丧失[1, 2], 植被恢复是入侵退化生态系统恢复与重建的关键措施[3, 4].原产于北美的外来入侵植物刺萼龙葵(Solanum rostratum)自1981年在我国辽宁朝阳发现以来, 随后扩散至吉林、山西、河北、北京、新疆和内蒙古等地, 并在荒地、农田和草原等本地生态系统形成优势群落, 造成本地生物多样性降低, 生态系统退化[5~8].针对刺萼龙葵入侵退化生态系统的恢复, 本课题组前期筛选出多种当地土著种和优质牧草组合替代修复刺萼龙葵, 取得了一定的可持续控制效果[9].
土壤微生物是土壤元素生物化学地球循环的主要驱动力, 对根际微生态的稳定及根际养分的供应起着重要作用[10, 11].土壤微生物与植物之间利用相互促进又相互制约的关系, 构建了一个稳定的系统, 植物分泌次生代谢物质影响着其根际土壤微生物的群落结构及多样性, 而植物根际土壤微生物区系环境, 又会显著影响植物生长发育[12~14].有研究发现, 较本土植物银毛龙葵(Solanum elaeagnifolium), 刺萼龙葵入侵我国后, 其根际土壤细菌如放线菌(Actinobacteria)多样性降低, 主要真菌菌群如子囊菌(Ascomycota)和担子菌(Basidiomycota)等真菌的丰度和多样性显著提升[15].然而植被恢复刺萼龙葵入侵退化生境后, 其根际土壤细菌群落结构及功能变化如何, 目前尚无相关报道.
近年来, 高通量测序(high throughput sequencing)技术已经被广泛应用在深入了解土壤环境中微生物群落组成和分布特征中[16~18].PICRUSt(phylogenetic investigation of communities by reconstruction of unobserved states)基于已测细菌16S rRNA基因的信息和对比Greengene数据库后近缘物种的OTU信息, 进而对菌群代谢功能进行预测.PICRUSt功能预测分析已在土壤、水体和人体内环境等多方面进行应用[19~22].本研究以此为出发点, 选取了前期研究中的2个植被组合, 采用16S rDNA MiSeq高通量测序技术, 研究刺萼龙葵入侵及植被修复后根际土壤细菌群落组成, 并结合PICRUSt分析预测其根际细菌的功能, 从微生物群落结构和功能分析不同植被恢复措施控制刺萼龙葵的差异变化, 以期为刺萼龙葵的入侵机制及入侵退化生态恢复提供理论依据.
1 材料与方法 1.1 样品采集本试验地点位于吉林省白城市洮北区平台镇(45°57′N, 122°85′E), 位于科尔沁自然草原北部, 海拔180 m, 全年平均气温4.9℃, 年平均降水量408 mm, 土壤类型为淡黑钙土.本试验设置4个处理, 分别为:本地自然植被(NP), 无刺萼龙葵发生, 以羊草(Leymus chinensis)为主.刺萼龙葵入侵区域(SR), 以刺萼龙葵为主, 伴生植物主要为羊草, 以及从课题组之前筛选的刺萼龙葵植物修复组合中选取2个处理, 分别为:沙打旺(Astragalus adsurgens)+披碱草(Elymus dahuricus)+无芒雀麦(Bromus inermis)(T1);沙打旺+苇状羊茅(Festuca arundinacea)+冰草(Agropyron cristatum)+羊草(T2).于2019年9月采用抖落法收集各处理中刺萼龙葵根际土壤样品, 每个处理设置4个生物学重复.将收集的土壤样品置于预先灭菌容器中存于干冰盒, 及时运回实验室提取土壤细菌总DNA, 剩余土壤样品用于土壤理化性质测定.
1.2 土壤细菌总DNA提取及理化性质测定土壤细菌总DNA提取, 参照MOBIO公司试剂盒(PowerSoil® DNA Isolation Kit)说明书进行.将提取得到的根际细菌总DNA通过微量紫外分光光度计(NanoDrop 2000, Thermo, USA)测定DNA浓度和纯度, 用琼脂糖凝胶电泳检测其完整性.
所采土壤样品带回实验室于阴凉处室温风干, 测定土壤样品pH、有机质(OM)、总氮(TN)、总磷(TP)、总钾(TK)、有效氮(AN)速效磷(AP)及速效钾(AK)等理化指标[23].
1.3 高通量测序采用通用引物(338F:5′-ACTCCTACGGGAGG CAGCAG-3′, 806R:5′-GGACTACHVGGGTWTCTAA T-3′)对细菌16S rRNA基因的V3~V4区进行PCR扩增, 扩增体系为5×FastPfu Buffer 4 μL、2.5 mmol·L-1 dNTPs 2 μL、Forward Primer (5 μmol·L-1)0.8 μL、Reverse Primer (5 μmol·L-1)0.8 μL、FastPfu Polymerase 0.4 μL、BSA 0.2 μL、DNA模板10 ng, 补充ddH2O至20 μL. PCR扩增反应条件为:95℃, 3 min; 30×(95℃, 30 s; 54℃, 30 s; 72℃, 45 s); 72℃, 10 min. MiSeq文库构建及Illumina测序委托上海美吉生物医药科技有限公司的MiSeq PE 300测序仪(Illumina Inc, San Diego, CA, USA)完成.
1.4 数据分析 1.4.1 土壤高通量测序数据分析对测序得到的原始数据进行拼接、过滤, 得到有效数据.高通量数据采用QIIME 1.9.1(quantitative insights into microbial ecology)进行生物信息学分析[24].根据序列的相似度, 将序列归为多个OTU(operational taxonomic unit), 为了得到每个OTU对应的物种分类信息, 采用Usearch(版本7.0)软件平台对97%相似水平的OTU代表序列进行分类学分析[25].为了得到每个OTU对应的物种分类信息, 采用RDP classifier贝叶斯算法对OTU序列进行分类学分析[26].从各个OTU中挑选出一条序列作为该OTU的代表序列, 将该代表序列与已知物种的16S数据库(Silva, http://www.arb-silva.de)进行物种注释分析; 根据每个OTU中序列的条数, 得到各个OTU的丰度, 为避免各样品微生物量的差异, 按最小样本序列数进行样本序列抽平处理, 得到标准化数据用于后续统计分析.Chao1及ACE指数通过Mothur软件(版本1.30.1)计算, Shannon指数和Simpson指数通过α多样性分析获得[27].利用Canoco(版本4.5)软件对细菌OTUs水平群落丰度与环境因子进行冗余分析(redundancy analysis, RDA).利用Bray-Curtis算法进行细菌群落聚类分析[28].
1.4.2 PICRUSt功能预测分析16S功能和代谢途径预测是通过PICRUSt对OTU丰度表进行标准化, 根据KEGG数据库的信息, 获得各功能基因的丰度, 推测微生物群落的功能信息[29].预测基因丰度利用HemI Heatmap Illustrator Program 1.0.3进行热图绘制[30].
1.4.3 统计分析土壤理化性质、高通量测序结果及多样性指数等数据分析在SPSS 22. 0中进行, 组间差异性检验采用t测验和单因素方差分析(One-way ANOVA).
2 结果与分析 2.1 不同处理土壤理化性质分析在本研究中, 刺萼龙葵入侵后, 相对于本地植被, pH、有机质、总氮、总磷及有效氮均显著提高(P < 0.05, 表 1), 而植被恢复后, 不同处理区土壤理化性质发生不同的变化, 其中沙打旺+披碱草+无芒雀麦(T1)相对于本地植被变化较小, 仅有机质及有效钾显著降低(P<0.05), 而沙打旺+苇状羊茅+冰草+羊草(T2)处理区有机质、总氮、总磷、总钾及有效氮均显著升高(P < 0.05), 而有效钾显著降低(P < 0.05).
|
|
表 1 各处理土壤主要理化参数1) Table 1 Main physical and chemical properties of the different samples |
2.2 高通量测序结果及多样性评估
高通量测序结果显示(表 2), 不同处理样品平均测序序列条数为51 296, 文库覆盖率(good coverage)均高于95%, 刺萼龙葵入侵(SR)后Simpson、ACE及Chao1指数均高于本地植被(NP), 但未达到显著水平, 而植被恢复(T1和T2)后, Shannon指数和Chao 1指数显著低于刺萼龙葵及本地植被区(P < 0.05).
|
|
表 2 不同处理样品根际细菌群落多样性 Table 2 Rhizobacterial community diversity in the different samples |
2.3 土壤细菌群落组成分析
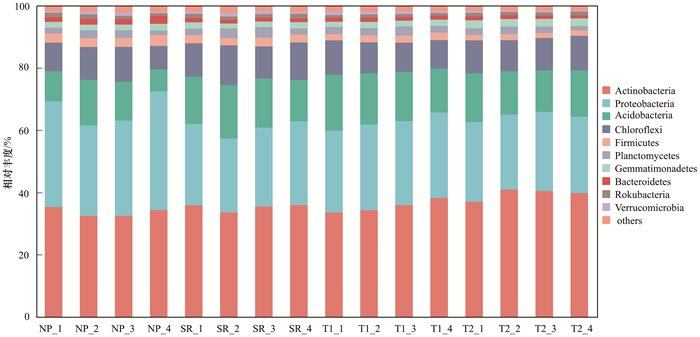
高通量结果显示, 经按最小样本序列数抽平后, 所有样品中细菌隶属33门92纲, 226目, 395科以及789属.其中不同处理区样品土壤细菌群落中优势菌群有放线菌门(Actinobacteria, 32.45%~40.91%)、变形菌门(Proteobacteria, 23.76%~38.26%)、酸杆菌门(Acidobacteria, 7.00%~18.01%)和绿弯菌门(Chloroflexi, 7.56%~12.76%), 这些优势门菌群占总菌数的86.73%~90.36%(图 1).
|
横坐标表示不同处理样品,每个样品有4个重复 图 1 不同样品门水平上物种相对丰度分布 Fig. 1 Relative read abundance of bacterial community structures at the phylum level in different samples |
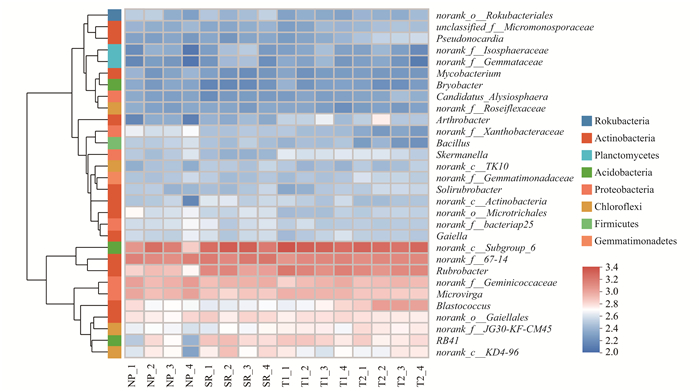
根据所有样品在属分类水平的物种注释及丰度信息, 选取隶属于8个门的丰度排名前30的属, 根据其在每个样品中的丰度信息, 分别从物种和样品两个层面进行聚类, 并绘制成热图, 不同颜色代表细菌的相对丰度对数lg值(图 2), 不同处理区样品土壤细菌群落中优势属为norank_c_Subgroup_6(5.73%~15.97%)、norank_ f_67-14(7.84%~11.86%)、红色杆菌属(Rubrobacter, 4.34%~10.79%)、norank_ f_Geminicoccaceae(4.62%~8.80%)及微枝形杆菌属(Microvirga, 4.52%~8.38%).
|
不同颜色代表细菌的相对丰度对数lg值 图 2 属水平下各样品土壤细菌群落组成 Fig. 2 Composition of the bacterial community at the genus level |
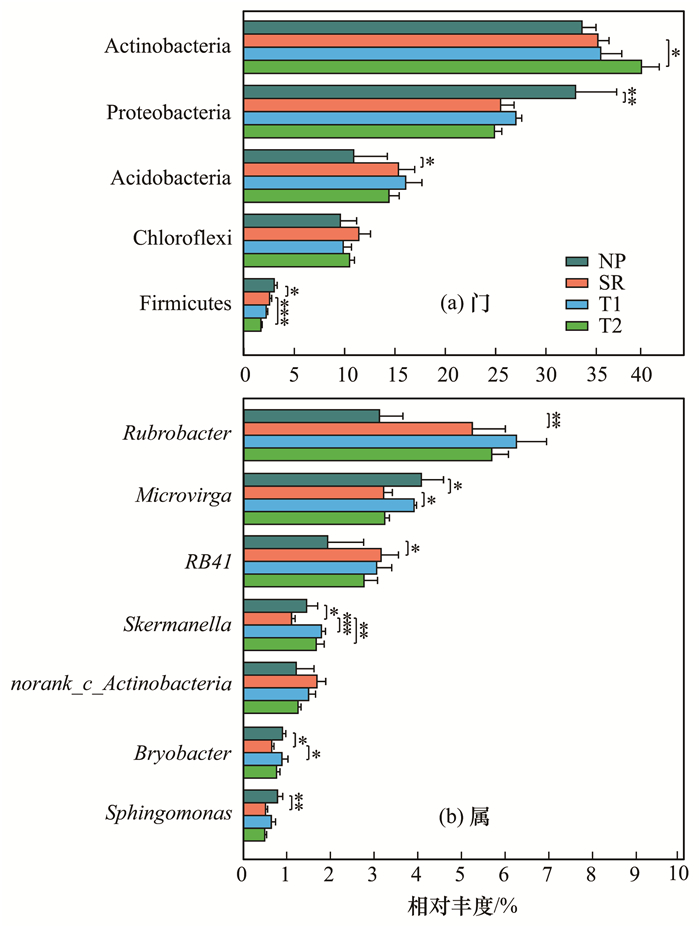
基于不同样本中群落丰度数据, 利用单因素方差分析(one-way ANOVA)比较了刺萼龙葵入侵及植被恢复对土壤细菌群落分布的影响, 结果发现, 相比本地植被, 刺萼龙葵入侵(SR)显著降低了变形菌门及厚壁菌门(Firmicutes)菌群丰度, 而酸杆菌门菌群丰度显著升高.而植被恢复以后, 变形菌门及酸杆菌门菌群丰度与刺萼龙葵入侵区差异不显著, 但厚壁菌门丰度却显著降低[图 3(a)].在属水平上, 相比本地植被群落, 刺萼龙葵入侵后, 其根际变形菌门的微枝形杆菌属(Microvirga)、斯科曼氏菌属(Skermanella)、鞘氨醇单胞菌属(Sphingomonas)及酸杆菌门的Bryobacter属均表现出下降趋势(P < 0.05), 而植被恢复后, 这些菌属丰度也随之上升.而放线菌门红色杆菌属(Rubrobacter)的丰度在刺萼龙葵入侵后较本地植被群落显著升高(P < 0.01), 植被恢复后, 其丰度依然高于刺萼龙葵入侵区域及本地植被.
|
*表示0.01 < P≤0.05, **表示0.001 < P≤0.01, ***表示P≤0.001 图 3 不同样品在门和属水平上差异显著的细菌分布 Fig. 3 Taxa with significant differences at the phylum and genus levels in different samples |
由相关性分析来看(表 3), Shannon指数与TK呈显著负相关(P =0.019), 与AK呈极显著正相关(P =0.001), Simpson指数与OM(P =0.039)、TP(P =0.048)和TK(P =0.001)呈显著正相关, 与AK呈极显著负相关(P =0.008).
|
|
表 3 土壤细菌群落多样性指数与土壤理化性质相关性1) Table 3 Person's rank correlation coefficients between soil bacterial diversity indices and measured soil characteristics |
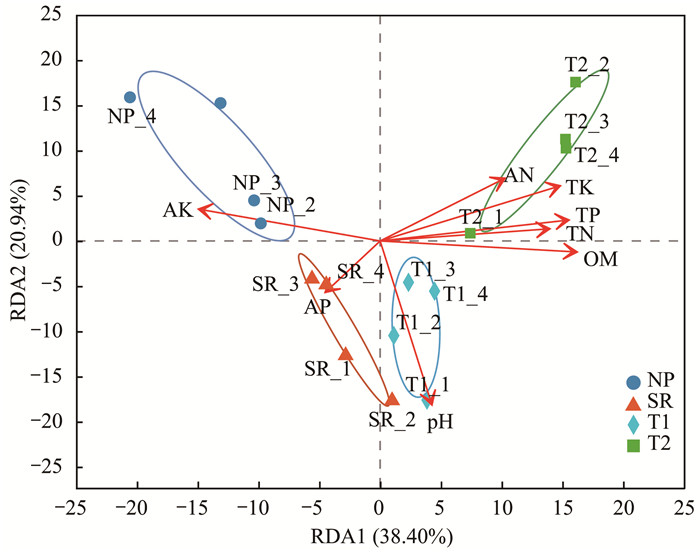
利用Canoco 4.5软件对细菌OTUs水平群落丰度进行除趋势对应分析方法(detrended correspon-dence analysis, DCA)分析, 结果显示, 4个轴中的特征值最大为0.0503, 小于3.0, 选择线性模型中冗余分析(RDA)用于后续分析.从图 4可以看出, 与第1排序轴相关性高的理化性质为:OM(R=0.998 8, P=0.004)、TN(R=0.995 7, P=0.02)、TP(R=0.990 9, P=0.003)、TK(R=0.943 4, P=0.001)和AK(R=-0.983 8, P=0.008), 说明这些环境因子与细菌群落组成具有显著的相关性, 是影响细菌群落组成的重要因素.
|
图 4 基于OTU水平细菌群落与环境因子RDA分析 Fig. 4 RDA analysis of bacterial community species and environment factors at the OTU level |
通过Pearson相关性热图分析土壤理化性质对不同处理土壤细菌属水平群落组成的影响(图 5).结果显示:OM、TN、TP、TK及AN与Solirubrobacter、芽球菌属(Blastococcus)和假诺卡氏菌(Pseudonocardia)呈正相关, 与微枝形杆菌属、Bryobacter、芽孢杆菌属(Bacillus)和norank_ f__Xanthobacteraceae呈负相关, 另外AK也显著影响多个属菌群的丰度.
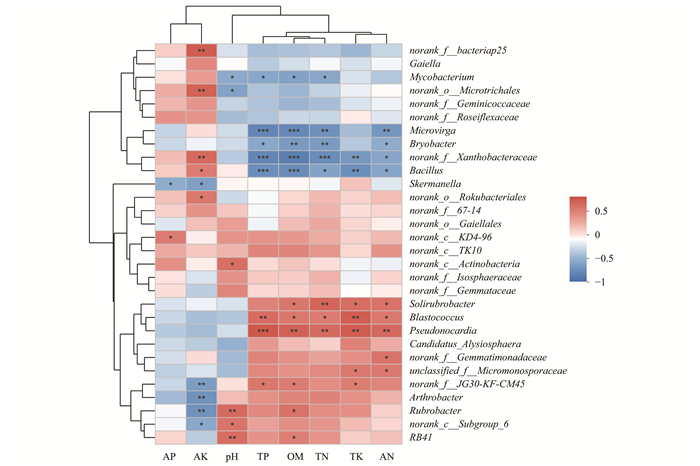
|
X轴和Y轴分别为环境因子和属水平菌群, 通过计算获得相关性R值和P值; 不同颜色表示R值, 图例表示不同R值的颜色区间; *表示0.01 < P≤0.05, **表示0.001 < P≤0.01, ***表示P≤0.001 图 5 属水平菌群与土壤理化性质相关性热图 Fig. 5 Correlation heatmap of the top fifteen phyla and soil properties |
为了预测不同处理区土壤细菌的功能, 本研究采用PICRUSt软件进行菌群预测分析.通过16S rRNA基因拷贝数归一化相对丰度来预测本研究中不同处理细菌的功能, PICRUSt预测的KEGG途径相关功能可分为代谢、遗传信息处理、环境信息处理和细胞过程等功能组.在三级功能层水平上结果显示, 不同处理区样品表现出相似的基因功能(图 6), 主要为代谢方面的功能, 包括氨基酸代谢、碳水化合物代谢、能量代谢和核酸代谢等, 另外还涉及到翻译、信号转导、膜转运及原核生物细胞群落感应等方面的功能.方差分析结果显示, 氨基酸合成、嘌呤代谢、嘧啶代谢、核糖体和氨酰-tRNA合成功能相对丰度均是刺萼龙葵入侵区>植被恢复区>本地植被区(P < 0.05), 可见刺萼龙葵入侵, 提高了该功能相对丰度, 而植被恢复以后其相对丰度显著降低.而丙酮酸代谢、原核生物碳固定途径、甘氨酸、丝氨酸和苏氨酸代谢以及糖酵解/糖异生等4个代谢相关功能, 刺萼龙葵入侵后, 较本地植被区该功能相对丰度显著升高(P < 0.05), 但植被恢复后, 并未显著降低(P>0.05).
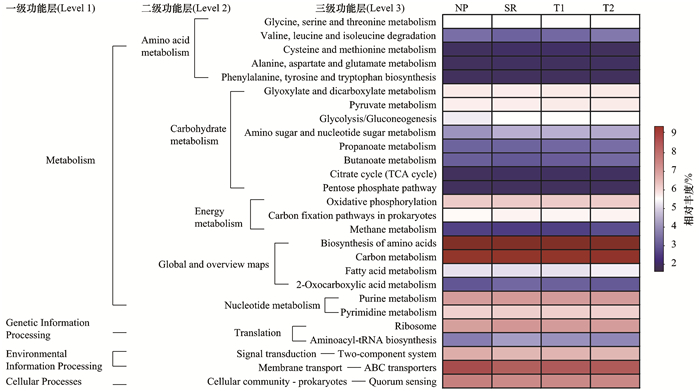
|
一级功能层:Metabolism代谢; Genetic Information Processing遗传信息处理; Environmental Information Processing环境信息处理; Cellular Processes细胞过程.二级功能层: Amino acid metabolism氨基酸代谢; Carbohydrate metabolism碳水化合物代谢; Energy metabolism能量代谢; Global and overview maps全局和概览通路; Nucleotide metabolism核苷酸代谢; Translation翻译; Signal transduction信号转导; Membrane transport膜转运; Cellular community-prokaryotes原核生物细胞群落感应.三级功能层: Glycine, serine and threonine metabolism甘氨酸、丝氨酸和苏氨酸代谢; Valine, leucine and isoleucine degradation缬氨酸、亮氨酸和异亮氨酸降解; Cysteine and methionine metabolism半胱氨酸和蛋氨酸代谢; Alanine, aspartate and glutamate metabolism丙氨酸、天冬氨酸和谷氨酸代谢; Phenylalanine, tyrosine and tryptophan biosynthesis苯丙氨酸、酪氨酸和色氨酸的生物合成; Glyoxylate and dicarboxylate metabolism乙醛酸和二元酸代谢; Pyruvate metabolism丙酮酸代谢; Glycolysis/Gluconeogenesis糖酵解/糖异生; Amino sugar and nucleotide sugar metabolism氨基糖和核苷酸糖代谢; Propanoate metabolism丙酸代谢; Butanoate metabolism丁酸盐代谢; Citrate cycle (TCA cycle)柠檬酸循环(TCA循环); Pentose phosphate pathway磷酸戊糖途径; Oxidative phosphorylation氧化磷酸化; Carbon fixation pathways in prokaryotes原核生物碳固定途径; Methane metabolism甲烷代谢; Biosynthesis of amino acids氨基酸合成; Carbon metabolism碳代谢; Fatty acid metabolism脂肪酸代谢; 2-Oxocarboxylic acid metabolism 2-氧代甲酸代谢; Purine metabolism嘌呤代谢; Pyrimidine metabolism嘧啶代谢; Ribosome核糖体; Aminoacyl-tRNA biosynthesis氨酰-tRNA生物合成; Two-component system双组分系统; ABC transporters ABC转运蛋白; Quorum sensing群体感应 图 6 不同样品预测功能基因相对丰度热图 Fig. 6 Heatmap of the relative abundances of PICRUSt predicted genes for the different samples |
国内外研究发现, 土壤微生物组成、功能与地上植物群落的组成、功能存在密切联系[31~33], 外来植物的入侵引起入侵地土壤微生物群落的改变, 反过来这种变化通过影响外来植物与当地植物间的竞争来重新布局地上植物群落[34, 35].有研究发现, 外来植物黄顶菊(Flaveria bidentis)入侵降低了土壤细菌群落多样性, 而利用4种替代植物与黄顶菊混种后, 提高土壤细菌群落多样性[36].在本研究中, 刺萼龙葵入侵(SR)后ACE及Chao1指数升高, 即刺萼龙葵入侵后, 提高了根际细菌丰度, 但并未达到显著水平(表 2).而植被恢复(T1和T2)后, Shannon指数和Chao1指数显著低于刺萼龙葵入侵区, 即植被恢复降低了刺萼龙葵根际细菌群落多样性及丰度.可见, 刺萼龙葵入侵使得本地生态系统地上群落发生变化, 而植被的恢复能够显著增加凋落物及根系分泌物的数量, 形成了不利于刺萼龙葵生长的土壤环境, 实现了对刺萼龙葵的控制.
王慧敏等[15]的研究发现, 刺萼龙葵入侵后, 其苗期根际土壤主要优势菌主要为:变形菌门、放线菌门、蓝藻门(Cyanobacteria)、拟杆菌门(Bacteroidetes)、酸杆菌门及绿弯菌门等菌群.在本研究中, 刺萼龙葵根际主要优势菌群发生较为明显的变化, 其中酸杆菌门和绿弯菌门相对丰度升高(图 1), 这种差异可能是不同生长时期及不同土壤类型造成的.RDA结果显示, OM、TN、TP、TK及AN显著影响土壤中菌群的分布(图 4).Pearson相关分析表明, 微枝形杆菌属及鞘氨醇单胞菌属与OM、TN、TK及AN显著负相关(P≤0.01), 斯科曼氏菌属与AP、AK及AN负相关, 但与AN并未达到显著水平(图 5), 这3个菌群在刺萼龙葵入侵后, 呈显著下调趋势(P < 0.05), 而植被恢复后, 这些菌属丰度也随之上升[图 3(b)].有研究发现, 微枝形杆菌属是一类根瘤固氮菌, 已在多种植物根际被分离出[37~39], 斯科曼氏菌属也在基于nifH基因高通量测序的沙地土壤中被检测到具有较高的丰度[40], 而本文中, 这2种与固氮相关的菌属与土壤氮呈负相关, 这可能因为其中刺萼龙葵入侵后, 提高了土壤中氮素含量, 而这种高浓度的氮素对固氮菌生长反而具有抑制性[41].
研究发现, 刺萼龙葵植株体内含有大量黄酮等芳香族次生代谢物质[42], 这些物质可能作为化感物质[43, 44], 在刺萼龙葵入侵中起重要作用.鞘氨醇单胞菌(Sphingomonas)存在于水体、土壤等多种环境中, 可广泛降解环境中的芳香族化合物[45, 46].在本研究中, 刺萼龙葵入侵后, 鞘氨醇单胞菌丰度显著降低[图 3(b)], 以减少刺萼龙葵次生代谢产物的降解, 从而更有利于其入侵, 而植被恢复后, 鞘氨醇单胞菌丰度稍有升高, 改变了刺萼龙葵根际细菌群落结构, 从而有助与恢复植物在与刺萼龙葵的竞争中取得优势.
目前针对外来植物入侵及植被恢复后土壤微生物菌群功能的研究开展较少, 刺萼龙葵入侵及植被恢复过程中地上植物群落和地下土壤理化性质的变化不可避免地会引起土壤微生物群落的变化, 进而影响微生物群落功能的改变.PICRUSt分析通过与数据库比对, 将微生物的变化情况和生物功能联系起来[47].氨基酸不仅是蛋白质的组成单位, 而且在许多生理过程中起着至关重要的作用, 另外氨基酸是许多次生代谢产物的前体, 具有重要的功能, 如植物信号、胁迫防御及与其他生物的互作等[48].糖酵解和原核生物碳固定等途径是碳水化合物代谢的关键途径, 它们为植物和细菌提供生长发育所需的能量和代谢产物[49].在本文中, 刺萼龙葵入侵显著提高了氨基酸及蛋白质方面代谢相关途径基因丰度, 如氨基酸合成、嘌呤代谢、嘧啶代谢、核糖体、氨酰-tRNA合成、糖酵解/糖异生和原核生物碳固定等方面的功能, 这表明刺萼龙葵根际土壤细菌参与了植物氨基酸和碳水化合物的合成, 为其生长提供营养和能量, 以满足刺萼龙葵生长需要, 而植被恢复后, 除糖酵解和原核生物碳固定途径外, 其余丰度随之降低, 这表明植被恢复影响了刺萼龙葵入侵后土壤细菌氨基酸以及蛋白质翻译等相关功能, 使其更接近本地植被群落细菌功能.目前PICRUSt初步预测相关细菌的功能, 仍有较大的局限性, 后续可结合传统分离培养方法研究与之相关的功能细菌[50]以及宏基因组学等[51]研究方法, 全面分析刺萼龙葵入侵及植被恢复过程中细菌的群落功能.
4 结论(1) 本文采用16S rDNA MiSeq高通量测序技术研究刺萼龙葵入侵及植被恢复后细菌群落组成, 刺萼龙葵入侵(SR)后Simpson指数和Chao1指数均高于本地植被(NP), 而植被恢复(T1和T2)后, Shannon指数和Chao1指数显著降低(P < 0.05), 即植被恢复降低了刺萼龙葵根际细菌群落多样性及丰度.
(2) 刺萼龙葵入侵(SR)显著降低了变形菌门(Proteobacteria)中的微枝形杆菌属(Microvirga)、斯科曼氏菌属(Skermanella)、鞘氨醇单胞菌属(Sphingomonas)及酸杆菌门(Acidobacteria)的Bryobacter属相对丰度(P < 0.05), 而植被恢复后, 这些菌属丰度也随之上升.土壤有机质、总氮、总磷、总钾和速效钾是影响细菌群落组成的重要因素.
(3) PICRUSt功能预测分析表明, 刺萼龙葵入侵显著提高了氨基酸及蛋白质方面代谢相关途径基因丰度, 如氨基酸合成、嘌呤代谢、嘧啶代谢和核糖体和氨酰-tRNA合成等方面的功能, 而植被恢复以后其相对丰度显著降低.
| [1] | Bajwa A A, Chauhan B S, Farooq M, et al. What do we really know about alien plant invasion? A review of the invasion mechanism of one of the world's worst weeds[J]. Planta, 2016, 244(1): 39-57. |
| [2] | Rai P K, Singh J S. Invasive alien plant species:their impact on environment, ecosystem services and human health[J]. Ecological Indicators, 2020, 111. DOI:10.1016/j.ecolind.2019.106020 |
| [3] |
乔文静, 戴银月, 张伟, 等. 黄土丘陵区撂荒恢复过程中植物群落组成与土壤养分及酶活性变化的关系[J]. 环境科学, 2018, 39(12): 5687-5698. Qiao W J, Dai Y Y, Zhang W, et al. Relationship between the vegetation community and soil nutrient and enzyme activity during the restoration of abandoned land in the Loess Hilly region[J]. Environmental Science, 2018, 39(12): 5687-5698. |
| [4] | Mostert E, Gaertner M, Holmes P M, et al. A multi-criterion approach for prioritizing areas in urban ecosystems for active restoration following invasive plant control[J]. Environmental Management, 2018, 62(6): 1150-1167. |
| [5] | Singh R P, Bagnall R H. Solanum rostratum Dunal., a new test plant for the potato spindle tuber virus[J]. American Potato Journal, 1968, 45(9): 335-336. |
| [6] | 车晋滇, 刘全儒, 胡彬. 外来入侵杂草刺萼龙葵[J]. 杂草科学, 2006(3): 58-60. |
| [7] |
贺俊英, 哈斯巴根, 孟根其其格, 等. 内蒙古新外来入侵植物——黄花刺茄(Solanum rostratum Dunal)[J]. 内蒙古师范大学学报(自然科学汉文版), 2011, 40(3): 288-290. He J Y, Khasbagan, Mongenqiqig, et al. Solanum rostratum Dunal:a newly invaded alien plant of Inner Mongolia[J]. Journal of Inner Mongolia Normal University (Natural Science Edition), 2011, 40(3): 288-290. |
| [8] |
向俊, 李翠妮, 刘全儒, 等. 北京外来入侵植物刺萼龙葵的生态状况[J]. 生态学杂志, 2011, 30(3): 453-458. Xiang J, Li C N, Liu Q R, et al. Ecological state of invasive alien plant Solanum rostratum in Beijing[J]. Chinese Journal of Ecology, 2011, 30(3): 453-458. |
| [9] |
张瑞海, 宋振, 张国良, 等. 刺萼龙葵土壤种子库特征及其对替代控制的响应[J]. 中国生态农业学报, 2019, 27(3): 337-347. Zhang R H, Song Z, Zhang G L, et al. Characteristics of soil seed bank for Solanum rostratum Dunal. and its response to control with replaceable plants[J]. Chinese Journal of Eco-Agriculture, 2019, 27(3): 337-347. |
| [10] | Cui Y X, Fang L C, Guo X B, et al. Responses of soil microbial communities to nutrient limitation in the desert-grassland ecological transition zone[J]. Science of the Total Environment, 2018, 642: 45-55. |
| [11] | Hawley A K, Nobu M K, Wright J J, et al. Diverse Marinimicrobia bacteria may mediate coupled biogeochemical cycles along eco-thermodynamic gradients[J]. Nature Communications, 2017, 8(1): 1507. |
| [12] | Huang R L, McGrath S P, Hirsch P R, et al. Plant-microbe networks in soil are weakened by century-long use of inorganic fertilizers[J]. Microbial Biotechnology, 2019, 12(6): 1464-1475. |
| [13] | Xiao X, Fan M C, Wang E T, et al. Interactions of plant growth-promoting rhizobacteria and soil factors in two leguminous plants[J]. Applied Microbiology and Biotechnology, 2017, 101(23): 8485-8497. |
| [14] | Kama R, Sun J, Afzal M U. Soil microorganism impact on the native plant growth under single and co-invasion with invasive plants:responses through plant-soil feedback[J]. Biomedical Letters, 2020, 6(2): 104-111. |
| [15] |
王慧敏.刺萼龙葵和银毛龙葵对根际土壤微生物群落的影响[D].北京: 中国农业科学院, 2018. Wang H M. The effects of Solanum rostratum and Solanum elaeagnifolium on rhizosphere soil microbial community[D]. Beijing: Chinese Academy of Agricultural Sciences, 2018. |
| [16] | Czechowski P, Clarke L J, Breen J, et al. Antarctic eukaryotic soil diversity of the Prince Charles Mountains revealed by high-throughput sequencing[J]. Soil Biology and Biochemistry, 2016, 95: 112-121. |
| [17] | Song Z, Zhang R H, Fu W D, et al. High-throughput sequencing reveals bacterial community composition in the rhizosphere of the invasive plant Flaveria bidentis[J]. Weed Research, 2017, 57(3): 204-211. |
| [18] | Nilsson R H, Anslan S, Bahram M, et al. Mycobiome diversity:high-throughput sequencing and identification of fungi[J]. Nature Reviews Microbiology, 2019, 17(2): 95-109. |
| [19] |
厉桂香, 马克明. 北京东灵山树线处土壤细菌的PICRUSt基因预测分析[J]. 生态学报, 2018, 38(6): 2180-2186. Li G X, Ma K M. PICRUSt-based predicted metagenomic analysis of treeline soil bacteria on Mount Dongling, Beijing[J]. Acta Ecologica Sinica, 2018, 38(6): 2180-2186. |
| [20] |
孙峰, 田伟, 张菲, 等. 丹江口库区库滨带植被土壤细菌群落多样性及PICRUSt功能预测分析[J]. 环境科学, 2019, 40(1): 421-429. Sun F, Tian W, Zhang F, et al. Composition and predictive functional analysis of rhizosphere bacterial communities in riparian buffer strips in the Danjiangkou Reservoir, China[J]. Environmental Science, 2019, 40(1): 421-429. |
| [21] | Wang L, Zhang J, Li H L, et al. Shift in the microbial community composition of surface water and sediment along an urban river[J]. Science of the Total Environment, 2018, 627: 600-612. |
| [22] | Wilkinson T J, Huws S A, Edwards J E, et al. CowPI:a rumen microbiome focussed version of the PICRUSt functional inference software[J]. Frontiers in Microbiology, 2018, 9: 1095. |
| [23] | 鲍士旦. 土壤农化分析[M]. ((第三版)). 北京: 中国农业出版社, 2000. |
| [24] | Caporaso J G, Kuczynski J, Stombaugh J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. |
| [25] | Edgar R C. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461. |
| [26] | Cole J R, Wang Q, Cardenas E, et al. The ribosomal database project:improved alignments and new tools for rRNA analysis[J]. Nucleic Acids Research, 2009, 37(S1): D141-D145. |
| [27] | Kozich J J, Westcott S L, Baxter N T, et al. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform[J]. Applied and Environmental Microbiology, 2013, 79(17): 5112-5120. |
| [28] | Bray J R, Curtis J T. An ordination of the upland forest communities of southern Wisconsin[J]. Ecological Monographs, 1957, 27(4): 325-349. |
| [29] | Langille M G I, Zaneveld J, Caporaso J G, et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J]. Nature Biotechnology, 2013, 31(9): 814-821. |
| [30] | Deng W K, Wang Y B, Liu Z X, et al. HemI:a toolkit for illustrating heatmaps[J]. PLoS One, 2014, 9(11). DOI:10.1371/journal.pone.0111988 |
| [31] |
宋明华, 刘丽萍, 陈锦, 等. 草地生态系统生物和功能多样性及其优化管理[J]. 生态环境学报, 2018, 27(6): 1179-1188. Song M H, Liu L P, Chen J, et al. Biology, multi-function and optimized management in grassland ecosystem[J]. Ecology and Environment Sciences, 2018, 27(6): 1179-1188. |
| [32] | Leff J W, Bardgett R D, Wilkinson A, et al. Predicting the structure of soil communities from plant community taxonomy, phylogeny, and traits[J]. The ISME Journal, 2018, 12(7): 1794-1805. |
| [33] | Chen Y L, Xu T L, Veresoglou S D, et al. Plant diversity represents the prevalent determinant of soil fungal community structure across temperate grasslands in northern China[J]. Soil Biology and Biochemistry, 2017, 110: 12-21. |
| [34] | Niu H B, Liu W X, Wan F H, et al. An invasive aster (Ageratina adenophora) invades and dominates forest understories in China:altered soil microbial communities facilitate the invader and inhibit natives[J]. Plant and Soil, 2007, 294(1-2): 73-85. |
| [35] | Ravichandran K R, Thangavelu M. Role and influence of soil microbial communities on plant invasion[J]. Ecological Questions, 2017, 27: 9-23. |
| [36] |
闫素丽, 皇甫超河, 李刚, 等. 四种牧草植物替代控制对黄顶菊入侵土壤细菌多样性的影响[J]. 植物生态学报, 2011, 35(1): 45-55. Yan S L, Huangfu C H, Li G, et al. Effects of replacement control with four forage species on bacterial diversity of soil invaded by Flaveria bidentis[J]. Chinese Journal of Plant Ecology, 2011, 35(1): 45-55. |
| [37] | Reeve W, Parker M, Tian R, et al. Genome sequence of Microvirga lupini strain LUT6T, a novel Lupinus alphaproteobacterial microsymbiont from Texas[J]. Standards in Genomic Sciences, 2014, 9(3): 1159-1167. |
| [38] | Rejili M, Msaddak A, Filali I, et al. New chromosomal lineages within Microvirga and Bradyrhizobium genera nodulate Lupinus angustifolius growing on different Tunisian soils[J]. FEMS Microbiology Ecology, 2019, 95(9). DOI:10.1093/femsec/fiz118 |
| [39] | Msaddak A, Rejili M, Durán D, et al. Microvirga tunisiensis sp. nov., a root nodule symbiotic bacterium isolated from Lupinus micranthus and L. luteus grown in Northern Tunisia[J]. Systematic and Applied Microbiology, 2019, 42(6). DOI:10.1016/j.syapm.2019.126015 |
| [40] |
唐凯, 高晓丹, 贾丽娟, 等. 浑善达克沙地生物土壤结皮及其下层土壤中固氮细菌群落结构和多样性[J]. 微生物学通报, 2018, 45(2): 293-301. Tang K, Gao X D, Jia L J, et al. Community structure and diversity of diazotrophs in biological soil crusts and soil underneath crust of Hunshandake deserts[J]. Microbiology China, 2018, 45(2): 293-301. |
| [41] | Zhang X M, Liu W, Schloter M, et al. Response of the abundance of key soil microbial nitrogen-cycling genes to multi-factorial global changes[J]. PLoS One, 2013, 8(10). DOI:10.1371/journal.pone.0076500 |
| [42] |
郝丽娟, 王珊, 朱晶晶, 等. 刺萼龙葵化学成分研究[J]. 中国中药杂志, 2014, 39(11): 2034-2038. Hao L J, Wang S, Zhu J J, et al. Chemical constituents from Solanum rostratum[J]. China Journal of Chinese Materia Medica, 2014, 39(11): 2034-2038. |
| [43] |
邵华. 外来入侵植物刺萼龙葵的化感作用研究[J]. 种子, 2015, 34(8): 101-104. Shao H. Study on the allelopathic effect of Solanum rostratum Dunal[J]. Seed, 2015, 34(8): 101-104. |
| [44] | Shao Q, Chang L, Wei Z Y, et al. Separation of four flavonol glycosides from Solanum rostratum Dunal using solvent sublation followed by HSCCC and low column temperature preparative HPLC[J]. Journal of Chromatographic Science, 2018, 56(8): 695-701. |
| [45] |
苟敏, 曲媛媛, 杨桦, 等. 鞘氨醇单胞菌:降解芳香化合物的新型微生物资源[J]. 应用与环境生物学报, 2008, 14(2): 276-282. Gou M, Qu Y Y, Yang H, et al. Sphingomonas sp.:a novel microbial resource for biodegradation of aromatic compounds[J]. Chinese Journal of Applied & Environmental Biology, 2008, 14(2): 276-282. |
| [46] |
张梦露, 党志, 伍凤姬, 等. 利用流式细胞术研究鞘氨醇单胞菌GY2B降解菲过程中细菌表面特性的变化[J]. 环境科学, 2014, 34(4): 1449-1456. Zhang M L, Dang Z, Wu F J, et al. Using flow cytometry to explore the changes of Sphingomonas sp. GY2B bacterial surface characteristics in the process of degrading phenanthrene[J]. Environmental Science, 2014, 34(4): 1449-1456. |
| [47] | Kim M J, Do H, Cho G, et al. Comparison of microbial community of rhizosphere and endosphere in kiwifruit[J]. The Plant Pathology Journal, 2019, 35(6): 705-711. |
| [48] | Florencio-Ortiz V, Sellés-Marchart S, Zubcoff-Vallejo J, et al. Changes in the free amino acid composition of Capsicum annuum (pepper) leaves in response to Myzus persicae (green peach aphid) infestation. A comparison with water stress[J]. PLoS One, 2018, 13(6). DOI:10.1371/journal.pone.0198093 |
| [49] | Megguer C A, Fugate K K, Lafta A M, et al. Glycolysis is dynamic and relates closely to respiration rate in stored sugarbeet roots[J]. Frontiers in Plant Science, 2017, 8. DOI:10.3389/fpls.2017.00861 |
| [50] |
丛建民, 刘小雯, 于文倩, 等. 刺萼龙葵土壤微生物区系及相关功能菌研究[J]. 中国农机化学报, 2017, 38(3): 87-92. Cong J M, Liu X W, Yu W Q, et al. Study on microbial flora and functional bacteria from Solanum rostratum soil[J]. Journal of Chinese Agricultural Mechanization, 2017, 38(3): 87-92. |
| [51] | Gibbons S M, Lekberg Y, Mummey D L, et al. Invasive plants rapidly reshape soil properties in a grassland ecosystem[J]. Msystems, 2017, 2(2). DOI:10.1128/mSystems.00178-16 |
 2021, Vol. 42
2021, Vol. 42


