2. 中国科学院生态环境研究中心, 饮用水科学与技术重点实验室, 北京 100085;
3. 生态环境部对外合作与交流中心, 北京 100035
2. Key Laboratory of Drinking Water Science and Technology, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China;
3. Foreign Economic Cooperation Office, Ministry of Ecology and Environment, Beijing 100035, China
锰元素(Mn)在环境中广泛存在, 溶解态的二价锰[Mn(Ⅱ)]被氧气氧化后生成锰氧化物沉淀(主要是二氧化锰, MnO2), 其具有较高的氧化性和吸附性.Mn(Ⅱ)氧化过程不仅影响Mn元素的形态和迁移, 还会影响其他元素(如有机物及重金属)的价态和迁移[1, 2], 因此在生物地球化学循环和水处理的研究领域有着广泛的研究.在无催化剂存在条件下, 氧气氧化Mn(Ⅱ)的速率非常低, 而部分微生物能够通过催化作用大幅提高该反应速率[3~7].有研究发现锰氧化微生物的种属分类广泛, 其可能的两种锰氧化机制为:锰氧化酶直接参与锰氧化[8~11]及微生物通过产生活性氧间接氧化Mn(Ⅱ)[11~14].
以往个别研究表明, 纯培养体系中没有锰氧化活性的菌株在与其它菌株混合培养时也能够产生锰氧化活性, 如Arthrobacter与Flavobacterium菌株之间的相互作用[15~17].本实验室此前的研究发现1株节杆菌(Arthrobacter sp. QXT-31, 后简称A. QXT-31)的锰氧化基因(boxA)在单独培养时不转录表达, 也不产生锰氧化活性, 但是在与1株鞘氨醇盒菌(Sphingopyxis sp. QXT-31, 后简称S. QXT-31)混合培养时能够产生锰氧化活性[11].笔者推测其中的机制是鞘氨醇盒菌通过拮抗作用刺激了节杆菌中锰氧化基因的表达.然而研究还并不清楚:①其它鞘氨醇盒菌能否刺激节杆菌中锰氧化基因的表达, 即是否具备普遍性; ②具备刺激锰氧化作用的鞘氨醇盒菌有何功能独特性; ③将boxA序列与美国国家生物技术信息中心(NCBI)的NR数据库比对, 很多Arthrobacter菌株的基因组中有高度同源的蛋白序列, 但是这些菌株的大部分并未报道具有锰氧化活性, 因此笔者猜测这些Arthrobacter菌株中的boxA同源基因可能处于沉默状态, 并能被Sphingopyxis sp. QXT-31菌株触发转录, 产生锰氧化活性.由于节杆菌广泛存在于自然界中[18], 包含水处理系统中, 可否在处理系统中加入鞘氨醇盒菌, 强化微生物种间相互作用, 进而强化锰氧化过程.
基于上述科学问题, 本文结合纯培养、比较基因组学分析、转录组分析及反应器研究, 对微生物相互作用产生锰氧化活性的必要条件及普适性进行了进一步的探究.在此基础上, 在滤柱反应器中探讨利用生物强化(加入Sphingopyxis菌株)间接促进节杆菌的锰氧化.本研究结果将有助于人们进一步认识“微生物种间相互作用产生锰氧化”的自然普适性, 并在此基础上指导含锰水体处理的工程应用.
1 材料与方法 1.1 菌株及培养基本实验研究包括5株菌, 除本实验室分离的菌株Arthrobacter sp. QXT-31和Sphingopyxis sp. QXT-31之外, 从中国普通微生物菌种保藏管理中心(China General Microbiological Culture Collection Center, CGMCC)购得3株菌, 分别为Sphingopyxis ginsengisoli(S. ginsengisoli; CGMCC号1.9098)、Sphingopyxis chilensis(S. chilensis; CGMCC号1.9099)和Sphingopyxis witflariensis(S. witflariensis; CGMCC号1.9093).微生物纯培养采用PYG培养基[19], 包含0.5 g ·L-1的MgSO4 ·7H2 O、60 mg ·L-1的CaCl2 ·2H2 O、0.25 g ·L-1的葡萄糖、蛋白胨和酵母浸膏, 上述成分经高温(115℃, 15 min)灭菌后过滤添加10 mmol ·L-1(pH 7.2)的无菌缓冲液HEPES[4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid]和100 μmol ·L-1的MnCl2.
1.2 菌株混合培养及Mn(Ⅱ)浓度测定将4株Sphingopyxis菌分别与A. QXT-31混合培养, 考察是否能触发后者产生锰氧化活性.在固体培养基上保存的菌落先接种到PYG培养基中活化培养两次, 每次活化培养24 h, 然后将活化后的菌液按照设定的体积比添加到新鲜的PYG培养基中继续培养, 菌液的接种比例为3%.菌株在150 mL锥形瓶中培养, 培养基体积为30 mL, MnCl2初始浓度为100 μmol ·L-1.因Mn(Ⅱ)氧化生成的锰氧化物(主要是MnO2)在水中不溶, 所以可以用溶解态Mn(Ⅱ)浓度的降低来评估锰氧化.测定溶解态Mn(Ⅱ)浓度时, 先高速离心(10 000 g, 5 min)菌液, 然后用孔径为0.22 μm的滤头过滤上清液, 用电感耦合等离子体光谱仪(G8015A, 美国安捷伦5110 VDV)测定溶解态Mn(Ⅱ)的浓度.测定菌液在600 nm吸光系数, 用于建立菌株的生长曲线.
1.3 细菌基因组测序菌株A. QXT-31和S. QXT-31的全基因组序列在NCBI的获取号分别是PRJNA361372、PRJNA361373.分别提取菌株S. witflariensis和S. ginsengisoli的DNA, 通过Illumina PE250建库策略(插入片段~400 bp)进行高通量测序, 测序平台为Illumina HiSeq 4000.高通量测序得到的序列首先用测序公司的标准质控流程处理, 质控后的序列被组装基因组, 质控后的序列和基因组组装结果详见表 1.利用SPAdes(v3.13.2)[20]组装菌株基因组, 参数设置为spades.py-o spades_output-careful-pe1-1 pair_1.fastq-pe1-2 pair_2.fastq-pe1-s unpaired.fastq-k 21, 33, 55, 77, 99, 127-cov-cutoff auto.此外, 提取菌株S. chilensis的DNA, 利用第三代测序技术(PacBio RS Ⅱ)完成了基因组完成图测序, 测序和组装由上海美吉生物医药科技有限公司完成.用checkM[21]评估组装基因组的完整度.具体组装信息见表 1.
|
|
表 1 菌株基因组测序与组装信息 Table 1 Genomic sequencing and genome assembly |
1.4 系统发育及基因组相似性分析
用Barrnap(v0.9; https://github.com/tseemann/barrnap)从组装得到的3个基因组序列中预测编码16S rRNA的基因序列.从NCBI数据库中下载(下载日期为2019年11月21日)Sphingopyxis属中仅有的8个完成基因组序列和16S rRNA编码基因序列.将上述11个菌株的16S rRNA序列用ClustalX(v2.1)[22]进行多序列对齐, 然后从起始和末尾的对齐序列位置截取所有序列共有的可变区域, 将上述截取的可变区域用ClustalX做对齐处理, 并将对齐结果文件用FastTree(v2.1.11)[23]生成系统发育树文件, 最后用iTOL(v4)[24]可视化系统发育树.用fastANI[25]分析11个基因组核酸序列的相似性, 并用R的hclust脚本可视化.
1.5 转录组数据分析之前研究了A. QXT-31和S. QXT-31混合培养第0、1.5、3、4和7 h的转录组数据(NCBI获取号PRJNA290005).上述转录组数据和2个菌株的全部基因(由NCBI Prokaryotic Genome Annotation Pipeline根据基因组序列预测并做功能注释)被用来定量所有基因的转录丰度, 转录丰度的计算利用Trinity软件[26]中的脚本align_and_estimate_abundance.pl(-est_method RSEM-aln_method bowtie)完成; 用脚本abundance_estimates_to_matrix.pl(-est_method RSEM-cross_sample_norm TMM)将样品间丰度值均一化, 以便分析基因在样品间的表达差异.
1.6 代谢通路分析利用KEGG(www.kegg.jp)提供的S. QXT-31基因组中的开放编码框和代谢通路注释信息统计该菌株的代谢通路信息.用Prodigal软件[27]识别其它3株Sphingopyxis菌株基因组序列中的开放阅读框, 然后将其对应的蛋白序列分别提交到KEGG网站, 利用在线的BlastKOALA工具[28]对蛋白序列进行KO注释并获取代谢通路和模块(MODULE)完整度信息.
1.7 比较基因组分析根据能否触发A. QXT-31产生锰氧化活性, 可将4株Sphingopyxis菌株分成2组, 阳性组包含S. chilensis和S. QXT-31, 阴性组包含S. ginsengisoli和S. witflariensis.通过比对阳性组两菌株的蛋白序列获取同源基因列表:用BLASTP(v2.9.0+)[29]将菌株S. chilensis、S. ginsengisoli和S. witflariensis编码的蛋白序列分别与S. QXT-31比对(关键参数-evalue 1e-10-max_target_seqs 1-outfmt 6), 然后从比对结果中筛选对齐序列长度大于查询序列的50%、并且相同匹配百分比大于60%的比对结果作为同源基因.根据比对结果获取阳性组中2个菌株共有的同源基因列表, 获取阴性组两菌株相比S. QXT-31均缺失的蛋白列表, 最后上述2个列表的交集, 作为可能参与触发A. QXT-31产生锰氧化活性的基因.
1.8 柱反应器实验柱反应器采用有机玻璃材质制作, 高50 cm, 内径3 cm.承托层高3 cm, 填料为7~8 mm的石英砂.滤料层高30 cm, 填料为直径2~4 mm的石英砂.共使用6个柱反应器.柱反应器实验进水为地下水原水, 原水水质参数见表 2.向原水中添加100 μmol ·L-1的MnCl2, 然后用一台多通道蠕动泵(BT100-1L, 兰格, 中国)将添加了MnCl2的地下水分别泵入相应的柱反应器, 流速为1 mL ·min-1.前30 d向柱反应器泵入添加了MnCl2(100 μmol ·L-1)的地下水, 使土著微生物在滤料上挂膜.为了加速柱体内部滤料中生物膜的形成, 向进水中添加稀释20倍的灭菌PYG培养基.柱反应器滤料挂膜后(约14 d), 开始向4个反应器中分别添加2个菌株, 其中2个反应器中接种S. QXT-31, 另外2个反应器中接种S. chilensis.菌株添加方式如下:离心收集培养24 h的菌体, 并用缓冲液(MgSO4 ·7H2 O, 0.5 g ·L-1; CaCl2 ·2H2 O, 60 mg ·L-1; HEPES, 10 mmol ·L-1, pH 7.2)洗3次, 然后悬浮在上述缓冲液中, 并用蠕动泵慢速(3 mL ·min-1)将菌体悬浮液分别泵入相应的柱反应器中.用电感耦合等离子体光谱仪测定进水和出水中溶解态Mn(Ⅱ)的浓度.
|
|
表 2 柱反应器进水水质参数 Table 2 Influent water quality in the column experiment |
2 结果与讨论 2.1 触发A. QXT-31产生锰氧化活性的菌株
之前的研究发现, 虽然A. QXT-31在单独培养时不表现出锰氧化活性, 但是与S. QXT-31混合培养时, 节杆菌原本沉默的锰氧化基因开始转录并伴随有胞外超氧自由基的产生, 产生了锰氧化活性[11].据此, 笔者购买了3个Sphingopyxis菌株, 分别与A. QXT-31混合培养, 考察其是否能够触发A. QXT-31产生锰氧化活性.研究将Sphingopyxis菌株与A. QXT-31等体积混合培养72 h, 检测溶解态Mn(Ⅱ)的残留浓度.结果显示, S. chilensis/S. QXT-31与A. QXT-31的混合培养体系中检测到锰氧化活性, 而菌株S. ginsengisoli和S. witflariensis均不能触发A. QXT-31产生锰氧化活性(图 1).接着, 本研究改变接种时S. chilensis与A. QXT-31的菌液体积比例, 考察其对锰氧化活性影响.结果显示, 较高比例的S. chilensis会使混合培养体系中提前产生锰氧化活性, 并且提高锰氧化活性的强度[图 2(a)], 且对混合培养体系中菌株生长没有明显抑制[图 2(b)].这个结果与菌株S. QXT-31相似[19, 30], 说明这两个菌株刺激A. QXT-31产生锰氧化活性的机制可能也是相同的.
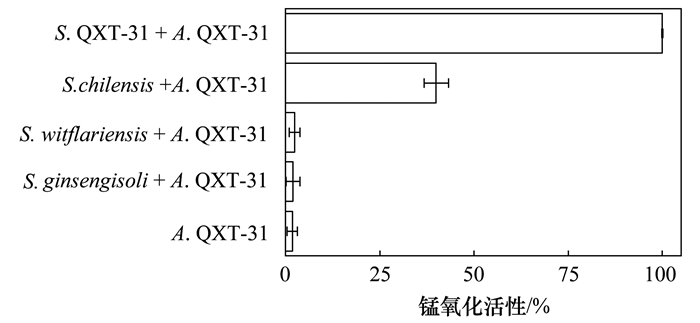
|
图 1 Sphingopyxis菌株与A. QXT-31混合培养体系中的锰氧化 Fig. 1 Mn(Ⅱ) oxidation in coculture of Sphingopyxis strains and A. QXT-31 |
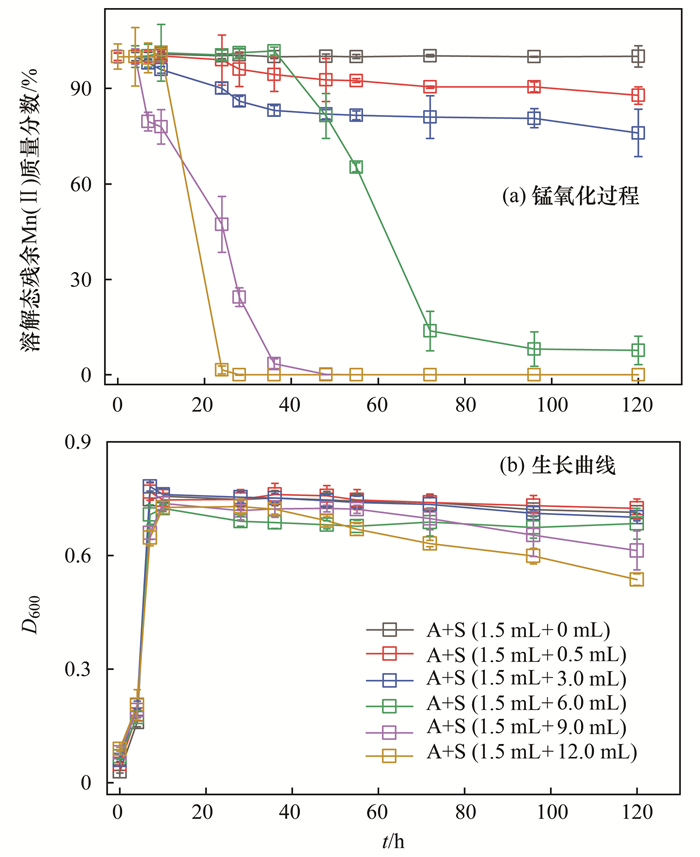
|
图 2 菌株S. chilensis与A. QXT-31混合培养体系中的锰氧化过程和生长曲线 Fig. 2 Mn(Ⅱ) oxidation and bacterial growth curve in a coculture of S. chilensis and A. QXT-31 |
从表 1可以看出组装得到的3个Sphingopyxis菌株的基因组完整度均大于99%.本研究通过基于16S rRNA基因序列和基因组序列分析, 考察了能够促使A. QXT-31产生锰氧化活性的菌株是否具备更近的系统发育关系和基因组相似性.基于16S rRNA基因的系统发育分析显示阳性组(S. chilensis和S. QXT-31)和阴性组(S. ginsengisoli和S. witflariensis)的菌株与组内菌株的系统发育关系更近[图 3(a)].但是, 每组内两菌株的基因组序列均非最相似[图 3(b)].上述结果表明, 能够触发A. QXT-31产生锰氧化活性的Sphingopyxis菌株可能有更近的基于16S rRNA基因序列的系统发育关系.
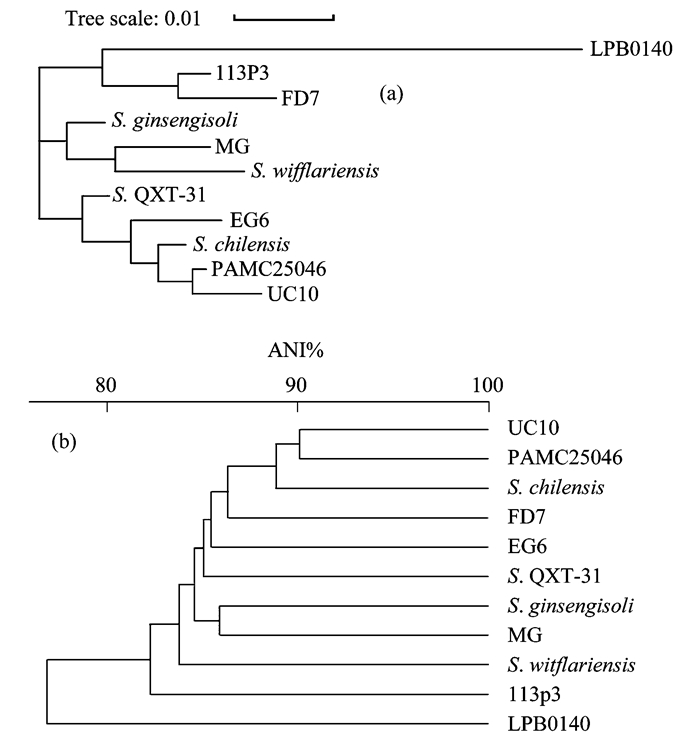
|
(a)基于16S rRNA基因序列的系统发育分析; (b)基于基因组平均核酸序列一致性的基因组相似性分析 图 3 基于16S rRNA基因序列和基因组序列的分析 Fig. 3 Analysis based on 16S rRNA gene and genomic sequence |
本研究比较了4个Sphingopyxis菌株的代谢通路, 考察导致A. QXT-31产生锰氧化活性的可能的代谢通路.在4个基因组中均发现394个KEGG MODULE, 而只有35个KEGG MODULE的完整度不完全相同, 其中组内(阳性组和阴性组)完整度相同而组间不同的MODULE只有2个, 编号分别是M00008(Entner-Doudoroff pathway, glucose-6P glyceraldehyde-3P+pyruvate)和M00017(Methionine biosynthesis, apartate homoserine methionine).之前的研究发现, 鞘氨醇盒菌是通过菌体的直接接触才能触发A. QXT-31产生锰氧化活性, 但是上述两个MODULE似乎与菌体间接触并不相关, 因此不是触发A. QXT-31产生锰氧化活性的直接因素.
2.4 同源基因比较及转录组分析本研究进一步通过基因组同源基因分析, 识别可能参与种间相互作用的基因.最终发现阳性组内2菌株共有但阴性组内都缺少的基因有193个.基于这193个基因在KEGG中的K编码(K number; 一共64个), 通过KEGG的映射工具(KEGG Mapper)注释这些基因参与的代谢通路类型.表 3中是匹配数目最高的5个代谢通路信息, 从中可以看出抗生素合成通路(Biosynthesis of antibiotics)的匹配数目仅次于代谢类型通路(Metabolic pathways), 推测抗生素合成通路可能参与了2菌株的种间相互作用.为了验证上述推测, 通过RNA-Seq数据计算并筛选了在2菌株混合培养1.5 h后转录丰度上升50%的基因列表(共20个), 考察了其编码蛋白质参与的代谢通路.结果显示这20个基因所编码蛋白质在23个代谢通路上的总匹配数(Hit)为220, 其中抗生素合成通路匹配数为23(表 4排第二), 仍占较大比例; 共映射到4个与抗生素合成相关的同源蛋白(K number), 其注释和代谢反应信息如下:acetyl-CoA C-acetyltransferase(K number:K00626;代谢通路及反应:pyrrolnitrin biosynthesis, tryptophan pyrrolnitrin; rebeccamycin biosynthesis, tryptophan rebeccamycin)、tryptophan 7-halogenase(K number:K14266;代谢通路及反应:rebeccamycin biosynthesis, tryptophan rebeccamycin; pyrrolnitrin biosynthesis, tryptophan pyrrolnitrin)、glutamine-fructose-6-phosphate transaminase(K number:K00820;代谢通路及反应:biosynthesis of antibiotics, KEGG无代谢反应的信息)、N-acetylglucosamine-6-phosphate deacetylase(K number:K01443;代谢通路及反应:biosynthesis of antibiotics, KEGG无代谢反应的信息).上述结果说明在S. QXT-31与A. QXT-31混合后1.5 h, S. QXT-31与抗生素合成基因转录水平普遍上升.因为抗生素能够调节细菌基因的转录[31], 上述分析结果表明鞘氨醇盒菌合成的抗生素可能参与两菌株相互作用, 并触发A. QXT-31转录锰氧化基因.
|
|
表 3 差异基因所属的代谢通路 Table 3 KEGG pathways of differential genes |
|
|
表 4 差异基因中转录水平上调基因所属的代谢通路 Table 4 KEGG pathways of upregulated differential genes |
2.5 反应器实验
通过向柱反应器中分别添加S. QXT-31和S. chilensis菌株, 考察能否利用微生物种间相互作用提高水处理过程中Mn(Ⅱ)的氧化沉淀去除.结果如图 4, 在前17 d内, 所有反应器中Mn(Ⅱ)的氧化去除率都有所上升(约20%的去除率), 接种菌株的反应器中Mn(Ⅱ)的氧化去除率并没有显著高于对照体系(未接菌).而从第18 d起, 添加菌株的2个处理中Mn(Ⅱ)的氧化去除率快速升高, 明显高于未接菌的对照组.上述结果表明, 在反应器中添加上述菌株能够加快Mn(Ⅱ)的氧化去除.
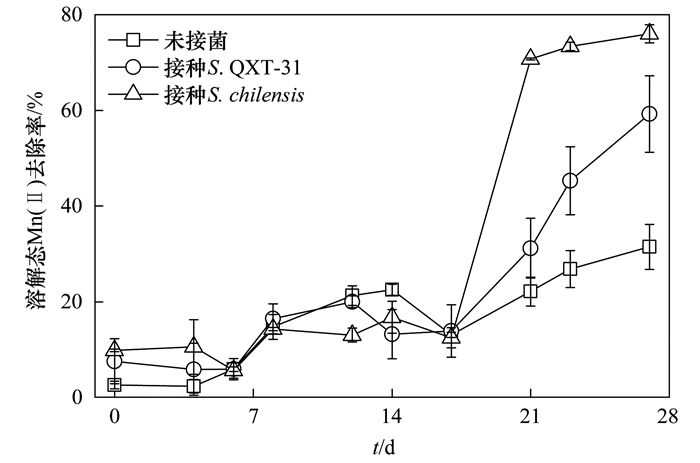
|
图 4 接种和不接种Sphingopyxis菌株的反应器出水中的Mn(Ⅱ)氧化去除 Fig. 4 Mn(Ⅱ) removal in reactors with or without inoculation of Sphingopyxis strains |
(1) S. chilensis也能够触发A. QXT-31产生锰氧化活性, 说明Sphingopyxis菌株触发A. QXT-31产生锰氧化活性这一现象具备一定的普适性.能够触发A. QXT-31产生锰氧化活性的2个Sphingopyxis菌株(S. chilensis和S. QXT-31)有更近的基于16S rRNA基因序列的系统发育关系, 但是并不具备更高的基因组相似性(与不能触发A. QXT-31产生锰氧化活性的两个菌株相比).
(2) 能够触发A. QXT-31产生锰氧化活性的2个Sphingopyxis菌株(S. QXT-31和S. chilensis)的基因组序列中, 含有特有(与不能触发A. QXT-31产生锰氧化活性的两个菌株相比)的抗生素合成的基因, 并且这些抗生素抗性基因的转录水平在与A. QXT-31混合培养后上调, 说明S. QXT-31的相关抗生素合成的代谢通路可能参与刺激A. QXT-31产生锰氧化活性.
(3) 向除锰的反应器中添加菌株S. QXT-31或S. chilensis均能够加速滤池中Mn(Ⅱ)的氧化除去, 说明利用微生物种间相互作用强化Mn(Ⅱ)的氧化去除具有潜在的工程可行性.由于生成的锰氧化物能氧化去除水中部分有机及无机污染物, 因此具有深度净化水体的作用.
| [1] | Gude J C J, Rietveld L C, van Halem D. As(Ⅲ) oxidation by MnO2 during groundwater treatment[J]. Water Research, 2017, 111: 41-51. DOI:10.1016/j.watres.2016.12.041 |
| [2] | Forrez I, Carballa M, Verbeken K, et al. Diclofenac oxidation by biogenic manganese oxides[J]. Environmental Science & Technology, 2010, 44(9): 3449-3454. |
| [3] | Cerrato J M, Falkinham Ⅲ J O, Dietrich A M, et al. Manganese-oxidizing and-reducing microorganisms isolated from biofilms in chlorinated drinking water systems[J]. Water Research, 2010, 44(13): 3935-3945. DOI:10.1016/j.watres.2010.04.037 |
| [4] | Morgan J J. Kinetics of reaction between O2 and Mn(Ⅱ) species in aqueous solutions[J]. Geochimica et Cosmochimica Acta, 2005, 69(1): 35-48. DOI:10.1016/j.gca.2004.06.013 |
| [5] | Zhao X, Liu B F, Wang X H, et al. Single molecule sequencing reveals response of manganese-oxidizing microbiome to different biofilter media in drinking water systems[J]. Water Research, 2020, 171: 115424. DOI:10.1016/j.watres.2019.115424 |
| [6] | Dangeti S, McBeth J M, Roshani B, et al. Microbial communities and biogenic Mn-oxides in an on-site biofiltration system for cold Fe-(Ⅱ)-and Mn (Ⅱ)-rich groundwater treatment[J]. Science of the Total Environment, 2020. DOI:10.1016/j.scitotenv.2019.136386 |
| [7] | Piazza A, Casalini L C, Pacini V A, et al. Environmental bacteria involved in Manganese (Ⅱ) oxidation and removal from groundwater[J]. Frontiers in Microbiology, 2019, 10: 119. DOI:10.3389/fmicb.2019.00119 |
| [8] | Corstjens P L A M, De Vrind J P M, Goosen T, et al. Identification and molecular analysis of the Leptothrix discophora SS-1mofA gene, a gene putatively encoding a manganese-oxidizing protein with copper domains[J]. Geomicrobiology Journal, 1997, 14(2): 91-108. DOI:10.1080/01490459709378037 |
| [9] | Brouwers G J, de Vrind J P M, Corstjens P L A M, et al. cumA, a gene encoding a multicopper oxidase, is involved in Mn2+ oxidation in Pseudomonas putida GB-1[J]. Applied and Environmental Microbiology, 1999, 65(4): 1762-1768. DOI:10.1128/AEM.65.4.1762-1768.1999 |
| [10] | Dick G J, Torpey J W, Beveridge T J, et al. Direct identification of a bacterial Manganese(Ⅱ) oxidase, the multicopper oxidase MnxG, from spores of several different marine Bacillus species[J]. Applied and Environmental Microbiology, 2008, 74(5): 1527-1534. DOI:10.1128/AEM.01240-07 |
| [11] | Liang J S, Bai Y H, Men Y J, et al. Microbe-microbe interactions trigger Mn(Ⅱ)-oxidizing gene expression[J]. The ISME Journal, 2017, 11(1): 67-77. DOI:10.1038/ismej.2016.106 |
| [12] | Andeer P F, Learman D R, McIlvin M, et al. Extracellular haem peroxidases mediate Mn(Ⅱ) oxidation in a marine Roseobacter bacterium via superoxide production[J]. Environmental Microbiology, 2015, 17(10): 3925-3936. DOI:10.1111/1462-2920.12893 |
| [13] | Learman D R, Voelker B M, Vazquez-Rodriguez A I, et al. Formation of manganese oxides by bacterially generated superoxide[J]. Nature Geoscience, 2011, 4(2): 95-98. |
| [14] | Chaput D L, Fowler A J, Seo O, et al. Mn oxide formation by phototrophs:spatial and temporal patterns, with evidence of an enzymatic superoxide-mediated pathway[J]. Scientific Reports, 2019, 9(1): 18244. DOI:10.1038/s41598-019-54403-8 |
| [15] | Clement B G. Biological Mn(Ⅱ) oxidation in freshwater and marine systems: new perspectives on reactants, mechanisms and microbial catalysts of Mn cycling in the environment[D]. San Diego: University of California, 2006. 59-87. |
| [16] | Bromfield S M, Skerman V B D. Biological oxidation of manganese in soils[J]. Soil Science, 1950, 69(5): 337-418. DOI:10.1097/00010694-195005000-00019 |
| [17] | Bromfield S M. Oxidation of manganese by soil microorganisms[J]. Australian Journal of Biological Sciences, 1956, 9(2): 238-252. DOI:10.1071/BI9560238 |
| [18] | Jones D, Keddie R M. The Genus Arthrobacter[A]. In: Dworkin M, Falkow S, Rosenberg E, et al (Eds.). The Prokaryotes: Volume 3: Archaea. Bacteria: Firmicutes, Actinomycetes[M]. New York: Springer, 2006. 945-960. |
| [19] | Liang J S, Bai Y H, Hu C Z, et al. Cooperative Mn(Ⅱ) oxidation between two bacterial strains in an aquatic environment[J]. Water Research, 2016, 89: 252-260. DOI:10.1016/j.watres.2015.11.062 |
| [20] | Nurk S, Bankevich A, Antipov D, et al. Assembling genomes and mini-metagenomes from highly chimeric reads[A]. In: Proceedings of the 17th Annual International Conference on Research in Computational Molecular Biology[C]. Beijing, China: Springer, 2013. 158-170. |
| [21] | Parks D H, Imelfort M, Skennerton C T, et al. CheckM:assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes[J]. Genome Research, 2015, 25(7): 1043-1055. DOI:10.1101/gr.186072.114 |
| [22] | Larkin M A, Blackshields G, Brown N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 |
| [23] | Price M N, Dehal P S, Arkin A P. FastTree 2-approximately maximum-likelihood trees for large alignments[J]. PLoS One, 2010, 5(3): e9490. DOI:10.1371/journal.pone.0009490 |
| [24] | Letunic I, Bork P. Interactive tree of life (iTOL) v4:recent updates and new developments[J]. Nucleic Acids Research, 2019, 47(W1): W256-W259. DOI:10.1093/nar/gkz239 |
| [25] | Jain C, Rodriguez-R L M, Phillippy A M, et al. High throughput ANI analysis of 90K prokaryotic genomes reveals clear species boundaries[J]. Nature Communications, 2018, 9(1): 5114. DOI:10.1038/s41467-018-07641-9 |
| [26] | Haas B J, Papanicolaou A, Yassour M, et al. De novo transcript sequence reconstruction from RNA-seq using the Trinity platform for reference generation and analysis[J]. Nature Protocols, 2013, 8(8): 1494-1512. DOI:10.1038/nprot.2013.084 |
| [27] | Hyatt D, Chen G L, LoCascio P F, et al. Prodigal:prokaryotic gene recognition and translation initiation site identification[J]. BMC Bioinformatics, 2010, 11(1): 119. DOI:10.1186/1471-2105-11-119 |
| [28] | Kanehisa M, Sato Y, Morishima K. BlastKOALA and GhostKOALA:KEGG tools for functional characterization of genome and metagenome sequences[J]. Journal of Molecular Biology, 2016, 428(4): 726-731. DOI:10.1016/j.jmb.2015.11.006 |
| [29] | Altschul S F, Gish W, Miller W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2 |
| [30] | Liang J S, Bai Y H, Qu J H. Microbial interspecies interactions affect arsenic fate in the presence of MnⅡ[J]. Microbial Ecology, 2017, 74(4): 788-794. DOI:10.1007/s00248-017-1008-9 |
| [31] | Goh E B, Yim G, Tsui W, et al. Transcriptional modulation of bacterial gene expression by subinhibitory concentrations of antibiotics[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(26): 17025-17030. DOI:10.1073/pnas.252607699 |
 2020, Vol. 41
2020, Vol. 41


