2. 中国科学院大学, 北京 100049;
3. 重庆大学三峡库区生态环境教育部重点实验室, 重庆 400045;
4. 重庆工商大学国家智能制造服务国际科技合作基地, 重庆 400067
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Key Laboratory of the Three Gorges Reservoir Region's Eco-Environments(Ministry of Education), Chongqing University, Chongqing 400045, China;
4. National Base of International Science and Technology Cooperation for Intelligent Manufacturing Service, Chongqing Technology and Business University, Chongqing 400067, China
据估计, 每年海洋中固定氮损失的50%左右可归因于厌氧氨氧化(anaerobic ammonium oxidation, ANAMMOX)的作用[1].厌氧氨氧化作用是由两种不同氮化合物, 亚硝酸盐(NO2-)和氨(NH4+)结合生成氮气(N2)的过程[2].在厌氧条件下, 厌氧氨氧化细菌以亚硝酸盐为电子受体, 直接将铵态氮转化为氮气, 如下式所示[3].
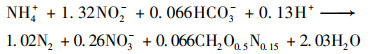
|
目前有一种公认的氨氧化细菌的代谢途径:首先, 由亚硝酸氧化还原酶(NIR)催化亚硝酸还原为一氧化氮(NO).然后一氧化氮与氨结合生成肼, 这一步反应由肼合成酶(hydrazine synthetase, HZS)催化.而通常认为HZS酶催化此反应由两步组成:首先, 由HZS酶的γ亚基将一氧化氮还原为羟胺(NH2OH), 然后由HZS酶的α亚基进一步将羟胺和氨合成肼.最后由肼氧化酶(hydrazine oxidase, HZO)将肼氧化为氮气[4].
根据目前的推测, 羟胺是厌氧氨氧化细菌的基本代谢底物, 但同时羟胺也是一种抑制剂, 厌氧氨氧化细菌对包括羟胺在内的多种抑制化合物高度敏感[5, 6].厌氧氨氧化细菌对羟胺毒性的敏感性及其生长速度的缓慢可能导致厌氧氨氧化过程在羟胺休克后失效.而目前关于厌氧氨氧化细菌颗粒如何应对羟胺的压力还没有很好的解释.通过测定特定底物(如氨和亚硝酸盐)的去除率, 可以研究羟胺对厌氧氨氧化活性的影响.有研究通过实验和数学建模研究了羟胺对厌氧氨氧化细菌肼代谢的影响[7].结果表明, 在无亚硝酸根的情况下, 羟胺发生了生物歧化反应, 生成了二硝基气体和铵根.并且发现随着羟胺含量的增加, 肼的浓度会达到峰值, 然后下降, 同时会产生少量的氨.结果表明, 羟胺能抑制肼转变为氮气, 从而抑制厌氧氨氧化的活性[6].然而, 其他研究表明, 添加微量羟胺促进了SBR体系中NO2-抑制的恢复[8, 9], 并且NH2OH能够刺激重要中间物质N2 H4的产生[10], 说明羟胺也可以作为厌氧氨氧化反应器运行的促进剂.因此, 为了准确、灵敏和特异地评价羟胺对厌氧氨氧化活性的影响, 迫切需要进一步地研究.
目前羟胺的添加被用于一些厌氧氨氧化细菌富集实验研究[11~15], 由于在厌氧氨氧化细菌中添加羟胺产生肼是独特的, 因此, 羟胺添加后肼的测量可以作为厌氧氨氧化细菌活性的“基准”[16].但由于肼是一种有毒易爆物质, 对于实验操作会提出很高的要求, 增加许多不必要的实验麻烦.同时外界扰动会导致细菌新陈代谢的不平衡, 从而导致中间产物浓度的可测量变化, 因此需要研究更为有效的方法检测羟胺加入后对厌氧氨氧化细菌的影响.由于mRNA水平对环境条件变化的快速响应, 基于mRNA的技术已被用于监测生物量活动[17].大多数研究表明, 生物量活动与相关的mRNA水平呈正相关[18, 19].虽然厌氧氨氧化应对羟胺休克反应的活性在一些研究中得到了评估, 但很少有研究全面评估关键功能基因在转录水平对羟胺休克反应的影响, 特别是HZO酶mRNA转录产物的丰度与厌氧氨氧化活性之间的关系尚未得到很好地研究.同时由于厌氧氨氧化细菌颗粒在实验室条件下难以进行纯化培养, 纯厌氧氨氧化细菌培养物的缺乏使得对其代谢相关的研究特别具有挑战性.目前多数研究主要用了多种互补的方法来阐明羟胺参与的相关的新陈代谢[6, 8, 20~24], 而羟胺对于厌氧氨氧化微生物群落结构的影响目前研究得比较少.
因此本研究采用羟胺添加批次实验, 利用逆转录酶定量聚合酶链反应(RT-qPCR)的方法, 定量测定了厌氧氨氧化细菌主要代谢途径中HZO酶的基因转录水平变化, 从而确定NH2OH冲击对厌氧氨氧化性能的影响.另外, 基于16S rRNA高通量测序的方法, 探究了在短期羟胺暴露实验中, 厌氧氨氧化细菌群落应对羟胺压力的调控机制.通过本研究, 可以更详细和深入地了解羟胺在厌氧氨氧化反应中的作用.
1 材料与方法 1.1 羟胺添加批次实验本研究设置了一系列不同浓度NH2OH, 来观察羟胺对厌氧氨氧化活性的影响.采用含有成熟厌氧氨氧化细菌的颗粒污泥进行3次分批实验, 颗粒污泥挥发性悬浮固体(volatile suspended solid, VSS)浓度为3.12 g ·L-1.实验前, 用去离子水冲洗生物载体4~5次.采用文献报道的人工合成废水来培养厌氧氨氧化细菌[25], 其中氨氮与亚硝酸盐比例为1.58 :1.在反应开始前, 用氮气或Ar冲洗间歇反应器的液相约15 min, 以消除液相和气相中的氧气, 也有利于厌氧氨氧化细菌的驯化.然后用丁基橡胶塞子封住间歇反应器, 在95r ·min-1左右的摇浴中搅拌, 摇浴温度保持在(37±0.5)℃.利用HCO3-缓冲系统, 使反应器中溶液pH值始终保持在8.0~8.5.并按照以前报道的方法[9], 每1 h监测一次氨氮和亚硝氮的浓度.
1.2 RNA提取和实时荧光定量聚合酶链反应(RT-qPCR)技术采用TRIzol试剂(天根, 中国)从厌氧氨氧化细菌中分离得到总RNA.为了进行RT-qPCR分析, 根据试剂盒的说明, 使用逆转录酶(Thermo ScientificTMEP0733)从经过DNaseI处理的总RNA中合成第一链cDNAs.RT-qPCR扩增在96孔板上用Biorad IQ5实时PCR系统进行.每个反应包含10 μL SYBR Green Master Mix试剂(Takara, Japan)、1.0 μL cDNA样品和200 nmol ·L-1基因特异性引物(见表 1), 最终体积为20 μL.以厌氧氨氧化细菌16S rRNA基因为内参基因.相对表达量的测定按照文献[26]报道的方法.根据批次实验安排, 提取总RNA的样品共分为6个样品, 分别为反应器1(R1)、反应器2(R2)、反应器3(R3)、反应器4(R4)、反应器5(R5)和反应器6(R6), 在不同羟胺浓度(0、40、50、60、70和80 mg ·L-1, 以N计)刺激下, 厌氧氨氧化细菌的总RNA, 其中反应器R1为对照组, 其他为实验组, 每组进行3次重复.并分别检测其纯度和浓度(图 1和表 2), 均符合实验要求.
|
|
表 1 RT-qPCR所用的特异性引物及核苷酸序列 Table 1 Specific primers and nucleotide sequences for RT-qPCR |
|
M为DNA Marker, 其大小由下到上依次为100、200、300、400、500、600、700、800、900、1 000、1 200、1 500、2 000、2 500、3 000、3 500、4 000、5 000、6 000、8 000、10 000 bp, 其只为检测基因组污染, 并不代表RNA条带大小 图 1 细菌总RNA提取电泳结果 Fig. 1 Bacteria total RNA electrophoresis results |
|
|
表 2 细菌总RNA浓度和吸光度值 Table 2 Bacteria total RNA concentrations and optical density (OD) values |
1.3 16S rRNA高通量测序方法
反应器运行结束后, 将实验用的厌氧氨氧化颗粒污泥进行研磨处理, 添加PBS缓冲液, 然后于-80℃冰箱低温保存.采用生工DNA分离试剂盒进行DNA提取, 使用生工DNA试剂盒对原始DNA进行定量, 然后扩增DNA样本, 引物序列使用靶向16S rRNA的V3~V4高变区, 后续通过干冰运输送至上海生工生物公司进行高通量测序分析, 将测序后采集的数据采用RDP classifier贝叶斯算法对相似水平97%的OTU代表序列进行分类学分析, 主要包括α多样性分析、物种多样性分析、菌群差异分析、物种分类树分析、β多样性分析和功能预测分析.
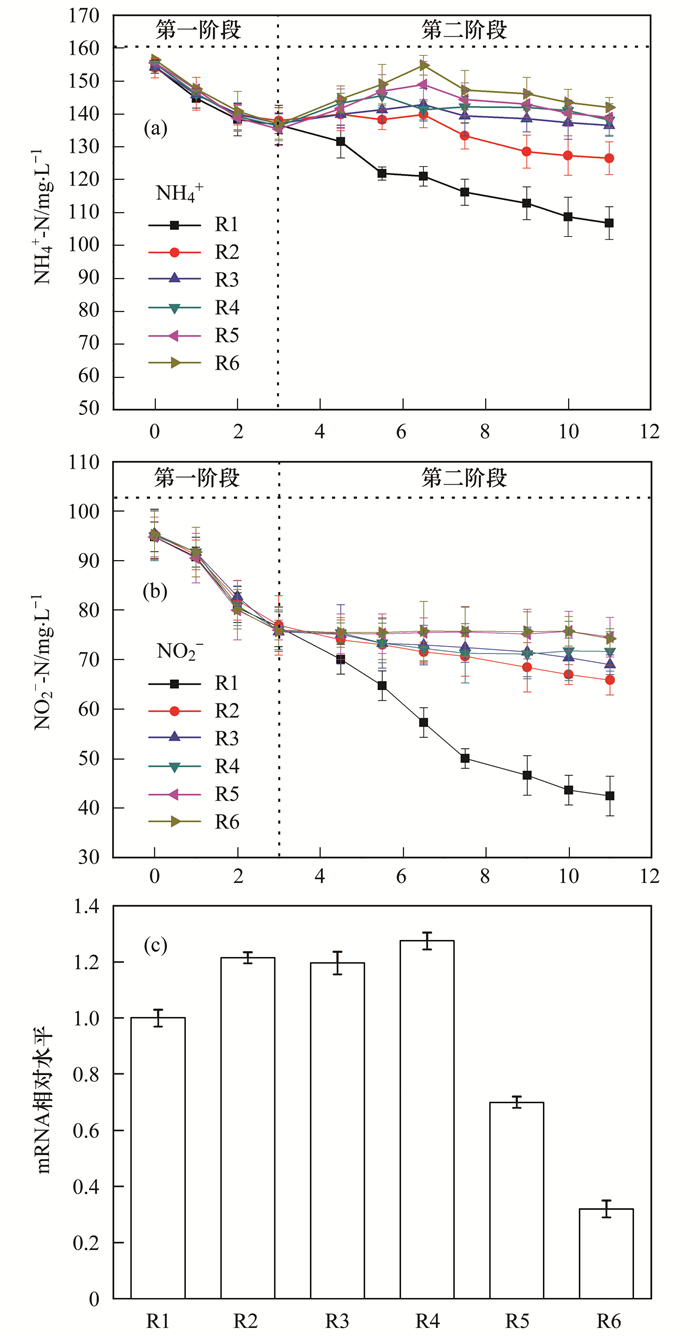
2 结果与讨论 2.1 羟胺的投加对厌氧氨氧化反应的影响为了探究NH2OH对厌氧氨氧化活性的影响, 将厌氧氨氧化颗粒污泥与NH2OH、亚硝酸盐(NO2-)和铵(NH4+)进行孵育.所有实验反应器的进水NO2-和NH4+的初始浓度都一样, 总共设置了6组实验, 分别为反应器1(R1)、反应器2(R2)、反应器3(R3)、反应器4(R4)、反应器5(R5)和反应器6(R6), 其中反应器R1为对照组, 其他为实验组, 每组进行3次重复.如图 2(a)和2(b)所示, 在开始的第一阶段, 所有的实验反应器中都不添加羟胺, 结果发现实验组的厌氧氨氧化活性均与对照组接近(R1).在第二阶段, 对照组R1不添加羟胺, 但分别在R2、R3、R4、R5和R6中添加40、50、60、70和80 mg ·L-1 NH2OH, 结果发现相比对照组R1, 其他实验组NO2--N和NH4+-N降解速率都变慢.对照组R1在羟胺添加后的亚硝酸盐的脱氮效率(以N/VSS计)为0.032 8 g ·d-1, 而R2、R3、R4、R5和R6的亚硝酸盐脱氮率分别为对照组的31.5%、22.5%、14.6%、6.8%和6.1%, 说明羟胺的投加对厌氧氨氧化反应产生了抑制作用.
|
图 2 羟胺对厌氧氨氧化活性的影响 Fig. 2 Effect of hydroxylamine on ANAMMOX activity |
经过前面的实验, 可以看出羟胺会对厌氧氨氧化活性产生抑制作用, 但无法判断多大量的羟胺会对厌氧氨氧化活性产生不可逆的抑制.为了探究厌氧氨氧化细菌对羟胺的耐受程度, 本研究通过RT-qPCR实验测定了不同反应器中的HZO酶mRNA的相对表达量, 以R1作为对照.如图 2(c)所示, R2、R3和R4反应器中HZO酶mRNA的相对表达量要远高于R1对照组, 说明当羟胺的添加量介于40~60 mg ·L-1时, 羟胺能够对厌氧氨氧化细菌产生抑制作用, 但不足以使厌氧氨氧化细菌失去活性.而厌氧氨氧化细菌会通过提高HZO酶的表达量, 来抵抗羟胺压力.但是R5和R6反应器中HZO酶mRNA的相对表达量要远低于R1对照组, 说明当羟胺的添加量高于70 mg ·L-1时, 羟胺浓度超出了厌氧氨氧化细菌的承受范围, 导致厌氧氨氧化细菌失去活性.这也印证文献[7]中高浓度羟胺可能会对厌氧氨氧化过程产生抑制作用.由此笔者推测3.12 g ·L-1的厌氧氨氧化细菌颗粒对羟胺的承受压力介于60~70 mg ·L-1之间.Jetten等[16]的研究报道可以根据羟胺添加后肼的生成量作为厌氧氨氧化活性的“基准”, 但还不够精确, 无法判断厌氧氨氧化细菌对羟胺的耐受极限, 本文的实验证实HZO的转录水平变化可以更准确地反映厌氧氨氧化活性.
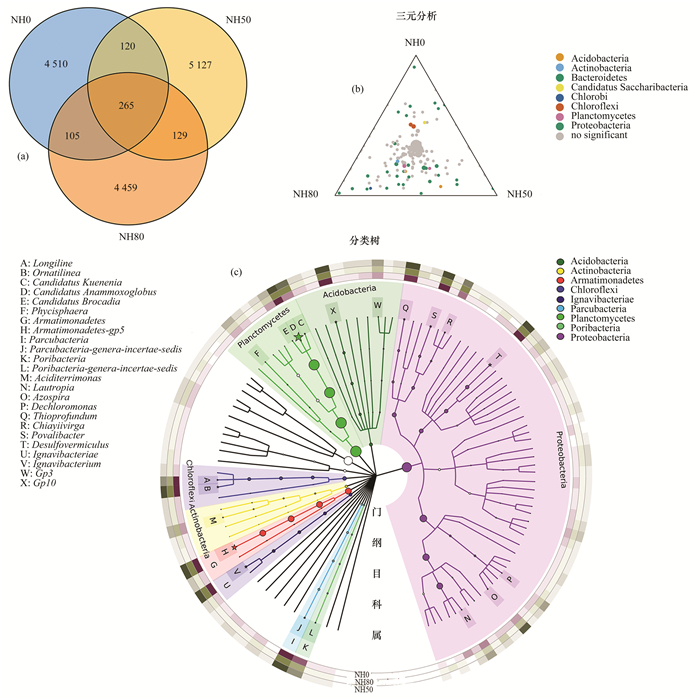
2.2 羟胺对厌氧氨氧化颗粒内部菌群的影响根据前面的实验结果, 本研究选择3种不同浓度羟胺进行批次实验, 分为对照组(NH0, 不添加羟胺)、低羟胺浓度组(NH50, 添加50 mg ·L-1羟胺)和高羟胺浓度组(NH80, 添加80 mg ·L-1羟胺), 然后对厌氧氨氧化细菌颗粒内部菌群作结构分析. 16S rRNA测序显示, 在3组厌氧氨氧化细菌颗粒DNA样本中, NH0、NH50和NH80的微生物种类结构差异较大. 3组样品中共有的菌种有265个, 而NH0独有的菌种有4 510个, NH50独有的菌种有5 127个, NH80独有的菌种有4 459个[图 3(a)], 说明羟胺对厌氧氨氧化细菌颗粒内部菌群结构产生了较大的影响.通过三元分析[图 3(b)], 可以看出在3个样品中主要富集的物种包括:酸杆菌门(Acidobacteria)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、未命名(Candidatus Saccharibacteria)、绿菌门(Chlorobi)、绿弯菌门(Chloroflexi)和浮霉菌门(Planctomycetes).结合系统发育进化树分析[图 3(c)], 在属水平上, 变形菌门的细菌种类最多, 共有7种, 分别为Thioprofundum、Chiayiivirga、Povalibacter、Desulfovermiculus、Dechloromonas、Azospira和Lautropia.虽然变形菌门在反应器中的细菌种类最多, 但在功能上并没有优势, 这可能与厌氧氨氧化颗粒独特厌氧环境有关.此外, 有文献报道, 变形菌门的主要作用在于维持厌氧氨氧化反应器内菌群的生态平衡[27].而在功能上最有优势的是浮霉菌门, 浮霉菌门主要包含4类细菌, 除了Phycisphaera其余3类都是厌氧氨氧化细菌, 分别为Candidatus Kuenenia、Candidatus Anammoxoglobus和Candidatus Brocadia, 这3类厌氧氨氧化细菌承担了主要的代谢功能.此外, 绿菌门含有两类细菌, 分别为Longiline和Ornatilinea.绿菌门的细菌总是与厌氧氨氧化的细菌一起存在, 二者在功能上相辅相成, 这与之前的报道相符[27], 其研究报道厌氧氨氧化细菌能够固定CO2, 合成氨基酸, 形成胞外聚合物, 而绿菌门的细菌能够分解胞外聚合物, 运输氨基酸进入胞内.此外, 厌氧氨氧化细菌和绿菌门的细菌能够分别氧化和还原亚硝酸盐, 形成一个闭合的亚硝酸盐环路.酸杆菌门主要包含Gp3和Gp10, 由于酸性细菌很难在实验室中生长, 因此关于它们的代谢作用知之甚少, 后续可以进行更多地研究.
|
图 3 厌氧氨氧化颗粒群落的结构变化 Fig. 3 Structural changes of ANAMMOX granules in microbial communities |
然后本研究进一步对3组反应器所含的细菌在属水平上的种类组成进行详细分析[图 4(a)], 并进行了不同组别之间对比分析.通过对比NH0组、NH50组和NH80组所含的菌群[图 4(b)和4(c)], 可以看出, 相比较NH0组和NH80组, NH50组都显著增加了Candidatus Anammoxoglobus, 说明添加适量羟胺(50 mg ·L-1, 以N计)可以促进厌氧氨氧化细菌的富集.同时相比较NH0组, NH50组都显著减少了unclassified和Ornatilinea, 这个结果也进一步证明了羟胺可以通过抑制其他细菌的生长来促进厌氧氨氧化细菌的活性[28].笔者发现相比较NH0组和NH80组, NH50组也显著增加了Armatimonadetes_gp5.Zhao等[29]的研究发现, 装甲菌门(Armatimonadetes)可为厌氧氨氧化细菌提供生长因子叶酸和钼辅因子, 这也侧面说明适量羟胺添加也有助于与厌氧氨氧化细菌共生菌的增加, 维持较好的生态平衡.但是相比较NH0组, 厌氧氨氧化细菌并没有在NH80组中富集[图 4(d)], 说明高剂量的羟胺(80 mg ·L-1)浓度抑制了所有细菌的活性, 这与批次实验的结果也是一致的.根据文献[30], 厌氧氨氧化细菌由于对环境极其敏感, 因此目前还无法在实验室获得纯培养.根据本研究的实验结果, 通过在反应器中添加适量(50 mg ·L-1)的羟胺, 利用羟胺的毒性, 可以抑制其他细菌的生长, 但并不影响厌氧氨氧化细菌的生长, 从而让反应器内厌氧氨氧化细菌处于更优的状态.
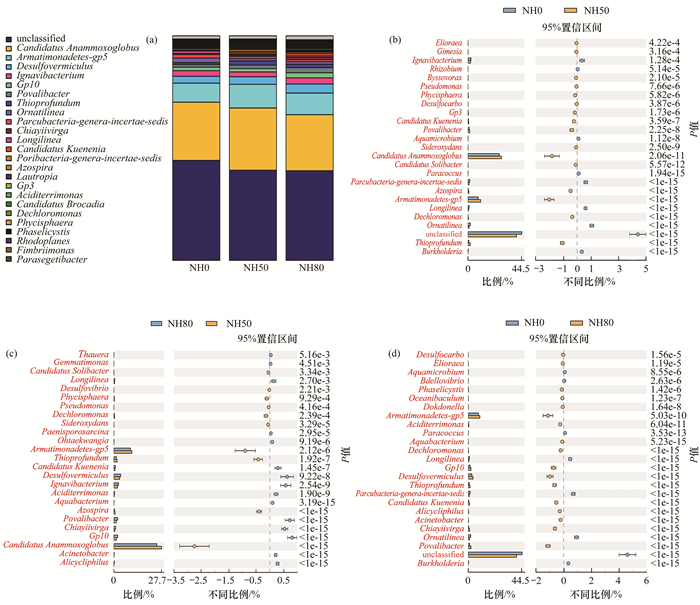
|
图 4 属水平微生物群落种类组成分析 Fig. 4 Analysis of the species composition of the microbial community (genus level) |
经过前期实验, 可以看出添加低浓度羟胺会对厌氧氨氧化活性产生抑制作用, 但不会使厌氧氨氧化细菌失去活性, 说明厌氧氨氧化细菌可能通过某些方式在抵抗羟胺压力.有研究结果表明[7], 厌氧氨氧化细菌会通过相关代谢酶将羟胺歧化生成N2和NH4+, 并进一步生成肼.而对于厌氧氨氧化颗粒细菌群落如何抵抗压力的机制还知之甚少, 因此本研究又进行了KEGG代谢功能分析.KEGG代谢功能主要包括代谢、膜转运、信号传递、细胞周期等相关细胞生化过程分析.图 5(a)显示在3组样品中所含的主要代谢功能分布, 而后进行不同组别之间的比对分析.通过对比NH0和NH50组可以发现[图 5(b)], 在细胞运动性、新陈代谢、细胞运动过程和信号等方面, NH50组要明显优于NH0组, 说明虽然低剂量的羟胺可能会对厌氧氨氧化代谢产生一定抑制作用, 但同时也在促进细胞产生更强的运动性, 这可能也是细胞抵抗羟胺压力的一种方式.而膜运输、碳水化合物代谢等方面NH50组要明显低于NH0组, 这也从深层面水平说明细菌在面对羟胺压力时, 羟胺可能通过抑制膜相关蛋白运输和能量代谢的方式来抑制细菌的活性.通过对比NH50和NH80组可以看出[图 5(c)], 在能量代谢方面NH80组要低于NH50组, 这与羟胺批次实验结果一致, 羟胺浓度越高对细菌的抑制作用越强.结合图 5(d), 可以发现在异种生物降解和代谢方面NH80组要同时优于NH50和NH0组, 说明在高浓度羟胺的压力下, 细菌可能会通过分解其他物质来应对羟胺压力.
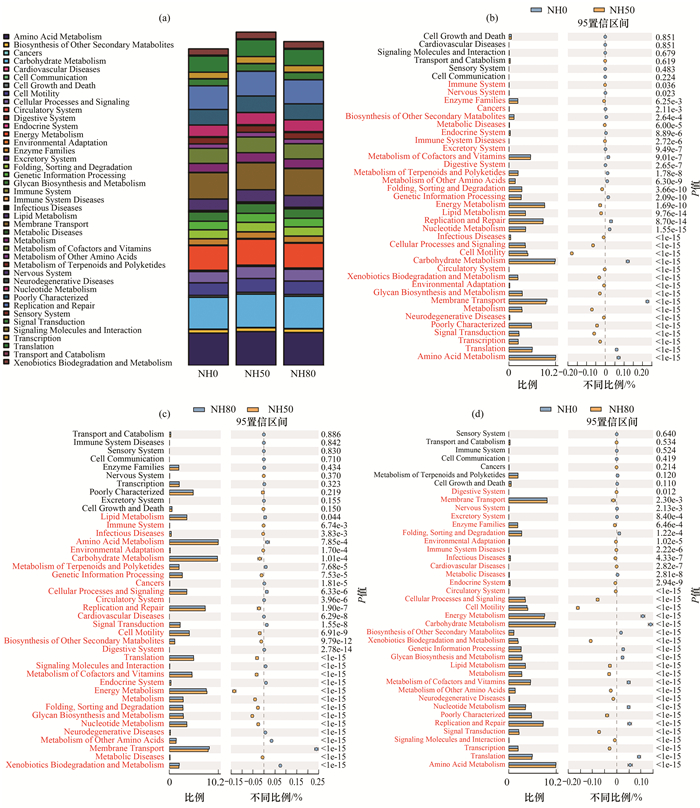
|
图 5 KEGG代谢功能分析 Fig. 5 Metabolic function analysis of KEGG |
(1) HZO酶的转录水平变化可以更精确地测定厌氧氨氧化颗粒对羟胺的耐受范围.相对于3.12 g ·L-1的厌氧氨氧化颗粒污泥, 其承受的羟胺浓度的阈值介于60~70 mg ·L-1.
(2) 羟胺的投加可以有效地抑制颗粒污泥内细菌的活性, 适量的羟胺(50 mg ·L-1, 以N计)能够增强厌氧氨氧化细菌的组成, 提供一个更佳的生态平衡.
(3) 厌氧氨氧化颗粒污泥可能通过提高细胞运动性、新陈代谢、细胞运动过程和信号以及异种生物降解和代谢等方面代谢活性来抵抗羟胺的压力.
| [1] | Schmid M C, Maas B, Dapena A, et al. Biomarkers for in situ detection of anaerobic ammonium-oxidizing (anammox) bacteria[J]. Applied and Environmental Microbiology, 2005, 71(4): 1677-1684. DOI:10.1128/AEM.71.4.1677-1684.2005 |
| [2] | Peng M W, Yu X L, Guan Y, et al. Underlying promotion mechanism of high concentration of silver nanoparticles on anammox process[J]. ACS Nano, 2019, 13(12): 14500-14510. DOI:10.1021/acsnano.9b08263 |
| [3] | Strous M, Heijnen J J, Kuenen J G, et al. The sequencing batch reactor as a powerful tool for the study of slowly growing anaerobic ammonium-oxidizing microorganisms[J]. Applied Microbiology and Biotechnology, 1998, 50(5): 589-596. DOI:10.1007/s002530051340 |
| [4] | Dietl A, Ferousi C, Maalcke W J, et al. The inner workings of the hydrazine synthase multiprotein complex[J]. Nature, 2015, 527(7578): 394-397. DOI:10.1038/nature15517 |
| [5] | Maalcke W J, Reimann J, de Vries S, et al. Characterization of anammox hydrazine dehydrogenase, a key N2-producing enzyme in the global nitrogen cycle[J]. Journal of Biological Chemistry, 2016, 291(33): 17077-17092. DOI:10.1074/jbc.M116.735530 |
| [6] | Shimamura M, Nishiyama T, Shigetomo H, et al. Isolation of a multiheme protein with features of a hydrazine-oxidizing enzyme from an anaerobic ammonium-oxidizing enrichment culture[J]. Applied and Environmental Microbiology, 2007, 73(4): 1065-1072. DOI:10.1128/AEM.01978-06 |
| [7] | van der Star W R L, van de Graaf M J, Kartal B, et al. Response of anaerobic ammonium-oxidizing bacteria to hydroxylamine[J]. Applied and Environmental Microbiology, 2008, 74(14): 4417-4426. DOI:10.1128/AEM.00042-08 |
| [8] | Strous M, Kuenen J G, Jetten M S M. Key physiology of anaerobic ammonium oxidation[J]. Applied and Environmental Microbiology, 1999, 65(7): 3248-3250. DOI:10.1128/AEM.65.7.3248-3250.1999 |
| [9] | Zekker I, Kroon K, Rikmann E, et al. Accelerating effect of hydroxylamine and hydrazine on nitrogen removal rate in moving bed biofilm reactor[J]. Biodegradation, 2012, 23(5): 739-749. DOI:10.1007/s10532-012-9549-6 |
| [10] | Hu A H, Zheng P, Mahmood Q, et al. Characteristics of nitrogenous substrate conversion by anammox enrichment[J]. Bioresource Technology, 2011, 102(2): 536-542. DOI:10.1016/j.biortech.2010.07.015 |
| [11] | Egli K, Fanger U, Alvarez P J J, et al. Enrichment and characterization of an anammox bacterium from a rotating biological contactor treating ammonium-rich leachate[J]. Archives of Microbiology, 2001, 175(3): 198-207. DOI:10.1007/s002030100255 |
| [12] | Kartal B, Rattray J, van Niftrik L A, et al. Candidatus "Anammoxoglobus propionicus" a new propionate oxidizing species of anaerobic ammonium oxidizing bacteria[J]. Systematic and Applied Microbiology, 2007, 30(1): 39-49. DOI:10.1016/j.syapm.2006.03.004 |
| [13] | Kartal B, van Niftrik L, Rattray J, et al. Candidatus 'Brocadia fulgida':an autofluorescent anaerobic ammonium oxidizing bacterium[J]. FEMS Microbiology Ecology, 2008, 63(1): 46-55. DOI:10.1111/j.1574-6941.2007.00408.x |
| [14] | Schmid M, Walsh K, Webb R, et al. Candidatus "Scalindua brodae", sp. nov., Candidatus "Scalindua wagneri", sp. nov., two new species of anaerobic ammonium oxidizing bacteria[J]. Systematic and Applied Microbiology, 2003, 26(4): 529-538. DOI:10.1078/072320203770865837 |
| [15] | van Dongen U, Jetten M S M, van Loosdrecht M C M. The SHARON®-Anammox® process for treatment of ammonium rich wastewater[J]. Water Science & Technology, 2001, 44(1): 153-160. |
| [16] | Jetten M, Schmid M, van de Pas-Schoonen K, et al. Anammox organisms:enrichment, cultivation, and environmental analysis[J]. Methods in Enzymology, 2005, 397: 34-57. DOI:10.1016/S0076-6879(05)97003-1 |
| [17] | Park H, Rosenthal A, Ramalingam K, et al. Linking community profiles, gene expression and N-removal in anammox bioreactors treating municipal anaerobic digestion reject water[J]. Environmental Science & Technology, 2010, 44(16): 6110-6116. |
| [18] | Saleh-Lakha S, Shannon K E, Henderson S L, et al. Effect of nitrate and acetylene on nirS, cnorB, and nosZ expression and denitrification activity in Pseudomonas mandelii[J]. Applied and Environmental Microbiology, 2009, 75(15): 5082-5087. DOI:10.1128/AEM.00777-09 |
| [19] | Bollmann A, Schmidt I, Saunders A M, et al. Influence of starvation on potential ammonia-oxidizing activity and amoA mRNA levels of Nitrosospira briensis[J]. Applied and Environmental Microbiology, 2005, 71(3): 1276-1282. DOI:10.1128/AEM.71.3.1276-1282.2005 |
| [20] | Cirpus I E Y, de Been M, den Camp H J M O, et al. A new soluble 10 kDa monoheme cytochrome c-552 from the anammox bacterium Candidatus "Kuenenia stuttgartiensis"[J]. FEMS Microbiology Letters, 2005, 252(2): 273-278. DOI:10.1016/j.femsle.2005.09.007 |
| [21] | Schalk J, de Vries S, Kuenen J G, et al. Involvement of a novel hydroxylamine oxidoreductase in anaerobic ammonium oxidation[J]. Biochemistry, 2000, 39(18): 5405-5412. DOI:10.1021/bi992721k |
| [22] | Huston W M, Harhangi H R, Leech A P, et al. Expression and characterisation of a major c-type cytochrome encoded by gene kustc0563 from Kuenenia stuttgartiensis as a recombinant protein in Escherichia coli[J]. Protein Expression and Purification, 2007, 51(1): 28-33. |
| [23] | Strous M, Pelletier E, Mangenot S, et al. Deciphering the evolution and metabolism of an anammox bacterium from a community genome[J]. Nature, 2006, 440(7085): 790-794. DOI:10.1038/nature04647 |
| [24] | Kartal B, Kuypers M M M, Lavik G, et al. Anammox bacteria disguised as denitrifiers:nitrate reduction to dinitrogen gas via nitrite and ammonium[J]. Environmental Microbiology, 2007, 9(3): 635-642. DOI:10.1111/j.1462-2920.2006.01183.x |
| [25] | Strous M, Fuerst J A, Kramer E H M, et al. Missing lithotroph identified as new planctomycete[J]. Nature, 1999, 400(6743): 446-449. DOI:10.1038/22749 |
| [26] | Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2 method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [27] | Lawson C E, Wu S, Bhattacharjee A S, et al. Metabolic network analysis reveals microbial community interactions in anammox granules[J]. Nature Communications, 2017, 8(1): 15416. DOI:10.1038/ncomms15416 |
| [28] | Ganesan S, Vadivelu V M. Effect of external hydrazine addition on anammox reactor start-up time[J]. Chemosphere, 2019, 223: 668-674. DOI:10.1016/j.chemosphere.2019.02.104 |
| [29] | Zhao Y P, Liu S F, Jiang B, et al. Genome-centered metagenomics analysis reveals the symbiotic organisms possessing ability to cross-feed with anammox bacteria in anammox consortia[J]. Environmental Science & Technology, 2018, 52(19): 11285-11296. |
| [30] | Lackner S, Terada A, Smets B F. Heterotrophic activity compromises autotrophic nitrogen removal in membrane-aerated biofilms:results of a modeling study[J]. Water Research, 2008, 42(4-5): 1102-1112. DOI:10.1016/j.watres.2007.08.025 |
 2020, Vol. 41
2020, Vol. 41


