2. 江苏省厌氧生物技术重点实验室, 无锡 214122;
3. 江苏省水处理技术与材料协同创新中心, 苏州 215011
2. Jiangsu Key Laboratory of Anaerobic Biotechnology, Wuxi 214122, China;
3. Jiangsu Collaborative Innovation Center of Water Treatment Technology and Material, Suzhou 215011, China
环境中抗生素抗性基因(antibiotic resistance genes, ARGs)快速发展, 抗生素耐药性问题日趋严重.污水处理厂是ARGs重要的“汇”与“源”[1]:一方面, 它汇集了污水中大量的ARGs;另一方面, 一旦其对污水中ARGs去除效率不足, 其排水将是受纳环境ARGs的重要来源.此外, 污水生化处理将大量ARGs汇集到剩余污泥, 使其ARGs丰度(以干污泥计)高达109 copies·g-1[2], 加强剩余污泥处理处置过程中ARGs的控制至关重要.
污泥厌氧消化具有污泥无害化、减量化和资源化的作用, 是国际上广泛应用的一项污泥处理技术[3].研究表明厌氧消化能一定程度上削减污泥中的ARGs[4, 5], 但也存在部分ARGs丰度上升的现象[6].学者们通过微生物群落分析, 并将之与ARGs变化进行相关性研究[7], 以此来解析厌氧消化过程中ARGs变化的内在规律.目前, 研究主要采用提取污泥总DNA, 随后进行定量PCR(qPCR)和高通量测序的方法展开[8, 9].但厌氧消化过程伴随着污泥微生物细胞裂解和细胞物质溶出等过程, 厌氧消化污泥中通常存在大量死亡菌体及其DNA, 将死菌DNA纳入后续分析可能会对ARGs和微生物群落分析产生影响.一方面, 死菌DNA所携带的ARGs与活菌携带ARGs的传播途径不同[7, 10~12], 将其纳入总DNA分析可能造成ARGs传播风险评估的偏差;另一方面, 死菌DNA的存在可能会对ARGs与微生物群落相关性分析产生影响, 不利于污泥厌氧消化过程中ARGs变化规律的解析.但死菌DNA对厌氧消化污泥中ARGs和微生物群落分析的具体影响尚未得到研究.
叠氮溴化丙锭(PMA)是一种叠氮类染料, 能够穿过死菌受损的细胞膜, 在强光诱导下与DNA形成共价结合, 抑制其在PCR反应中的扩增.采用PMA处理可以消除样品中死菌DNA对PCR和后续检测分析的影响[13~15].因此, 本研究对3种不同类型厌氧消化污泥进行PMA处理并开展后续ARGs和微生物群落分析, 将之与未处理样品进行对照, 以解析PMA处理对厌氧消化污泥ARGs和微生物群落的影响, 评估死菌DNA对厌氧消化污泥ARGs和微生物群落分析的潜在干扰.
1 材料与方法 1.1 样品来源选取3个不同类型厌氧消化系统采集污泥样品, 系统1为剩余污泥自厌氧消化体系, 由3.9 g污水处理厂脱水污泥和6.1 mL去离子水组成, 于厌氧管中自消化72 d后采集样品(S1);系统2为污泥水解液厌氧消化体系, 体系由400 mL经离心去除固体物质的剩余污泥水解液和100 mL厌氧颗粒污泥种泥组成, 厌氧消化15 d后采集样品(S2);系统3为处理规模1 t·d-1高含固污泥厌氧消化示范工程, 污泥停留时间35 d, 污泥含固率15.7%, 采集厌氧消化罐出泥口污泥(S3).
1.2 PMA处理方法及对死菌DNA屏蔽效率验证所用PMA购自Biotium公司.参考文献[16, 17], 本研究取适量样品置于离心管, 10 000 r·min-1下离心10 min收集固形物, 使用0.9% NaCl溶液振荡离心清洗3遍后, 重悬至D600处于0.700~0.900之间.取两等份, 一份进行PMA处理, 一份作为不进行PMA处理的对照样品. 3个污泥经PMA处理的样品分别编号为:S1P、S2P和S3P, 对照样品分别编号为S1N、S2N和S3N.PMA反应于1.5 mL离心管中进行, 管内置入995 μL重悬后的污泥样品以及5 μL 20 mmol·L-1的PMA溶液, 于黑暗处反应30 min后, 在冰浴中用500 W卤素灯光照反应10 min, 随后再于黑暗处静置15 min.对照样品不添加PMA而用5 μL 0.9% NaCl溶液补足体系完成相同操作.
PMA对死菌DNA PCR扩增抑制效率的验证实验设计如下:选用原污泥中未检出的Nitrospira amoA基因作为标记物, 通过克隆构建携带Nitrospira amoA的大肠杆菌JM 109克隆子;随后将克隆子扩培至对数期, 均匀取两等份, 一份置于95℃水浴10 min灭活;分别将10 μL活菌和死菌投加到985 μL稀释后的污泥样品, 加入5 μL PMA进行处理, 并设置不进行PMA处理对照组.相应的实验组设置如表 1所示, 每个组别设置两个平行反应体系.
|
|
表 1 PMA处理效率验证实验分组 Table 1 Groups of study of verification of PMA treatment efficiency |
1.3 DNA提取及定量PCR方法
样品于10 000 r·min-1下离心10 min, 收集底部污泥使用PowerSoil® DNA Isolation Kit提取DNA, 操作步骤按照试剂盒说明书进行.
本研究选择了tetA、tetO、sulI、sulII和ermB共5种ARGs进行定量检测.ARGs及Nitrospira amoA基因qPCR检测所使用的引物和温度程序见表 2.qPCR反应在QuantStudio® 3(美国, Thermo Fisher)仪器上完成, 反应体系为20 μL, 包含SYBR Green Ⅰ(SYBR® Green qPCR Mastermixes, QIAGEN)10 μL, 前后引物各1 μL, DNA模板1 μL, ddH2O 7 μL.将已知拷贝数标准质粒以10倍梯度稀释进行qPCR反应构建标准曲线, 标曲R2均大于0.994, 扩增效率为97%~104%. qPCR反应均采用3个平行测试.
|
|
表 2 引物序列和PCR温度程序 Table 2 Primers and PCR thermal procedures |
1.4 Illumina高通量测序
测序采用细菌古菌通用引物515FmodF(5′-GTGYCAGCMGCCGCGGTAA-3′)和806RmodR(5′- GGACTACNVGGGTWTCTAAT-3′)完成[23, 24].PCR反应条件如下:95℃预变性3 min, 95℃变性30 s, 55℃退火30 s, 72℃延伸45 s, 共30个循环;72℃最终延伸10 min.PCR产物胶纯化回收建库, 建库成功后使用Illumina MiSeq平台对扩增子文库进行测序.对测序数据进行Paired-end raw reads数据过滤、拼接, Raw Tags序列质量过滤.数据处理后进行OTU划分, 并使用Ⅰ-Sanger云平台进行微生物群落α和β多样性等分析.测序工作委托上海美吉生物医药科技有限公司完成.
1.5 显著性分析采用SPSS 22.0软件进行卡方检验完成显著性分析.
2 结果与讨论 2.1 PMA对死菌DNA PCR扩增的抑制效果PMA对死菌DNA PCR扩增的抑制效率验证实验结果如图 1所示.“PMA处理+活菌”和“无PMA处理+活菌”实验组的amoA基因拷贝值处于同一数量级且较为接近, PMA处理在厌氧消化污泥样品中活菌的检测影响不大.“无PMA处理+死菌”实验组中amoA基因拷贝值与“无PMA处理+活菌”相比仍在同一数量级, 丰度上略有下降, 这可能是由于高温灭活过程中菌体死亡后有少量DNA发生降解造成的.“PMA处理+死菌”实验组的amoA拷贝值与“无PMA处理+死菌”相比下降了两个数量级.由此可知PMA处理能够有效抑制厌氧消化污泥中死菌DNA的PCR扩增, 抑制效率为98.1%.以往PMA-qPCR技术大多运用在纯菌[25]或者污水厂出水[18]等悬浮物浓度较低、干扰物质较少的样品, 死菌DNA PCR扩增的抑制效率在97%~99%[17, 25, 26], 本研究得到的PMA抑制效率与上述研究相仿, 证明了PMA处理对抑制厌氧消化污泥样品中死菌DNA扩增的有效性, 满足本研究后续实验的要求.本研究的目的在于探究死菌DNA是否会对厌氧消化污泥中ARGs及微生物群落的分析产生干扰, 在死菌DNA抑制效率未达到100%的情况下, 若发现死菌DNA对ARGs和微生物群落分析的影响, 更加能够说明死菌DNA会对厌氧消化污泥中ARGs及微生物群落的分析产生干扰.
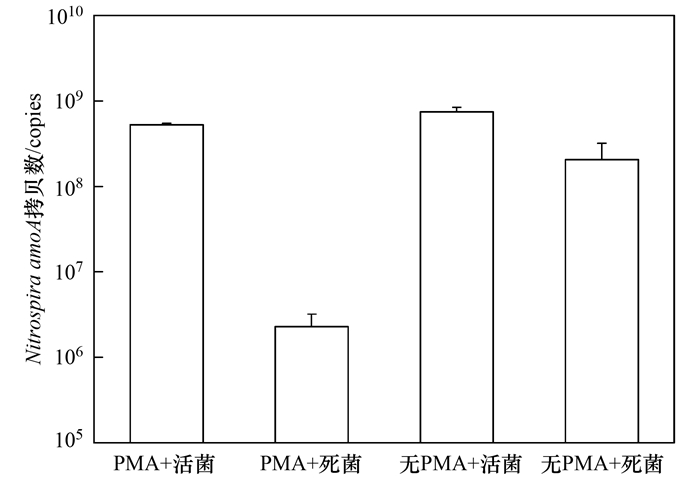
|
图 1 Nitrospira amoA基因拷贝值变化 Fig. 1 Variation of the gene abundance of Nitrospira amoA |
经PMA处理和未经PMA处理厌氧消化污泥中ARGs丰度的测定结果如图 2所示.可以看出, 绝大多数PMA处理组的ARGs丰度都比未经PMA染料处理组有所降低.其中, S1样品经PMA处理后各个ARGs丰度较未经PMA处理组下降了41%~86%;S3样品经PMA处理后各个ARGs丰度下降幅度更大, 达到74%~98%.而S2样品经PMA处理后ARGs丰度变化相对较小.
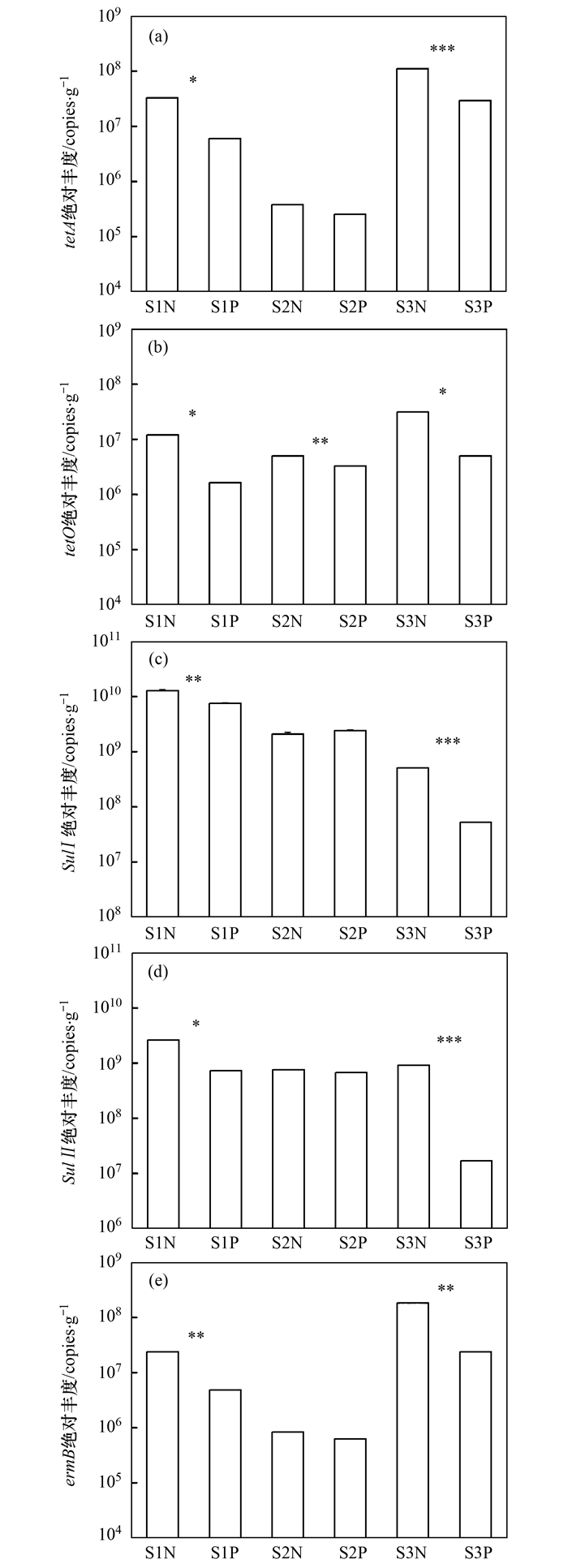
|
经PMA处理与未经PMA处理样品间的差异显著性用*表示: *表示0.01 < P≤0.05, **表示0.001 < P≤0.01, ***表示P≤0.001 图 2 ARGs绝对丰度 Fig. 2 Absolute abundance of the ARGs |
这一结果说明, S3中死菌ARGs在污泥ARGs总量中所占的比例最大, S1中死菌ARGs的比例也较高, 而S2中死菌ARGs的量较小.分析污泥来源可知, S2取自污泥水解液厌氧消化体系, 体系由液态的污泥水解液和厌氧颗粒污泥组成, 所采的S2样品实际为高活性的厌氧颗粒污泥, 因此S2中所携带的死菌较少.S1取自剩余污泥自厌氧消化体系, 剩余污泥在进入厌氧环境后, 部分不适应厌氧条件的微生物大量死亡, 所以S1样品中死菌的比例也较高, 这与PMA处理大幅降低了ARGs检测丰度的结果一致.S3是高含固污泥厌氧消化罐出泥口采集的污泥样品, 高含固污泥厌氧消化进泥浓度高、厌氧消化处理时间长, 经消化后污泥中微生物死亡裂解残余物多, 所以S3经PMA处理后ARGs检测丰度下降量也最大.
比较5种ARGs的丰度发现, 整体上厌氧消化污泥中sulI的丰度(以干污泥计, 下同)最高, 在未经PMA处理的污泥样品中达到5.08×108~1.27×1010 copies·g-1;sulII的丰度次之, 为7.59×108~2.59×109 copies·g-1;其余3个ARGs丰度相对较低.除S2P中的sulI基因丰度略有上升之外, tetA、tetO、sulI、sulII和ermB在污泥PMA处理后丰度各下降了33%~82%、34%~86%、41%~90%、11%~98%和26~87%, 说明这5种ARGs在死菌和活菌中均各有分布.此外, 经PMA处理后, S3中ARGs高低顺序由未经PMA处理时的sulII>sulI>ermB> tetA>tetO变为sulI> tetA> ermB>sulII>tetO.可见, 厌氧消化污泥中活菌携带的各ARGs丰度高低顺序可能与死菌携带ARGs的高低顺序不同, PMA处理可以更加准确分析厌氧消化污泥中活菌ARGs高低.
2.3 PMA处理对不同厌氧消化污泥中微生物群落分析的影响3类厌氧消化污泥经PMA处理与未经PMA处理的6个微生物群落文库信息如表 3所示.可以看出, 文库的覆盖度(coverage)达到0.995 3~0.999 3, 能够很好反映污泥中微生物群落的信息.比较发现, 无论是否经过PMA处理, S1样品中测定的OTUs数量均最大, S2样品中测定的OTUs数量最小.而Chao 1指数的高低顺序也为S1>S3>S2, 表明S1的微生物群落丰富度最高, 而S2最低.从Shannon指数看, S1污泥中微生物群落多样性最高, S2和S3比较接近, 均低于S1.比较PMA处理对群落多样性指数的影响, 发现S3样品经PMA处理后Shannon指数有较大幅度的下降;而S1和S2样品Shannon指数变化相对较小.
|
|
表 3 样品高通量测序文库信息 Table 3 Information of the high throughput sequencing libraries |
6个样品在门和纲水平上的微生物群落组成如图 3所示.变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和绿弯菌门(Chloroflexi)为主要的门:其中, Proteobacteria是S1和S2中丰度最高的门(45.2%~47.5%);Firmicutes在S3中占绝对优势(39.8%~68.3%);Bacteroidetes在各样品中的相对丰度较为接近(8.0%~24.3%);Chloroflexi在S1与S3中稍低(4.0%~6.6%), 而在S2中相对较高(16.1%~19.6%).从纲水平看, S1中主要为β-变形菌纲(β-Proteobacteria)和鞘脂杆菌纲(Sphingobacteriia)等;S2中主要为ε-变形菌纲(ε-Proteobacteria)、γ-变形菌纲(γ-Proteobacteria)和拟杆菌纲(Bacteroidia)等;而S3中梭菌纲(Clostridia)是最主要的优势纲.可见, 3个污泥微生物群落差异明显, 其中S3与其他两个污泥的差异最大.S1为剩余污泥自消化后的样品, 其β-Proteobacteria、Sphingobacteriia和γ-Proteobacteria等主要菌群与活性污泥中常见的菌群[27]较为相似.厌氧颗粒污泥中常含有Proteobacteria、放线菌门(Actinobacteria)、Bacteroidetes和Firmicutes等[28], 也在S2中能够找到.而S3来源于高含固污泥厌氧消化系统, 其占主要地位的Clostridia具有有机质高效降解、强化有机物发酵产酸等功能[29, 30], 能够促进高含固污泥厌氧消化过程的进行.
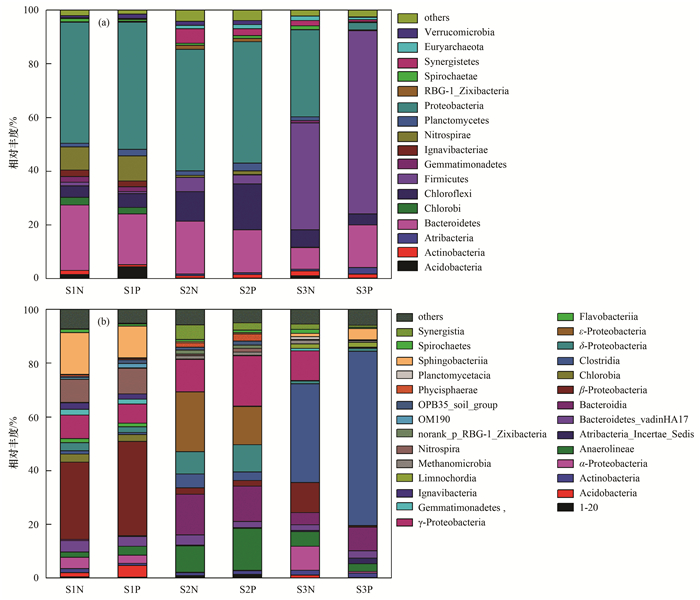
|
(a)门水平; (b)纲水平 图 3 样品中的微生物群落组成 Fig. 3 Microbial community composition in all samples |
比较PMA处理对样品微生物群落组成分析结果的影响, 发现S1P、S2P较S1N、S2N相比, Bacteroidetes相对丰度明显下降;Chloroflexi相对丰度在S1P、S2P样品中有不同程度的提高.在纲水平上, S1P和S2P群落组成较S1N和S2N差异更大, Sphingobacteriia相对丰度在S1P中明显降低, 而β-Proteobacteria和酸杆菌纲(Acidobacteria)相对丰度则大幅升高;S2P中ε-Proteobacteria、Bacteroidia、互营养菌纲(Synergistia)和Clostridia等丰度下降, 而γ-Proteobacteria、厌氧绳菌纲(Anaerolineae)和δ-变形菌纲(δ-Proteobacteria)等丰度上升.S3经PMA处理后微生物群落分析结果变化更为明显, 在门水平中Firmicutes相对丰度上升了28.5%, 而Proteobacteria相应地下降了29.7%.在纲水平中γ-Proteobacteria、β-Proteobacteria、α-变形菌纲(α-Proteobacteria)和Anaerolineae的相对丰度分别降低了10.7%、10.6%、8.2%和2.7%, 而Clostridia、Bacteroidia和Sphingobacteriia相对丰度分别上升28.3%、4.2%和3.1%.与门和纲水平结果类似, 经PMA处理后, S1和S2主要科相对丰度的变化相对较小, 而S3的变化则较大(图 4).S1和S2中的主要优势科在经PMA处理和未经处理PMA处理样品中检测到的相对丰度变化超过1%的为6个, 而在S3中主要优势科在经PMA处理后相对丰度变化超过1%达到18个.
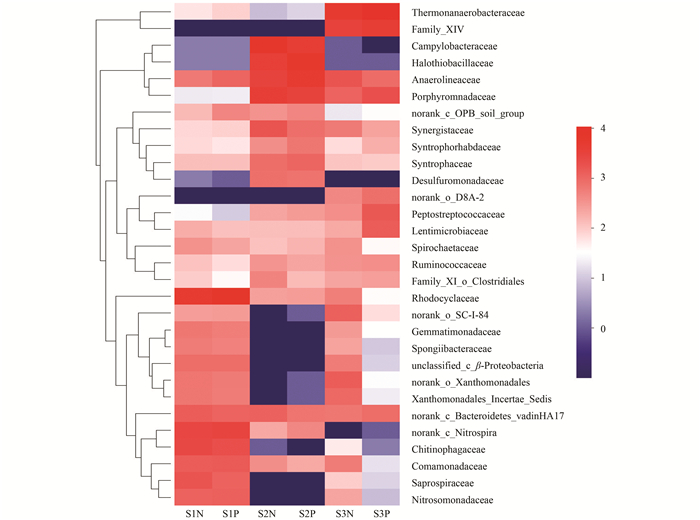
|
图 4 科水平下的30个主要物种相对丰度 Fig. 4 Relative abundance of the top 30 families in the samples |
对经PMA处理和未经PMA处理样品采用Fisher' exact test方法进行样本物种丰度差异显著性检验(图 5), 结果显示在S1N和S1P、S2N和S2P、S3N和S3P之间, 丰度最高的15个纲中分别有14、14和13个纲的相对丰度存在显著差异, 体现了PMA处理与否样品分析得到的微生物群落组成的显著差异.微生物作为ARGs的宿主, 微生物群落的差异必然会引起ARGs分析结果的差异, 但由于不同ARGs存在于不同的微生物菌株中, 这些ARGs宿主菌在死菌菌群中的存在情况又可能有所不同, 所以PMA处理与未PMA处理的污泥中不同ARGs丰度差异也各不相同(图 2).正是由于死菌DNA对微生物群落分析的干扰, 同时会对ARGs的分析产生影响, 所以排除死菌DNA的干扰, 能更加准确地反映出厌氧消化污泥中微生物群落及ARGs的特征.
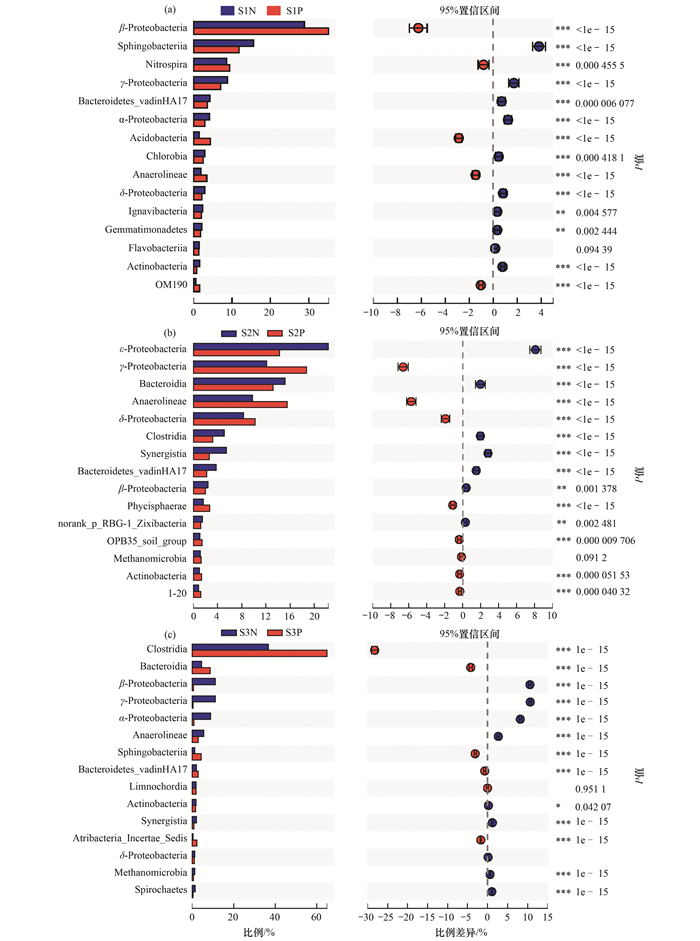
|
*表示0.01 < P≤0.05, **表示0.001 < P≤0.01, ***表示 P≤0.001 图 5 两样本差异显著性比较 Fig. 5 Significance comparison of the two samples |
进一步通过Bray-Curtis距离算法、共有OTUs分析和样本差异显著性比较等方法, 来评估各个样品经PMA处理和未经PMA处理微生物群落之间的差异性.基于Bray-Curtis距离算法可知(图 6), S1N与S1P、S2N与S2P以及S3N与S3P的距离系数分别为0.174 0、0.215 3和0.461 6, PMA处理与未经PMA处理样品之间均存在较大的组内距离, 其中S3N与S3P组内距离最大.
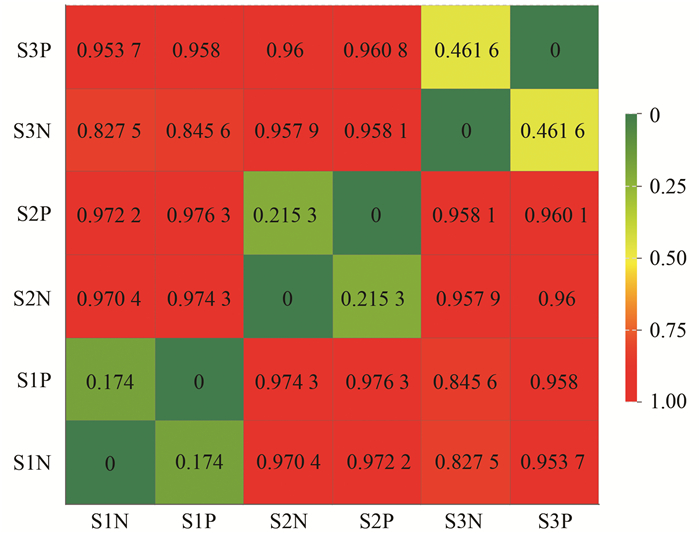
|
色块带为Bray-Curtis距离系数刻度, 系数越大表示微生物群落之间的差异性越大 图 6 样本间相对丰度距离 Fig. 6 Distance heatmap based on the relative abundance of OTUs in all samples |
共有OTUs分析显示(表 4), S1、S2样本中PMA处理独有和未经PMA处理独有的OTUs占比在6.5%~10.0%之间, 共有OTUs占比分别为81.6%和83.5%;而S3样本中共有OTUs占比只有58.7%, PMA处理独有OTUs为6.6%, 而未经PMA处理独有的OTUs占比达到34.7%.Bray-Curtis距离算法和共有OTUs分析均显示了PMA处理对厌氧消化污泥微生物群落分析的影响, 其中S3样品经PMA处理前后微生物群落分析结果差异最大.值得关注的是, S3中未经PMA处理独有的OTUs占比达到34.7%, 说明污泥中存在大量不同种类的死亡菌体.而这些OTUs仅在未经PMA处理的样品中被检出, 说明PMA能够有效抑制厌氧消化污泥中死菌DNA的PCR扩增, 更准确地分析污泥中存活的微生物群落组成.
|
|
表 4 PMA处理样品与未PMA处理样品共有OTUs分析 Table 4 Shared OTUs analysis of PMA treated samples and non-PMA treated samples |
2.4 PMA处理对ARGs与微生物群落组成相关性分析的影响
目前对与ARGs携带菌的分析, 主要通过将ARGs与微生物群落进行各种相关性分析来推测[7, 31, 32].本研究进一步比较了经PMA处理和未经PMA处理的3类厌氧消化污泥中ARGs丰度与微生物群落主要纲的相关性, 结果如图 7所示.在未经PMA处理情况下, sulI与硝化螺旋菌纲(Nitrospira)和unclassified_p_Bacteroidetes呈显著正相关, 与甲烷微菌纲(Methanomicrobia)呈显著负相关.sulII与纤维黏网菌纲(Cytophagia)、黄杆菌纲(Flavobacteriia)、Sphingobacteriia和绿菌纲(Chlorobia)呈显著正相关;而在经PMA处理情况下, sulI则与Actinobacteria呈显著负相关;sulII与OPB35_soil_group呈显著正相关, 与Clostridia和Limnochordia呈显著负相关, 此外tetA也与多个纲分别呈现相关性.可以看出, 经PMA处理情况下的ARGs与微生物群落相关性与未经PMA处理相比差异巨大, 原因可能是在厌氧消化体系中死菌DNA上的16S rRNA与ARGs的降解速率可能存在差异, 使得死菌中ARGs与微生物群落的相关性与活菌菌群中的分析结果不同, 死菌DNA对ARGs与微生物群落相关性分析存在干扰.
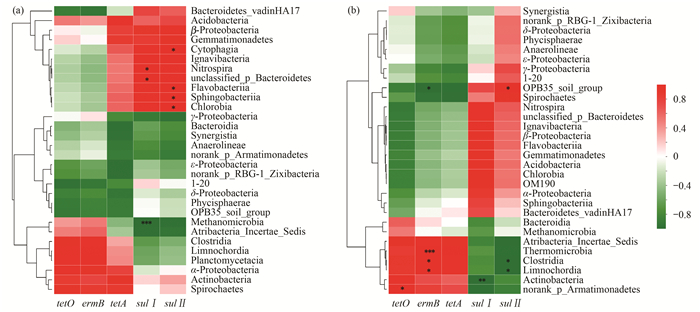
|
*表示0.01 < P≤0.05, **表示0.001 < P≤0.01, ***表示P≤0.001;(a)基于未经PMA处理的样品, (b)基于经PMA处理的样品 图 7 ARGs与微生物群落的Pearson相关性分析 Fig. 7 Pearson correlation analysis between ARGs and microbial communities |
综上所述, 经PMA处理后3个厌氧消化污泥样品中ARGs检测丰度有所下降, 微生物群落分析结果也发生了变化, ARGs与微生物群落组成相关性分析结果更是截然不同.可知, PMA处理会对厌氧消化污泥中ARGs和微生物群落分析结果产生影响, 而这正是由于PMA能够抑制厌氧消化污泥中死菌DNA的PCR扩增造成的.可以看出, 死菌DNA对厌氧消化污泥中ARGs与微生物群落分析存在较大干扰, PMA处理后进行后续分析能够更准确地反映厌氧消化污泥中的微生物群落及ARGs特征.
3 结论(1) PMA处理能够有效抑制厌氧消化污泥中死菌DNA在PCR中的扩增, 抑制效率达98.1%.
(2) 经PMA处理, 厌氧消化污泥的ARGs定量检测丰度相比于未经PMA处理对照组明显降低, 微生物群落分析结果与未经PMA处理样品呈现不同程度的差异.
(3) 在经PMA处理和未经PMA处理情况下, 厌氧消化污泥中ARGs与微生物群落组成的相关性分析呈现截然不同的结果.
(4) 死菌DNA对厌氧消化污泥ARGs和微生物群落分析存在干扰, 对于死菌含量较高的厌氧消化污泥, 采用PMA预处理能够更准确地反映厌氧消化污泥中的微生物群落及ARGs的特征.
| [1] | Czekalski N, Díez E G, Bürgmann H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake[J]. The ISME Journal, 2014, 8(7): 1381-1390. DOI:10.1038/ismej.2014.8 |
| [2] | Munir M, Wong K, Xagoraraki I. Release of antibiotic resistant bacteria and genes in the effluent and biosolids of five wastewater utilities in Michigan[J]. Water Research, 2011, 45(2): 681-693. DOI:10.1016/j.watres.2010.08.033 |
| [3] | Elalami D, Carrère H, Monlau F, et al. Pretreatment and co-digestion of wastewater sludge for biogas production:recent research advances and trends[J]. Renewable and Sustainable Energy Reviews, 2019, 114: 109287. DOI:10.1016/j.rser.2019.109287 |
| [4] | Tian Z, Chi Y Z, Yu B, et al. Thermophilic anaerobic digestion reduces ARGs in excess sludge even under high oxytetracycline concentrations[J]. Chemosphere, 2019, 222: 305-313. DOI:10.1016/j.chemosphere.2019.01.139 |
| [5] | Huang H N, Chen Y G, Zheng X, et al. Distribution of tetracycline resistance genes in anaerobic treatment of waste sludge:the role of pH in regulating tetracycline resistant bacteria and horizontal gene transfer[J]. Bioresource Technology, 2016, 218: 1284-1289. DOI:10.1016/j.biortech.2016.07.097 |
| [6] | Xu R, Yang Z H, Zheng Y, et al. Metagenomic analysis reveals the effects of long-term antibiotic pressure on sludge anaerobic digestion and antimicrobial resistance risk[J]. Bioresource Technology, 2019, 282: 179-188. DOI:10.1016/j.biortech.2019.02.120 |
| [7] | Zhang Y, Li A L, Dai T J, et al. Cell-free DNA:a neglected source for antibiotic resistance genes spreading from WWTPs[J]. Environmental Science & Technology, 2018, 52(1): 248-257. |
| [8] | Tong J, Fang P, Zhang J Y, et al. Microbial community evolution and fate of antibiotic resistance genes during sludge treatment in two full-scale anaerobic digestion plants with thermal hydrolysis pretreatment[J]. Bioresource Technology, 2019, 288: 121575. DOI:10.1016/j.biortech.2019.121575 |
| [9] | Zhang J Y, Lu T D, Shen P H, et al. The role of substrate types and substrate microbial community on the fate of antibiotic resistance genes during anaerobic digestion[J]. Chemosphere, 2019, 229: 461-470. DOI:10.1016/j.chemosphere.2019.05.036 |
| [10] | Karkman A, Do T T, Walsh F, et al. Antibiotic-resistance genes in waste water[J]. Trends in Microbiology, 2018, 26(3): 220-228. |
| [11] | Mao D Q, Luo Y, Mathieu J, et al. Persistence of extracellular DNA in river sediment facilitates antibiotic resistance gene propagation[J]. Environmental Science & Technology, 2014, 48(1): 71-78. |
| [12] | Miller J H, Novak J T, Knocke W R, et al. Survival of antibiotic resistant bacteria and horizontal gene transfer control antibiotic resistance gene content in anaerobic digesters[J]. Frontiers in Microbiology, 2016, 7: 263. |
| [13] | Kim M, Wuertz S. Survival and persistence of host-associated Bacteroidales cells and DNA in comparison with Escherichia coli and Enterococcus in freshwater sediments as quantified by PMA-qPCR and qPCR[J]. Water Research, 2015, 87: 182-192. DOI:10.1016/j.watres.2015.09.014 |
| [14] | Askar M, Sajid M, Nassif Y, et al. Propidium monoazide-polymerase chain reaction for detection of residual periprosthetic joint infection in two-stage revision[J]. Molecular Biology Reports, 2019, 46(6): 6463-6470. DOI:10.1007/s11033-019-05092-z |
| [15] | Dorn-In S, Gareis M, Schwaiger K. Differentiation of live and dead Mycobacterium tuberculosis complex in meat samples using PMA qPCR[J]. Food Microbiology, 2019, 84: 103275. DOI:10.1016/j.fm.2019.103275 |
| [16] |
仝铁铮. PMA-qPCR选择性检测水中活性菌方法的研究与应用[D].北京: 清华大学, 2010. Tong T Z. Selective detection of viable pathogens in water using propidium monoazide combined with qPCR and its application[D]. Beijing: Tsinghua University, 2010. http://cdmd.cnki.com.cn/Article/CDMD-10003-1011280856.htm |
| [17] | Guo F, Zhang T. Detecting the nonviable and heat-tolerant bacteria in activated sludge by minimizing DNA from dead cells[J]. Microbial Ecology, 2014, 67(4): 829-836. DOI:10.1007/s00248-014-0389-2 |
| [18] | Fowler S J, Palomo A, Dechesne A, et al. Comammox Nitrospira are abundant ammonia oxidizers in diverse groundwater-fed rapid sand filter communities[J]. Environmental Microbiology, 2018, 20(3): 1002-1015. DOI:10.1111/1462-2920.14033 |
| [19] | Chen B W, Liang X M, Huang X P, et al. Differentiating anthropogenic impacts on ARGs in the Pearl River Estuary by using suitable gene indicators[J]. Water Research, 2013, 47(8): 2811-2820. DOI:10.1016/j.watres.2013.02.042 |
| [20] | Luo Y, Mao D Q, Rysz M, et al. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environmental Science & Technology, 2010, 44(19): 7220-7225. |
| [21] | Jiang L, Hu X L, Xu T, et al. Prevalence of antibiotic resistance genes and their relationship with antibiotics in the Huangpu River and the drinking water sources, Shanghai, China[J]. Science of the Total Environment, 2013, 458-460: 267-272. DOI:10.1016/j.scitotenv.2013.04.038 |
| [22] | Huerta B, Marti E, Gros M, et al. Exploring the links between antibiotic occurrence, antibiotic resistance, and bacterial communities in water supply reservoirs[J]. Science of the Total Environment, 2013, 456-457: 161-170. DOI:10.1016/j.scitotenv.2013.03.071 |
| [23] | Walters W, Hyde E R, Berg-Lyons D, et al. Improved bacterial 16S rRNA gene (V4 and V4-5) and fungal internal transcribed spacer marker gene primers for microbial community surveys[J]. Msystems, 2016, 1(1): e00009-15. |
| [24] | Sampson T R, Debelius J W, Thron T, et al. Gut microbiota regulate motor deficits and neuroinflammation in a model of Parkinson's disease[J]. Cell, 2016, 167(6): 1469-1480. DOI:10.1016/j.cell.2016.11.018 |
| [25] | Li H Y, Xin H Y, Li S F Y. Multiplex PMA-qPCR Assay with internal amplification control for simultaneous detection of viable Legionella pneumophila, Salmonella typhimurium, and Staphylococcus aureus in environmental waters[J]. Environmental Science & Technology, 2015, 49(24): 14249-14256. |
| [26] | Li D, Tong T Z, Zeng S Y, et al. Quantification of viable bacteria in wastewater treatment plants by using propidium monoazide combined with quantitative PCR (PMA-qPCR)[J]. Journal of Environmental Sciences, 2014, 26(2): 299-306. DOI:10.1016/S1001-0742(13)60425-8 |
| [27] |
赵婷婷, 乔凯, 王蕾, 等. 淀粉废水处理系统中活性污泥的微生物群落结构及多样性分析[J]. 环境科学, 2020, 41(1): 321-329. Zhao T T, Qiao K, Wang L, et al. Measurements of bacterial community and biodiversity from activated sludge for a wastewater treatment containing starch[J]. Environmental Science, 2020, 41(1): 321-329. |
| [28] |
宿程远, 郑鹏, 卢宇翔, 等. 磁性纳米铁对厌氧颗粒污泥特性及其微生物群落的影响[J]. 环境科学, 2018, 39(3): 1316-1324. Su C Y, Zheng P, Lu Y X, et al. Effects of magnetic Fe3O4 nanoparticles on the characteristics of anaerobic granular sludge and its interior microbial community[J]. Environmental Science, 2018, 39(3): 1316-1324. |
| [29] | Callejas C, Fernández A, Passeggi M, et al. Microbiota adaptation after an alkaline pH perturbation in a full-scale UASB anaerobic reactor treating dairy wastewater[J]. Bioprocess and Biosystems Engineering, 2019, 42(12): 2035-2046. DOI:10.1007/s00449-019-02198-3 |
| [30] | Su C Y, Lin X M, Zheng P, et al. Effect of cephalexin after heterogeneous fenton-like pretreatment on the performance of anaerobic granular sludge and activated sludge[J]. Chemosphere, 2019, 235: 84-95. DOI:10.1016/j.chemosphere.2019.06.136 |
| [31] | Liu C, Li H, Zhang Y Y, et al. Evolution of microbial community along with increasing solid concentration during high-solids anaerobic digestion of sewage sludge[J]. Bioresource Technology, 2016, 216: 87-94. DOI:10.1016/j.biortech.2016.05.048 |
| [32] | Chen Q L, An X L, Li H, et al. Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil[J]. Environment International, 2016, 92-93: 1-10. DOI:10.1016/j.envint.2016.03.026 |
 2020, Vol. 41
2020, Vol. 41


