2. 贵州喀斯特环境生态系统教育部野外科学观测研究站, 贵阳 550025
2. Field Scientific Observation and Research Station, Ministry of Education, Guiyang 550025, China
城镇污水厂尾水深度脱氮是废水处理领域的研究热点, 尾水中存在大量的氮污染物, 易造成水体富营养化, Yu等[1]的研究发现, 我国各省除西藏区域外均有流域污染问题, 京杭大运河在1980年、巢湖在1985年和滇池在1981年均已开始出现氮污染, 氮累积近40年.目前常见的深度脱氮有生物法和物化法, 如离子交换法、膜分离法、反硝化生物滤池(DNBF)、移动床生物膜反应器(MBBR)[2]和人工湿地法等.但深度脱氮技术均存在碳源不足的现象, 通常补充外加碳源, 例甲醇、葡萄糖、乙醇和乙酸钠等[3], 然而外加碳源存在成本增加、资源浪费等问题.有研究者于1975年在Bardenpho工艺基础上提出发展带有前置厌氧段的Phoredox系列同步脱氮除磷工艺[4], 认为随着人们对污水处理生物原理认识的加深, 完全可以设计出可靠的系统实现高标准出水, 即TN < 3 mg·L-1.此外, 北京、昆明、巢湖和太湖等重点区域及流域将TN排放标准从20 mg·L-1(一级B)和15 mg·L-1(一级A), 提升为10 mg·L-1, 甚至5 mg·L-1(昆明A标), 逐渐向极限脱氮迈进.
甲烷氧化菌能氧化甲烷并产生有机物, 并在氮存在时, 甲烷氧化菌氧化甲烷的过程伴随着氮污染物的去除, 该过程分别为好氧甲烷氧化耦合反硝化(AME-D)[2]和厌氧甲烷氧化耦合反硝化(ANME-D)[5].其中, 好氧甲烷氧化菌最早于1906年被发现[6], 但AME-D过程最初是由Harremoes等于20世纪70年代发现[7].此外, 1978年Rhee等[8]首次证实了好氧条件下的甲烷氧化耦合反硝化是由甲烷氧化菌和反硝化菌共同完成的, 即甲烷氧化过程中产生的有机物被反硝化菌利用进行反硝化;Sun等[9]利用膜生物反应器(MBfR)进行污水脱氮实验, 得到最大反硝化率为97%;李彦澄等[10]采用生物膜反应器进行实验, 可实现硝酸盐氮去除率最高为98.93%.
目前, 好氧甲烷氧化耦合反硝化(AME-D)的机制有两种解释:①好氧甲烷氧化菌单独脱氮, 有关研究从基因组和转录方面分析, 发现好氧甲烷氧化菌中存在nirS、nirK和norB等能产生脱氮过程的基因, 但至今仍未发现1株具有全部反硝化功能基因的好氧甲烷氧化菌, 由于好氧甲烷氧化菌体内的反硝化功能基因广泛分布, 其在高硝酸盐氮或亚硝酸盐氮的环境中具有生存优势[11];②协同脱氮, 甲烷氧化菌氧化过程中会产生中间产物(有机物), 如甲醇、柠檬酸盐和乙酸盐等, 反硝化菌会利用中间产物作为碳源进行反硝化脱氮[12].
好氧甲烷耦合反硝化(AME-D)在城镇污水厂尾水深度脱氮方面具有巨大的应用潜力, 所需甲烷为可再生能源, 可由污水处理厂的厌氧处理工艺产生.本研究采用改良型反硝化生物滤池, 在人工模拟城镇污水厂尾水的情况下, 构建出AME-D极限脱氮系统, 考察了运行方式对系统效能的影响, 对出水中有机物的官能团进行拉曼光谱分析, 并采用16S rRNA基因测序技术分析微生物群落结构, 以期为城镇污水厂尾水深度脱氮提供技术支持和理论支撑.
1 材料与方法 1.1 实验水质本研究采用人工模拟城镇污水厂尾水, 模拟废水水质参数分别为:COD 20 mg·L-1、总氮20 mg·L-1、硝酸盐氮12 mg·L-1、氨氮8 mg·L-1和总磷1 mg·L-1.其中硝氮采用NaNO3模拟, 氨氮采用NH4Cl模拟, COD采用葡萄糖模拟, 总磷采用KH2PO4模拟.为保证微生物的正常生长, 添加微生物必需的微量元素, 微量元素的浓度分别为(mg·L-1)[13]:MgSO4·7H2O, 1 000; CaCl2·2H2O, 200; Fe-EDTA, 0.38; Na2MoO4·H2O, 0.26; FeSO4·7H2O, 0.90; MnCl2·4H2O, 0.02; CoCl2·6H2O, 0.05; H3BO3, 0.015; Na-EDTA, 0.25; ZnSO4·7H2O, 0.40; NiCl2·6H2O, 0.01; CuSO4·5H2O, 1.25.
1.2 实验装置本实验所采用生物膜反应器如图 1所示, 该反应器与反硝化生物滤池构造类似, 主体为双层构造, 反应器内层的有效体积为2 L, 底部设置曝气管, 并铺设鹅卵石(6~8 mm)垫层5 cm, 采用填料为石英砂(2~4 mm), 外层为水浴保温层, 通过循环水控制温度, 反应器可分为连续式和间歇式两种运行方式.本实验所需气体为甲烷(3%)与空气(97%)形成的混合气体, 并在反应器出气管上设置甲烷在线监测仪.
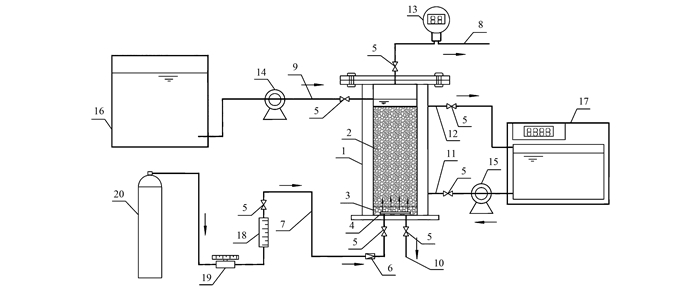
|
1.反应器主体; 2.填料(2~4 mm石英砂); 3.垫层(6~8 mm鹅卵石); 4.曝气管或曝气头; 5.阀门; 6.单向阀; 7.进气管; 8.出气管; 9.废水进水管; 10.废水出水管; 11.热水进水管; 12.热水出水管; 13.甲烷在线监测仪; 14.进水蠕动泵; 15.热水循环泵; 16.进水箱; 17.恒温水箱; 18.气体流量计; 19.气体流量调节阀; 20.混合气体 图 1 实验装置示意 Fig. 1 Experimental reactor |
反应器启动时接种贵阳市某污水处理厂浓缩池污泥, 污泥接种浓度为7.92 g·L-1, 温度控制为(30±1)℃.反应器运行初期采用下方进水上方出水的连续式运行方式, 采用蠕动泵控制进水流量, 流量控制为5 L·d-1.通过气体流量调节阀调节混合气体进气流量, 不间断进气, 进气流量为30 mL·min-1.反应器运行过程中采用甲烷在线监测仪监测出气中甲烷浓度比, 定期检测出水中的温度、DO、pH、氨氮、亚硝酸盐氮、硝酸盐氮和COD等水质指标.
连续式运行结束后, 采用上方进水下方出的间歇式运行方式, 温度仍控制为(30±1)℃, 运行周期为进水0.5 h→运行23 h→出水0.5 h, 每个周期结束后更换反应器内部所有液体, 其水量为1.2 L·d-1.进水和出水时停止进气, 运行时进气流量为30 mL·min-1.反应器运行过程中采用甲烷在线监测仪监测出气中甲烷浓度比, 定期检测出水中的温度、DO、pH、氨氮、亚硝酸盐氮、硝酸盐氮和COD等水质指标.
1.4 分析测试方法 1.4.1 理化指标分析氨氮的分析采用水杨酸分光光度法(HJ 536-2009), 亚硝酸盐氮的分析采用N-(1-萘基)-乙二胺光度法(GB 7493-87), 硝酸盐氮的分析采用紫外可见分光光度法(HJ/T 346-2007), COD采用HACH-COD快速消解分析法[14], pH、DO分别用pH便携式分析仪和DO便携式分析仪, 甲烷的监测采用SK-600-CH4型甲烷在线监测仪.
1.4.2 拉曼光谱分析样品预处理:取约15 mL样品放于离心管中, 冷冻成冰, 利用真空冷冻干燥机将样品制成干燥的粉末并冷藏保存.采用LRS-8共焦显微拉曼光谱仪对干燥的样品进行分析.具体分析方法如下:用硅片放入载物台进行波长矫正, 干燥的粉末放入硅片后放入载物台, 开启监视器推入显微镜上部的观察镜, 开启控制箱上白光光源, 调节电压旋钮到合适光强, 根据不同样品选择显微物镜, 上下左右调整载物台, 使硅片在显示屏上得到清晰像, 调节电压旋钮到0, 关闭白光光源, 打入激光光源再微调载物台, 使光源在TV显示屏上得到点像, 再拔出观察镜, 使用50倍物镜, 激发波长为520 cm-1, “狭缝”值为200 μm, 扫描范围100~3 000 cm-1, 仪器开始运行测量, 并记录光谱图像.
1.4.3 微生物多样性分析采集接种污泥和构建完成后系统的微生物进行16S rRAN基因测序分析, 将采集的混合液置于离心管中进行离心1 min, 转速5 000 r·min-1, 去除上清液后立即储存于- 20℃条件下冷冻, 然后送至上海美吉生物医药科技有限公司(Majorbio)进行16S rRNA基因测序分析, 进行V3-V4高变区测序[15], 选用细菌通用引物为:338F(5′-ACTCCT ACGGGAGGCAGCAG- 3′)和806R(5′-GGACTACHV GGGTWTCTAAT-3′), 分析测试方法与李彦澄等[10]的研究方法相同.
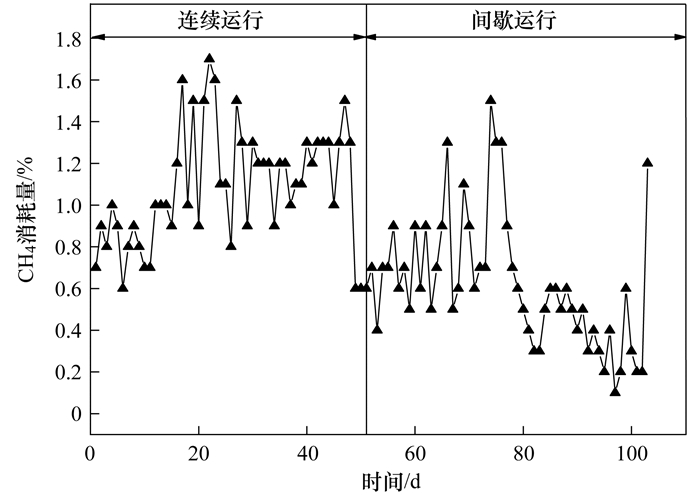
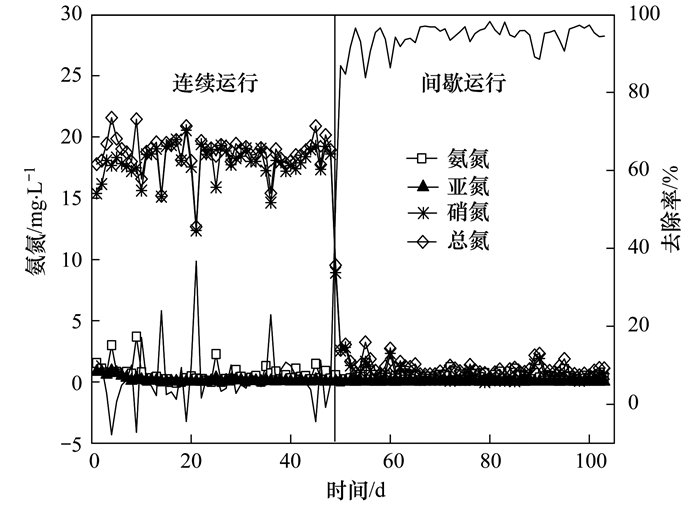
2 结果与讨论 2.1 AME-D极限脱氮系统构建反应器接种污泥后, 在连续进出水的方式下运行, 由图 2可知, 反应器启动初期对甲烷的消耗量呈上升趋势, 并在从第20 d开始趋于稳定, 其平均甲烷消耗量为1.22%, 但系统对总氮的去除率较低, 其平均去除率仅为6.47%(图 3), 对氨氮的平均去除率为92.11%, 说明该系统能在20 d内富集得到甲烷氧化菌和自养硝化菌, 系统中能发生硝化作用, 但不能发生反硝化作用, 连续运行的方式不利于氮污染的去除.
|
图 2 甲烷消耗量日变化 Fig. 2 Daily variation of the methane consumption |
|
图 3 不同运行条件下反应器出水TN、NH4+-N、NO3--N、NO2--N和去除率 Fig. 3 TN, NH4+-N, NO3--N, and NO2--N concentration and efficiency of the effluent under the different methods of operation |
从第49 d开始, 调整为间歇运行的方式, 停留时间变长, 两种运行方式下出水的pH值、DO和COD如表 1所示, 系统出水均呈中性, pH值相差不大, 出水中COD值均比进水高, 主要由于甲烷被氧化过程中会产生中间产物, 而间歇运行方式的DO比连续运行方式的DO低1.34 mg·L-1, 更有利于反硝化菌的生长繁殖.由图 2和图 3可知, 经过10 d左右, 系统趋于稳定, 其平均甲烷消耗量为0.62%, 总氮和氨氮的平均去除率分别为94.77%和93.30%, 出水中总氮和氨氮的平均浓度分别为1.05 mg·L-1和0.54 mg·L-1, 说明在该运行方式下能构建出AME-D极限脱氮系统.田宇心等[16]通过模型模拟与情景分析方法, 探讨了改良Bardenpho工艺的极限脱氮效果, 在低C/N进水条件下, 提高碳源(甲醇)加药量为3.0 m3·d-1, 出水的TN低于极限脱氮目标的3 mg·L-1, 系统TN去除率达96%.美国应用极限脱氮技术的污水厂中位于佛罗里达州的Bay point污水厂利用MLE活性污泥工艺, 升级曝气和化学处理, 将TN出水平均浓度从6.33 mg·L-1改造为3.99 mg·L-1, 宾夕法尼亚的Hempden Twp污水厂采用序列间歇式活性污泥(CSR)进行结构、流程优化, 将TN出水浓度从4.66 mg·L-1改造到3.64 mg·L-1.通过对比可知, 所构建出的AME-D极限脱氮系统具有较高的出水水质.
|
|
表 1 系统出水的pH、DO和COD Table 1 The pH, DO, and COD of the effluent in the methanotrophic system |
2.2 进出水拉曼光谱分析
采集间歇运行方式下的进出水进行拉曼光谱分析, 其图谱如图 4所示, 拉曼光谱图解析如表 2所示, 通过比对图谱可知, 进水中波数为416.46 cm-1和出水中波数为421.25 cm-1的峰, 该峰由PO43-的PO4对称变角振动引起, 主要是由于实验用水中含有磷酸盐等营养物质.进水经过处理后, 波数为1 051.57 cm-1的峰明显消失, 而该峰由NO3-对称伸缩引起, 进一步说明系统对硝酸盐氮具有较高的去除能力.出水中明显增强的峰为497.94 cm-1, 说明系统中产生了具有官能团P— Cl的物质.此外, 出水中明显增强的峰分别为608.81、623.46和672.90 cm-1, 醇COH面外弯曲或C—H面外弯曲振动吸收引起, 说明甲烷被氧化形成的中间产物可能主要为醇类物质.Kauffmann等[17]用拉曼光谱测定硝酸盐溶液, 在1 047 cm-1波长处得到特定NO3-的阴离子.占新华等[18]进行水溶性有机物和多环芳烃结合特征的红外光谱研究, 得到在610~690 cm-1吸收峰之间含有酚基功能团的结论.

|
图 4 系统进出水拉曼光谱图 Fig. 4 Raman spectrum of the inflow and effluent in the methanotrophic system |
|
|
表 2 拉曼光谱图解析 Table 2 Results of Raman spectroscopic analysis |
2.3 微生物多样性分析
对接种污泥和AME-D极限脱氮系统中的微生物进行16S rRNA基因测序分析, 如图 5所示, 在门水平上[图 5(a)], 接种污泥中检测出26门, AME-D极限脱氮系统中检测出17门.接种污泥中的优势门分别为变形菌门(Proteobacteria, 21.88%)、绿弯菌门(Chloroflexi, 33.48%)、拟杆菌门(Bacteroidetes, 10.19%)、硝化螺旋菌门(Nitrospirae, 5.11%)和浮霉菌门(Planctomycetes, 1.15%).AME-D极限脱氮系统中优势门分别为变形菌门(Proteobacteria, 76.10%)、绿弯菌门(Chloroflexi, 3.59%)、拟杆菌门(Bacteroidetes, 2.81%)、硝化螺旋菌门(Nitrospirae, 0.10%)和浮霉菌门(Planctomycetes, 0.17%), 其中, 丰度变化最大的为变形菌门, 1994年就有研究者发现微生物用于水处理方面最主要的为变形菌门[19], 且大部分反硝化细菌和甲烷氧化菌均属于变形菌门.说明该系统对变形菌门生长有促进作用, 而对绿弯菌门、拟杆菌门、浮霉菌门和硝化螺旋菌门有抑制作用.

|
S1 :接种污泥: S2:AME-D极限脱氮系统; (a)门水平; (b)科水平: (c)属水平. 图 5 门、科和属水平上的微生物分类 Fig. 5 Respective microbial classification at the phylum, family, and genus level |
在科水平上[图 5(b)], AME-D极限脱氮系统中出现新的假单胞菌科(Pseudomonadaceae)和芽孢杆菌科(Bacillaceae), 其相对丰度分别为56.92%和1.99%, 同时嗜甲基菌科(Methylophilaceae)、甲基球菌科(Methylococcaceae)和甲基囊孢菌科(Methylocystaceae)的相对丰度分别从接种污泥中的0.01%、0.08%和0.01%增长为1.91%、0.21%和0.27%.其中, 甲基球菌科和甲基囊孢菌科均属于好氧甲烷氧化菌[20], 具有氧化甲烷的能力[21], 是该系统中的主要甲烷氧化菌.而嗜甲基菌科是一种甲基营养菌, 不能直接氧化甲烷[22], 但能以甲醇和甲基胺类为生长基质[23], 嗜甲基菌科在系统中明显增长可能是甲烷氧化过程的中间产物主要为醇类等有机物[3].Kalyuhznaya等[24]发现嗜甲基菌科在湖泊沉积物中的反硝化脱氮时具有一定作用.Xia等[25]在富集培养甲烷氧化菌时, 分离出的主要微生物属于嗜甲基菌科, 同时表明甲烷氧化菌氧化甲烷时产生的中间有机物能被其他微生物利用[23].其他相对丰度变化较大的为腐螺旋菌科(Saprospiraceae)、多囊粘菌科(Polyangiaceae)和厌氧绳菌科(Anaerolineaceae)分别从4.73%、0.10%和1.45%下降为0.09%、0.06%和0%.
在属水平上[图 5(c)], 接种污泥中相对丰度较高的为Roseiflexus(25.59%)和Nitrospira(5.11%), 其中Nitrospira属于硝化菌, 而反硝化菌属占优势的分别为Hyphomicrobium(0.75%)、Pseudomonas(0.24%)和Paracoccus(0.30%).AME-D极限脱氮系统中相对丰度高的为Pseudomonas(56.92%)、Paenibacillus(3.52%)和Lysinibacillus(3.00%), 均属于反硝化菌, 其脱氮性能被广泛关注[26], 是主要的生物脱氮微生物, 系统中并还出现其他丰度较小的反硝化菌, 如Hyphomicrobium(0.65%)和Bacillus(1.92%).此外, AME-D极限脱氮系统中的硝化细菌主要为Nitrospira, 其相对丰度为0.1%.
此外, 接种污泥中检测出的甲烷氧化菌属分别为Methylobacteriaceae_norank(0.18%)、Methyloparacoccus(0.04%)和Methylosarcina(0.04%).AME-D极限脱氮系统中检测出的甲烷氧化菌属分别为Methyloparacoccus(0.12%)、Methylosarcina(0.10%)和Methylocaldum(0.09%)[27], 属于兼性甲烷氧化菌属的为Methylocystis(0.27%), 经过富集培养后, 系统中的甲烷氧化菌的种类增多, 且相对丰度升高, 其中, Methylocaldum、Methylosarcina和Methyloparacoccus属于甲基球菌科, 而Methylocystis属于甲基囊孢菌科, Belova等[28]发现Methylocystis的其他嗜酸甲烷氧化菌能够利用甲烷和乙酸生长, 并且具有sMMO和pMMO功能基因.而属于嗜甲基菌科的微生物主要为Methylophilaceae_unclassified(1.75%)和Methylophilus(0.12%).
由上述可知, AME-D极限脱氮系统中同时存在硝化菌、反硝化菌和好氧甲烷氧化菌, 主要的甲烷氧化菌为Methylocystis、Methylosarcina和Methyloparacoccus, 主要的反硝化菌为Pseudomonas、Paenibacillus和Lysinibacillus, 主要的硝化菌为Nitrospira.说明AME-D极限脱氮系统的脱氮作用是由好氧甲烷氧化菌、反硝化菌和硝化菌协同实现的, 其中, 好氧甲烷氧化菌氧化甲烷所产生的醇类等中间物质可能为反硝化细菌提供所需的碳源.
3 结论(1) 在间歇式运行方式下, 构建出AME-D极限脱氮系统, 其总氮和氨氮的平均去除率分别为94.77%和93.30%, 出水中总氮和氨氮的平均浓度分别为1.05 mg·L-1和0.54 mg·L-1.
(2) 通过拉曼光谱分析可知, 由NO3-对称伸缩引起的峰(1 051.57 cm-1)明显消失, 由醇COH面外弯曲或C—H面外弯曲振动吸收引起峰(608.81、623.46和672.90 cm-1)明显增强, 系统对硝酸盐氮具有较高的去除能力, 此外, 甲烷被氧化成的中间产物可能主要为醇类物质.
(3) 16S rRNA基因测序结果表明, AME-D极限脱氮系统同时存在硝化菌、反硝化菌和好氧甲烷氧化菌, 其中, 甲烷氧化菌主要为Methylocystis (0.27%)、Methylosarcina(0.10%)和Methyloparacoccus(0.12%), 反硝化菌主要为Pseudomonas(56.92%)、Paenibacillus(3.52%)和Lysinibacillus(3.00%), 硝化菌主要为Nitrospira(0.1%).
| [1] | Yu C Q, Huang X, Chen H, et al. Managing nitrogen to restore water quality in China[J]. Nature, 2019, 567(7749): 516-520. DOI:10.1038/s41586-019-1001-1 |
| [2] | Buitrón G, Moreno-Andrade I. Biodegradation kinetics of a mixture of phenols in a sequencing batch moving bed biofilm reactor under starvation and shock loads[J]. Journal of Chemical Technology and Biotechnology, 2011, 86(5): 669-674. DOI:10.1002/jctb.2566 |
| [3] | Zhu J, Wang Q, Yuan M D, et al. Microbiology and potential applications of aerobic methane oxidation coupled to denitrification (AME-D) process:a review[J]. Water Research, 2016, 90: 203-215. DOI:10.1016/j.watres.2015.12.020 |
| [4] |
刘智晓. 生物除磷理论及实践新突破——从主流EBPR到侧流EBPR[J]. 中国给水排水, 2018, 34(24): 19-25. Liu Z X. New breakthrough in the theory and practice of biological phosphorus removal:from mainstream EBPR to side-stream EBPR[J]. China Water & Wastewater, 2018, 34(24): 19-25. |
| [5] | Haroon M F, Hu S H, Shi Y, et al. Anaerobic oxidation of methane coupled to nitrate reduction in a novel archaeal lineage[J]. Nature, 2013, 500(7464): 567-570. DOI:10.1038/nature12375 |
| [6] | Söhngen N L. Vber bakterien, welche methan als kohlenstoffnahrung und energiequelle gebrauchen[J]. Zentrabl Bakteriol Parasitenk Infektionskr, 1906, 15: 513-517. |
| [7] | Harremoes P, Christensen M H. Denitrification with methane denitrification med methane[J]. Vand, 1971, 1: 7-11. |
| [8] | Rhee G Y, Fuhs G W. Wastewater denitrification with one-carbon compounds as energy source[J]. Journal (Water Pollution Control Federation), 1978, 50(9): 2111-2119. |
| [9] | Sun F Y, Dong W Y, Shao M F, et al. Aerobic methane oxidation coupled to denitrification in a membrane biofilm reactor:treatment performance and the effect of oxygen ventilation[J]. Bioresource Technology, 2013, 145: 2-9. DOI:10.1016/j.biortech.2013.03.115 |
| [10] |
李彦澄, 杨娅男, 刘邓平, 等. 基于好氧甲烷氧化菌的反硝化效能及微生物群落研究[J]. 中国环境科学, 2019, 39(10): 4387-4393. Li Y C, Yang Y N, Liu D P, et al. Denitrification efficiency and microbial community research in an aerobic methanotroph-based system[J]. China Environmental Science, 2019, 39(10): 4387-4393. DOI:10.3969/j.issn.1000-6923.2019.10.043 |
| [11] | Stein L Y, Klotz M G. Nitrifying and denitrifying pathways of methanotrophic bacteria[J]. Biochemical Society Transactions, 2011, 39(6): 1826-1831. DOI:10.1042/BST20110712 |
| [12] |
周祥玉, 赵云飞, 李东, 等. 微氧条件下以甲烷为碳源的反硝化实验研究[J]. 环境科学学报, 2017, 37(5): 1704-1710. Zhou X Y, Zhao Y F, Li D, et al. Experimental study on denitrification using methane as carbon source under microaerobic condition[J]. Acta Scientiae Circumstantiae, 2017, 37(5): 1704-1710. |
| [13] | Whittenbury R, Phillips K C, Wilkinson J F. Enrichment, isolation and some properties of methane-utilizing bacteria[J]. Journal of General Microbiology, 1970, 61(2): 205-218. DOI:10.1099/00221287-61-2-205 |
| [14] |
吉芳英, 杨琴, 罗固源. 实验室自配HACH-COD替代试剂研究[J]. 给水排水, 2003, 29(1): 17-20. Ji F Y, Yang Q, Luo G Y. Laboratory prepared substitutes for HACH-COD meter[J]. Water & Wastewater Engineering, 2003, 29(1): 17-20. |
| [15] | Xu N, Tan G C, Wang H Y, et al. Effect of biochar additions to soil on nitrogen leaching, microbial biomass and bacterial community structure[J]. European Journal of Soil Biology, 2016, 74: 1-8. DOI:10.1016/j.ejsobi.2016.02.004 |
| [16] | 田宇心, 李冰, 胡志荣, 等.改良Bardenpho污水处理工艺优化设计与极限脱氮分析[A].见: 2017中国环境科学学会科学与技术年会论文集(第二卷)[C].北京: 中国环境科学学会, 2017. |
| [17] | Kauffmann T H, Fontana M D. Inorganic salts diluted in water probed by Raman spectrometry:data processing and performance evaluation[J]. Sensors and Actuators B:Chemical, 2015, 209: 154-161. DOI:10.1016/j.snb.2014.11.108 |
| [18] |
占新华, 周立祥, 杨红, 等. 水溶性有机物与多环芳烃结合特征的红外光谱学研究[J]. 土壤学报, 2007, 44(1): 47-53. Zhan X H, Zhou L X, Yang H, et al. Infrared spectroscopy of Dom-PAHs complexes[J]. Acta Pedologica Sinica, 2007, 44(1): 47-53. DOI:10.3321/j.issn:0564-3929.2007.01.008 |
| [19] | Manz W, Wagner M, Amann R, et al. In situ characterization of the microbial consortia active in two wastewater treatment plants[J]. Water Research, 1994, 28(8): 1715-1723. DOI:10.1016/0043-1354(94)90243-7 |
| [20] |
贠娟莉, 王艳芬, 张洪勋. 好氧甲烷氧化菌生态学研究进展[J]. 生态学报, 2013, 33(21): 6774-6785. Yun J L, Wang Y F, Zhang H X. Ecology of aerobic methane oxidizing bacteria (methanotrophs)[J]. Acta Ecologica Sinica, 2013, 33(21): 6774-6785. |
| [21] | Kalyuzhnaya M G, Puri A W, Lidstrom M E. Metabolic engineering in methanotrophic bacteria[J]. Metabolic Engineering, 2015, 29: 142-152. DOI:10.1016/j.ymben.2015.03.010 |
| [22] | Doronina N, Kaparullina E, Trotsenko Y. The family methylophilaceae[A]. In: Rosenberg E, DeLong E F, Lory S, et al (Eds.). The Prokaryotes: Alphaproteobacteria and Betaproteobacteria (4th ed.)[M]. Berlin: Springer, 2014. 869-880. |
| [23] | Beck D A C, McTaggart T L, Setboonsarng U, et al. The expanded diversity of methylophilaceae from lake washington through cultivation and genomic sequencing of novel ecotypes[J]. PLoS One, 2014, 9(7): e102458. DOI:10.1371/journal.pone.0102458 |
| [24] | Kalyuhznaya M G, Martens-Habbena W, Wang T S, et al. Methylophilaceae link methanol oxidation to denitrification in freshwater lake sediment as suggested by stable isotope probing and pure culture analysis[J]. Environmental Microbiology Reports, 2009, 1(5): 385-392. DOI:10.1111/j.1758-2229.2009.00046.x |
| [25] | Xia F, Zou B, Shen C, et al. Complete genome sequence of Methylophilus sp. TWE2 isolated from methane oxidation enrichment culture of tap-water[J]. Journal of Biotechnology, 2015, 211: 121-122. DOI:10.1016/j.jbiotec.2015.07.023 |
| [26] |
周晓黎, 孙迎雪, 沈丹丹, 等. 反硝化生物滤池反冲洗前后的生物膜特征研究[J]. 环境科学与技术, 2015, 38(S1): 26-29, 65. Zhou X L, Sun Y X, Shen D D, et al. Characteristics of biofilm in a denitrification biological filter before and after backwashing[J]. Environmental Science and Technology, 2015, 38(S1): 26-29, 65. |
| [27] | Bowman J P. The family methylococcaceae[A]. In: Rosenberg E, DeLong E F, Lory S, et al (Eds.). The Prokaryotes: Alphaproteobacteria and Betaproteobacteria (4th ed.)[M]. Berlin: Springer, 2014. 411-440. |
| [28] | Belova S E, Baani M, Suzina N E, et al. Acetate utilization as a survival strategy of peat-inhabiting Methylocystis spp.[J]. Environmental Microbiology Reports, 2011, 3(1): 36-46. DOI:10.1111/j.1758-2229.2010.00180.x |
 2020, Vol. 41
2020, Vol. 41


