2. 清华大学环境学院, 北京 100084;
3. 北京大学环境科学与工程学院, 北京 100871
2. School of Environment, Tsinghua University, Beijing 100084, China;
3. College of Environmental Sciences and Engineering, Peking University, Beijing 100871, China
抗生素是20世纪最重要的医学发现之一, 对控制人类感染性疾病发挥了巨大的作用.目前, 抗生素主要被用于人类医疗[1].人类使用的抗生素大多不能被充分利用而直接排入环境中.废水处理厂则是抗生素直接进入到水环境的重要途径[2].长期接触抗生素而产生的选择性压力促使抗生素抗性基因(antibiotic resistance genes, ARGs)广泛分布[3].即使是在低水平的抗生素环境下也可能导致ARGs的出现.在河流[4~6]、湖泊[7, 8]、土壤[9]和废水处理厂(wastewater treatment plants, WWTPs)[10]甚至人类影响较少的地区, 如永冻层沉积物[11]中都找到ARGs的踪迹. ARGs除了自身遗传外, 还可以通过从一种细菌到另一种细菌的基因水平转移获得, 移动基因元件在ARGs的水平转移过程中发挥着重大作用, 如质粒、整合子和转座子[12]. ARGs对人类公共卫生造成了越来越大的威胁.
在城市水循环系统中, 废水处理厂发挥着重要的作用.它既是各种污水的汇集地, 又是污染地表水的源头, 而地表水又是饮用水的重要来源[2].人类使用的抗生素中, 约50%~90%是通过尿液和粪便排出体外, 然后这些化合物被带到WWTPs[13].废水处理厂汇集了人类生活、生产中的大量废水[14].其污水和污泥被认为是向环境中释放抗生素抗性基因的主要人为污染来源[15].
通过废水处理厂处理单元, 废水中大部分ARGs绝对丰度降低, 其中ARGs降低的主要原因是细菌在二沉池中的沉降[16].由于废水处理厂处理工艺的不同, ARGs在WWTPs中可能受到不同的影响, 加拿大某废水处理厂通过MBR工艺, ARGs丰度下降1.77~2.69个数量级[17], 浙江某市政废水处理厂经过A/O工艺, ARGs丰度降低1~3个数量级[18], 安徽某废水处理厂经过A2/O工艺, ARGs丰度降低0.41~1.60个数量级[19].有研究发现, 在Fe2+/H2O2摩尔比为0.1, H2O2浓度为0.01 mol ·L-1, pH为3.0, 反应时间为2 h的最优条件下, 目标ARGs基因下降2.58~3.79个数量级[20].
本研究选取以生产抗生素为主导行业的某化工园区废水处理系统为研究对象, 该厂进水为工业废水, 主要为化工废水、印染废水和制药废水等.沿污水处理工艺流程采集污水和污泥样品, 对其中8类共26种ARGs进行定性检测, 并对其中11种ARGs、一类整合子及16S rDNA进行定量检测, 分析ARGs在该废水处理厂中的分布特征及其对ARGs的去除效果, 研究含有高抗生素、抗生素抗性基因的污水对废水处理厂处理水体中ARGs的影响.本研究对了解高抗生素浓度废水对废水处理厂沿程ARGs分布的影响具有突出的典型意义.
1 材料与方法 1.1 园区污水简介选取以生产抗生素为主导行业的某化工园区废水处理厂为研究对象, 其进水状况复杂, 医药、染料等难处理废水比重高.园区内共有42家染料制造企业, 20家医药制造企业, 8家农药制造企业, 14家纺织印染企业, 7家日用化妆品制造企业, 还有17家其他化工企业, 这些企业的废水最终排入该废水处理厂.园区医药制造企业以合成大环内酯类抗药菌为主, 其中克拉霉素、罗红霉素等产品在我国市场占优势地位, 医药企业、农药制造业、纺织印染业和染料制造业废水各占园区总废水的16%、5%、15%和30%.废水处理厂的进水水质如表 1所示.
|
|
表 1 废水处理厂进水水质 Table 1 Water quality in the influents of the WWTPs |
1.2 样品采集及预处理
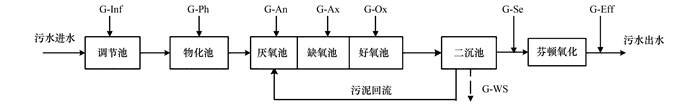
样品于2018年4月采集, 该废水处理厂的工艺流程以及采样点见图 1, 设计处理水量22.5万m3 ·d-1, 目前实际处理水量11.5万m3 ·d-1, 废水处理厂的进水来自于园区的工业废水.该厂主要处理单元包括调节池(酸碱调节)、物化处理单元(絮凝沉淀)、生物处理单元以及深度处理单元(芬顿工艺), 主要生物处理单元为厌氧池, 氧化沟(缺氧段, 好氧段), 水力停留时间为11 h.
|
图 1 废水处理系统工艺流程和采样点示意 Fig. 1 Processes and sampling points at the wastewater treatment plant |
采样点包括:进水(G-Inf)、物化池水(G-Ph)、二沉池出水(G-Se)、芬顿出水(G-Eff)、生物处理单元不同区段的泥水混合物(G-An、G-Ax和G-Ox), 以及二沉池剩余污泥(G-WS).每个采样点采集了1 L样品, 水样存放于预先清洗干净的无菌聚乙烯塑料瓶中, 泥样存放于预先清洗干净的铝盒中, 冷藏运输, 回到实验室后水样置于4℃保存, 泥样置于-20℃保存.
1.3 样品DNA提取以0.22 μm混合纤维素酯滤膜(Millipore, 德国)过滤样品, 再使用PowerSoil DNA Isolation Kit(Mo Bio, 美国)试剂盒, 根据试剂盒方法对滤样提取DNA.使用质量浓度为1%的琼脂糖凝胶电泳和NanoDrop 2000(Thermo Fisher, 美国)对所提DNA纯度和浓度进行检测.
1.4 普通PCR检测本研究选择了8类共26种ARGs进行定性检测, 具体包括6种四环素类ARGs (tetA、tetB、tetC、 tetM、 tetO和tetW), 4种磺胺类ARGs (sulⅠ、sulⅡ、sulⅢ和sulA), 3种甲氧苄啶ARGs (dfrA1、dfrA12和dfrA13), 3种β-内酰胺类ARGs (ampC、blaSHV和blaPSE-1), 2种大环内酯类ARGs (ermA、ermB), 3种氯霉素类ARGs (catⅠ、catⅡ和floR), 2种氨基糖苷类ARGs[aac(3)-Ⅱa、aac(3)-Ⅳ]和3种万古霉素类ARGs (vanA、vanB和vanC). PCR引物序列如表 2所示, 本实验所使用的引物均由生工生物工程(上海)股份有限公司合成.普通PCR采用25 μL体系, 包含12.5 μL Premix Ex TaqTM Hot Start Version (TaKaRa, 中国), 各1 μL的上、下游引物, 1 μL的DNA模板及9.5 μL ddH2O. PCR产物置于4℃保存, 并使用质量浓度为1%~2%的琼脂糖凝胶电泳进行检测.
|
|
表 2 PCR引物序列 Table 2 Sequences of premiers in PCR reactions |
1.5 定量PCR(qPCR)检测
根据普通PCR定性检测的结果, 本研究对tetA、tetC、tetO、tetW、sulⅠ、sulⅡ、dfrA1、dfrA13、floR、 ermB和blaPSE-1共11种ARGs、一类整合子intI1及16S rDNA进行定量测定.采用SYBR Green Ⅰ方法, 使用实时荧光定量PCR仪(BioRad CFX96 Touch, 美国)对各目标基因进行定量测定. qPCR采用20 μL体系, 包含10.0 μL SYBR Premix Ex TaqTM (TaKaRa, 中国), 引物各0.4 μL, DNA模板1 μL及ddH2O 8.2 μL.反应程序为:95℃预变性1~2 min; 95℃变性30 s, 退火30 s, 72℃延伸30 s, 共40个循环[27].溶解曲线程序为55~95℃, 每0.5℃读数一次, 期间停留30 s.测定时, 将标准质粒以10倍梯度稀释, 再根据质粒浓度计算得到标准质粒的拷贝数, 与通过实时荧光定量PCR测定得的Ct值对应做出标准曲线, 标准质粒的构建方法参照张衍等[28]的研究.各目标基因标准曲线的线性相关系数在0.993~0.999之间.每个样品均设置3个平行样.
2 结果与讨论 2.1 废水处理系统进水中ARGs存在情况表 3显示了样品中ARGs的检出情况.
|
|
表 3 废水处理厂进水中ARGs检出情况1) Table 3 Presence of ARGs in the influents of the WWTPs |
在进水水样中共检出7大类16种ARGs, 并在进水中检出intI1, 进水中没有检出万古霉素类, 其中四环素类ARGs检出的种类最多, 共检出5种.四环素广泛应用于人类治疗、畜牧和水产养殖等领域, 在中国抗生素的使用中, 四环素的使用量已排名第一, 四环素难以被人和动物代谢和吸收[24], 这可能导致废水处理厂进水中四环素类ARGs检出种类多.
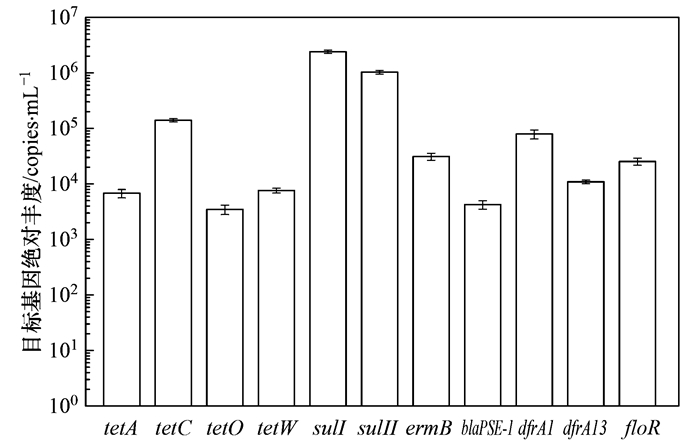
定量结果分析表明, 该废水处理厂进水中的绝对丰度最高的为磺胺类ARGs, 进水中的检测结果如图 2所示.
|
图 2 废水处理厂进水中的目标基因绝对丰度 Fig. 2 Absolute abundances of target genes in the WWTPs |
进水中磺胺类的绝对丰度为2.41×106 copies ·mL-1(sulⅠ)和1.03×106 copies ·mL-1(sulⅡ), 磺胺是应用最广泛的抗生素之一, 具有高溶解度和持久性的特点, 而磺胺耐药基因的持久性比抗生素本身要长, 因此, 磺胺类耐药基因可能是环境中占主导地位的ARG[29].而后绝对丰度依次递减的是tetC、dfrA1、ermB、floR、dfrA13、tetW、tetA、BlaPSE-1和tetO. 图 2显示tetA和tetC的绝对丰度高于tetO. tetA和tetC基因的抗性机制为编码外排泵蛋白, tetO基因的抗性机制为编码核糖体保护蛋白[27], 即在该园区的进水中编码外排泵蛋白抗性机制的四环素类ARGs的浓度远远高于编码核糖体保护蛋白抗性机制的四环素类ARGs.
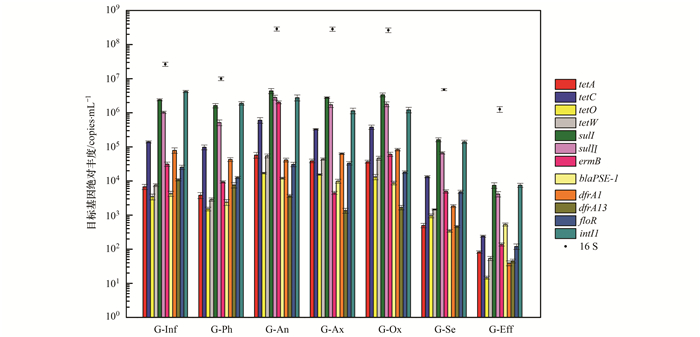
2.2 废水处理厂工艺沿程ARGs的分布特征废水处理厂采集的样品中目标基因的绝对丰度分布情况如图 3所示.通过废水处理厂各单元处理, 每个基因绝对丰度都发生了显著的变化.
|
图 3 废水处理厂中ARGs和16S rDNA的分布 Fig. 3 Abundance of ARGs and 16S rDNA in the WWTPs |
园区废水经过格栅、絮凝沉淀、生物处理和二沉池沉淀各个单元后, 水体中各ARGs的绝对丰度发生了巨大变化, 污水在曝气池中进行生物降解并且微生物在曝气池中不断生长, ARGs和16S rRNA基因在曝气池中大量增殖[16], 曝气池水体中ARGs增殖了0.16~2.34个数量级, ARG的去除与水体中微生物量的减少密切相关[30], 这意味着较低的细菌生长速度有利于去除ARG.有研究表明, 固液分离去除吸附在活性污泥上的ARGs是生物工艺去除ARG的主要机制[31], 污泥浓度、水力停留时间、污泥沉降比和污泥泥龄均对固液分离效果有影响. Fenton工艺通过过氧化氢和Fe2+在溶液中产生羟基自由基来降解细菌的外膜, 增加通透性, 诱发内部反应, 最终使细胞失去活性[32].
从图 3中可以看出, floR、tetO、tetW和ermB这4种ARGs, 经过絮凝沉淀、二沉池沉淀绝对丰度分别下降了floR(0.30、0.42)、tetO(0.36、0.20)、tetW(0.42、0.29)和ermB(0.43、0.28)个数量级, 经过絮凝沉淀去除的ARGs丰度与二沉池沉淀去除的ARGs丰度基本一致, 说明这4种ARGs不能被生物处理单元有效去除, 均是通过微生物的沉淀去除的; 而芬顿工艺对该4种ARGs的去除为1.60、1.56、1.43和1.82个数量级, 去除效果均优于絮凝沉淀和二沉池沉淀. tetA、tetC、sulⅠ、sulⅡ、dfrA1和dfrA13这6种抗性基因通过混凝沉淀后, 绝对丰度分别下降了0.25、0.16、0.17、0.30、0.27和0.15个数量级, 这6种ARGs经过生物处理单元, 绝对丰度下降了0.88、0.86、1.00、0.89、1.37和1.22个数量级, 生物处理对该6种ARGs的去除效果显著地优于絮凝沉淀.经过芬顿工艺处理后, 水体中ARGs的绝对丰度分别下降了0.78、1.74、1.34、1.21、1.67和1.01个数量级, 这6种ARGs在该废水处理厂各工艺段中, 生物处理工艺与芬顿工艺对ARGs的去除效果显著优于絮凝沉淀.前4种ARGs与后6种ARGs在不同工艺中被去除的效果明显不同, 可能是由于ARGs对抗生素发挥抗药性的机制不同导致. floR、tetO、tetW和ermB主要是通过改变抗生素的作用位点遏制抗生素[33]; sulⅠ、sulⅡ、dfrA1和dfrA13通过使抗生素无法作用于目标酶来抑制抗生素[34]; tetA和tetC能改变细胞膜通透性使抗生素的胞内浓度降低[35].该废水处理厂各工艺对blaPSE-1去除效果不佳, 物化池的絮凝沉淀以及芬顿工艺均没有对该抗性基因起到很好地去除作用, 经过絮凝沉淀和芬顿工艺, 该ARG的绝对丰度分别下降了0.25和-0.19个数量级.
尽管该废水处理厂去除ARGs效果明显, 但由于园区污水中ARGs的本底值高, 废水处理厂最终出水中的ARGs的绝对丰度远远高于纳污环境中ARGs的本底丰度, 出水直接排入纳污水体中, 可能引起ARGs的传播, 影响纳污水体中的生态环境.
相关性分析结果显示, 废水处理厂水样中16S rDNA绝对丰度与四环素类抗性基因的绝对丰度显著正相关[tetA(P < 0.05, r=0.967)、tetC(P < 0.05, r=0.967)、tetO(P < 0.05, r=0.967)和tetW(P < 0.05, r=0.967)].说明废水中四环素类抗性基因丰度变化受微生物浓度变化的影响[36].而甲氧苄啶类抗性基因绝对丰度沿废水处理厂的工艺流程不断降低, 与16S rDNA绝对丰度无显著相关(P=0.52), 说明微生物对甲氧苄啶类抗性基因的影响不明显.甲氧苄啶类抗性基因存在于印染废水中, 园区中有多家印染和染料企业.印染废水很大程度上影响甲氧苄啶类抗性基因的分布和转移[37].
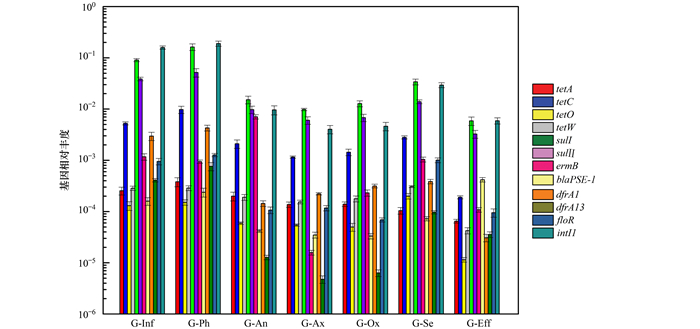
有研究表明, 一个细菌平均约有4个16S rDNA基因拷贝[38].从单位细菌角度分析ARGs在废水处理厂沿程分布, 结果如图 4所示.相对丰度最高的ARGs为sulⅠ, intI 1、sulⅡ、tetC和ermB依次递降, intI 1相对丰度与sulⅠ (P < 0.05, r=0.965)、sulⅡ (P < 0.05, r=0.988)存在显著正相关, I类整合子可能参与磺胺类抗性基因的传播, 整合子基因序列能够通过横向转移促进磺胺类抗性基因的传播[39].废水处理厂进水中ARGs的相对丰度为1.30×10-4~9.06×10-2, 进水中的微生物与曝气池中大量微生物结合, 最终在二沉池沉降, 出水中ARGs的相对丰度下降至1.10×10-5~5.89×10-4, 而曝气池污泥中ARGs的绝对丰度为1.70×104~4.38×106 copies ·g-1, 远低于剩余污泥中ARGs的绝对丰度(1.59×105~9.96×107 copies ·g-1), 说明大量ARGs由剩余污泥的形式排入环境.
|
图 4 废水处理厂中ARGs的相对丰度 Fig. 4 Relative abundance of ARGs in the WWTPs |
表 4所示, W1废水处理厂的工艺为厌氧-缺氧-好氧MBR, 进水为生活污水; W2废水处理厂的工艺为A2/O, 进水为生活污水; W3废水处理厂的工艺为CASS, 进水为生活污水; W4废水处理厂的工艺为缺氧-好氧-好氧-混凝砂滤, 处理的是焦化废水.
|
|
表 4 各废水处理厂工艺及ARGs检出情况 Table 4 Processing of WWTPs and detection of ARGs |
以生产抗生素为主导行业的某化工园区废水处理厂(W5)在进水中检出16种ARGs, 甲氧苄啶类、氯霉素类和氨基糖苷类ARGs均在W5废水处理厂中被检出. W1废水处理厂[tetC(3.7×104copies ·mL-1)、sulⅡ (5.4×107 copies ·mL-1)]、W2废水处理厂[tetA(2.82×104 copies ·mL-1)、tetW (2.63×106 copies ·mL-1)、sulⅠ (9.33×105 copies ·mL-1)和sulⅡ (4.68×106 copies ·mL-1)];W3废水处理厂[tetA(3.31×104 copies ·mL-1)、tetO(4.57×104 copies ·mL-1)、sulⅠ (4.47×105 copies ·mL-1)和sulⅡ (5.62×106 copies ·mL-1)]和W5废水处理厂[tetA(6.78×104 copies ·mL-1)、tetO(3.47×104 copies ·mL-1)、sulⅠ (2.51×105 copies ·mL-1)和sulⅡ (1.03×106 copies ·mL-1)]进水中四环素类、磺胺类ARGs相似, 四环素类ARGs均在104 copies ·mL-1丰度水平, 磺胺类ARGs在106 copies ·mL-1丰度水平.城镇污水处理系统汇集了居民生活、工业生产、农业生产、医疗卫生等各方面的废水, 有研究表明, 居民生活、农业生产、医疗卫生废水以及畜禽类动物粪便中四环素类、磺胺类ARGs检出丰度高[41].本研究中, 园区的医药企业以生产大环内酯类抗生素为主, 四环素类和磺胺类抗生素为辅, 有研究表明, 含有高浓度四环素类、磺胺类抗生素的医疗废水与tet和sul类ARGs呈不相关甚至反相关关系[42].因此W1、W2和W3废水中四环素类、磺胺类ARGs丰度与W5废水中四环素类、磺胺类ARGs相似.
W1废水处理厂[blaPSE-1 (3.40×105 copies ·mL-1)]、W2废水处理厂[blaCTX-M(9.77×103 copies ·mL-1)]和W3废水处理厂[blaCTX-M(6.31×105 copies ·mL-1)]进水中β-内酰胺类抗性基因绝对丰度均高于W5废水处理厂[blaPSE-1 (4.24×103 copies ·mL-1)]进水中β-内酰胺类抗性基因, W5废水处理厂进水中β-内酰胺类抗性基因绝对丰度仅达到W1和W3废水处理厂的1%.由于青霉素、头孢菌素类抗生素均属于β-内酰胺类抗生素[43], 三代头孢、青霉素类和二代头孢分别位列计算抗菌药物使用强度(AUD)排名的前3位[44], β-内酰胺类抗生素在居民生活以及医疗卫生废水中浓度较高[45], 由于选择性压力, 进水为生活污水的W1、W2和W3废水处理厂中β-内酰胺类ARGs绝对丰度高于W5废水处理厂和W4废水处理厂进水中β-内酰胺类ARGs绝对丰度.
W5废水处理厂进水中ermB(3.12×104 copies ·mL-1)的绝对丰度远远高于W1(2.14×102 copies ·mL-1)和W4(4.14×103 copies ·mL-1)废水处理厂中ermB的绝对丰度.园区共有20家医药企业, W5废水处理厂进水中包含园区制药废水中大量大环内酯类抗生素, ermB的绝对丰度较高可能受进水中占16%的医药废水影响, 长期在高浓度大环内酯类抗生素环境中, 废水中ARGs显著高于其他废水处理厂.
5座废水处理厂进水中的ARGs经过生物处理后, 总ARGs丰度明显下降, 在出水中磺胺类ARGs的绝对丰度显著高于其他ARGs的绝对丰度. W1、W2和W3废水处理厂进水经过生物处理, 四环素类ARGs丰度下降了1.50~3.10 log, 由于进水中四环素类ARGs绝对丰度大, 最终W1废水处理厂[tetC(1.58×103 copies ·mL-1)]、W2废水处理厂[tetA(2.80×102 copies ·mL-1)]、W3废水处理厂[tetA(2.18×102 copies ·mL-1)]出水中四环素类ARGs绝对丰度高于W4废水处理厂以及W5废水处理厂[tetA(8.2×101 copies ·mL-1)]出水中四环素类ARGs.经过生物处理后, 5座污水处理厂出水中β-内酰胺类ARGs绝对丰度(1.01×102~6.16×102 copies ·mL-1)基本一致.
3 结论(1) 在以生产抗生素为主导行业的某化工园区废水处理厂进水中检出7大类16种ARGs, 其中磺胺类抗性基因绝对丰度最高, 绝对丰度分别为sulⅠ (2.41×106 copies ·mL-1), sulⅡ (1.03×106 copies ·mL-1).
(2) 芬顿氧化工艺对ARGs的去除效果明显优于絮凝沉淀和生物处理工艺对ARGs的去除效果, 含有高丰度ARGs的水体有必要增加芬顿工艺.
(3) 以生产大环内酯类抗生素为主导行业的某化工园区废水处理厂进水中四环素类、磺胺类、β-内酰胺类ARGs绝对丰度明显低于W1、W2和W3废水处理厂, 大环内酯类ARGs绝对丰度明显高于其他废水处理厂.
致谢: 苏志国、李菲菲、颉亚玮、赵雅楠、郑宇涵等在采样方面提供帮助, 在此一并致谢.| [1] |
窦春玲, 郭雪萍, 尹大强. 污水处理厂抗生素抗性基因分布和去除研究进展[J]. 环境化学, 2013, 32(10): 1885-1893. Dou C L, Guo X P, Yin D Q. Review on distribution and removal of antibiotic resistance genes (ARGs) in wastewater treatment plants (WWTPs)[J]. Environmental Chemistry, 2013, 32(10): 1885-1893. DOI:10.7524/j.issn.0254-6108.2013.10.010 |
| [2] |
张治国, 李斌绪, 李娜, 等. 污水深度处理工艺对抗生素抗性菌和抗性基因去除研究进展[J]. 农业环境科学学报, 2018, 37(10): 2091-2100. Zhang Z G, Li B X, Li N, et al. Removal of antibiotic-resistant bacteria(ARB)and antibiotic-resistance genes(ARGs)in the effluent of wastewater treatment plants (WWTPs)via advanced treatment processes[J]. Journal of Agro-Environment Science, 2018, 37(10): 2091-2100. DOI:10.11654/jaes.2018-0098 |
| [3] | Chen H, Zhang M M. Effects of advanced treatment systems on the removal of antibiotic resistance genes in wastewater treatment plants from Hangzhou, China[J]. Environmental Science & Technology, 2013, 47(15): 8157-8163. |
| [4] | Ling Z H, Yang Y, Huang Y L, et al. A preliminary investigation on the occurrence and distribution of antibiotic resistance genes in the Beijiang river, South China[J]. Journal of Environmental Sciences, 2013, 25(8): 1656-1661. DOI:10.1016/S1001-0742(12)60223-X |
| [5] | Brown P C, Borowska E, Schwartz T, et al. Impact of the particulate matter from wastewater discharge on the abundance of antibiotic resistance genes and facultative pathogenic bacteria in downstream river sediments[J]. Science of the Total Environment, 2019, 649: 1171-1178. DOI:10.1016/j.scitotenv.2018.08.394 |
| [6] | Luo Y, Mao D Q, Rysz M, et al. Trends in antibiotic resistance genes occurrence in the Haihe river, China[J]. Environmental Science & Technology, 2010, 44(19): 7220-7225. |
| [7] | Di Cesare A, Eckert E M, Teruggi A, et al. Constitutive presence of antibiotic resistance genes within the bacterial community of a large subalpine lake[J]. Molecular Ecology, 2015, 24(15): 3888-3900. DOI:10.1111/mec.13293 |
| [8] | Thevenon F, Adatte T, Wildi W, et al. Antibiotic resistant bacteria/genes dissemination in lacustrine sediments highly increased following cultural eutrophication of Lake Geneva (Switzerland)[J]. Chemosphere, 2012, 86(5): 468-476. DOI:10.1016/j.chemosphere.2011.09.048 |
| [9] | Czekalski N, Díez E G, Bürgmann H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake[J]. The ISME Journal, 2014, 8(7): 1381-1390. DOI:10.1038/ismej.2014.8 |
| [10] | Le T H, Ng C, Tran N H, et al. Removal of antibiotic residues, antibiotic resistant bacteria and antibiotic resistance genes in municipal wastewater by membrane bioreactor systems[J]. Water Research, 2018, 145: 498-508. DOI:10.1016/j.watres.2018.08.060 |
| [11] | Yang Y, Zhou R J, Chen B W, et al. Characterization of airborne antibiotic resistance genes from typical bioaerosol emission sources in the urban environment using metagenomic approach[J]. Chemosphere, 2018, 213: 463-471. DOI:10.1016/j.chemosphere.2018.09.066 |
| [12] |
苏志国, 张衍, 代天娇, 等. 环境中抗生素抗性基因与Ⅰ型整合子的研究进展[J]. 微生物学通报, 2018, 45(10): 2217-2233. Su Z G, Zhang Y, Dai T J, et al. Antibiotic resistance genes and class 1 integron in the environment:research progress[J]. Microbiology China, 2018, 45(10): 2217-2233. |
| [13] | Tran N H, Reinhard M, Gin K Y H. Occurrence and fate of emerging contaminants in municipal wastewater treatment plants from different geographical regions-a review[J]. Water Research, 2018, 133: 182-207. DOI:10.1016/j.watres.2017.12.029 |
| [14] | Zhang Y, Li A L, Dai T J, et al. Cell-free DNA:a neglected source for antibiotic resistance genes spreading from WWTPs[J]. Environmental Science & Technology, 2018, 52(1): 248-257. |
| [15] | Czekalski N, Berthold T, Caucci S, et al. Increased levels of multiresistant bacteria and resistance genes after wastewater treatment and their dissemination into Lake Geneva, Switzerland[J]. Frontiers in Microbiology, 2012, 3: 106. |
| [16] | McConnell M M, Hansen L T, Jamieson R C, et al. Removal of antibiotic resistance genes in two tertiary level municipal wastewater treatment plants[J]. Science of the Total Environment, 2018, 643: 292-300. DOI:10.1016/j.scitotenv.2018.06.212 |
| [17] | Mao D Q, Yu S, Rysz M, et al. Prevalence and proliferation of antibiotic resistance genes in two municipal wastewater treatment plants[J]. Water Research, 2015, 85: 458-466. DOI:10.1016/j.watres.2015.09.010 |
| [18] | Guo M T, Yuan Q B, Yang J. Distinguishing effects of ultraviolet exposure and chlorination on the horizontal transfer of antibiotic resistance genes in municipal wastewater[J]. Environmental Science & Technology, 2015, 49(9): 5771-5778. |
| [19] | Chen H, Zhang M M. Occurrence and removal of antibiotic resistance genes in municipal wastewater and rural domestic sewage treatment systems in eastern China[J]. Environment International, 2013, 55: 9-14. DOI:10.1016/j.envint.2013.01.019 |
| [20] | Zhang Y Y, Zhuang Y, Geng J J, et al. Reduction of antibiotic resistance genes in municipal wastewater effluent by advanced oxidation processes[J]. Science of the Total Environment, 2016, 550: 184-191. DOI:10.1016/j.scitotenv.2016.01.078 |
| [21] | Maynard C, Fairbrother J M, Bekal S, et al. Antimicrobial resistance genes in enterotoxigenic Escherichia coli O149:K91 isolates obtained over a 23-year period from pigs[J]. Antimicrobial Agents and Chemotherapy, 2003, 47(10): 3214-3221. DOI:10.1128/AAC.47.10.3214-3221.2003 |
| [22] | Ji X L, Shen Q H, Liu F, et al. Antibiotic resistance gene abundances associated with antibiotics and heavy metals in animal manures and agricultural soils adjacent to feedlots in Shanghai; China[J]. Journal of Hazardous Materials, 2012, 235-236: 178-185. DOI:10.1016/j.jhazmat.2012.07.040 |
| [23] | Chen S, Zhao S H, White D G, et al. Characterization of multiple-antimicrobial-resistant salmonella serovars isolated from retail meats[J]. Applied and Environmental Microbiology, 2004, 70(1): 1-7. DOI:10.1128/AEM.70.1.1-7.2004 |
| [24] | Schwartz T, Kohnen W, Jansen B, et al. Detection of antibiotic-resistant bacteria and their resistance genes in wastewater, surface water, and drinking water biofilms[J]. FEMS Microbiology Ecology, 2003, 43(3): 325-335. DOI:10.1111/j.1574-6941.2003.tb01073.x |
| [25] | Stoll C, Sidhu J P S, Tiehm A, et al. Prevalence of clinically relevant antibiotic resistance genes in surface water samples collected from Germany and Australia[J]. Environmental Science & Technology, 2012, 46(17): 9716-9726. |
| [26] | Zhang Y, Chen L J, Sun R H, et al. Effect of wastewater disposal on the bacterial and archaeal community of sea sediment in an industrial area in China[J]. FEMS Microbiology Ecology, 2014, 88(2): 320-332. DOI:10.1111/1574-6941.12298 |
| [27] |
李奥林, 陈吕军, 张衍, 等. 抗生素抗性基因在两级废水处理系统中的分布和去除[J]. 环境科学, 2018, 39(10): 4593-4600. Li A L, Chen L J, Zhang Y, et al. Distribution and removal of antibiotic resistance genes in two sequential wastewater treatment plants[J]. Environmental Science, 2018, 39(10): 4593-4600. |
| [28] |
张衍, 陈吕军, 谢辉, 等. 两座污水处理系统中细胞态和游离态抗生素抗性基因的丰度特征[J]. 环境科学, 2017, 38(9): 3823-3830. Zhang Y, Chen L J, Xie H, et al. Abundance of cell-associated and cell-free antibiotic resistance genes in two wastewater treatment systems[J]. Environmental Science, 2017, 38(9): 3823-3830. |
| [29] | Chen J Y, Su Z G, Dai T J, et al. Occurrence and distribution of antibiotic resistance genes in the sediments of the East China Sea bays[J]. Journal of Environmental Sciences, 2019, 81(7): 156-167. |
| [30] | Zhang X M, Yue X P, Liu Z Q, et al. Impacts of sludge retention time on sludge characteristics and membrane fouling in a submerged anaerobic-oxic membrane bioreactor[J]. Applied Microbiology and Biotechnology, 2015, 99(11): 4893-4903. DOI:10.1007/s00253-015-6383-x |
| [31] | Li B, Qiu Y, Li J, et al. Removal of antibiotic resistance genes in four full-scale membrane bioreactors[J]. Science of the Total Environment, 2019, 653: 112-119. DOI:10.1016/j.scitotenv.2018.10.305 |
| [32] | Giannakis S, Le M T T, Manuel Entenza J, et al. Solar photo-Fenton disinfection of 11 antibiotic-resistant bacteria (ARB) and elimination of representative AR genes. Evidence that antibiotic resistance does not imply resistance to oxidative treatment[J]. Water Research, 2018, 143: 334-345. DOI:10.1016/j.watres.2018.06.062 |
| [33] | Lambert P A. Bacterial resistance to antibiotics:modified target sites[J]. Advanced Drug Delivery Reviews, 2005, 57(10): 1471-1485. DOI:10.1016/j.addr.2005.04.003 |
| [34] | Huovinen P, Sundström L, Swedberg G, et al. Trimethoprim and sulfonamide resistance[J]. Antimicrobial Agents and Chemotherapy, 1995, 39(2): 279. DOI:10.1128/AAC.39.2.279 |
| [35] | Kumar A, Schweizer H P. Bacterial resistance to antibiotics:active efflux and reduced uptake[J]. Advanced Drug Delivery Reviews, 2005, 57(10): 1486-1513. DOI:10.1016/j.addr.2005.04.004 |
| [36] | Guo X Y, Yan Z, Zhang Y, et al. Behavior of antibiotic resistance genes under extremely high-level antibiotic selection pressures in pharmaceutical wastewater treatment plants[J]. Science of the Total Environment, 2018, 612: 119-128. DOI:10.1016/j.scitotenv.2017.08.229 |
| [37] |
焦雅楠.印染废水与生活污水中抗生素抗性基因分布差异及机理研究[D].杭州: 浙江大学, 2017. 10-15. Jiao Y N. The distribution characteristic and mechanism of antibiotic resistome between dyeing and domestic wastewater[D]. Hangzhou: Zhejiang University, 2017. 10-15. |
| [38] |
吴韵斐, 何义亮, 袁其懿, 等. 水源型水库抗生素抗性基因赋存特征研究[J]. 环境科学学报, 2019, 39(6): 1834-1841. Wu Y F, He Y L, Yuan Q Y, et al. Study on antibiotic resistance genes characteristics in a drinking water reservoir[J]. Acta Scientiae Circumstantiae, 2019, 39(6): 1834-1841. |
| [39] | Luo Y, Xu L, Rysz M, et al. Occurrence and transport of tetracycline, sulfonamide, quinolone, and macrolide antibiotics in the Haihe river basin, China[J]. Environmental Science & Technology, 2011, 45(5): 1827-1833. |
| [40] | Wen Q X, Yang L, Duan R, et al. Monitoring and evaluation of antibiotic resistance genes in four municipal wastewater treatment plants in Harbin, Northeast China[J]. Environmental Pollution, 2016, 212: 34-40. DOI:10.1016/j.envpol.2016.01.043 |
| [41] |
张宁, 李淼, 刘翔. 土壤中抗生素抗性基因的分布及迁移转化[J]. 中国环境科学, 2018, 38(7): 2609-2617. Zhang N, Li M, Liu X. Distribution and transformation of antibiotic resistance genes in Soil[J]. China Environmental Science, 2018, 38(7): 2609-2617. DOI:10.3969/j.issn.1000-6923.2018.07.029 |
| [42] | Wang Q, Wang P L, Yang Q X. Occurrence and diversity of antibiotic resistance in untreated hospital wastewater[J]. Science of the Total Environment, 2018, 621: 990-999. DOI:10.1016/j.scitotenv.2017.10.128 |
| [43] |
狄彩霞, 杨永青, 莎娜, 等. 历年生鲜乳中舒巴坦敏感β-内酰胺酶类药物检验考核及注意事项分析[J]. 畜牧与兽医, 2019, 51(1): 131-135. Di C X, Yang Y Q, Sha N, et al. Inspection and key points for testing of sulbactam sensitive β-lactamases in raw milk over the years[J]. Animal Husbandry & Veterinary Medicine, 2019, 51(1): 131-135. |
| [44] |
杜德才, 周书明, 沈爱宗, 等. 医院抗菌药物使用强度分析[J]. 中华医院感染学杂志, 2010, 20(6): 848-851. Du D C, Zhou S M, Shen A Z, et al. Antibacterial use density in a hospital[J]. Chinese Journal of Nosocomiology, 2010, 20(6): 848-851. |
| [45] |
张佳奇.湘江流域β内酰胺抗性基因污染特征与影响因素研究[D].天津: 天津大学, 2015. Zhang J Q. Research on pollution characteristics and impact factors on β-lactam ARGs in Xiangjiang river basin[D]. Tianjin: Tianjin University, 2015. |
 2019, Vol. 40
2019, Vol. 40


