随着人们对海洋经济产品需求的增加, 海水养殖面积扩大, 残余的饲料和代谢物随养殖尾水排入近海, 致使海水养殖成为近海污染的重要原因之一[1].鱼类饲料和排泄物积聚沉积物中, 使得这些沉积物有机质浓度较高, 营养物质积累较多, 特别是磷和氮化合物[2].这些有机物积累改变了沉积物的理化性质, 影响了生态过程, 从而改变了沉积物微生物群落的结构[3].养殖业的污染已经开始制约渔业生产的持续健康发展.近年来, 抗生素通常与饲料混合以治疗不同的养殖生物疾病.抗生素的使用改变了沉积物的微生物群落, 导致抗生素抗性病原体广泛传播[4], 可能培养出具有抗生素抗性的鱼类病原体[5], 甚至是人类病原体[6].众多研究表明, 海水养殖也显著降低了沉积物中的物种多样性, 表明沉积物微生物生态系统的基本生物过程受到了破坏[7].
微生物在物质循环和能量流动中发挥着基础作用, 因此, 微生物群落结构和多样性有可能作为评价海水养殖生态系统健康和稳定的重要指标[8].细菌等微生物群落结构和多样性通常对环境变动较为敏感, 与环境的相关性较大[9].在近海水产养殖中, 沉积物微生物依赖于多种环境条件, 包括沉积物基质、营养盐、盐度和pH[10, 11].沉积物细菌群落多样性与人类活动的影响密切相关, 例如富营养化的加剧, 化学物质的污染, 浮游动植物的变化以及挖掘开垦等行为[12].因此, 海水养殖对细菌等微生物群落结构的影响显著, 其可能是与海洋环境污染问题有关的重要因素.
养殖场尾水直排河道的环境影响可通过沉积物中细菌种类和群落结构变化反映.本研究通过Illumina MiSeq测序技术和磷脂脂肪酸(PLFA)分析养殖场区进出水河道不同位点沉积物的微生物群落结构与功能, 通过比较进、出水渠沉积物中的各类细菌组成来探究海水养殖尾水对河道自净能力及近海环境造成的影响, 以期为海水养殖污染的防控提供科学依据.
1 材料与方法 1.1 实验设计与采样本实验样品于2016年7月采自浙江省宁波市鄞州养殖场区(N 29.72° E 121.86°).养殖场区靠近象山港, 主要进行三疣梭子蟹和南美白对虾的养殖.养殖场区所有养殖塘的海水来自同一条河道(进水渠), 各养殖塘分布在另一条河道(出水渠)两侧, 并将含有大量饵料、抗菌物质和代谢产物的养殖废水直接排入出水渠中.本研究选取了养殖场区1个进水渠位点、出水口和2个出水渠位点(相距约1.5 km)进行沉积物细菌群落结构和多样性的研究, 每个位点设置3个采样点作为平行.采集各采样点表层5 cm的沉积物, 2 h内带回实验室, 保存于-80℃.
1.2 沉积物理化分析沉积物理化指标按照文献[13]中所阐述的方法进行测定:总有机碳(TOC)用重铬酸钾容量法;氨氮(NH4+)用2 mol·L-1 KCl浸提-靛酚蓝比色法;硝氮(NO3-)用酚二磺酸比色法.
1.3 磷脂脂肪酸(PLFA)分析将土样带回实验室进行PLFA的提取[14]:称取3 g冷冻干燥土样, 加入3.2 mL柠檬酸缓冲液, 4 mL氯仿, 8 mL甲醇, 水平振荡2 h(250 r·min-1), 离心(10 min, 1 500 r·min-1), 转移至大试管, 剩余土样加入之前一半剂量的试剂, 然后重复之前步骤, 往大试管中加入4.8 mL柠檬酸缓冲液和6 mL氯仿, 加盖, 振荡1 min, 间隔放气, 4℃隔夜遮光静置, 将下层氯仿相转移至中号试管, 氮气吹干, 样品上柱(提前用5 mL氯仿洗柱, 样品3 mL氯仿2次溶解), 加10 mL氯仿洗中性酯, 10 mL丙酮洗糖酯, 倒掉底液, 加8 mL甲醇, 洗磷脂入小号试管, 氮气吹干, 加200 μL内标(涡旋振荡), 氮气吹干, 1 mL甲醇甲苯混合液溶解磷脂, 加1 mL 0.2 mol·L-1氢氧化钾甲醇溶液, 37℃水浴15 min, 加2 mL正己烷氯仿混合液、0.3 mL 1 mol·L-1醋酸和2 mL去离子水, 振荡分离10 min, 离心(3 000 r·min-1, 5 min), 转移上层有机相至小号试管, 加2 mL正己烷氯仿混合液至原试管, 重复之前振荡离心步骤, 转移上层有机相, 氮气吹干, 加1 mL正己烷溶解转移至GC瓶.
提取完成后, 用气相色谱法测定(Gas chromatography), 用正己烷溶解磷脂脂肪酸甲酯, 转移至GC内插管, 氮气吹干, 加60 μL正己烷上机.
1.4 DNA的提取, PCR扩增及测序所采泥样用FastDNA SPIN Kit for Soil试剂盒(MP Biomedicals)提取总DNA, 分光光度计(NanoDrop, ND-1000)测定浓度和纯度后, 用16S rRNA基因V3-V4区引物341F(5′-CCTAYGGGRBG CASCAG-3′)和806R(5′-GGACTACNNGGGTATCT AAT-3′)扩增基因片段, 产物经试剂盒(Takara)纯化后测定浓度, 每个样品取等量的PCR产物混合, 并用MiSeq双末端测序平台测序(Illumina, USA).
1.5 Illumina数据处理测序数据用FLASH软件拼接二端序列[15], 利用Qiime流程进行分析, 去嵌合体和质控.步骤概括为, 剪接掉3个以上的连续且质控分小于20的碱基, 保留大于300 bp的长序列, 然后用UCHIME去除嵌合体, 用UCLUST在97%的相似性水平聚类为分类操作单元(OTUs).以每个OTU中丰度和覆盖度最高的序列为代表序列, 用PyNAST在Greengenes数据库中比对, 获得OTUs的物种分类信息, 去除不属于细菌的古菌和叶绿体序列以及在所有样品中只出现一次的序列.每个样品随机选取17100条序列(最低测序深度)进行后续分析.
1.6 统计分析基于Bray-Curits距离使用非度量多维尺度分析不同位点底泥沉积物细菌群落间的差异.用冗余分析表征环境因子与细菌群落变异的相关性.门水平的分析细菌群落时将不同处理平均相对丰度小于1%的均归为其它(others), 纲水平的分析细菌群落时将不同处理平均相对丰度小于1.5%的均归为其它(others).基于测序数据分别计算纲水平的香农多样性指数, 变形菌门和酸杆菌门相对丰度的比值, 寡营养型细菌和富营养型细菌的比值(酸杆菌门与疣微菌门序列之和与放线菌门、β-变形菌纲和厚壁菌门序列之和的比值), 革兰氏阳性细菌的相对丰度(放线菌门和厚壁菌门的相对丰度之和), 革兰氏阴性细菌的相对丰度(存在Hsp60插入的细菌门[16]相对丰度之和)及其两者的比值[17];基于PLFA数据分别计算细菌香农多样性指数, 真菌和细菌的比值, 革兰氏阳性细菌的比例, 革兰氏阴性细菌的比例及其两者的比值.
2 结果与分析 2.1 沉积物理化性质理化分析结果(表 1)显示进水渠中有机碳和无机氮的含量均要显著低于出水口和出水渠(P < 0.05).出水渠与出水口相比, 氨氮含量降低, 反之硝氮含量增高, 有机碳含量也增加.出水渠两位点间各理化参数无显著差异.
|
|
表 1 独立样本t检验分析养殖场区各位点理化参数的差异1)/mg·kg-1 Table 1 Independent sample t test analysis of the difference of environmental parameters in the culture area/mg·kg-1 |
2.2 沉积物中微生物的PLFA组成
不同位点沉积物中微生物磷脂脂肪酸的分析结果显示, 去除丰度低于1%且只在个别样品中出现的磷脂脂肪酸后, 剩余的25类磷脂脂肪酸均在不同位点的沉积物中有一定含量(图 1).其中PLFA 16:0为细菌常见磷脂脂肪酸, 在所有位点样品中均含量最高.其次为PLFAs 18:1ω7c和16:1ω7c, 分别属于Ⅱ型和Ⅰ型甲烷氧化细菌的特征磷脂脂肪酸[14]. PLFA 18:2ω6c在进水渠沉积物中含量并不高, 但是在出水渠及出水口沉积物中大幅度增加, 其在出水渠沉积物中的含量达到进水渠中的40倍.沉积物中含有的PLFA总量在不同位点也有显著差别(图 2), 进水渠最少, 出水口约为进水渠的两倍, 两个出水渠位点的PLFA总量(以鲜土计)则超过进水渠的6倍, 达到18 μg·g-1.
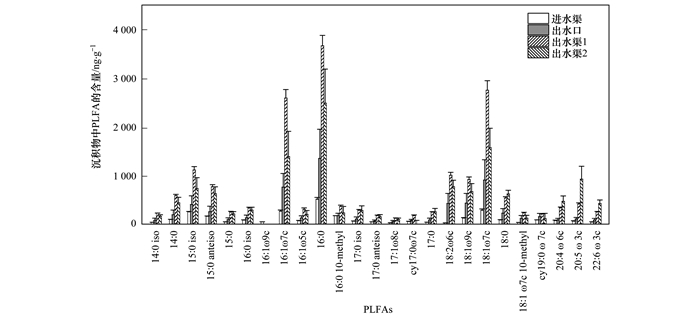
|
图 1 不同位点的PLFAs组成 Fig. 1 Concentrations of individual phospholipid fatty acids (PLFAs) at different sites |
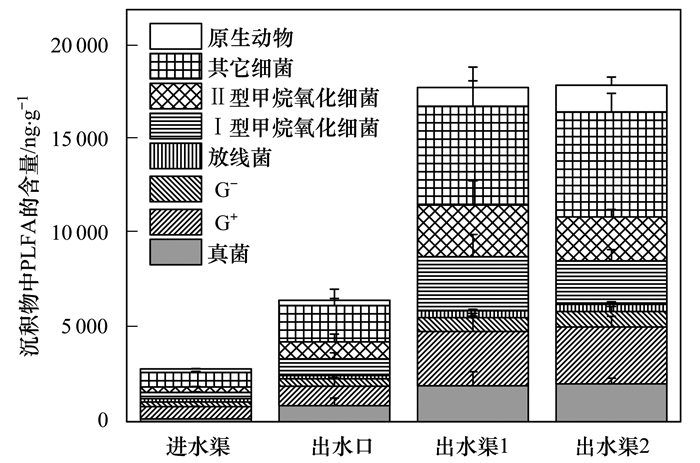
|
图 2 不同位点PLFAs总量及各类微生物的组成 Fig. 2 Total phospholipid fatty acids (PLFAs) and microbial compositions at different sites |
根据文献[18~20]对PLFA的生物表征, 将本实验检测到的PLFA种类进行以下分类:Ⅰ型甲烷氧化细菌(Meth Ⅰ):16:1ω7c和16:1ω5c;Ⅱ型甲烷氧化细菌(Meth Ⅱ):18:1ω7c;放线菌(Actinomycetes):16:0 10-methyl;真菌(Fungi):18:2ω6c和18:1ω9c;革兰氏阳性菌(G+):14:0 iso、15:0 iso、15:0 anteiso、16:0 iso、17:0 iso和17:0 anteiso;除甲烷氧化菌外的革兰氏阴性菌(G-):16:1ω9c、cy17:0ω7c、cy19:0ω7c、18:1ω7c 10-methyl和17:1ω8c;其它通用的细菌(other Bacteria):14:0、15:0、16:0、17:0和18:0;原生动物(protozoa):20:4ω6c、20:5ω3c和22:6ω3c.结果如图 2所示, 真菌的PLFAs在进水渠沉积物中仅占5.35%, 但在出水口和出水渠的沉积物中已超过10%, 是数量增长最多的微生物类群.而放线菌的PLFA含量在进水渠和出水口的沉积物中无显著差异, 在出水渠沉积物中的含量也仅为进水渠的2倍, 使其在出水渠沉积物中的相对丰度比进水渠中降低.
2.3 沉积物细菌群落组成和结构分析对测序的序列进行随机抽样, 以抽到的序列数与其所能代表的数目构建稀释性曲线(图 3).从图 3中可以看出, 4个位点沉积物样品的稀释曲线在0.97相似性水平下趋于平坦但仍未达到饱和, 说明4个位点沉积物样品获取了绝大多数样本信息, 基本能够反映河道沉积物的微生物群落组成, 但还有必要在后续研究中增加测序深度.
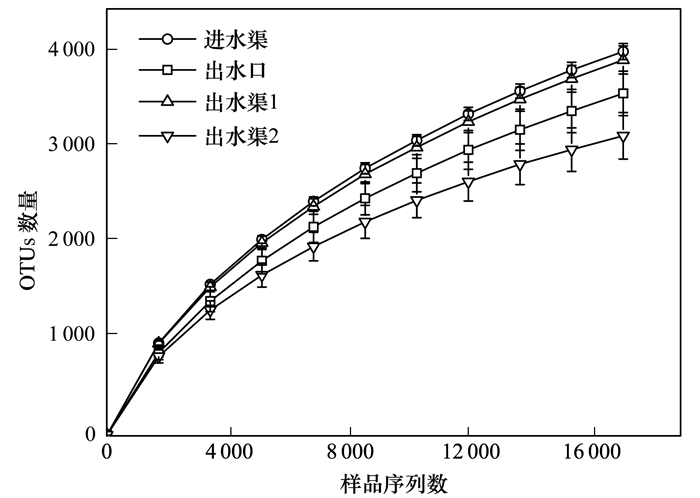
|
图 3 OTUs稀释曲线 Fig. 3 Rarefaction curves of OTUs |
沉积物中细菌类群丰富, 主要包括变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteria)、厚壁菌门(Firmicutes)、芽单胞菌门(Gemmatimonadetes)、蓝细菌(Cyanobacteria)、硝化螺旋菌门(Nitrospirae)、螺旋原虫(Spirochaetes)和WS3等[图 4(a)].其中变形菌门相对丰度最高, 在各位点均占一半以上.进水渠沉积物中的拟杆菌门相对丰度显著低于出水口和出水渠(P < 0.05), 而酸杆菌门的相对丰度则显著高于出水口和出水渠(P < 0.05).分析拟杆菌门的群落组成, 其中黄杆菌纲、拟杆菌纲和纤维粘网菌(Cytophagia)是其优势菌群[图 4(d)].黄杆菌纲和拟杆菌纲在进水渠的平均相对丰度要低于出水口和出水渠.放线菌在进水渠和出水口的沉积物中无显著差异, 在出水渠中则显著降低(P < 0.05), 这与PLFA的分析结果相一致.放线菌纲和酸微菌纲是放线菌门的优势菌群[图 4(c)].放线菌纲(Actinobacteria)在进水渠、出水口和出水渠中的平均相对丰度分别为0.51%、2.24%和1.45%, 且均属于放线菌目(Actinomycetales), 包括常见的单孢菌科(Micromonosporaceae)、假诺卡氏科(Pseudonocardiaceae)、诺卡氏菌科(Nocardiaceae)、链霉菌科(Streptomycetaceae)和链孢囊菌科(Streptosporangiaceae)等.酸微菌纲在进水渠中的相对丰度明显高于其余3个位点(P < 0.05).进水渠沉积物中, 硝化螺旋菌门(Nitrospirae)的相对丰度为2.83%±0.30%, 在出水口和出水渠中显著降低(P < 0.05), 在出水渠沉积物中的平均相对丰度低于0.5%.
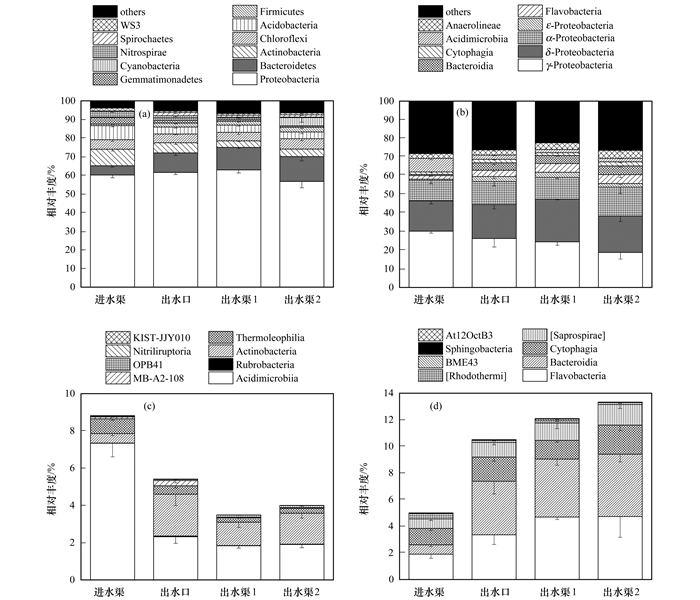
|
(a)门水平, (b)纲水平, (c)放线菌门, (d)拟杆菌门 图 4 各位点主要优势菌群在门水平和纲水平的相对丰度及放线菌门和拟杆菌门的群落组成 Fig. 4 Relative abundances of the dominant communities at phylum and class levels at different sites, and the community compositions of Actinobacteria and Bacteroidetes |
细菌群落组成和结构在纲水平上的进一步分析, 去除平均丰度小于1.5%的纲后, 得到主要细菌类群为α-变形菌纲(α-Proteobacteria)、γ-变形菌纲(γ-Proteobacteria)、δ-变形菌纲(δ-Proteobacteria)、ε-变形菌纲(ε-Proteobacteria)、黄杆菌纲(Flavobacteria)、拟杆菌纲(Bacteroidia)、酸微菌纲(Acidimicrobiia)、厌氧绳菌纲(Anaerolineae)和噬纤维菌纲(Cytophagia)[图 4(b)].其中拟杆菌纲在出水渠中的相对丰度显著高于进水渠(P < 0.05), 厌氧绳菌纲、黄杆菌纲和δ-变形菌纲的相对丰度也略有增高.而γ-变形菌纲在出水渠中的相对丰度则有所降低.另外, 厌氧的梭菌纲(Clostridia)在进水渠、出水口和出水渠中的平均相对丰度分别为0.46%、1.68%和1.94%.硝化螺菌纲(Nitrospira)在进水渠中的平均丰度为2.83%, 在出水口和出水渠中则分别降为1.20%和0.38%.其中具有好氧氨氧化作用的硝化螺旋菌属(Nitrospira)在进水渠沉积物中占0.27%±0.07%, 在出水口和出水渠中的相对丰度也显著降低(P < 0.05), 出水口为0.07%±0.02%, 出水渠中的平均相对丰度则不足0.02%.
2.4 沉积物细菌群落分布及其与环境因子的关系基于沉积物样品的细菌OTUs的丰度以及纲和门水平的分析数据进行了非度量多维尺度(NMDS)分析, 结果表明细菌群落按进水和出水聚类(图 5).出水口和出水渠沉积物中的细菌群落组成在OTU、纲和门水平都比较相似, 与进水渠的有显著差异(P < 0.05).
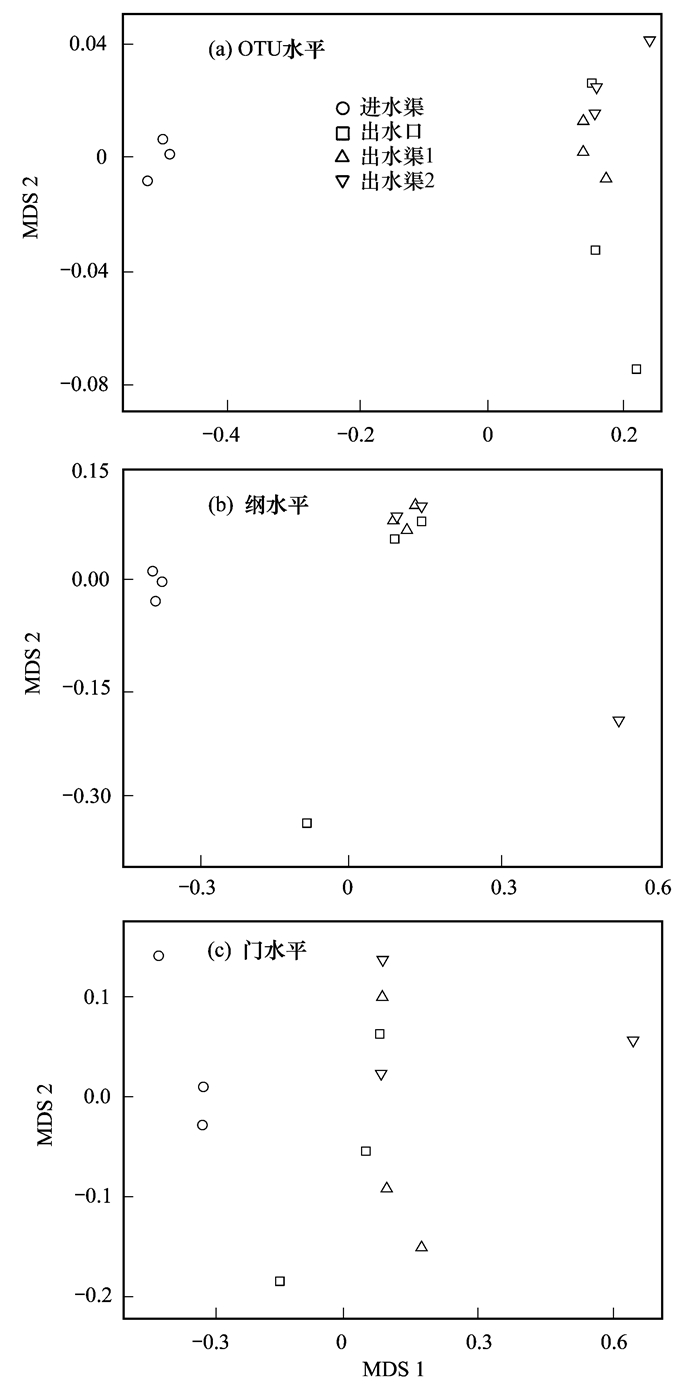
|
图 5 基于细菌OTU的丰度、纲水平和门水平的非度量多维尺度分析沉积物细菌群落分布 Fig. 5 Non-metric multidimensional scaling analysis of bacterial community composition based on the relative abundance of bacterial OTU-, class-, and phylum-level metabarcoding data |
比较不同位点的细菌群落多样性, 包括Chao1、物种多样性(observed species)、香农多样性(Shannon diversity)和系统发育多样性(phylogenetic diversity)(表 2), 可以看出, 进水渠沉积物的细菌多样性均较高.尽管如此, 出水渠沉积物中细菌的多样性也较高, 且不同位点也存在差异.
|
|
表 2 沉积物细菌群落多样性指数1) Table 2 Diversity indices of sediment bacterial communities |
分析无机氮和有机碳含量对不同位点底泥沉积物细菌群落结构变化的影响, 将铵态氮、硝态氮和总有机碳与高通量测序结果进行了冗余分析(RDA, 图 6).第一排序轴RDA 1解释量为22.3%, 第二排序轴RDA 2解释量为20.2%.冗余分析第一排序RDA 1的主要环境因子为总有机碳(正相关, r=0.58), 冗余分析第二排序RDA 2的主要环境因子为铵态氮(负相关, r=-0.85).这些因子共同解释了44.8%的群落变异.
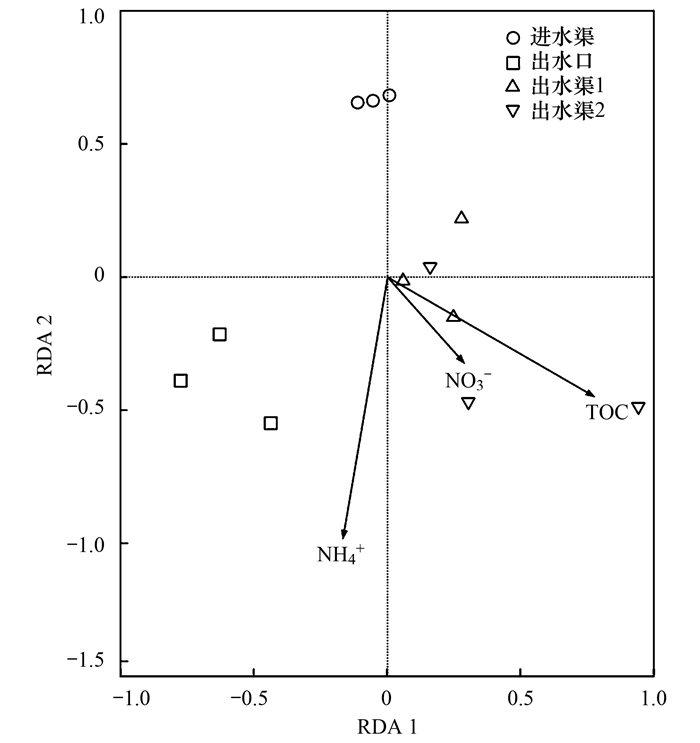
|
图 6 沉积物细菌群落及环境因子RDA双向排序 Fig. 6 Redundancy analysis (RDA) ordination biplot showing the driving factors of the bacterial community variations |
基于细菌PLFA数据和16S rRNA基因测序数据进行相关性分析, 结果显示两者相关性较好(图 7).革兰氏阴性细菌的百分含量(Gram neg%)尽管在PLFA数据和16S rRNA基因测序数据中不同, 但两者显著相关(R2=0.50, P =0.006 2).革兰氏阳性细菌的百分含量(Gram pos%)在PLFA数据和16S rRNA基因测序数据中的相关性并未达到显著水平, 但两种数据中革兰氏阳性细菌与阴性细菌的比值(Gram pos:neg)则是相关性最好的(R2=0.80, P < 0.001).细菌PLFA中真菌与细菌的比值(F:B ratio)与16S rRNA基因测序数据中革兰氏阳性细菌与阴性细菌的比值(Gram pos:neg-DNA)也存在显著的相关性(R2=0.64, P =0.001).细菌PLFA中真菌与细菌的比值、革兰氏阳性细菌与阴性细菌的比值(Gram pos:neg-PLFA)可以指示微生物的生长速率, 它们与16S rRNA基因测序数据中指示营养状况的寡营养与富营养的比值(Oligo:Copio)的相关性未达到显著水平.基于细菌PLFA与16S rRNA基因测序数据在纲水平上的香农多样性指数H′(Class H′)或均匀度指数E均显著相关, R2值为0.48(P=0.007), 而与门水平上的香农多样性指数H′或均匀度指数E均无相关性. 16S rRNA基因测序数据中变形细菌与酸杆菌门的比值(Proteo:Acido)与沉积物的营养状态有关[21], 它与PLFA中革兰氏阳性细菌与阴性细菌的比值、真菌与细菌的比值都呈显著相关, R2值分别为0.60(P=0.002)和0.43(P =0.012).
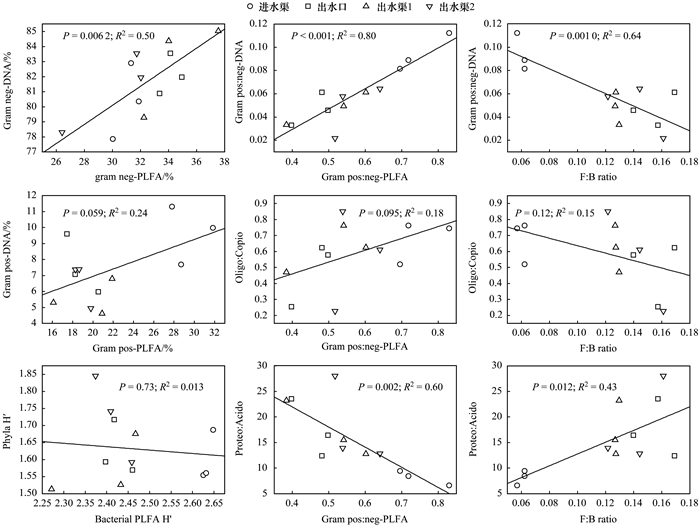
|
图 7 基于细菌PLFA(x轴)和16S rRNA基因测序数据(y轴)的相关性分析 Fig. 7 Correlations among metrics based on PLFA (x axes) and 16S rRNA gene metabarcoding (y axes) |
沉积物是河道水体生物自净的主要生物栖息地, 同时又是有机质沉淀等水体物理净化过程中污染物质的受体.有机碳和无机氮是三疣梭子蟹和南美白对虾等海水养殖尾水中主要的污染物质, 养殖尾水的长期排放, 导致养殖场区排水河道沉积物中有机碳和无机氮的含量显著增高(表 1), 影响了沉积物中微生物群落的分布(图 6).与此同时, 沉积物与河道水体交界面存在大量的化学物质交换, 尤其是无机氮, 沉积物中的铵态氮会重新溶到水体中[22], 导致排入近海的养殖尾水含氮量增加.铵态氮在出水口沉积物中含量最高, 出水渠中含量有所降低, 硝态氮则相反, 表明出水渠河道中存在铵态氮向硝态氮的转化.沉积物中有机碳含量沿尾水排放距离呈增加趋势, 这可能是尾水中有机物质在排水河道不断物理沉降的结果.有机物的好氧降解消耗大量氧气, 耗氧速率大于水体复氧速率.若氧气含量低于临界点, 河道将会丧失自净能力.出水渠中厌氧的梭菌纲和厌氧绳菌纲的相对丰度增加说明沉积物氧气含量低, 可能已局部厌氧.
3.2 养殖尾水对沉积物细菌群落组成的影响在受养殖尾水直排影响的沉积物中, 变形细菌门、拟杆菌门和放线菌门是主要的细菌门类, 这和其它受养殖尾水影响沉积物中细菌群落的研究结果相类似[23].变形菌门和拟杆菌门是海洋异养细菌中最大的两个门类[24].本研究沉积物中变形细菌以δ-、γ-和α-变形细菌纲为主[图 4(b)], 这与戴欣等[25]对南沙海区沉积物中细菌群落的研究结果相类似.变形菌门是革兰氏阴性细菌的主要门类, 在活性污泥、废水处理系统以及沉积物中含量丰富, 在污水处理系统中碳络合物的降解和氮去除等过程中起关键作用[26], 在水生生态系统中也参与不同的生物地球化学过程(例如:碳、氮以及硫的循环)[27, 28].放线菌门不仅因为他们的分类学和生态学意义还因它们独特的代谢产物和酶类, 在海洋微生物研究中具有重要的价值.海洋放线菌在纤维素水解[29]、蛋白水解[30]、淀粉分解[31]、脂肪分解[30]、几丁质分解[31]和溶磷[32]等方面的活性均有报道.此外众多研究表明放线菌对不同的病原菌存在抑菌特性[33], 是潜在的益生菌, 对有机化合物的降解和循环也有贡献[20].尽管磷脂脂肪酸的分析表明出水渠沉积物中放线菌的数量比进水渠有所增加(图 2), 但其相对丰度在出水渠沉积物中显著降低[图 4(a)].这表明出水渠沉积物受养殖尾水中污染物的影响, 抗菌能力下降, 其它细菌迅速增加.细菌群落结构分析显示, 取而代之的是拟杆菌门和蓝细菌等相对丰度增高.其中, 拟杆菌门的相对丰度受养殖尾水排放的影响最为显著, 主要包括黄杆菌纲(Flavobacteria)、鞘脂杆菌纲(Sphingobacteria)和噬纤维菌纲(Cytophagia)等[图 4(b)、4(d)], 是一类严格的化能异养细菌类群, 能够降解复杂有机物, 是有机物质的主要降解者之一[34], 在海洋碳循环中有着重要地位.如黄杆菌纲在沉积物有机物质降解初始阶段中起着非常重要的作用, 是生态系统同化能力的重要决定因素[35].拟杆菌门中的很多种类是生活在人或者动物的肠道中, 专性厌氧, 是粪便中主要的微生物种类.研究表明, 高密度无循环的养殖相比循环水养殖, 沉积物中拟杆菌的相对丰度更高[36].在鲑鱼、金头鲷等鱼类的网箱中也发现了拟杆菌门相对丰度的增长[37].养殖尾水作为养殖动物排泄物的受纳水体, 在出水渠中排泄物沉降形成新的沉积物, 增加了拟杆菌门在沉积物中的相对丰度, 其中的黄杆菌属(Flavobacterium, 0.05%)、弓形菌属(Arcobacter, 0.08%)均是潜在的细菌类病原菌[36].此外, 放线菌门的分枝杆菌属(Mycobacterium)也属于潜在病原菌[38], 在出水渠中的相对丰度也显著增高(P < 0.05).表明养殖尾水直排会增加沉积物中潜在病原菌的丰度, 对近海养殖生物存在较高的致病风险, 进而影响近海养殖和池塘海水养殖的持续健康发展.
3.3 沉积物细菌生物量的变化及其生态意义养殖尾水中含有残余饵料和养殖动物排泄物等, 营养丰富, 显著增加了沉积物中微生物的生物量(图 2).由PLFA测定得到的结果显示, 出水口沉积物中微生物总PLFA为进水渠沉积物中的2倍多, 出水渠沉积物中的微生物总PLFA均为进水渠的6倍左右.在翟海佳等对对虾养殖塘沉积物微生物群落的研究中也发现, 沉积物PLFA组成以饱和脂肪酸、支链脂肪酸和单不饱和脂肪酸为主, 多不饱和脂肪酸与环丙烷脂肪酸含量较少, 微生物总生物量、细菌生物量和真菌生物量伴随着养殖的进行都明显增加[39].相比于细菌总生物量的增加, 放线菌生物量仅增加了2倍左右, 以致放线菌门相对丰度反而降低[图 4(a)].真菌细菌比是指示微生物群落结构的良好指标[17], 在森林土和牧草土中可以反映土壤环境中营养物质的可利用情况, 与革兰氏阳性与阴性细菌的比值负相关[40], 这与本研究的结果相类似.但是在森林土和牧草土研究中发现, 真菌细菌比与寡营养型细菌和富营养型的细菌比值呈正相关[41].这是因为氮肥施加降低了微生物的生物量, 高氮环境可能会使土壤中可利用碳成为微生物生长繁殖的限制因素[42].但在养殖尾水排放河道里, 尽管无机氮含量较高, 有机碳的沉淀同时为微生物的生长提供了足够的碳源, 微生物生物量增加.由于真菌个体比细菌大得多, 真菌细菌比也随之增加(图 7).因此, 在沉积物中其与寡营养型细菌和富营养型的细菌比值并无显著相关性.
基于测序数据计算的参数(变形菌门和酸杆菌门的比值、寡营养型细菌和富营养型细菌的比值、革兰氏阳性细菌的相对丰度、革兰氏阴性细菌的相对丰度及其两者的比值)可以用以反映细菌潜在的生长速度、碳使用率和营养需求.变形菌门与酸杆菌门的比值可以反映沉积物的养分状况, 比值越高, 沉积物中的营养水平就越高[21], 这与本研究的结果相似.本研究中, 受养殖尾水影响的沉积物中的变形菌门与酸杆菌门的比值显著高于进水渠(图 7).基于细菌PLFA数据和16S rRNA基因测序数据的革兰氏阳性细菌与阴性细菌的比值的相关性极显著, 这在一定程度上证明了可以将PLFA作为革兰氏阳性细菌和阴性细菌相对丰度的指标.尽管多种微生物中含有同样的PLFA, 采用细菌PLFA来计算多样性指标存在很多争议.在本研究中, 细菌PLFA多样性和均匀度与基因测序数据纲水平的多样性和均匀度显著相关, 表明基于PLFA的多样性指标可以在较广泛的分类水平上表征细菌的多样性.
本研究结果表明PLFA和16S rRNA基因的测序数据具有广泛的可比性, 两种方法对不同位点的细菌群落呈现了相似的差异性(图 5), 两者在不同的微生物指标方面也有较好的相关性(图 7).
3.4 沉积物细菌群落生态功能氨氮与沉积物细菌群落结构的相关性最大(图 6), 其对细菌群落的影响最明显.硝化螺旋菌门是海洋环境中主要氨氧化细菌[43], 出水渠沉积物中的平均相对丰度显著低于进水渠, 这可能是由于沉积物氧气含量低, 甚至局部厌氧, 硝化螺旋菌属于好氧氨氧化细菌, 缺少氧气难以生长繁殖.细菌群落分析结果也显示出水渠中红螺菌科(Rhodospirillaceae)等好氧反硝化菌的相对丰度显著降低, 厌氧的梭菌纲、兼性厌氧的绿弯菌门和厌氧绳菌纲的相对丰度增加, 进一步说明出水渠沉积物的环境条件更利于厌氧微生物的生长.绿弯菌门是一种可以在厌氧条件下进行光合作用的好氧兼性细菌, 在海洋、潮间带和淡水表面以及地下沉积物中都很丰富.厌氧绳菌纲既能够进行糖和氨基酸的厌氧发酵, 也能够进行有氧糖呼吸, 其生理的灵活性使得其成为在厌氧/有氧变化的环境中的重要竞争者[44].
环境因子对沉积物的细菌群落产生显著影响[45], 反之, 微生物能够综合地反映环境的污染情况[46].由于海水养殖通常养殖密度较高, 并且投入大量饵料和抗菌药物等物质, 使养殖水体和排水渠沉积物营养盐和有机质含量升高, 超过河道的自净能力, 影响沉积物细菌的群落结构及其生态功能, 进而影响近海环境和渔业生产的健康发展.
4 结论(1) 大量有机质的不断沉降和分解使排水渠沉积物趋于厌氧, 氨氧化微生物相对丰度低, 河道自净能力弱.
(2) 养殖尾水直排影响了沉积物中的细菌群落组成, 生物量和相对丰度均发生显著变化.抑菌特性的放线菌门相对丰度降低, 而能够降解复杂有机物的拟杆菌门相对丰度升高, 说明沉积物有机质和潜在病原菌含量升高, 可能威胁近海的水产养殖.
(3) 磷脂脂肪酸分析和16S rRNA基因的测序数据具有广泛的可比性, 不同微生物类群的比值能较好地反映环境中各营养成分的变化, 对环境具有一定的指示作用.
| [1] | Cao L, Wang W M, Yang Y, et al. Environmental impact of aquaculture and countermeasures to aquaculture pollution in China[J]. Environmental Science and Pollution Research-International, 2007, 14(7): 452-462. DOI:10.1065/espr2007.05.426 |
| [2] | Karakassis I, Tsapakis M, Hatziyanni E. Seasonal variability in sediment profiles beneath fish farm cages in the Mediterranean[J]. Marine Ecology Progress Series, 1998, 162: 243-252. DOI:10.3354/meps162243 |
| [3] | Hornick K M, Buschmann A H. Insights into the diversity and metabolic function of bacterial communities in sediments from Chilean salmon aquaculture sites[J]. Annals of Microbiology, 2018, 68(2): 63-77. DOI:10.1007/s13213-017-1317-8 |
| [4] | Xiong W G, Sun Y X, Zhang T, et al. Antibiotics, antibiotic resistance genes, and bacterial community composition in fresh water aquaculture environment in China[J]. Microbial Ecology, 2015, 70(2): 425-432. |
| [5] | Pulkkinen K, Suomalainen L R, Read A F, et al. Intensive fish farming and the evolution of pathogen virulence: the case of columnaris disease in Finland[J]. Proceedings of the Royal Society B, 2010, 277(1681): 593-600. DOI:10.1098/rspb.2009.1659 |
| [6] | Høi L, Larsen J L, Dalsgaard I, et al. Occurrence of Vibrio vulnificus biotypes in Danish marine environments[J]. Applied and Environmental Microbiology, 1998, 64(1): 7-13. |
| [7] | Bissett L. Reducing the risk of acquiring antimicrobial-resistant bacteria[J]. British Journal of Nursing, 2006, 15(2): 68-71. DOI:10.12968/bjon.2006.15.2.20365 |
| [8] | van der Gucht K, Cottenie K, Muylaert K, et al. The power of species sorting: local factors drive bacterial community composition over a wide range of spatial scales[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(51): 20404-20409. DOI:10.1073/pnas.0707200104 |
| [9] | Langenheder S, Ragnarsson H. The role of environmental and spatial factors for the composition of aquatic bacterial communities[J]. Ecology, 2007, 88(9): 2154-2161. DOI:10.1890/06-2098.1 |
| [10] | Dale N G. Bacteria in intertidal sediments: factors related to their distribution[J]. Limnology and Oceanography, 1974, 19(3): 509-518. DOI:10.4319/lo.1974.19.3.0509 |
| [11] | Jackson C R, Vallaire S C. Effects of salinity and nutrients on microbial assemblages in Louisiana wetland sediments[J]. Wetlands, 2009, 29(1): 277-287. |
| [12] | Li J L, Li F C, Yu S X, et al. Impacts of mariculture on the diversity of bacterial communities within intertidal sediments in the northeast of China[J]. Microbial Ecology, 2013, 66(4): 861-870. DOI:10.1007/s00248-013-0272-6 |
| [13] | 鲍士旦. 土壤农化分析[M]. 北京: 中国农业出版社, 1981: 30-35, 50-52, 54-56. |
| [14] | Qiu Q F, Noll M, Abraham W R, et al. Applying stable isotope probing of phospholipid fatty acids and rRNA in a Chinese rice field to study activity and composition of the methanotrophic bacterial communities in situ[J]. The ISME Journal, 2008, 2(6): 602-614. DOI:10.1038/ismej.2008.34 |
| [15] | Magoč T, Salzberg S L. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [16] | Gupta R S. Origin of diderm (Gram-negative) bacteria: antibiotic selection pressure rather than endosymbiosis likely led to the evolution of bacterial cells with two membranes[J]. Antonie van Leeuwenhoek, 2011, 100(2): 171-182. DOI:10.1007/s10482-011-9616-8 |
| [17] | Orwin K H, Dickie I A, Holdaway R, et al. A comparison of the ability of PLFA and 16S rRNA gene metabarcoding to resolve soil community change and predict ecosystem functions[J]. Soil Biology and Biochemistry, 2018, 117: 27-35. DOI:10.1016/j.soilbio.2017.10.036 |
| [18] | Pisano M A, Sommer M J, Taras L. Bioactivity of chitinolytic actinomycetes of marine origin[J]. Applied Microbiology and Biotechnology, 1992, 36(4): 553-555. |
| [19] | Sahu M K, Sivakumar K, Thangaradjou T, et al. Phosphate solubilizing actinomycetes in the estuarine environment: an inventory[J]. Journal of Environmental Biology, 2007, 28(4): 795-798. |
| [20] | Weyland H. Actinomycetes in north sea and Atlantic ocean sediments[J]. Nature, 1969, 223(5208): 858. |
| [21] | Smit E, Leeflang P, Gommans S, et al. Diversity and seasonal fluctuations of the dominant members of the bacterial soil community in a wheat field as determined by cultivation and molecular methods[J]. Applied and Environmental Microbiology, 2001, 67(5): 2284-2291. DOI:10.1128/AEM.67.5.2284-2291.2001 |
| [22] | Cornwell J C, Owens M S, Boynton W R, et al. Sediment-water nitrogen exchange along the Potomac river estuarine salinity gradient[J]. Journal of Coastal Research, 2016, 32(4): 776-787. |
| [23] | Santander-de Leon S M S, Reichardt W, Peralta-Milan S, et al. Bacterial community composition of sediments from a milkfish Chanos chanos Forsskål farm[J]. Aquaculture Research, 2016, 47(8): 2569-2581. DOI:10.1111/are.12705 |
| [24] | Kirchman D L, Cottrell M T, Lovejoy C. The structure of bacterial communities in the western Arctic ocean as revealed by pyrosequencing of 16S rRNA genes[J]. Environmental Microbiology, 2010, 12(5): 1132-1143. DOI:10.1111/j.1462-2920.2010.02154.x |
| [25] | 戴欣, 周惠, 陈月琴, 等. 中国南海南沙海区沉积物中细菌16S rDNA多样性的初步研究[J]. 自然科学进展, 2002, 12(5): 479-484. DOI:10.3321/j.issn:1002-008X.2002.05.006 |
| [26] | Shu D T, He Y L, Yue H, et al. Microbial structures and community functions of anaerobic sludge in six full-scale wastewater treatment plants as revealed by 454 high-throughput pyrosequencing[J]. Bioresource Technology, 2015, 186: 163-172. DOI:10.1016/j.biortech.2015.03.072 |
| [27] | Klase G, Lee S, Liang S, et al. The microbiome and antibiotic resistance in integrated fishfarm water: implications of environmental public health[J]. Science of the Total Environment, 2019, 649: 1491-1501. DOI:10.1016/j.scitotenv.2018.08.288 |
| [28] | Zhang J X, Yang Y Y, Zhao L, et al. Distribution of sediment bacterial and archaeal communities in plateau freshwater lakes[J]. Applied Microbiology and Biotechnology, 2015, 99(7): 3291-3302. DOI:10.1007/s00253-014-6262-x |
| [29] | Murugan M, Srinivasan M, Sivakumar K, et al. Characterization of an actinomycete isolated from the estuarine finfish, Mugil cephalus Lin. (1758) and its optimization for cellulase production[J]. Journal of Scientific & Industrial Research, 2007, 66(5): 388-393. |
| [30] | Ellaiah P, Ramana T, Bapi Raju K V V S N, et al. Investigation on marine actinomycetes from bay of Bengal near Kakinada coast of Andhra Pradesh[J]. Asian Journal of Microbiology, Biotechnology and Environmental Sciences, 2004, 6(1): 53-56. |
| [31] | Subramani R, Aalbersberg W. Marine actinomycetes: an ongoing source of novel bioactive metabolites[J]. Microbiological Research, 2012, 167(10): 571-580. DOI:10.1016/j.micres.2012.06.005 |
| [32] | Sabarathnam B, Manilal A, Sujith S, et al. Role of sponge associated actinomycetes in the marine phosphorous biogeochemical cycle[J]. American-Eurasian Journal of Agriculture and Environmental Sciences, 2010, 8(3): 253-256. |
| [33] | Sivakumar K, Sahu M K, Thangaradjou T, et al. Research on marine actinobacteria in India[J]. Indian Journal of Microbiology, 2007, 47(3): 186-196. DOI:10.1007/s12088-007-0039-1 |
| [34] | Fernández-Gómez B, Richter M, Schüler M, et al. Ecology of marine Bacteroidetes: a comparative genomics approach[J]. The ISME Journal, 2013, 7(5): 1026-1037. DOI:10.1038/ismej.2012.169 |
| [35] | Bissett A, Bowman J P, Burke C M. Flavobacterial response to organic pollution[J]. Aquatic Microbial Ecology, 2008, 51(1): 31-43. |
| [36] | Wang J H, Lu J, Zhang Y X, et al. High-throughput sequencing analysis of the microbial community in coastal intensive mariculture systems[J]. Aquacultural Engineering, 2018, 83: 93-102. DOI:10.1016/j.aquaeng.2018.10.001 |
| [37] | Vezzulli L, Chelossi E, Riccardi G, et al. Bacterial community structure and activity in fish farm sediments of the Ligurian sea (western Mediterranean)[J]. Aquaculture International, 2002, 10(2): 123-141. DOI:10.1023/A:1021365829687 |
| [38] | Taylor L H, Latham S M, Woolhouse M E J. Risk factors for human disease emergence[J]. Philosophical Transactions of the Royal Society B, 2001, 356(1411): 983-989. DOI:10.1098/rstb.2001.0888 |
| [39] |
翟海佳, 郑忠明, 金中文, 等. 凡纳滨对虾覆膜养殖池塘沉积物微生物群落的磷脂脂肪酸谱图分析[J]. 水产学报, 2011, 35(12): 1865-1872. Zhai H J, Zheng Z M, Jin Z W, et al. Study on sediment microbial community structure in Litopenaeus vannamei ponds covered with a bottom plastic sheet using phospholipid fatty acid (PLFA) analysis[J]. Journal of Fisheries of China, 2011, 35(12): 1865-1872. |
| [40] | Wardle D A, Bardgett R D, Klironomos J N, et al. Ecological linkages between aboveground and belowground biota[J]. Science, 2004, 304(5677): 1629-1633. DOI:10.1126/science.1094875 |
| [41] | De Vries F T, Van Groenigen J W, Hoffland E, et al. Nitrogen losses from two grassland soils with different fungal biomass[J]. Soil Biology and Biochemistry, 2011, 43(5): 997-1005. DOI:10.1016/j.soilbio.2011.01.016 |
| [42] | Demoling F, Nilsson L O, Bååth E. Bacterial and fungal response to nitrogen fertilization in three coniferous forest soils[J]. Soil Biology and Biochemistry, 2008, 40(2): 370-379. DOI:10.1016/j.soilbio.2007.08.019 |
| [43] | Satoh H, Nakamura Y, Okabe S. Influences of infaunal burrows on the community structure and activity of ammonia-oxidizing bacteria in intertidal sediments[J]. Applied and Environmental Microbiology, 2007, 73(4): 1341-1348. DOI:10.1128/AEM.02073-06 |
| [44] | Hug L A, Castelle C J, Wrighton K C, et al. Community genomic analyses constrain the distribution of metabolic traits across the Chloroflexi phylum and indicate roles in sediment carbon cycling[J]. Microbiome, 2013, 1: 22. DOI:10.1186/2049-2618-1-22 |
| [45] |
宋洪宁, 杜秉海, 张明岩, 等. 环境因素对东平湖沉积物细菌群落结构的影响[J]. 微生物学报, 2010, 50(8): 1065-1071. Song H N, Du B H, Zhang M Y, et al. Effect of environmental factors on bacterial community in Lake Dongping sediment[J]. Acta Microbiologica Sinica, 2010, 50(8): 1065-1071. |
| [46] | Dai W F, Zhang J J, Tu Q C, et al. Bacterioplankton assembly and interspecies interaction indicating increasing coastal eutrophication[J]. Chemosphere, 2017, 177: 317-325. DOI:10.1016/j.chemosphere.2017.03.034 |
 2019, Vol. 40
2019, Vol. 40


