2. 俄克拉荷马大学环境基因组学研究所, 诺曼, 美国
2. Institute for Environmental Genomics, University of Oklahoma, Norman, OK, USA
根据《中国统计年鉴2017》[1]的数据:2016年全国废水排放总量为711.10亿m3, 废水中的化学需氧量(COD)总排放量高达1 046.53万t, 氨氮、总氮和总磷的排放总量分别为141.78、212.11和13.94万t.城市污水处理厂(wastetwater treatment plants, WWTPs)是控制水污染, 保证人类可持续发展的重要基础设施.城市WWTPs中应用最为普遍的是生物处理法, 而微生物是污水生物处理系统实现污染物去除和稳定运行的基础[2, 3].深入了解活性污泥(activated sludge, AS)中微生物群落的特性是理解污水生物处理本质的关键, 同时也可为污水处理系统的设计、优化及稳定运行提供理论依据及合理建议[4].
近年来, 高通量技术的发展极大地扩充了对AS群落多样性的认识[5~7], 但对AS群落的组成特征目前仍缺乏深入理解. WWTPs本质上是一个开放系统[8], 人类肠道、管网、空气和降水等来源的微生物都可能对AS群落产生一定的影响, 解析AS群落微生物来源能够在一定程度上揭示群落的组成机制, 有利于更加全面深入地认识AS群落组成特征.目前, 关于WWTPs中微生物来源的报道十分缺乏.仅有的一些研究通过分析WWTPs中进水微生物群落来源后发现, 12.1%的进水微生物源于粪便, 而81.4%的微生物则与城市的污水管网有关, 不同城市WWTPs进水微生物群落的地理分布差异主要来自高比例的管网微生物[9].此外, 降水能够通过地表径流或渗透携带土壤微生物进入污水管道中从而增加进水微生物群落的多样性[10]. Saunders等[11]通过对比进水微生物群落与AS群落后发现, 进水微生物的迁移对AS群落的组成具有一定影响.因此, 不同环境来源的微生物或许能够在AS群落组成中得以反映.不同城市WWTPs由于所处地理环境及接收污水特性的差异, 其AS群落微生物来源可能也存在不同, 明晰各城市AS群落微生物来源可更加深入地认识AS群落的组成, 为分析不同地域AS群落间存在差异提供新的角度, 为WWTPs的精准调控提供理论依据.然而, 目前的研究主要关注WWTPs中进水微生物的可能来源, 对AS群落的溯源分析还未见报道.
此外, 关于微生物的溯源分析, 目前使用较多的方法是, 将不同环境来源的微生物群落进行对比后, 分析共有微生物的种类与数量或根据几种预先确定好的指示微生物进行判断[12, 13], 具有极大的局限性.依据贝叶斯理论的溯源模型将已知的不同来源的微生物和待分析的微生物群落作为一个整体, 通过贝叶斯理论确定不同环境来源的微生物以及未知环境来源的微生物的相对占比[14], 能够较好地避免上述方法的缺陷. Knights等[14]已成功利用基于贝叶斯理论的SourceTracker分析工具对重症监护室(NICUs), 办公室以及分子生物实验室中的微生物污染的来源进行了解析, 发现源于肠道及皮肤的微生物是实验室中PCR实验用水的主要微生物污染源; NICUs中皮肤是主要的微生物污染物来源, 此外还有大量未知污染物来源; 办公室电话上的微生物主要来自皮肤与口腔.
基于以上背景, 本研究在全国15个城市的WWTPs开展采样工作, 采用高通量16S rRNA基因测序技术, 运用生态学、统计学等相关分析方法并结合基于贝叶斯理论的溯源模型, 解析我国城市WWTPs中AS群落的组成以及微生物来源, 深化对污水处理系统中AS群落组成特征认识, 以期为WWTPs的优化设计和精准调控提供理论依据.
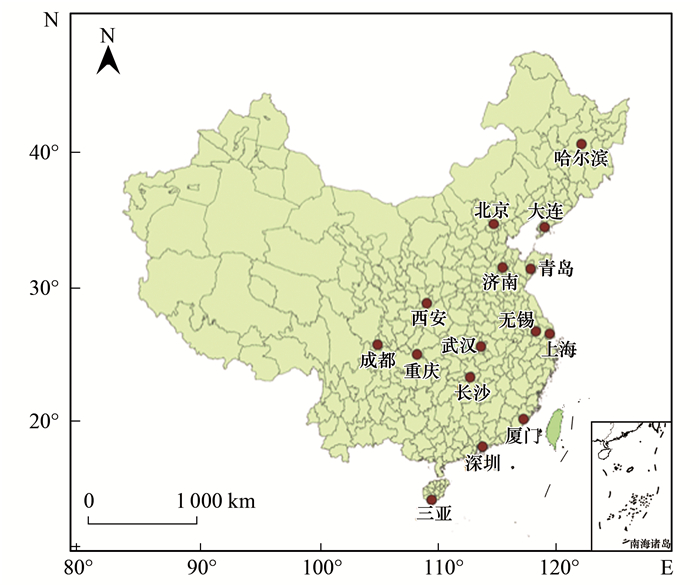
1 材料与方法 1.1 样品采集本研究于2014年7~8月期间采用统一方法对中国从南至北的15个城市, 包括三亚(SY)、深圳(SZ)、厦门(XM)、长沙(CS)、重庆(CQ)、武汉(WH)、成都(CD)、上海(SH)、无锡(WX)、西安(XA)、青岛(QD)、济南(JN)、大连(DL)、北京(BJ)和哈尔滨(HB)的WWTPs进行采样, 采样点地理分布如图 1所示.为保证后续相关数据的统计学分析结果更加可靠, 采样遵循每个城市至少采集4个WWTPs(其中DL只联系到2个WWTPs, 因此在DL的同一个WWTP不同曝气池中按照同样标准分别取样两次), 每个WWTP至少采集3个曝气池AS样品(分别在曝气池的前、中、后端), 若同一WWTP具有不同的处理工艺, 则在不同处理工艺的曝气池中都进行AS样品的采集.采样共获得来自全国60个WWTPs的211个AS样品.
|
图 1 采样城市地理位置分布示意 Fig. 1 Distribution of sampling cities |
使用美国MoBio公司(MoBio Laboratories, Carlsbad, CA, USA)的PowerSoil® DNA提取试剂盒对211个AS样品离心后的污泥沉淀物进行DNA提取.在确定提取的DNA样品满足上机测序的用量需求(DNA浓度保持在50~200 ng ·mL-1)和纯度要求(A260/A280在1.8附近, A260/A230大于1.7)后, 对其进行两步法PCR扩增[15].其中第一步PCR使用的是16S rRNA基因V4区通用引物515F(5′-GTGCCAGCMGCCGCGGTAA-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)[16].之后对PCR产物进行凝胶纯化, 纯化后的产物通过MiSeq平台(Illumina, San Diego, CA, USA), 利用基于Caporaso等[17]提出的测序方法完成16S rRNA基因测序.测序得到的原始数据通过Galaxy平台(http://zhoulab5.rccc.ou.edu:8080/)完成, 预处理过程中参数的设置详见文献[15, 18].
1.3 数据处理及分析方法在进行不同城市独特OTUs的分析前, 先对211个样品的OTU表格进行城市水平上的数据处理:只保留城市水平样品检出率大于50%的OTUs, 并将其在该城市所有样品中的平均丰度作为此OTU在城市水平上的丰度, 而其他城市水平样品检出率小于50%的OTUs的相对丰度记为0.
使用Knights等提出的溯源方法以及所提供的SourceTracker软件包, 在Qiime平台下完成AS微生物群落的溯源分析.软件包的安装及使用步骤详见文献[14].
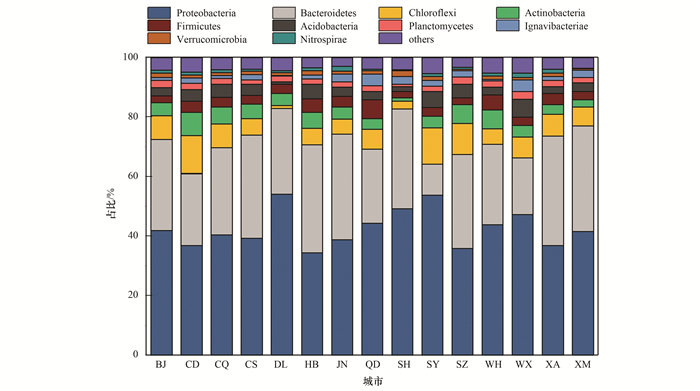
2 结果与讨论 2.1 不同城市的AS群落组成211个AS样品共获得了源于58个门的33 849个OTUs. 58个门中相对丰度超过0.1%的门(优势门)有10个, 其相对丰度之和占AS群落总丰度的94.53%~97.04%.除HB外, 其余城市AS群落中相对丰度最高的门均为Proteobacteria(34.42%~54.09%), 其次为Bacteroidetes(10.49%~36.72%)和Chloroflexi(0.90%~12.83%).此外, Actinobacteria, Firmicutes, Acidobacteria, 和Planctomycetes的相对丰度也较高(图 2).这与已报道的AS群落中的优势门类基本保持一致[19, 20], 但除了Proteobacteria和Bacteroidetes外, 其他优势门类的顺序略有不同.这些高丰度微生物或与WWTPs的功能有着密切联系, 例如:来自Proteobacteria的Dokdonella和Thauera, 与污水处理系统中的反硝化过程相关[21, 22].来自Bacteroidetes的Ferruginibacter是AS中常见的核心物种, 能够降解某些有机物[23]; Lewinella能够降解淀粉等多种有机碳[24]; 而Solitalea同样和反硝化过程有关[25].此外, Nitrospirae在各城市AS群落中均属于优势门, 具有较高的相对丰度(0.27%~1.61%)(图 2), 与Guo等[26]利用宏基因组测序技术探究WWTPs中优势菌所获得的结论相一致. Nitrospirae是WWTPs广泛存在的一类微生物, 隶属于该门的Nitrospira属, 能够参与污水处理系统的硝化作用[27, 28].因此, 各城市AS群落中较高丰度的Nitrospirae有益于WWTPs硝化作用的顺利进行.
|
相对丰度小于0.1%的门统一划归到others 图 2 全国不同城市的AS群落组成(门水平) Fig. 2 Composition of AS microbial communities in each city at the phylum level |
对比10个优势门在不同城市AS群落组成中的占比后发现:来自DL的AS群落中Proteobacteria相对丰度高达54.09%, 但Chloroflexi仅占群落总丰度的0.90%, 低于Actinobacteria(4.18%)、Firmicutes(3.08%)、Ignavibacteriae(2.01%)和Verrucomicrobia(0.93%).来自SY的AS群落中Bacteroidetes的相对丰度仅有10.49%, 远低于Proteobacteria的相对丰度(53.73%); 而来自HB的AS群落中Bacteroidetes(36.33%)的相对丰度则略高于Proteobacteria(34.42%).相比于其他城市的AS群落, Ignavibacteriae在QD和WX的AS系统中丰度相对较高.优势菌(门水平)在各城市AS群落中的分布情况表明, 全国不同城市AS群落中的优势门类相似, 但相对丰度或许受地理位置等因素(如靠近中国两端、沿海等)的影响而有所不同.
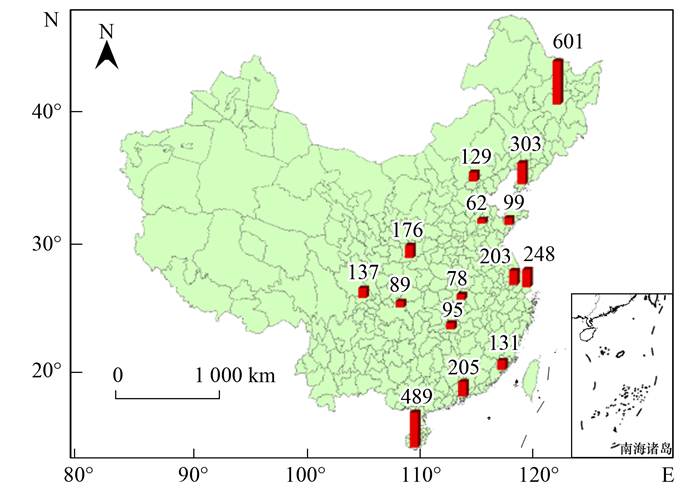
2.2 不同城市的独特OTUs研究表明, 虽然各个地区WWTPs执行功能基本一致, 运行条件基本相似, AS群落中存在着共有核心物种[11, 20, 29], 但不同地区WWTPs中的微生物群落组成同时存在一定地域差异[19]; 此外, 有报道指出, WWTPs之间距离相距越远, 其微生物群落及功能基因组成相似性均越小, 呈现出距离-衰减规律[30].为了进一步探究AS群落组成的地理分布特性, 随后分析了各城市WWTPs中所含有的独特OTUs(仅在某一个城市的样品检出率大于50%的OTUs).结果显示, 超过3 000个OTUs只广泛存在于某一个城市的WWTPs中.这表明, 不同城市因其地理位置或所处自然环境的差异, 如气候炎热或寒冷, 临近海洋或靠近内陆等, 其AS群落中可能存在一些其他城市稀有, 但在当地分布较广的OTUs, 即独特OTUs.独特OTUs在不同城市的数量分布同样受到地理因素的影响(图 3).此次采样最北端的HB(601个)和最南端的SY(489个)被检测出具有最多的独特OTUs.其中一个可能的原因是, 和其他采样城市相比, HB和SY具有较低和较高的年平均气温, 温度能够直接影响AS群落中微生物的生长代谢速率, 进而影响物种的进化速率、群落结构以及多种生态系统过程[31~33].此外, 一些沿海城市如DL(303个)、SH(248个)和SZ(205个)等也倾向于具有更多的独特OTUs, 这些独特OTUs或许和海洋微生物有关, 随后的AS群落溯源分析结果也证实了这一推测.相反, 中西部地区, 尤其是WH、CS和CQ的WWTPs中被检测出的独特OTUs相对较少(图 3).
|
图中柱状体上方的数字代表该城市含有的特殊OTUs个数 图 3 独特OTUs在采样城市的数量分布 Fig. 3 Numbers of unique OTUs in the WWTPs of each city |
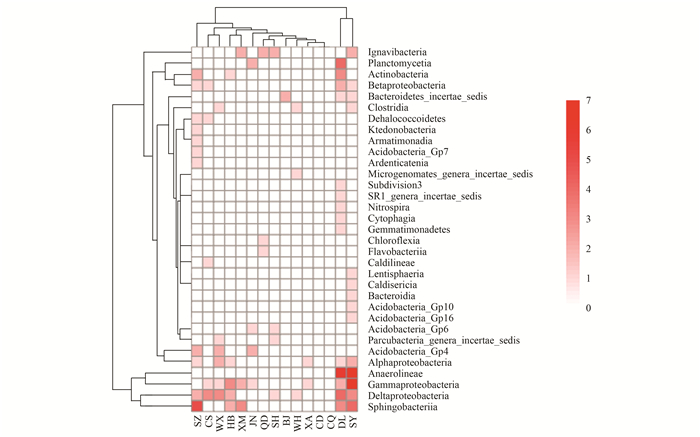
在城市特殊OTUs的研究基础上, 进一步分析了其中相对丰度较高(相对丰度大于0.1%)的微生物(纲水平), 即城市的特征微生物.整体来看, SY的特征微生物最多, 有33个, 主要来自Deltaproteobacteria纲和Anaerolineae纲, 其次为DL和SZ, 分别有31个和18个; 而CD和CQ未检测到特征微生物.由城市的特征微生物(纲水平)分布可以看出(图 4), 各城市的特征微生物主要来自Sphingobacteriia、Deltaproteobacteria、Gammaproteobacteria等, 而属于Anaerolineae的特征微生物仅来自于SY和DL.此外, SY和DL的特征微生物组成最为相似, 被归为一组, 而CD和CQ由于未检测到特征微生物而同样被归为一组(图 4), 表明地理环境因素可能是不同城市特征微生物存在差异的最大原因.
|
其中方框内颜色分为8个等级(0~7), 颜色越接近红色, 表明属于该纲的特有微生物数量越多(最多为7个), 白色表示个数为0 图 4 不同城市的特征微生物(纲水平)热图 Fig. 4 Heat map of AS indicators for each city |
由上述研究结果得知, 每个城市的WWTPs中具有一些独特OTUs或特征微生物, 且这些微生物的种类和数量可能与城市的地理位置及环境条件有关.利用SourceTracker对不同城市WWTPs中AS群落进行溯源分析后发现, 不同城市AS群落的微生物来源(属水平)存在一定差异(图 5):仅在沿海城市, 如DL、QD、SY、SZ和XM的AS群落中检测到海洋微生物源, 但比例都不高, 约占2%.水系发达的城市, 如SY、QD、SZ、CS、WH、WX和XM的AS群落中淡水来源(包括地下水、湖和河)微生物的占比接近或高于土壤, 其中SY的AS群落中有31%的微生物来自淡水.相反, 在城市水系不太发达的地区如CD和CQ等, WWTPs中AS群落检测到的土壤微生物来源高于淡水.因此, 淡水微生物来源在淡水资源相对较多的南方区域AS群落中所占比例较高, 这与人们的认知是一致的.此外, 植物及根际来源的微生物在不同城市的AS群落中也占据了一定的比例(4%~12%), 可能的原因是采样发生在降雨量较大的夏季, 雨水冲刷植物、土壤后, 大量携带有植物微生物或根际微生物的雨水汇入污水管网进入WWTPs.除SY外, 每个城市都有一定量的工业废水来源微生物(4%~14%), 这是由于所采样的大部分WWTPs都处理一定量的工业废水; 而SY未检测到工业废水来源微生物, 这与采样时工作人员所填写的SY市内WWTPs不处理任何工业废水的问卷结果相一致.
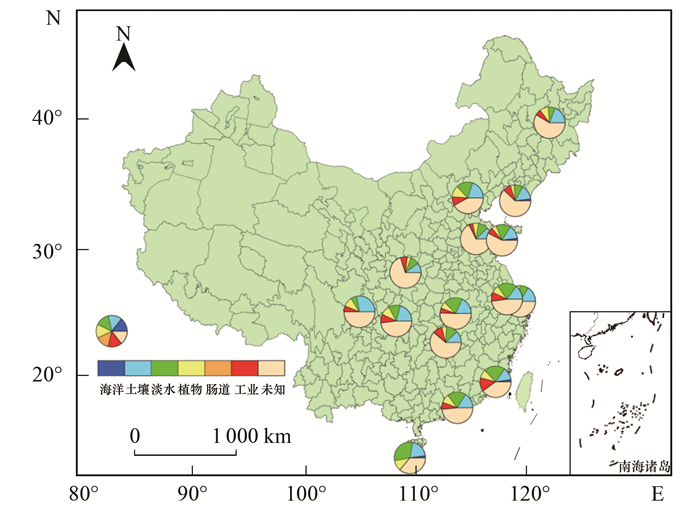
|
图 5 不同城市AS群落的微生物源比例 Fig. 5 Proportions of source environment microbes in AS microbial communities in each city |
从溯源分析的结果可以看出, AS群落结构能在一定程度上反映该城市的进水性质, 证明了进水微生物的迁移的确对AS群落的组成具有一定影响[11].此外, 城市的部分地理环境特征也能通过AS群落结构得以体现.但值得注意的是, 人体肠道微生物的占比非常低, 一方面可能由于人体肠道微生物随污水的处理过程被去除, 另一方面可能是数据库中的人体肠道微生物多来自欧美人群, 而肠道微生物的组成主要由环境以及饮食决定的[34], 因此数据库所提供的肠道微生物源对中国不太适用, 但具体原因还需进一步研究.此外, 每个城市将近一半的微生物源未知, 表明AS群落可能还有大量源于污水管网中的微生物以及在AS系统操作参数条件下被驯化的微生物.
3 结论(1) 全国不同城市AS群落中的优势微生物(相对丰度超过0.1%的门)种类相似, 包括Proteobacteria、Bacteroidetes以及和WWTPs硝化作用相关的Nitrospirae等.各城市AS群落中优势门的丰度之和占群落总丰度的94.53%~97.04%.
(2) 城市的地理位置及环境条件能够影响AS群落组成:不同城市WWTPs中各优势门的相对丰度, 独特OTUs个数以及特征微生物种类受地理位置等因素的影响而存在差异.接近中国大陆两端的哈尔滨和三亚, 具有较多的独特OTUs; 沿海城市WWTPs中的独特OTUs也相对较多.所处地理环境相似的城市, 其AS群落中的特征微生物也更为相似.
(3) AS群落结构能在一定程度上反映该城市的进水性质及地理环境特征.植物及土壤来源微生物在不同城市的AS群落中均占据了较高比例; 淡水来源微生物在淡水资源相对较多的南方区域中所占比例较高; 除SY外, 每个城市都有一定量的工业废水来源; 但人体肠道微生物的占比非常低.除此之外, 还有大量微生物来源未知.
| [1] | 中华人民共和国国家统计局. 中国统计年鉴[M]. 北京: 中国统计出版社, 2017. |
| [2] | Rittmann B E, Hausner M, Löffler F, et al. A vista for microbial ecology and environmental biotechnology[J]. Environmental Science & Technology, 2006, 40(4): 1096-1103. |
| [3] | Limpiyakorn T, Kurisu F, Sakamoto Y, et al. Effects of ammonium and nitrite on communities and populations of ammonia-oxidizing bacteria in laboratory-scale continuous-flow reactors[J]. FEMS Microbiology Ecology, 2007, 60(3): 501-512. DOI:10.1111/fem.2007.60.issue-3 |
| [4] |
王晓慧.城市污水处理厂中氨氧化菌及细菌群落结构与功能研究[D].北京: 清华大学, 2010. Wang X H. The community structures and functions of ammonia oxidizing bacteria and bacteria in wastewater treatment plant[D]. Beijing: Tsinghua University, 2010. http://cdmd.cnki.com.cn/Article/CDMD-10003-1011280384.htm |
| [5] | Xia Y, Hu M, Wen X H, et al. Diversity and interactions of microbial functional genes under differing environmental conditions:insights from a membrane bioreactor and an oxidation ditch[J]. Scientific Reports, 2016, 6: 18509. DOI:10.1038/srep18509 |
| [6] | Cydzik-Kwiatkowska A, Zielińska M. Bacterial communities in full-scale wastewater treatment systems[J]. World Journal of Microbiology and Biotechnology, 2016, 32(4): 66. DOI:10.1007/s11274-016-2012-9 |
| [7] | Griffin J S, Wells G F. Regional synchrony in full-scale activated sludge bioreactors due to deterministic microbial community assembly[J]. The ISME Journal, 2017, 11(2): 500-511. DOI:10.1038/ismej.2016.121 |
| [8] | Ofiţeru I D, Lunn M, Curtis T P, et al. Combined niche and neutral effects in a microbial wastewater treatment community[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(35): 15345-15350. DOI:10.1073/pnas.1000604107 |
| [9] | Shanks O C, Newton R J, Kelty C A, et al. Comparison of the microbial community structures of untreated wastewaters from different geographic locales[J]. Applied and Environmental Microbiology, 2013, 79(9): 2906-2913. DOI:10.1128/AEM.03448-12 |
| [10] | McLellan S L, Huse S M, Mueller-Spitz S R, et al. Diversity and population structure of sewage-derived microorganisms in wastewater treatment plant influent[J]. Environmental Microbiology, 2010, 12(2): 378-392. DOI:10.1111/emi.2010.12.issue-2 |
| [11] | Saunders A M, Albertsen M, Vollertsen J, et al. The activated sludge ecosystem contains a core community of abundant organisms[J]. The ISME Journal, 2016, 10(1): 11-20. DOI:10.1038/ismej.2015.117 |
| [12] | Flores G E, Bates S T, Caporaso J G, et al. Diversity, distribution and sources of bacteria in residential kitchens[J]. Environmental Microbiology, 2013, 15(2): 588-596. DOI:10.1111/emi.2013.15.issue-2 |
| [13] | Barberán A, Ladau J, Leff J W, et al. Continental-scale distributions of dust-associated bacteria and fungi[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(18): 5756-5761. DOI:10.1073/pnas.1420815112 |
| [14] | Knights D, Kuczynski J, Charlson E S, et al. Bayesian community-wide culture-independent microbial source tracking[J]. Nature Methods, 2011, 8(9): 761-763. DOI:10.1038/nmeth.1650 |
| [15] | Wu L Y, Wen C Q, Qin Y J, et al. Phasing amplicon sequencing on Illumina Miseq for robust environmental microbial community analysis[J]. BMC Microbiology, 2015, 15: 125. DOI:10.1186/s12866-015-0450-4 |
| [16] | Shi S J, Nuccio E, Herman D J, et al. Successional trajectories of rhizosphere bacterial communities over consecutive seasons[J]. mBio, 2015, 6(4): :e00746-15. DOI:10.1128/mBio.00746-15 |
| [17] | Caporaso J G, Lauber C L, Walters W A, et al. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(Suppl 1): 4516-4522. |
| [18] | Guo X, Feng J J, Shi Z, et al. Climate warming leads to divergent succession of grassland microbial communities[J]. Nature Climate Change, 2018, 8(9): 813-818. DOI:10.1038/s41558-018-0254-2 |
| [19] | Zhang T, Shao M F, Ye L. 454 pyrosequencing reveals bacterial diversity of activated sludge from 14 sewage treatment plants[J]. The ISME Journal, 2012, 6(6): 1137-1147. DOI:10.1038/ismej.2011.188 |
| [20] | Wang X H, Hu M, Xia Y, et al. Pyrosequencing analysis of bacterial diversity in 14 wastewater treatment systems in China[J]. Applied and Environmental Microbiology, 2012, 78(19): 7042-7047. DOI:10.1128/AEM.01617-12 |
| [21] | Tian H L, Zhao J Y, Zhang H Y, et al. Bacterial community shift along with the changes in operational conditions in a membrane-aerated biofilm reactor[J]. Applied Microbiology and Biotechnology, 2015, 99(7): 3279-3290. DOI:10.1007/s00253-014-6204-7 |
| [22] | Mao Y P, Xia Y, Wang Z P, et al. Reconstructing a Thauera genome from a hydrogenotrophic-denitrifying consortium using metagenomic sequence data[J]. Applied Microbiology and Biotechnology, 2014, 98(15): 6885-6895. DOI:10.1007/s00253-014-5756-x |
| [23] | Lim J H, Baek S H, Lee S T. Ferruginibacter alkalilentus gen. nov., sp. Nov. and Ferruginibacter lapsinanis sp. nov., novel members of the family 'Chitinophagaceae' in the phylum Bacteroidetes, isolated from freshwater sediment[J]. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(10): 2394-2399. DOI:10.1099/ijs.0.009480-0 |
| [24] | Ju F, Guo F, Ye L, et al. Metagenomic analysis on seasonal microbial variations of activated sludge from a full-scale wastewater treatment plant over 4 years[J]. Environmental Microbiology Reports, 2014, 6(1): 80-89. DOI:10.1111/1758-2229.12110 |
| [25] | Tian W D, Li W G, An K J, et al. Denitrifying dephosphatation performance link to microbial community structure[J]. Journal of Water Sustainability, 2011, 1(3): 11-20. |
| [26] | Guo J H, Ni B J, Han X Y, et al. Unraveling microbial structure and diversity of activated sludge in a full-scale simultaneous nitrogen and phosphorus removal plant using metagenomic sequencing[J]. Enzyme and Microbial Technology, 2017, 102: 16-25. DOI:10.1016/j.enzmictec.2017.03.009 |
| [27] | Yao Q, Peng D C. Nitrite oxidizing bacteria (NOB) dominating in nitrifying community in full-scale biological nutrient removal wastewater treatment plants[J]. AMB Express, 2017, 7(1): 25. DOI:10.1186/s13568-017-0328-y |
| [28] | Siripong S, Rittmann B E. Diversity study of nitrifying bacteria in full-scale municipal wastewater treatment plants[J]. Water Research, 2007, 41(5): 1110-1120. DOI:10.1016/j.watres.2006.11.050 |
| [29] | Xia Y, Wen X H, Zhang B, et al. Diversity and assembly patterns of activated sludge microbial communities:a review[J]. Biotechnology Advances, 2018, 36(4): 1038-1047. DOI:10.1016/j.biotechadv.2018.03.005 |
| [30] | Wang X H, Wen X H, Deng Y, et al. Distance-Decay Relationship for Biological Wastewater Treatment Plants[J]. Applied and Environmental Microbiology, 2016, 82(16): 4860-4866. DOI:10.1128/AEM.01071-16 |
| [31] | Brown J H, Gillooly J F, Allen A P, et al. Toward a metabolic theory of ecology[J]. Ecology, 2004, 85(7): 1771-1789. DOI:10.1890/03-9000 |
| [32] | West G B, Brown J H, Enquist B J. A general model for the origin of allometric scaling laws in biology[J]. Science, 1997, 276(5309): 122-126. DOI:10.1126/science.276.5309.122 |
| [33] | Price C A, Weitz J S, Savage V M, et al. Testing the metabolic theory of ecology[J]. Ecology Letters, 2012, 15(12): 1465-1474. DOI:10.1111/j.1461-0248.2012.01860.x |
| [34] | Rothschild D, Weissbrod O, Barkan E, et al. Environment dominates over host genetics in shaping human gut microbiota[J]. Nature, 2018, 555(7695): 210-215. DOI:10.1038/nature25973 |
 2019, Vol. 40
2019, Vol. 40


