2. 中国科学院大学, 北京 100049;
3. 河北大学化学与环境科学学院, 保定 071002
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Chemistry & Environmental Science, Hebei University, Baoding 071002, China
快速砂滤池广泛应用在城市饮用水处理中, 滤池中的微生物及其对水质的影响受到越来越多的关注和研究.尽管处于寡营养和频繁冲洗的条件下, 滤料表面仍然栖息着大量的微生物[1~3], 而且这些微生物参与了水中污染物的去除[4].已有研究发现了滤池中对铁、锰、砷和氨氮具有氧化作用的微生物或功能基因[5~9], 异养微生物的代谢活动也消耗着水中的有机物质[10].在实验室研究中, 发现快速砂滤池中的微生物对一些农药具有降解作用[11].为了加深微生物对饮用水水质作用的普遍认识, 需要对快速砂滤池微生物整体上的构成和功能进行研究.
滤池中优势微生物的构成情况中, 细菌占绝对优势, 古菌和真核生物相对丰度较低[2, 12].在门分类水平上, Proteobacteria占主导, 其它优势门类因不同水源有所差别, 地下水滤池中优势门类为Nitrospirae和Acidobacteria[2, 13], 地表水滤池中优势门类为Bacteroidetes和Firmicutes[14, 15].在优势微生物的功能特征上, Gülay等[2]使用16S rRNA方法研究了一系列地下水滤池, 发现相对丰度大于1%的共有菌, 大多是以氨氮、甲烷、硫化氢和还原性铁锰作为电子供体, 证明了这些优势菌对地下水滤池污染物去除的主导作用.因为饮用水源遭受多种微量有机物的污染[16], 大家更关注微生物对这些物质的去除作用. Oh等[14]对地表水滤池使用宏基因组方法研究, 发现在活性炭滤池中Rhizobiales具有较多的芳香类化合物降解基因.本研究证明滤池中的微生物对灭草松、草甘膦等农药或卡马西平代谢物具有降解作用, 其降解需要较长的水力停留时间及特定化合物结构[11, 17], Gu等[18]从饮用水滤池中分离培养一株红球菌属(Rhodococcus sp.CS-1), 并用转录组方法验证其降解苯酚的能力, 但这种培养筛选的方法并不适于对不可培养微生物能力的发掘.另外, 添加农药2, 6-二氯苯甲酰胺的降解菌实现污染物去除的尝试, 因为目标菌不能适应滤池的生态环境而流失[19, 20].
本研究假定在饮用水快滤池中, 存在着特定的核心群落, 它们对水中的物质转化起到主导作用.据此, 笔者使用宏基因组技术对11个饮用水快滤池进行研究, 明确滤池中优势菌群的构成及其功能特征, 通过其对异生物质降解潜能的评估识别相关的关键菌属, 并引述相关研究佐证其降解能力.本文完善了对滤池微生物及其功能的认识, 以期为研究或指导饮用水滤池利用生物(强化)去除各类污染物, 特别是异生物质提供理论基础.
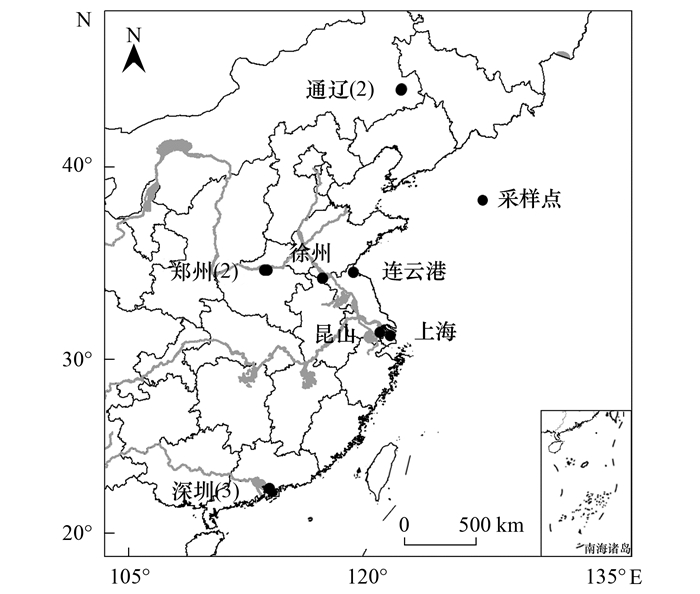
1 材料与方法 1.1 采样点和采样时间为了研究饮用水快滤池中微生物的共性特征, 需要尽可能研究多种类型的快滤池.选取国内从南至北7个城市的11座饮用水处理快滤池进行采样(图 1), 使用的水源包括为地下水(3座)和地表水(8座), 其中所有地下水滤池和6座地表水滤池使用石英砂滤料, 2座地表水滤池使用煤炭(上层)和石英砂(下层)双层滤料.各滤池水力停留时间均在20 min以内, 反冲洗周期1~3 d.分别在9月和12月对所有滤池进行共两次采样.
|
图 1 饮用水快滤池采样点示意 Fig. 1 Locations of the rapid sand filter sampling sites |
分别对滤池的进出水和滤料进行采集.对于水样, 从管道上的采样龙头(采集流出3 min后的水)或进出水渠中, 使用5 L干净的聚乙烯桶涮洗两遍后采集, 随后将部分水样转移至250 mL蓝盖瓶中, 并使用硫酸酸化至pH<2后置于4℃保存.前期研究发现, 滤池中微生物群落随深度增加只有生物量的变化, 生物种类和功能并没有大的变化[1], 因此, 本研究仅对滤池表面5~15 cm处的滤料进行采集.为保证足够的生物量, 在反冲洗前5 h的范围内采样.将滤池水面降至滤料层以下后, 使用涮洗干净的铁锹或带上丁腈手套手动采集.对滤池多个地点的滤料混合, 将两份约25 g混合样品分装在两个50 mL离心管中作为平行.所有样品在冰袋中运送至实验室, 检测前, 水样保存在4℃, 滤料样品保存在-80℃.
1.3 测定方法水样分析:将过0.45 μm滤膜(津隆)后的水样用于DOC、NH4+-N和NO3--N分析, 将水样直接进行TN和浊度检测.使用总有机碳分析仪(Shimadzu TOC-VCPH, 日本)分析DOC, 对2个平行样品分别测定3次.使用纳氏试剂光度法、紫外分光光度法和过硫酸钾氧化紫外分光光度法分别对NH4+-N、NO3--N和TN进行分析[21], 每项检测做3组平行.使用浊度计(WGZ-2000, 申光)对混匀后的样品进行浊度检测.
滤料分析:称取10 g滤料, 使用强力土壤大提试剂盒(QIAGEN, Germany)提取滤料中所有微生物的DNA, 所有DNA样品送至华大基因公司进行宏基因组建库和测序.使用Illumina(HiSeq X-Ten)平台测序, 插入片段长度为300bp或350bp, 测序长度为双端150bp, 最终成功获得39个宏基因组数据.
1.4 宏基因组数据分析数据预处理:原始测序序列的质量控制由公司完成, 包括去除接头序列、重复序列、含N的碱基数目总和达到10%的reads以及质量值连续≤32的碱基数达到40%的reads, 最终产生约5.7 Gbp的高质量数据.利用MyTaxa[22]预测测序深度, 结果显示测序数据覆盖了滤池中54.16%~99.36%(平均80.24%)的生物, 证明实验获得了大多数的生物信息.
宏基因组装和非冗余基因集构建:首先, 用MEGAHIT (v1.1.3)[23]将每个样品的单独组装, 选取组装长度≥500 bp的contigs用Prodigal (v2.6.3)[24]预测开放阅读框(open reading frame, ORF), 并过滤掉长度小于100bp的ORF.然后将所有样品的ORFs合并, 使用CD-HIT (v4.7;-c 0.95 -aS 0.9 -g 1)[25]聚类获得非冗余基因集.最后使用Kallisto (v0.43.1)[26]将每个样品的reads和非冗余基因集比对, 获得基因在每个样品中的丰度, 并用edgeR (v3.20.2)[27]修正的M-值标准化方法(TMM)算法对比对结果进行标准化.
物种注释和功能注释:物种注释使用DIAMOND (v0.8.12;--evalue 1e-5 -query-cover 60 -id 30)[28]软件将非冗余基因集的蛋白序列比对NCBI-NR数据库, 使用最近公共祖先算法获得在门、纲、目、科和属水平上的注释结果.功能注释利用GhostKOALA (v2.0)[29]将非冗余基因集的蛋白序列和KEGG数据库(v87.1)进行比对, 获得基因功能注释.随后, 将这些功能基因信息匹配到KEGG通路图(pathway map)数据库获得通路注释.
优势种属获取:将相对丰度占前10%(14个属)的属作为优势属.然后, 根据注释为这些属的基因ID信息, 从功能注释结果中提取属于该物种的所有功能注释结果.物种相对丰度计算使用注释到物种的reads数除以该样品的所有reads数.功能相对丰度计算使用该物种所具有的某项功能的reads数除以该样品中比对上该项功能的所有reads数.分析使用的reads数为TMM标准化后的值.
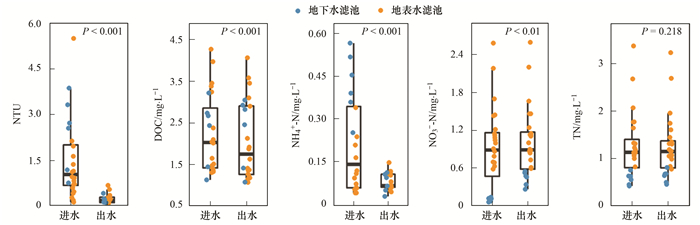
2 结果与讨论 2.1 快速砂滤池出水水质变化饮用水滤池主要设计功能是降低浊度, 出水浊度要求满足生活饮用水卫生标准(GB 5749-2006)[30], 本研究中所有滤池的出水浊度均小于1(图 2), 符合设计要求.然而, 在过滤过程中, 其它污染物也发生了降解或转化.如图 2, 有机物发生少量但显著(P < 0.001)的降解, 这些有机物的去除和滤池中的微生物活动有关[10], 异养微生物的生命活动需要以有机物作为电子供体获取能量, 从而消耗水中的有机物.另外, 水中总氮未发生明显变化(P=0.218), 说明反硝化及厌氧氨氧化作用在滤池中不显著. NH4+-N显著性降低(P < 0.001), 对应的NO3--N显著性增加(P < 0.01), 说明NH4+-N主要是转化为NO3--N.许多研究报道, 在饮用水滤池中生存着丰富的氨氧化细菌、氨氧化古菌以及完全氨氧化菌(comammox)[7, 9, 31~33], 这类微生物以NH4+或NO2-作为电子供体获取能量, 从而将氨氮最终转化为硝酸盐氮.滤池中微生物对水中有机物和无机物均有降解或转化作用, 说明快滤池中的微生物对水质的影响是不可忽视的.
|
图 2 滤后水水质指标变化 Fig. 2 Variation in water quality indices after filtration |
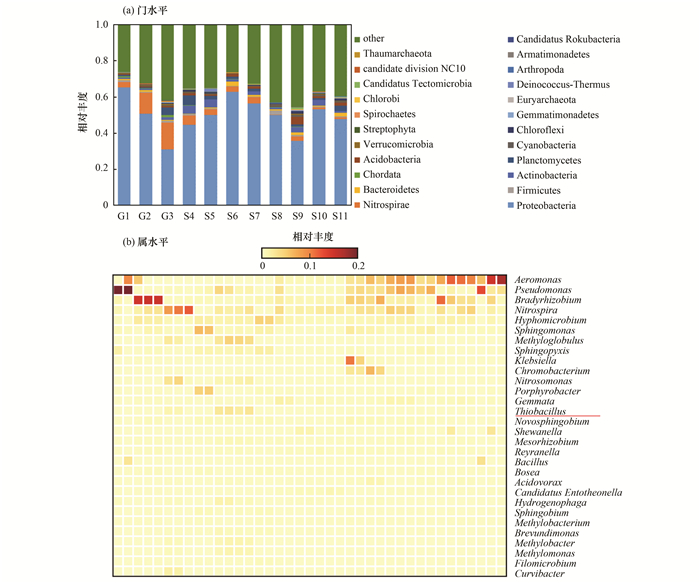
共有70.85%±6.37%的序列reads获得物种注释, 其中细菌、古菌和真核生物分别占70%±6.67%、0.13%±0.11%和0.72%±0.59%.在门分类水平上[图 3(a)], Proteobacteria在所有滤池中占绝对优势(49.8%±10.3%), 滤池中只检测到两类古菌, Thaumarchaeota(0.07%±0.11%)和Euryarchaeota(0.06%±0.03%), 古菌在饮用水滤池中的丰度普遍较低[34], 另外发现有三类真核生物检出, 分别是脊索动物门、节肢动物门和链型植物门.根据各物种丰度结果可以得出, 在饮用水滤池中, 细菌的生命活动, 特别是变形菌门, 是实现各类物质生物转化的主要途径, 而古菌和真核生物对水质的影响相对有限.
|
图中展示平均相对丰度前30的属, 红线以上部分为占前10%的属 图 3 饮用水快滤池微生物群落在门水平和属水平上的相对丰度 Fig. 3 Relative abundance of microbial communities (at the phylum and genus level) from rapid sand filters used in the treatment of drinking water |
在各种饮用水快滤池中, 环境类似而且稳定, 在这样的条件下会形成特定的优势微生物群落, 筛选这类微生物有多种理论方法[35], 考虑到高丰度物种的代谢活动会更大地影响水中的物质转化, 而且各样品中相对丰度较高的属比较一致[图 3(b)].因此, 本研究使用平均相对丰度占前10%的属作为优势属进行研究, 共获得14个优势属[图 3(b)虚线以上部分], 优势属总的相对丰度为22.01%±8.97%.在这些优势属中, 除了Nitrospira和Gemmata, 其它属在门类水平上都属于Proteobacteria.
2.3 优势属的功能特征在获得优势属的种类后, 本研究从宏基因组数据中提取了5种和物质转化相关的通路信息进行探究.总体上看[图 4(a)], 优势群落各项功能的相对丰度显著高于物种的相对丰度(P < 0.01碳水化合物代谢, P < 0.001其他), 这说明优势群落在对碳水化合物、氮、硫和异生物质的利用以及各种物质传输效率方面, 具有更大的优势, 这些优势可能是这些物种占据高丰度的原因.进一步分析优势属各自的相对丰度及其5种通路的相对丰度[图 4(b)].总体上看, 物种丰度越高, 功能基因越丰富(r=0.98, P < 0.001, 皮尔森相关性), 对水中各种物质转化的潜能越高.其中, Aeromonas拥有最高的膜转运基因和碳水化合物代谢基因, 说明该属在获取资源和能量方面潜能最大.而Bradyrhizobium在氮、硫和异生物质代谢功能上拥有最大的潜能.

|
P Wilcoxon符号秩检验, 表示属与5种通路在相对丰度上的差异显著性; 热图中用黑框标记出最高值 图 4 饮用水滤池中优势属相对丰度及其5种通路的相对丰度 Fig. 4 Relative abundance of dominant genera and the relative abundance of five pathways |
然而, 也存在不同的现象, 因为这些属对特定物质的代谢能力不同.比如Nitrosomonas的氮代谢相对丰度要高于几个相对丰度较高的属, Nitrosomonas属于氨氧化细菌, 以氨氮为电子供体来获取能量, 图 2显示过滤过程中氨氮被转化为硝酸盐氮, 这和Nitrosomonas的活动密切相关.优势菌中和Nitrosomonas有类似功能的是Nitrospira, Nitrospira在其它饮用水滤池中也有非常高的丰度[36], 这可能和它多样性的代谢功能相关, 包括常规的硝化和氨氧化作用[37, 38], 以及对尿素[39]和丙酮酸[40]的代谢.类似地, Thiobacillus的硫代谢丰度也要高于几种相对丰度较高的属, 因为Thiobacillus属于化能自养型菌, 以还原态的硫为电子供体获取能量进行二氧化碳固定[41].因此, 通过这种方式可以发现一些对特定物质转化有特殊贡献的物种, 比如Klebsiella对碳水化合物的转化, Hyphomicrobium对硫的转化以及Methyloglobulus对异生物质的转化.
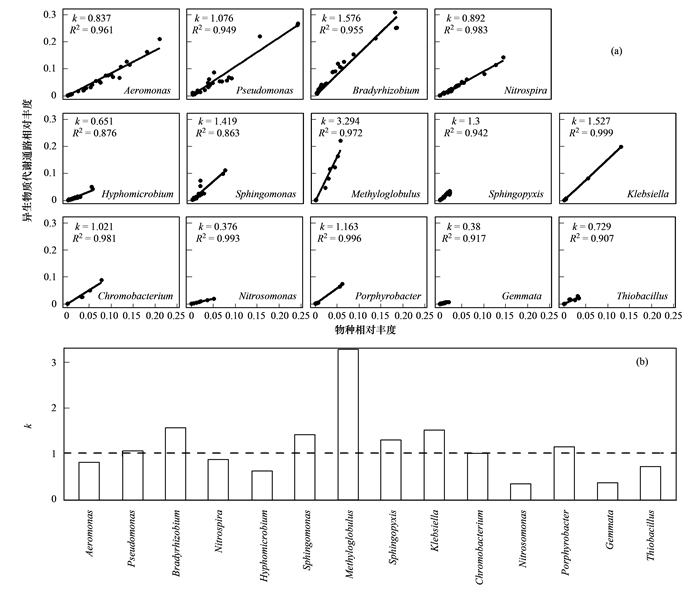
人类活动产生的微量污染物(如农药、多环芳烃及PPCPs)已经威胁到饮用水水源[16], 本研究通过评估滤池中的优势微生物对异生物质的降解潜能, 发掘降解异生物质的关键菌种.如图 5(a)所示, 通过单位物种的功能丰度大小, 即对物种相对丰度和该物种的异生物质代谢通路相对丰度进行线性拟合获得的斜率k值, 来表征该物种降解异生物质的潜能大小. 图 5(b)展现出滤池优势属的代谢潜能, 其中5种具有较大潜能(k≥1.3), 分别是Bradyrhizobium、Sphingomonas、Methyloglobulus、Sphingopyxis及Klebsiella. Bradyrhizobium同时也是滤池中异生物质代谢基因最丰富的属[图 4(b)], Oh等[14]使用宏基因组方法也发现Rhizobiales物种编码丰富的芳香族化合物降解基因, 该目中就包括Bradyrhizobium.在Bradyrhizobium菌种的基因组中发现了降解2, 4-二氯苯氧乙酸和卤代脂肪族污染物的基因[42, 43].在培养实验中, 已经证实Bradyrhizobium中的物种能够降解有机磷农药[44]、甲氧滴滴涕[45]及其它多种难降解有机物[46, 47].这些结果表明Bradyrhizobium是饮用水滤池降解微量有机物的关键属.
|
(a)线性关系; (b)k值分布; k值为线性拟合的斜率值, 其实际含义表示单位丰度物种的通路丰度大小 图 5 优势属的相对丰度与其异生物质代谢通路相对丰度的线性关系以及它们的k值分布 Fig. 5 Linear relationship between the relative abundance of dominant genera and the relative abundance of xenobiotic metabolic pathways and their k distribution |
有趣的是, Methyloglobulus作为专性的甲烷氧化菌[48], 却是降解异生物质潜能最大的属, 其k值高达3.294.这是因为甲烷代谢菌的关键性酶, 甲烷单加氧酶(methane monooxygenase, MMO), 尤其是溶解性MMO, 能够催化非特异性的氧化反应[49], Hedegaard等[50]在处理地下水的饮用水快滤池中, 分离并证实了这类微生物能共代谢降解灭草松.经进一步发掘该属对降解不同异生物质的基因丰度, 发现85.6%的基因是针对氯代烷烃、氯代烯烃降解和硝基甲苯降解这类具有甲烷类似结构的异生物质.
3 结论(1) 饮用水快速砂滤池中的微生物对水质有显著的影响, 包括氨氮和有机物的去除.细菌、尤其是变形菌门, 对水质转化的贡献较大.
(2) 饮用水快速砂滤池中存在特定的高丰度菌群, 它们对饮用水中的碳水化合物、氮、硫和异生物质的代谢转化具有更高的贡献以及更高的利用效率.而Bradyrhizobium和Aeromonas是影响滤池各种物质转化的关键物种.
(3) 饮用水快速砂滤池中存在降解异生物质的关键属, 具有丰度优势的关键属包括Bradyrhizobium、Sphingomonas、Methyloglobulus、Sphingopyxis和Klebsiella.
致谢: 感谢华大基因提供宏基因组测序服务, 以及各饮用水厂领导及员工在采样方面提供支持和帮助, 在此一并致谢!| [1] | Bai Y H, Liu R P, Liang J S, et al. Integrated metagenomic and physiochemical analyses to evaluate the potential role of microbes in the sand filter of a drinking water treatment system[J]. PLoS One, 2013, 8(4): e61011. DOI:10.1371/journal.pone.0061011 |
| [2] | Gülay A, Musovic S, Albrechtsen H J, et al. Ecological patterns, diversity and core taxa of microbial communities in groundwater-fed rapid gravity filters[J]. The ISME Journal, 2016, 10(9): 2209-2222. DOI:10.1038/ismej.2016.16 |
| [3] | Lautenschlager K, Hwang C, Ling F Q, et al. Abundance and composition of indigenous bacterial communities in a multi-step biofiltration-based drinking water treatment plant[J]. Water Research, 2014, 62: 40-52. DOI:10.1016/j.watres.2014.05.035 |
| [4] | Rittmann B E, Snoeyink V L. Achieving biologically stable drinking water[J]. Journal American Water Works Association, 1984, 76(10): 106-114. DOI:10.1002/j.1551-8833.1984.tb05427.x |
| [5] | Bruins J H, Petrusevski B, Slokar Y M, et al. Biological and physico-chemical formation of Birnessite during the ripening of manganese removal filters[J]. Water Research, 2015, 69: 154-161. DOI:10.1016/j.watres.2014.11.019 |
| [6] | Crognale S, Casentini B, Amalfitano S, et al. Biological As(Ⅲ) oxidation in biofilters by using native groundwater microorganisms[J]. Science of the Total Environment, 2019, 651: 93-102. DOI:10.1016/j.scitotenv.2018.09.176 |
| [7] | Fowler S J, Palomo A, Dechesne A, et al. Comammox Nitrospira are abundant ammonia oxidizers in diverse groundwater-fed rapid sand filter communities[J]. Environmental Microbiology, 2018, 20(3): 1002-1015. DOI:10.1111/emi.2018.20.issue-3 |
| [8] | Gülay A, Musovic S, Albrechtsen H J, et al. Neutrophilic iron-oxidizing bacteria:occurrence and relevance in biological drinking water treatment[J]. Water Supply, 2013, 13(5): 1295-1301. DOI:10.2166/ws.2013.113 |
| [9] | Tatari K, Musovic S, Gülay A, et al. Density and distribution of nitrifying guilds in rapid sand filters for drinking water production:Dominance of Nitrospira spp.[J]. Water Research, 2017, 127: 239-248. DOI:10.1016/j.watres.2017.10.023 |
| [10] | Terry L G, Summers R S. Biodegradable organic matter and rapid-rate biofilter performance:a review[J]. Water Research, 2018, 128: 234-245. DOI:10.1016/j.watres.2017.09.048 |
| [11] | Hedegaard M J, Albrechtsen H J. Microbial pesticide removal in rapid sand filters for drinking water treatment-potential and kinetics[J]. Water Research, 2014, 48: 71-81. DOI:10.1016/j.watres.2013.09.024 |
| [12] | Li Q, Yu S, Li L, et al. Microbial communities shaped by treatment processes in a drinking water treatment plant and their contribution and threat to drinking water safety[J]. Frontiers in Microbiology, 2017, 8: 2465. DOI:10.3389/fmicb.2017.02465 |
| [13] | Albers C N, Ellegaard-Jensen L, Harder C B, et al. Groundwater chemistry determines the prokaryotic community structure of waterworks sand filters[J]. Environmental Science & Technology, 2015, 49(2): 839-846. |
| [14] | Oh S, Hammes F, Liu W T. Metagenomic characterization of biofilter microbial communities in a full-scale drinking water treatment plant[J]. Water Research, 2018, 128: 278-285. DOI:10.1016/j.watres.2017.10.054 |
| [15] | Xu J J, Tang W, Ma J, et al. Comparison of microbial community shifts in two parallel multi-step drinking water treatment processes[J]. Applied Microbiology and Biotechnology, 2017, 101(13): 5531-5541. DOI:10.1007/s00253-017-8258-9 |
| [16] | Schwarzenbach R P, Escher B I, Fenner K, et al. The challenge of micropollutants in aquatic systems[J]. Science, 2006, 313(5790): 1072-1077. DOI:10.1126/science.1127291 |
| [17] | Brezina E, Prasse C, Wagner M, et al. Why small differences matter:elucidation of the mechanisms underlying the transformation of 2OH- and 3OH-carbamazepine in contact with sand filter material[J]. Environmental Science & Technology, 2015, 49(17): 10449-10456. |
| [18] | Gu Q H, Wu Q P, Zhang J M, et al. Isolation and transcriptome analysis of phenol-degrading bacterium from carbon-sand filters in a full-scale drinking water treatment plant[J]. Frontiers in Microbiology, 2018, 9: 2162. DOI:10.3389/fmicb.2018.02162 |
| [19] | Albers C N, Feld L, Ellegaard-Jensen L, et al. Degradation of trace concentrations of the persistent groundwater pollutant 2, 6-dichlorobenzamide (BAM) in bioaugmented rapid sand filters[J]. Water Research, 2015, 83: 61-70. DOI:10.1016/j.watres.2015.06.023 |
| [20] | Horemans B, Raes B, Vandermaesen J, et al. Biocarriers improve bioaugmentation efficiency of a rapid sand filter for the treatment of 2, 6-dichlorobenzamide-contaminated drinking water[J]. Environmental Science & Technology, 2017, 51(3): 1616-1625. |
| [21] | 国家环境保护总局. 水和废水监测分析方法[M]. (第四版). 北京: 中国环境科学出版社, 2002: 254-268. |
| [22] | Luo C W, Rodriguez-R L M, Konstantinidis K T. MyTaxa:an advanced taxonomic classifier for genomic and metagenomic sequences[J]. Nucleic Acids Research, 2014, 42(8): e73. DOI:10.1093/nar/gku169 |
| [23] | Li D H, Luo R B, Liu C M, et al. MEGAHIT v1.0:A fast and scalable metagenome assembler driven by advanced methodologies and community practices[J]. Methods, 2016, 102: 3-11. DOI:10.1016/j.ymeth.2016.02.020 |
| [24] | Hyatt D, Chen G L, LoCascio P F, et al. Prodigal:prokaryotic gene recognition and translation initiation site identification[J]. BMC Bioinformatics, 2010, 11: 119. DOI:10.1186/1471-2105-11-119 |
| [25] | Fu L M, Niu B F, Zhu Z W, et al. CD-HIT:accelerated for clustering the next-generation sequencing data[J]. Bioinformatics, 2012, 28(23): 3150-3152. DOI:10.1093/bioinformatics/bts565 |
| [26] | Bray N L, Pimentel H, Melsted P, et al. Near-optimal probabilistic RNA-seq quantification[J]. Nature Biotechnology, 2016, 34(5): 525-527. DOI:10.1038/nbt.3519 |
| [27] | Robinson M D, McCarthy D J, Smyth G K. edgeR:a Bioconductor package for differential expression analysis of digital gene expression data[J]. Bioinformatics, 2010, 26(1): 139-140. |
| [28] | Buchfink B, Xie C, Huson D H. Fast and sensitive protein alignment using DIAMOND[J]. Nature Methods, 2015, 12(1): 59-60. DOI:10.1038/nmeth.3176 |
| [29] | Kanehisa M, Sato Y, Morishima K. BlastKOALA and GhostKOALA:KEGG tools for functional characterization of genome and metagenome sequences[J]. Journal of Molecular Biology, 2016, 428(4): 726-731. DOI:10.1016/j.jmb.2015.11.006 |
| [30] | 严煦世, 范瑾初. 给水工程[M]. (第四版). 北京: 中国建筑工业出版社, 1999: 315-318. |
| [31] | de Vet W W J M, Kleerebezem R, van der Wielen P W J J, et al. Assessment of nitrification in groundwater filters for drinking water production by qPCR and activity measurement[J]. Water Research, 2011, 45(13): 4008-4018. DOI:10.1016/j.watres.2011.05.005 |
| [32] | Lee C O, Boe-Hansen R, Musovic S, et al. Effects of dynamic operating conditions on nitrification in biological rapid sand filters for drinking water treatment[J]. Water Research, 2014, 64: 226-236. DOI:10.1016/j.watres.2014.07.001 |
| [33] | Tatari K, Smets B F, Albrechtsen H J. Depth investigation of rapid sand filters for drinking water production reveals strong stratification in nitrification biokinetic behavior[J]. Water Research, 2016, 101: 402-410. DOI:10.1016/j.watres.2016.04.073 |
| [34] | Palomo A, Fowler S J, Gülay A, et al. Metagenomic analysis of rapid gravity sand filter microbial communities suggests novel physiology of Nitrospira spp.[J]. The ISME Journal, 2016, 10(11): 2569-2581. DOI:10.1038/ismej.2016.63 |
| [35] | Shade A, Handelsman J. Beyond the venn diagram:the hunt for a core microbiome[J]. Environmental Microbiology, 2012, 14(1): 4-12. DOI:10.1111/j.1462-2920.2011.02585.x |
| [36] | Feng S, Chen C, Wang Q, et al. Microbial community in a full-scale drinking water biosand filter[J]. Journal of Environmental Biology, 2013, 34(2): 321-324. |
| [37] | Daims H, Lebedeva E V, Pjevac P, et al. Complete nitrification by Nitrospira bacteria[J]. Nature, 2015, 528(7583): 504-509. DOI:10.1038/nature16461 |
| [38] | van Kessel M A H J, Speth D R, Albertsen M, et al. Complete nitrification by a single microorganism[J]. Nature, 2015, 528(7583): 555-559. DOI:10.1038/nature16459 |
| [39] | Koch H, Lucker S, Albertsen M, et al. Expanded metabolic versatility of ubiquitous nitrite-oxidizing bacteria from the genus Nitrospira[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(36): 11371-11376. DOI:10.1073/pnas.1506533112 |
| [40] | Daims H, Nielsen J L, Nielsen P H, et al. In situ characterization of Nitrospira-like nitrite-oxidizing bacteria active in wastewater treatment plants[J]. Applied and Environmental Microbiology, 2001, 67(11): 5273-5284. DOI:10.1128/AEM.67.11.5273-5284.2001 |
| [41] | Kelly D P, Wood A P, Stackebrandt E. Thiobacillus[EB/OL]. https://doi.org/10.1002/9781118960608.gbm00969, 2015-09-14. |
| [42] | Kitagawa W, Takami S, Miyauchi K, et al. Novel 2, 4-dichlorophenoxyacetic acid degradation genes from oligotrophic Bradyrhizobium sp. strain HW13 isolated from a pristine environment[J]. Journal of Bacteriology, 2002, 184(2): 509-518. DOI:10.1128/JB.184.2.509-518.2002 |
| [43] | Sato Y, Monincova M, Chaloupkova R, et al. Two rhizobial strains, Mesorhizobium loti MAFF303099 and Bradyrhizobium japonicum USDA110, encode haloalkane dehalogenases with novel structures and substrate specificities[J]. Applied and Environmental Microbiology, 2005, 71(8): 4372-4379. DOI:10.1128/AEM.71.8.4372-4379.2005 |
| [44] | Abd-Alla M H. Phosphodiesterase and phosphotriesterase in Rhizobium and Bradyrhizobium strains and their roles in the degradation of organophosphorus pesticides[J]. Letters in Applied Microbiology, 1994, 19(4): 240-243. DOI:10.1111/j.1472-765X.1994.tb00953.x |
| [45] | Satsuma K, Masuda M, Sato K. A role of Bradyrhizobium elkanii and closely related strains in the degradation of methoxychlor in soil and surface water environments[J]. Bioscience, Biotechnology, and Biochemistry, 2013, 77(11): 2222-2227. DOI:10.1271/bbb.130439 |
| [46] | Nguyen L N, Nghiem L D, Oh S. Aerobic biotransformation of the antibiotic ciprofloxacin by Bradyrhizobium sp. isolated from activated sludge[J]. Chemosphere, 2018, 211: 600-207. DOI:10.1016/j.chemosphere.2018.08.004 |
| [47] | Qu Y, Spain J C. Biodegradation of 5-nitroanthranilic acid by Bradyrhizobium sp. strain JS329[J]. Applied and Environmental Microbiology, 2010, 76(5): 1417-1422. DOI:10.1128/AEM.02816-09 |
| [48] | Schink B, Deutzmann J S. Methyloglobulus[EB/OL]. https://doi.org/10.1002/9781118960608.gbm01412, 2016-06-29. |
| [49] | Jiang H, Chen Y, Jiang P X, et al. Methanotrophs:multifunctional bacteria with promising applications in environmental bioengineering[J]. Biochemical Engineering Journal, 2010, 49(3): 277-288. |
| [50] | Hedegaard M J, Deliniere H, Prasse C, et al. Evidence of co-metabolic bentazone transformation by methanotrophic enrichment from a groundwater-fed rapid sand filter[J]. Water Research, 2018, 129: 105-114. DOI:10.1016/j.watres.2017.10.073 |
 2019, Vol. 40
2019, Vol. 40


