2. 中国科学院大学, 北京 100049;
3. 湖北工程学院生命科学与技术学院, 孝感 432000
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. College of Life Science and Technology, Hubei Engineering University, Xiaogan 432000, China
磷是地球生物生长和发育必需的营养元素, 不仅作为核酸和磷脂的结构成分, 还直接参与能量代谢和代谢中间体的活化[1].虽然土壤中全磷比较丰富, 但是绝大部分以Ca2-P、Fe-P和Al-P等固定态的磷存在, 难以被植物或微生物吸收利用.而能被植物或微生物直接吸收利用的有效磷较为缺乏[1, 2].土壤有效磷的缺乏不仅会限制植物的生长, 还会影响土壤微生物活性和功能, 尤其是根际微生物.根际定义为:在植物根系表面以及可以直接受根系影响的土壤区域, 是土壤-植物-微生物相互作用的活性界面[3].根际微生物作为土壤生态系统中最活跃的组分, 与土壤生态系统中的物质循环和能量流动密切相关, 对土壤氮素的转化与利用等具有重要作用[4, 5].
反硝化作用作为土壤氮素转化的一个重要过程, 是反硝化微生物在厌氧条件下以硝酸根为电子受体进行呼吸代谢并将其转化为N2O或N2的过程, 是土壤中氮素损失的重要途径[6].反硝化过程主要由4种不同的酶诱导:硝酸还原酶、亚硝酸还原酶、一氧化氮还原酶、氧化亚氮还原酶, 其中, 亚硝酸还原酶基因被广泛用作反硝化微生物的标记基因[7].编码亚硝酸还原酶有两种不同基因型的微生物:一种是nirK基因编码的铜型亚硝酸还原酶, 另一种是nirS基因编码的含细胞色素cd1型的亚硝酸还原酶[8].虽然还未明确这两种亚硝酸盐还原酶在功能上的差异, 但二者催化位点不同, 且不能共存于同种细胞中[9].Heylen等[10]发现nirK普遍存在于α-变形菌纲, 而nirS基因则在β-变形菌纲中较多, 且nirK易发生水平基因转移, nirK与16S rRNA基因系统发育不一致, 而nirS基因在科和属水平上与16S rRNA系统发生一致.Bárta等[11]在对云杉林土壤的研究中发现, nirK反硝化菌丰度与土壤有机碳含量成正比, 但是nirS反硝化菌并未发生这种现象.由此可见, 两种具有亚硝酸还原功能的反硝化类群生理生化特性及其对环境因子的响应是有差异的.
有研究表明, 反硝化作用受多种因素, 如土壤养分状态、水分以及温度等环境因子的影响[12~14].Chen等[12]的研究发现, 单施氮肥、均衡施氮磷钾肥和有机无机肥配施可显著提高nirK和nirS基因丰度, 同时改变其种群结构, 但nirK比nirS响应更敏感.与施化肥相比, 有机肥的施用会显著提高nirK和nirS型反硝化菌丰度和多样性[15].然而有关土壤磷素水平对反硝化微生物的影响, 尤其是同功能的nirK和nirS型反硝化微生物的了解较少.已有的研究表明, 有效磷的缺乏会限制反硝化微生物活性, 磷肥施用可以刺激含nirK基因的反硝化菌生长[16, 17].此外, 土壤磷素有效性还可能通过影响植物生长状况改变土壤氮素水平[18, 19], 从而间接影响反硝化微生物的群落结构及其功能.但是, 具有同种功能的nirK和nirS型反硝化菌对土壤磷水平变化的响应是否一致且其关键调控机制等问题均尚不明确.
因此, 本研究通过采集严重缺磷水稻土, 设置不同施磷水平, 利用水稻盆栽试验和现代微生物分子生物学技术, 探究在水稻种植条件下磷水平对亚硝酸还原酶基因组成与丰度的调控作用, 以期为深入探究施肥对土壤反硝化过程的影响提供理论依据.
1 材料与方法 1.1 供试土壤土壤取自中国科学院桃源农业生态试验站第四纪红色黏土发育的水稻土, 选取长期定位试验缺磷处理土壤(0~20 cm), 采样时间为2016年1月20日.经自然风干并过2 mm筛, 去除石子和植物残体, 室温保存备用.土壤基本化学性质为:有机质3.51%, 全氮2.00 g·kg-1, 全磷0.33 g·kg-1, 全钾10.25 g·kg-1, 速效氮193.20 mg·kg-1, 速效磷3.17 mg·kg-1, 速效钾59.23 mg·kg-1, pH 5.02.
1.2 水稻盆栽试验共设3个处理, 即对照(CK):不施磷肥; 低磷(P1):P添加量为15 mg·kg-1[(NH4)3PO4 70 mg·kg-1]; 高磷(P2):P添加量为30 mg·kg-1[(NH4)3PO4 140 mg·kg-1].所有处理均施加尿素100 mg·kg-1和钾素100 mg·kg-1, 所用肥料为尿素和氯化钾.将肥料与土壤充分混匀后分装到塑料桶(直径16 cm×高20 cm)中, 每桶装土3 kg, 设置4个重复.每桶中放置了两个尼龙网袋(孔径50 μm, 可固定根系生长范围, 但不影响土壤水肥正常扩散), 尺寸分别为直径8 cm×高15 cm和直径6 cm×高15 cm.前者作为根袋用于水稻种植, 后者不种水稻用于非根际土壤采集, 两袋间相隔1 cm.袋中均填充拌肥土壤.
淹水一周后移栽水稻(苗龄18 d)幼苗到各根袋中, 每袋移栽水稻苗一兜含5棵幼苗.水稻品种为五山丝苗(编号为粤审稻2009031).水稻生长至分蘖盛期(苗龄52 d), 破坏性采集根际和非根际土壤.根际土:将直径8 cm的根袋取出, 除去尼龙网袋后取根系密集分布区域土样为根际土, 分别标注为CK-R、P1-R和P2-R.非根际土:将直径6 cm的土柱取出, 去掉尼龙网和上下端各4 cm后用灭菌器具取土柱中心样品为非根际土, 分别标注为CK-B、P1-B和P2-B.采集的土壤样品一部分存放在4℃用于Olsen-P、铵态氮、硝态氮和溶解性有机碳(DOC)的检测, 另一部分存放于-80℃冰箱用于土壤DNA分析.
1.3 有效磷、硝态氮、铵态氮、DOC测定采用0.5 mol·L-1 K2SO4溶液共提法浸提, 使用连续流动注射分析仪(FIAstar 5000Analyzer)测定土壤硝态氮和铵态氮含量, 使用有机碳分析仪(TOC-VWP)测定土壤DOC含量[20].采用0.5 mol·L-1 NaHCO3浸提、钼锑抗比色法, 使用紫外可见分光光度计(UV-2600)测定土壤有效磷(Olsen-P)含量[20].
1.4 土壤DNA提取和荧光定量PCR土壤微生物DNA的提取方法参照SDS-GITC-PEG方法[12], 并进行了适当修改.用1%的琼脂糖凝胶电泳检测所提DNA片段大小, 并用核酸蛋白测定仪(Nanodrop-ND-1000 UV-vis分光光度计)测定DNA的浓度及质量.
分别利用引物对nirK-876F/1040R、nirS-cd3aF/R3cd进行nirK、nirS基因片段扩增(表 1), 仪器为ABI 7900(Massachusetts, USA).荧光定量PCR反应体系为10μL, 包括上下游引物(10 μmol·L-1)各0.3μL, SYBR Green Ⅱ(Takara)5μL, Rox Ⅰ(Takara)0.2μL, DNA模板1 μL(5 ng·μL-1), ddH2O 3.2μL.nirK基因扩增程序为:95℃预变性30 s; 40个循环(95℃, 5 s; 60℃, 30 s; 72℃, 10 s); 添加溶解曲线程序(95℃, 15 s; 60℃, 15 s; 95℃, 15 s).nirS基因扩增程序为:95℃预变性30 s; 40个循环(95℃, 5 s; 60℃, 30 s; 72℃, 30 s); 添加溶解曲线程序(95℃, 15 s; 60℃, 15 s; 95℃, 15 s).荧光定量PCR标准曲线的建立参照Chen[12]的方法.分别选择含有nirK和nirS目的片段的质粒, 以10倍梯度稀释法配制定量标准曲线.标准曲线范围在102~108之间, R2>0.99, 扩增效率在90%~110%之间.
|
|
表 1 nirK和nirS基因引物1) Table 1 Primers for nirK and nirS genes |
1.5 PCR扩增和高通量测序
利用带有8个碱基序列barcode的nirK-F1aCu/R3Cu和nirS-cd3aF/R3cd分别对nirK和nirS基因进行扩增(表 1), 所用仪器为Eppendorf Mastercycler(Model-5333).PCR反应体系为50μL, 包括上下游引物(10 μmol·L-1)各2μL, 2×PCR mix(Hot Start PCR Enzyme, Takara)25μL, DNA模板2 μL(20 ng·μL-1), ddH2O补至50μL.nirK基因扩增程序为:98℃预变性2 min, 30个循环(98℃, 10 s; 58℃, 30 s; 72℃, 45 s), 最后72℃延伸10 min.nirS基因扩增程序为:98℃预变性2 min, 30个循环(98℃, 10 s; 54℃, 30 s; 72℃, 45 s), 最后72℃延伸10 min.
PCR产物采用琼脂糖凝胶纯化回收试剂盒(Axygen Biosciences, Union City, CA, USA)进行纯化, 然后用QuantiFluorTM-ST(Promega, Wisconsin, USA)进行定量.将样品等质量混匀后建库, 利用Illumina MiseqMiseq PE300测序平台(Illumina, San Diego, USA)进行测序分析(上海美吉生物医药科技有限公司, 中国).
1.6 数据分析方法采用Excel 2016和SPSS 25.0对Olsen-P、铵态氮、硝态氮、DOC以及基因拷贝数进行数据统计分析, 采用单因素(one-way ANOVA)和Duncan法进行方差分析和多重检验(α=0.05), 并用Pearson法对土壤养分含量和基因拷贝数进行相关性分析.在美吉生物云平台(http://www. i-sanger.com/)进行高通量测序数据质控, 包括数据去杂和嵌合体去除, 然后分别以83%和82%序列相似性进行nirK和nirS基因的OTU划分[25], 并进行群落OTU水平上的相似性分析(ANOSIM分析).应用QIIME软件对得到的已划分OTU的去杂序列进行后续物种注释.每个OTU挑选一条代表序列与物种注释模板进行比对, 进行物种注释, 比对方法为Blast.所用模板为FunGene数据库(http://fungene.cme.msu.eds)下载的各个功能基因的序列及物种信息.利用Origin 9、Excel 2016以及Canoco 5.0软件制图
2 结果与分析 2.1 土壤养分含量变化不同磷水平的土壤养分含量变化存在差异(表 2).在非根际土壤中, 与严重缺磷的CK-B相比, 磷肥的施用显著增加了P1-B和P2-B土壤中的Olsen-P含量(P<0.05).根际土壤也呈现出同样的趋势, 表现为P2-R>P1-R>CK-R.与非根际相比, 各处理根际土壤中Olsen-P含量均呈现显著下降(P<0.05), 且随着磷水平的增加, 根际与非根际土壤之间的差异越来越大, 表现为CK-R的Olsen-P含量比CK-B低0.37 mg·kg-1, 而P1-R和P2-R处理的Olsen-P含量分别比P1-B和P2-B低0.45 mg·kg-1和2.08 mg·kg-1.
|
|
表 2 不同磷水平处理水稻土养分含量变化1) Table 2 Nutrient concentrations of three phosphorus levels in rhizosphere and bulk paddy soils |
在非根际土壤中, 2个磷添加处理的DOC含量显著低于CK处理(P<0.05), 说明施磷后, 土壤中的微生物消耗了更多的DOC.在根际土中, 仅P2-R的DOC含量显著低于CK-R(P<0.05), 而P1-R与CK-R无显著差异(P>0.05).植物根系的生长导致各处理根际土的DOC含量均高于非根际, 且随着磷水平的提高, 其增加的幅度也显著提高, 说明施磷后, 水稻的种植会显著提高根际土壤DOC含量.
NH4+-N含量在非根际的3个处理之间没有显著差异(P>0.05), 但在根际土中差异显著(P<0.05), 表现为P2-R<P1-R<CK-R.与非根际相比, 不同磷水平条件下根际土壤NH4+-N含量均显著降低, 且随着磷水平的提高, 其减少的幅度也越高, 说明磷的添加加速了根际土壤NH4+-N的转化.
NO3--N含量在根际和非根际的3个磷水平处理间都没有显著性差异(P>0.05).
2.2 nirK和nirS基因丰度变化非根际土中, 随着磷水平的提高, 水稻土中的亚硝酸还原酶基因丰度有不同程度地增加.相较于CK, 从低磷到高磷处理的nirK基因拷贝数增加幅度小, 即P2-B>P1-B>CK-B.其中P1-B与CK-B之间无显著差异(P>0.05), 但P2-B处理的nirK基因拷贝数平均约为CK-B的167%, 达到显著水平[P<0.05, 图 1(a)].与CK-B相比, P1-B的nirS基因拷贝数也无显著变化(P>0.05), 但是P2-B处理的nirS基因丰度是CK-B的2倍多[图 1(b)].不同磷水平的根际土相比, P2-R的nirK丰度最高, 其次是CK-R和P1-R, 但P2-R与CK-R以及CK-R和P1-R之间均无显著差异(P>0.05).与nirK不同, P1-R中的nirS丰度与CK-R无显著差异(P>0.05), 但P2-R的nirS丰度是CK-R的3倍以上[P<0.05, 图 1(b)].与其非根际相比, 水稻根系的生长导致CK根际土具有较高的nirK丰度, 但在低磷和高磷条件下, 根际和非根际之间无显著差异(P>0.05).与nirK不同, 在低磷和CK处理中根>际和非根际nirS的丰度无显著差异(P>0.05), 但在高磷处理中, 根际土nirS基因拷贝数是非根际的2.46倍.上述结果说明在高磷条件下根际环境对含nirK和nirS基因反硝化微生物丰度的影响更大, 且nirS种群响应更敏感.此外, nirK和nirS基因丰度与Olsen-P的相关性分析显示(表 3), 不管是根际还是非根际土, nirS基因的丰度与Olsen-P的相关性均明显高于nirK, nirK和nirS在根际土中的相关系数分别为0.273(P>0.05)和0.519(P>0.05), 在非根际土中的相关系数分别为0.791(P<0.01)和0.919(P<0.001), 进一步说明nirS种群丰度对土壤磷浓度的变化响应更敏感.
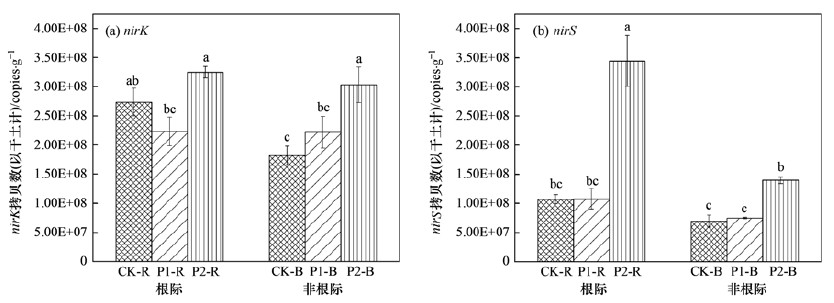
|
图柱代表每处理4个重复的测定平均值, 误差线代表标准误; 不同字母代表差异显著(P<0.05) 图 1 不同磷水平处理水稻土根际和非根际nirK、nirS基因丰度 Fig. 1 The nirK and nirS gene abundance for three phosphorus levels in rhizosphere and bulk paddy soils |
|
|
表 3 土壤养分含量与nirK和nirS基因丰度的Pearson相关系数1) Table 3 Pearson's correlation coefficients for the relationships between nutrient concentrations and the abundance of nirK and nirS in rhizosphere and bulk paddy soils |
2.3 含nirK和含nirS反硝化细菌种群组成结构变化
nirK和nirS有效序列分别为894 856和881 424条, 平均每个样品37 286和36 726条(表 4).分别以83%和82%序列相似性划分nirK和nirS基因的OTU, 获得750和248个OTU.通过计算各处理根际和非根际土壤nirK和nirS多样性指数发现, 各处理nirK和nirS种群的α-多样性指数无显著差异(P>0.05).
|
|
表 4 nirK和nirS基因高通量测序有效序列数和α-多样性指数 Table 4 High-throughput sequencing and α-diversity index of nirK and nirS genes |
由测序结果(图 2)可知, nirK种群的优势属Bradyrhizobium、Sinorhizobium、Alcaligenes、Unclassified-Proteobacteria、Phaeobacter、Pseudomonas和Rhizobium均归属于变形菌门, 占nirK型反硝化菌丰度的66.27%~88.22%.这些属在3个磷水平处理间以及根际与非根际间均存在显著差异(P<0.05).在非根际土中, P1-B的Alcaligenes和Proteobacteria的相对丰度分别从占比12.16%和8.29%减少到8.07%和4.00%.与P1-B相比, P2-B的Bradyrhizobium和Sinorhizobium的相对丰度略有上升, 从38.25%和8.36%增加到45.40%和13.02%.
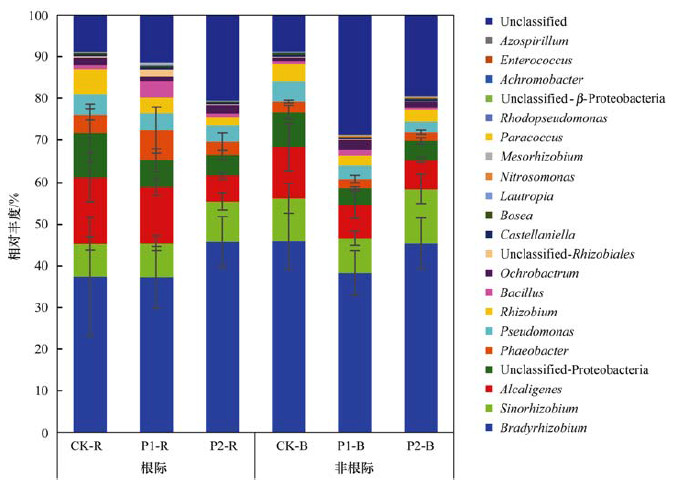
|
图柱代表每处理4个重复的测定平均值, 误差线代表标准差 图 2 不同磷水平水稻土nirK型反硝化细菌属分类水平物种组成 Fig. 2 Composition of nirK-containing bacterial genera at three phosphorus levels in rhizosphere and bulk paddy soils |
不同磷水平条件下的根际土nirK种群组成结构也存在差异(P<0.05), 尤其是P2-R处理.例如, 与CK-R相比, P2-R处理中的Bradyrhizobium和Sinorhizobium的相对丰度分别从37.3%和8%增加到45.75%和9.7%, 而Alcaligenes从15.82%降低到6.17%.但与CK-R相比, P1-R中nirK种群组成没有发生明显变化(P>0.05).
在严重缺磷条件下, 水稻根系生长导致根际CK-R中Bradyrhizobium和Sinorhizobium的相对丰度显著低于非根际土CK-B(P<0.05), 而Alcaligenes和Rhizobium显著增加(P<0.05).在低磷条件下, 根际土P1-R中Alcaligenes、Rhizobium和Phaeobacter相对丰度均显著高于非根际P1-B(P<0.05).但在高磷条件下, 含nirK基因反硝化微生物组成结构在根际和非根际之间无显著差异(P>0.05).
由nirS基因属水平上的注释结果可知(图 3), 磷的添加显著改变了nirS型反硝化菌的群落结构(P<0.05).在非根际土中, P1-B和P2-B群落组成结构相似, 但与CK-B存在显著差异(P<0.05).与CK-B相比, P2-B高磷处理的unclassified组分的相对丰度从24.19%增加到53.56%, 但Pseudomonas和Halomonas的相对丰度减少了将近一半, Dechlorospirillum从3.83%减少到了0.5%以下.其中, 部分菌属在高磷水平下的变化更明显, Polymorphum在CK-B中的相对丰度为11.02%, 而在P1-B和P2-B处理中仅占7.25%和4.39%, Unclssified-Proteobacteria由CK-B处理的10.14%分别减少到6.10%和3.71%.

|
图柱代表每处理4个重复的测定平均值, 误差线代表标准差 图 3 不同磷水平水稻土nirS型反硝化细菌属分类水平物种组成 Fig. 3 Composition of nirS-containing denitrifiers at the genus level for three phosphorus levels in rhizosphere and bulk paddy soils |
磷的添加同样导致根际土壤中nirS种群的结构发生了显著变化(P<0.05), 尤其是高磷处理.如与CK-R相比, P1-R和P2-R处理中Dechlorospirillum的相对丰度分别平均减少了9.15%和27.39%, 而unclassified组分相对丰度分别增加了4.81%和27.53%.
不同磷水平条件下的水稻根系生长均导致其根际土中的nirS种群组成结构显著不同于其非根际土壤(P<0.05), 主要体现在根际土中Pseudomonas和Dechlorospirillum的相对丰度大幅提高, 而Dinoroseobacter、Halomonas、Polymorphum和Unclassified-Proteobacteria等种群比例相对降低.但是在不同磷水平条件下根际与非根际间的差异有差别.与CK相比, P1和P2处理根际与非根际土之间Pseudomonas相对丰度的差值变大, 而随着磷含量的增加, Dechlorospirillum、Halomonas、Polymorphum和Unclassified-Proteobacteria在根际与非根际之间的差异越来越小.如CK-R的Dechlorospirillum相对丰度比CK-B高34.69%, 而在P1和P2处理中, 根际与非根际间仅分别相差28.86%和10.78%.
通过PCA分析进一步探讨在严重缺磷的稻田土壤中, 磷水平对nirK和nirS型反硝化细菌种群结构的影响(图 4).由图 4可知, nirK基因的根际和非根际群落样本聚集在一起[图 4(a)], 而nirS的根际与非根际样品在轴1上分开[图 4(b)], 说明水稻土含nirS基因的反硝化微生物群落结构在根际与非根际间有明显差异, 而含nirK基因的反硝化微生物根际与非根际间群落结构相似.同时, nirK基因的P1-B、P2-B与CK-B在轴1上分开, 但是P1-B与P2-B聚集在一起; P2-R与CK-R在轴1上分开, 但是P1-R与CK-R聚集在一起[图 4(a)].而不管是根际还是非根际, nirS基因的3个磷水平都能在轴2上较好地分开, 尤其是CK-B与P2-B, CK-R与P2-R[图 4(b)].说明不管是根际还是非根际, 含nirK和nirS基因微生物的群落结构对磷添加都有明显的响应, 尤其是高磷水平, 但是nirS基因对磷添加的响应比nirK更敏感.此外, nirK和nirS种群结构的相似性分析(ANOSIM)结果显示, 施磷后, 含nirK和nirS基因微生物的群落结构都发生了极显著的变化(P<0.01), 但是nirS基因处理间的差异(R=0.639 1)比nirK(R=0.329 1)更大, 进一步说明nirS种群结构对土壤磷浓度的变化响应更敏感.
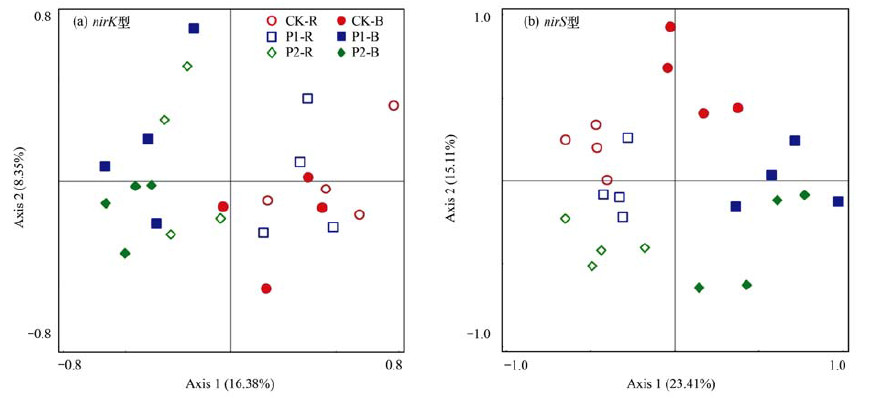
|
图 4 不同磷水平水稻土nirK和nirS型反硝化细菌OTU水平PCA分析 Fig. 4 Principal component analysis of nirK- and nirS- containing bacterial OTU at three phosphorus levels in rhizosphere and bulk paddy soils |
磷是重要的生命元素, 是核苷酸和能量代谢必不可少的成分, 所以土壤磷的缺乏不仅限制植物生长[1], 同时也影响土壤微生物繁衍和功能[26, 27].但是, 对于在缺磷环境下土壤磷如何调控功能微生物种群, 尤其是具有相同功能的微生物种群缺乏清晰的认识.本研究发现, 在严重缺磷的水稻土上施用磷肥, 土壤亚硝酸还原酶基因nirK和nirS所代表的反硝化微生物种群间存在明显的差异性响应.不管是根际还是非根际土, 含nirS的微生物种群均对施磷水平表现敏感, 相比之下, 含nirK基因的微生物组成结构和丰度变异较小.以往的研究发现, 不同环境下, 共存的nirK和nirS反硝化菌群落构建规则存在差异[28], nirK和nirS在土壤生态系统中可能占据着不同的生态位[29].其中, nirK种群对生境的特异选择性更强, 对环境变异的响应比nirS种群更敏感[28].但也有研究发现nirS对pH的改变比nirK敏感, 尤其是在pH=4.7的阈值下, nirS的转录过程受到严重抑制, 酶的数量及活性明显降低[30, 31].因此, nirK和nirS种群对不同生境变化的响应可能存在差异.本研究中, 增加土壤磷含量, 尤其是高磷(P2)水平引起根际和非根际土中nirS基因丰度和部分种群的相对丰度成倍增加或减少, 表明这些组分中的一个或多个物种对磷含量响应敏感, 但nirK的丰度和种群相对丰度的变化幅度明显小于nirS.虽然人们无法全部了解这些对磷响应敏感的物种, 但从已注释物种, 可以推测它们受磷调控可能是因为磷的添加促进部分反硝化菌的生长繁殖, 如nirS种群中的Halomonas, 在缺磷条件下严重制约其生长繁殖[32].Allers等[33]和Sánchez等[34]的研究也表明, 在海水中, 磷添加加快浮游细菌Rhodobacteraceae的生长速率, 而本研究发现添加磷肥显著增加该细菌目中Dinoroseobacter和Paracoccus等属的丰度.此外, 本实验还发现, 根际土中nirS基因对磷的响应比nirK更敏感, 尤其是在高磷(P2-R)水平下, nirS基因基因拷贝数甚至达到CK-R的3倍以上.Yin等[35]在对潮土棕壤的研究中也发现, nirS基因群落结构受到磷浓度的调控, 而nirK种群未发现这种影响.上述结果进一步说明淹水水稻土中含nirS反硝化种群对土壤磷含量响应更为敏感, 尤其是高磷水平, 且在水稻根际和非根际都表现出相同的趋势.
与非根际土壤主要受磷水平影响相比, 水稻根际土壤不仅受磷的影响, 同时还受水稻植株根系分泌物制约.一般情况下, 在缺磷土壤上施用磷肥显著促进植物生长的同时也改善根际环境和增加根系分泌物, 从而影响根际微生物组成与活性[36, 37].本研究发现, 含nirS基因种群受到根际的显著影响, 不论是基因丰度还是群落结构, 根际与非根际之间都存在差异, 而nirK几乎没有显著变化.研究表明, nirS型反硝化菌容易在好氧土壤中聚集[38], 而水稻根系细胞的通气组织可以分泌氧气促进nirS型反硝化菌数量.但是nirS基因丰度在根际与非根际间的显著性差异只发生在高磷处理, 这可能是因为, 高磷水平下植物生长最好, 根系活性最高, 既需要从土壤中吸收更多的养分, 又产生更多的根系分泌物, 造成了根际与非根际较大的养分差异和氧化还原条件[19].此外, 还有研究认为, 植物根系分泌物的量会影响根际微生物的数量, 而根系分泌物的类型则会影响根际微生物的群落结构[39].因此, 含nirS基因的群落结构在根际与非根际间具有显著性差异可能是因为水稻根系分泌物能诱导和刺激特殊种群的微生物在根际定殖[36], 以往的研究也发现, 根系分泌物提供的碳源和氮源能促进含nirS基因的微生物在根际生长, 如Pseudomonas和Dechlorospirillum[36, 40, 41].因此, 含nirS反硝化菌对水稻生长的响应比nirK反硝化菌更加敏感.
4 结论在严重缺磷水稻土中加施磷肥会显著提高反硝化菌数量, 并改变反硝化菌种群组成结构, 且根际土响应更加敏感.与含nirK反硝化细菌相比, 含nirS反硝化细菌种群对磷水平和水稻生长的响应更加敏感.
| [1] | Rausch C, Bucher M. Molecular mechanisms of phosphate transport in plants[J]. Planta, 2002, 216(1): 23-37. DOI:10.1007/s00425-002-0921-3 |
| [2] | Holford I C R. Soil phosphorus:its measurement, and its uptake by plants[J]. Australian Journal of Soil Research, 1997, 35(2): 227-240. DOI:10.1071/S96047 |
| [3] |
陆雅海, 张福锁. 根际微生物研究进展[J]. 土壤, 2006, 38(2): 113-121. Lu Y H, Zhang F S. The advances in rhizosphere microbiology[J]. Soils, 2006, 38(2): 113-121. DOI:10.3321/j.issn:0253-9829.2006.02.001 |
| [4] | Philippot L, Hallin S, B?rjesson G, et al. Biochemical cycling in the rhizosphere having an impact on global change[J]. Plant and Soil, 2009, 321(1-2): 61-81. DOI:10.1007/s11104-008-9796-9 |
| [5] | Dotaniya M L, Meena V D. Rhizosphere effect on nutrient availability in soil and its uptake by plants:a review[J]. Proceedings of the National Academy of Sciences, India Section B:Biological Sciences, 2015, 85(1): 1-12. DOI:10.1007/s40011-013-0297-0 |
| [6] |
贺纪正, 张丽梅. 土壤氮素转化的关键微生物过程及机制[J]. 微生物学通报, 2013, 40(1): 98-108. He J Z, Zhang L M. Key processes and microbial mechanisms of soil nitrogen transformation[J]. Microbiology China, 2013, 40(1): 98-108. |
| [7] | Zhou S L, Huang T L, Zhang C H, et al. Illumina MiSeq sequencing reveals the community composition of nirS-type and nirK-type denitrifiers in Zhoucun reservoir-a large shallow eutrophic reservoir in northern China[J]. RSC Advances, 2016, 6(94): 91517-91528. DOI:10.1039/C6RA18017E |
| [8] | Zumft W G. Cell biology and molecular basis of denitrification[J]. Microbiology and Molecular Biology Reviews, 1997, 61(4): 533-616. |
| [9] |
郭丽芸, 时飞, 杨柳燕. 反硝化菌功能基因及其分子生态学研究进展[J]. 微生物学通报, 2011, 38(4): 583-590. Guo L Y, Shi F, Yang L Y. Advances in functional genes and molecular ecology in denitrifiers[J]. Microbiology China, 2011, 38(4): 583-590. |
| [10] | Heylen K, Gevers D, Vanparys B, et al. The incidence of nirS and nirK and their genetic heterogeneity in cultivated denitrifiers[J]. Environmental Microbiology, 2006, 8(11): 2012-2021. DOI:10.1111/emi.2006.8.issue-11 |
| [11] | Bárta J, Melichová T, Vaněk D, et al. Effect of pH and dissolved organic matter on the abundance of nirK and nirS denitrifiers in spruce forest soil[J]. Biogeochemistry, 2010, 101(1-3): 123-132. DOI:10.1007/s10533-010-9430-9 |
| [12] | Chen Z, Luo X Q, Hu R G, et al. Impact of long-term fertilization on the composition of denitrifier communities based on nitrite reductase analyses in a paddy soil[J]. Microbial Ecology, 2010, 60(4): 850-861. |
| [13] | Chen Z, Liu J B, Wu M N, et al. Differentiated response of denitrifying communities to fertilization regime in paddy soil[J]. Microbial Ecology, 2012, 63(2): 446-459. |
| [14] | Liu J B, Hou H J, Sheng R, et al. Denitrifying communities differentially respond to flooding drying cycles in paddy soils[J]. Applied Soil Ecology, 2012, 62: 155-162. DOI:10.1016/j.apsoil.2012.06.010 |
| [15] |
尹昌, 范分良, 李兆君, 等. 长期施用有机和无机肥对黑土nirS型反硝化菌种群结构和丰度的影响[J]. 环境科学, 2012, 33(11): 3967-3975. Yin C, Fan F L, Li Z J, et al. Influences of long-term application of organic and inorganic fertilizers on the composition and abundance of nirS-type denitrifiers in black soil[J]. Environmental Science, 2012, 33(11): 3967-3975. |
| [16] | Sheng R, Meng D L, Wu M N, et al. Effect of agricultural land use change on community composition of bacteria and ammonia oxidizers[J]. Journal of Soils and Sediments, 2013, 13(7): 1246-1256. DOI:10.1007/s11368-013-0713-3 |
| [17] | He M Z, Dijkstra F A. Phosphorus addition enhances loss of nitrogen in a phosphorus-poor soil[J]. Soil Biology and Biochemistry, 2015, 82: 99-106. DOI:10.1016/j.soilbio.2014.12.015 |
| [18] | Mori T, Ohta S, Ishizuka S, et al. Phosphorus application reduces N2O emissions from tropical leguminous plantation soil when phosphorus uptake is occurring[J]. Biology and Fertility of Soils, 2014, 50(1): 45-51. DOI:10.1007/s00374-013-0824-4 |
| [19] | Duan Y H, Shi X J, Li S L, et al. Nitrogen use efficiency as affected by phosphorus and potassium in long-term rice and wheat experiments[J]. Journal of Integrative Agriculture, 2014, 13(3): 588-596. DOI:10.1016/S2095-3119(13)60716-9 |
| [20] | 鲍士旦. 土壤农化分析[M]. 第三版. 北京: 中国农业出版社, 2000: 49-56, 79-83. |
| [21] | Henry S, Baudoin E, López-Gutiérrez J C, et al. Quantification of denitrifying bacteria in soils by nirK gene targeted real-time PCR[J]. Journal of Microbiological Methods, 2004, 59(3): 327-335. DOI:10.1016/j.mimet.2004.07.002 |
| [22] | Hallin S, Lindgren P E. PCR detection of genes encoding nitrite reductase in denitrifying bacteria[J]. Applied and Environmental Microbiology, 1999, 65(4): 1652-1657. |
| [23] | Michotey V, Méjean V, Bonin P. Comparison of methods for quantification of cytochrome cd1-denitrifying bacteria in environmental marine samples[J]. Applied and Environmental Microbiology, 2000, 66(4): 1564-1571. DOI:10.1128/AEM.66.4.1564-1571.2000 |
| [24] | Throbäck I N, Enwall K, Jarvis Å, et al. Reassessing PCR primers targeting nirS, nirK and nosZ genes for community surveys of denitrifying bacteria with DGGE[J]. FEMS Microbiology Ecology, 2004, 49(3): 401-417. |
| [25] | Palmer K, Biasi C, Horn M A. Contrasting denitrifier communities relate to contrasting N2O emission patterns from acidic peat soils in arctic tundra[J]. The ISME Journal, 2012, 6(5): 1058-1077. DOI:10.1038/ismej.2011.172 |
| [26] | Cleveland C C, Townsend A R. Nutrient additions to a tropical rain forest drive substantial soil carbon dioxide losses to the atmosphere[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(27): 10316-10321. DOI:10.1073/pnas.0600989103 |
| [27] | Wei X M, Hu Y J, Peng P Q, et al. Effect of P stoichiometry on the abundance of nitrogen-cycle genes in phosphorus-limited paddy soil[J]. Biology and Fertility of Soils, 2017, 53(7): 767-776. DOI:10.1007/s00374-017-1221-1 |
| [28] | Jones C M, Hallin S. Ecological and evolutionary factors underlying global and local assembly of denitrifier communities[J]. The ISME Journal, 2010, 4(5): 633-641. DOI:10.1038/ismej.2009.152 |
| [29] | Tang Y Q, Zhang X Y, Li D D, et al. Impacts of nitrogen and phosphorus additions on the abundance and community structure of ammonia oxidizers and denitrifying bacteria in Chinese fir plantations[J]. Soil Biology and Biochemistry, 2016, 103: 284-293. DOI:10.1016/j.soilbio.2016.09.001 |
| [30] | Brenzinger K, Dörsch P, Braker G. pH-driven shifts in overall and transcriptionally active denitrifiers control gaseous product stoichiometry in growth experiments with extracted bacteria from soil[J]. Frontiers in Microbiology, 2015, 6: 961. |
| [31] | Herold M B, Giles M E, Alexander C J, et al. Variable response of nirK and nirS containing denitrifier communities to long-term pH manipulation and cultivation[J]. FEMS Microbiology Letters, 2018, 365(7), doi: 10.1093/femsle/fny035. https://www.ncbi.nlm.nih.gov/pubmed/29471521 |
| [32] | Sarilmiser H K, Ates O, Ozdemir G, et al. Effective stimulating factors for microbial levan production by Halomonas smyrnensis AAD6T[J]. Journal of Bioscience and Bioengineering, 2015, 119(4): 455-463. DOI:10.1016/j.jbiosc.2014.09.019 |
| [33] | Allers E, Gómez-Consarnau L, Pinhassi J, et al. Response of Alteromonadaceae and Rhodobacteriaceae to glucose and phosphorus manipulation in marine mesocosms[J]. Environmental Microbiology, 2007, 9(10): 2417-2429. DOI:10.1111/emi.2007.9.issue-10 |
| [34] | Sánchez O, Koblížek M, Gasol J M, et al. Effects of grazing, phosphorus and light on the growth rates of major bacterioplankton taxa in the coastal NW Mediterranean[J]. Environmental Microbiology Reports, 2017, 9(3): 300-309. DOI:10.1111/1758-2229.12535 |
| [35] | Yin C, Fan F L, Song A L, et al. Different denitrification potential of aquic brown soil in northeast China under inorganic and organic fertilization accompanied by distinct changes of nirS- and nirK-denitrifying bacterial community[J]. European Journal of Soil Biology, 2014, 65: 47-56. DOI:10.1016/j.ejsobi.2014.09.003 |
| [36] | Wu H L, Wang X Z, He X J, et al. Effects of root exudates on denitrifier gene abundance, community structure and activity in a micro-polluted constructed wetland[J]. Science of the Total Environment, 2017, 598: 697-703. DOI:10.1016/j.scitotenv.2017.04.150 |
| [37] |
罗永清, 赵学勇, 李美霞. 植物根系分泌物生态效应及其影响因素研究综述[J]. 应用生态学报, 2012, 23(12): 3496-3504. Luo Y Q, Zhao X Y, Li M X. Ecological effect of plant root exudates and related affecting factors:a review[J]. Chinese Journal of Applied Ecology, 2012, 23(12): 3496-3504. |
| [38] | Coyne M S, Arunakumari A, Averill B A, et al. Immunological identification and distribution of dissimilatory heme cd1 and nonheme copper nitrite reductases in denitrifying bacteria[J]. Applied and Environmental Microbiology, 1989, 55(11): 2924-2931. |
| [39] |
朱丽霞, 章家恩, 刘文高. 根系分泌物与根际微生物相互作用研究综述[J]. 生态环境, 2003, 12(1): 102-105. Zhu L X, Zhang J E, Liu W G. Review of studies on interactions between root exudates and rhizopheric microorganisms[J]. Ecology and Environment, 2003, 12(1): 102-105. DOI:10.3969/j.issn.1674-5906.2003.01.025 |
| [40] | Rovira D A. Interactions between plant roots and soil microorganisms[J]. Annual Review of Microbiology, 1965, 19: 241-266. DOI:10.1146/annurev.mi.19.100165.001325 |
| [41] | Phillips L A, Armstrong S A, Headley J V, et al. Shifts in root-associated microbial communities of Typha latifolia growing in naphthenic acids and relationship to plant health[J]. International Journal of Phytoremediation, 2010, 12(8): 745-760. DOI:10.1080/15226510903535106 |
 2019, Vol. 40
2019, Vol. 40


