2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
作为一种新型的环境污染物[1], 抗生素抗性基因(antibiotic resistance genes, ARGs)所导致的抗生素耐药性问题已在全世界范围内引起极大的关注[2, 3].抗生素耐药性是一种自然的现象[4], 然而自抗生素量产以来人类在医疗以及养殖业中无监管地滥用抗生素使得这一现象飞速发展, 最终导致严重的耐药性问题[2, 5].据报道, 引起2011年暴发的“毒黄瓜”事件的致病菌因携带多种耐药基因, 导致抗生素治疗失效, 最终造成33人死亡[3].如果不加以控制, 人类将进入无药可用的“后抗生素时代”[2].
除了治疗与预防疾病外, 抗生素因具有促进生长和提高饲料的转化率的作用而被广泛应用在养殖业中.抗生素在畜禽肠道内诱导出耐药菌株, 此外大部分抗生素难以被充分吸收利用, 耐药菌和未被代谢的抗生素(25%~70%)一并随排泄物进入周围环境[6, 7].残留的抗生素对环境微生物群落进化施加选择压力, 粪源抗性基因则有可能通过基因水平迁移(horizontal gene transfer, HGT)机制进入环境微生物[8, 9].环境微生物中的抗性基因还会迁移进入人类致病菌中, 对人类健康造成威胁[10].不同动物的生长周期和用药量不同, 粪便中ARGs的种类和丰度也不同[11, 12], 因而不同动物粪便对其受纳环境的影响程度也有可能不同[13].然而已有的研究大多针对单一动物[8, 14]或者有限的几种常报道的抗性基因[9, 11], 对不同类型养殖场土壤中抗生素抗性基因的整体情况还鲜见报道.
四川是我国的农业大省, 其肉猪出栏量居全国各省市之首.据报道, 2010年四川省畜禽粪便排放量为18.8亿t, 总污染量2.1亿t, 略低于河南省, 排在全国第二位[15].此外, 四川省也是我国兽用抗生素消费大省[16].紫色土丘陵区是四川盆地重要的农业区域, 其土层浅薄, 抗侵蚀能力弱, 易发生水土流失与污染物迁移[17].然而目前关于该地区养殖场土壤中抗生素抗性基因的相关研究鲜有报道.因此, 本研究选取紫色土丘陵区3个不同动物类型的养殖场土壤为研究对象, 采用高通量荧光定量技术对抗生素抗性基因丰度和多样性的整体情况进行分析, 以期为紫色土丘陵区养殖场土壤中耐药基因污染状况与生态风险的评价及其污染防治措施的制定提供基础数据和科学依据.
1 材料与方法 1.1 样品采集及处理所研究的养殖场位于四川省绵阳市盐亭县内, 均为小型养殖场.养牛场土壤为牛粪堆放地土壤, 仔鸡养殖场土壤为养殖场内仔鸡活动地土壤, 养猪场土壤为猪场排污沟渠周围土壤.样品采集时间为2017年9月, 运用5点采样法, 采集这3家畜禽养殖场表层0~10 cm土壤, 现场混匀, 放入冰盒内带回实验室保存于-20℃冰箱, 用于提取样品DNA.
1.2 土壤DNA提取称取0.25 g土壤鲜样按照PowerSoil DNA Isolation Kit (MoBio Laborataries Inc. CA, 美国)试剂盒的操作步骤提取土壤总DNA, 随后用1%的琼脂糖凝胶电泳检测. DNA样品浓度和纯度用超微量分光光度计(NanoDrop ND-1000, NanoDrop Technologies Inc., DE, 美国)检测, 所提取的DNA样品A260/A280均在1.8~2.0之间. DNA样品用无菌水统一稀释至20 ng· μL-1后置于-20℃冰箱冷冻保存待测.
1.3 抗生素抗性基因测定采样高通量荧光定量PCR(high-throughput QPCR, HT-QPCR)反应平台(SmartChip Real-time PCR system, Wafergen Inc., 美国)测定样品中ARGs的丰度和多样性.高通量定量PCR的反应体系和反应条件参照文献[18].本研究中所选取的296对引物均在已有文献中被有效验证过, 包括285对用于检测抗生素抗性基因, 10对检测可移动基因元件(mobile genetic elements, MGEs)和1对检测16S rDNA的引物[19].定量PCR的数据处理参照文献[18]进行.反应结束后, QPCR的数据通过Cycler预设的筛选条件(扩增效率介于1.8~2.2以及熔解曲线单峰)进行导出.根据SmartChip Real-Time PCR Systems的仪器性能, 确定CT值为31时作为仪器的检测限.每个样品进行3次技术重复实验, 3次技术重复都被扩增出来时认为是阳性扩增.
1.4 16S rRNA基因丰度测定本研究采用Roche 480 Ⅱ荧光定量PCR仪(Roche Diagnostics, 美国)对样品中16S rRNA基因丰度进行绝对定量[20]. QPCR反应体系为20μL, 包括10μL的2 × LightCycler 480 SYBR® Green I Master Mix, 1 μL的DNA模板, 10μmol·L-1的上下游引物各1 μL和7 μL灭菌超纯水. QPCR反应条件为:95℃预变性5 min; 95℃变性15 s, 60℃退火1 min, 72℃延伸15s, 总共进行40个循环; 反应结束后仪器附带的程序自动添加熔解曲线程序进行分析.阴性对照为灭菌的超纯水代替样品, 每个样品均做3次技术重复实验.
1.5 数据分析样品中ARGs的相对拷贝数计算参照已有研究的方法[18], 采用公式(1)进行计算. HT-QPCR得到的16S rRNA基因相对拷贝数与QPCR得到的该基因绝对拷贝数极显著相关(P < 0.01), 因此采用公式(2)计算样品中ARGs的绝对丰度.
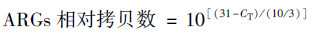
|
(1) |

|
(2) |
高通量定量PCR的数据采用Excel 2010进行计算, OriginPro 9.0进行相关作图.采用R3.5.0的vegan数据包进行主坐标分析和聚类分析, ggplot2数据包作图.
2 结果与讨论 2.1 紫色土丘陵区畜禽养殖场土壤中抗生素抗性基因的多样性和丰度采用HT-QPCR技术, 3种养殖场土壤中共检出79种抗性基因和5种可移动基因元件.根据其对应的抗生素类型, 可将检出的耐药基因分为9类[20], 包括:氟喹诺酮类/氯霉素类/胺酰醇类抗性基因(fluoroquinolone/chloramphenicol/amphenicol, FCA)、氨基糖苷类抗性基因(Aminoglycoside)、β-内酰胺类抗性基因(β-Lactamase)、大环内酯类-林肯酰胺类-链阳性菌素B类抗性基因(Macrolide-Lincosamide-Streptogramin B, MLSB)、多重耐药类(Multidrug)、磺胺类抗性基因(Sulfonamide)、四环素类抗性基因(Tetracycline)、万古霉素类抗性基因(Vancomycin)和其他类(other).紫色土丘陵区3种养殖场土壤中主要的耐药基因类型为氨基糖苷类、β-内酰胺类、多重耐药类和四环素类抗性基因, 这4类基因数量占样品中总检出数的77.27%~77.78% [图 1(a)].已有研究表明[21], 氨基糖苷类、大环内酯类、林肯酰胺类和四环素类抗生素是常用的饲料添加剂, 并不包含β-内酰胺类抗生素.相关分析表明, 养殖场土壤中β-内酰胺类抗性基因丰度与MLSB类(r=0.801, P < 0.01)和万古霉素类(r=0.831, P < 0.01)基因极显著正相关.不同类型抗性基因间显著的正相关性说明这些基因可能共现在相同的可移动基因元件上[9], 使得抗生素选择压力可以同时促进其对应的和其他种类的抗性基因富集[8].因此本研究中较高的β-内酰胺类抗性基因检出率可能与抗生素的共选择作用有关[22].已有的研究也发现了类似的现象, 如Looft等[6]研究了饲料中添加金霉素(100 g·t-1)、磺胺甲嘧啶(100 g·t-1)和青霉素(50 g·t-1)对猪肠道内抗性基因丰度的影响, 发现加药组中氨基糖苷类抗性基因丰度约是对照组的100倍. Zhu等[8]的研究发现未使用氨基糖苷类药物的猪场样品中氨基糖苷类抗性基因丰度相比于对照样品最高富集了10 000倍.
|
图 1 抗生素抗性基因种类型 Fig. 1 Profile of detected antibiotic resistance genes |
本研究中检出的抗性基因按照抗性机制还可分为抗生素失活机制(antibiotic deactivation)、外排泵机制(efflux pump)、细胞核糖体保护机制(cellular protection)和其他或未知机制(other/unknown) [图 1(b)], 其中抗生素失活机制(53.16%)和外排泵机制(29.11%)是养殖场土壤中主要的耐药机制.这与已有研究的结果一致[8, 20], 其原因可能是它们均能有效降低微生物胞内抗生素的浓度.抗生素失活酶可将胞内抗生素转换为低活性产物, 因此是微生物适应抗生素选择压力的最有效方式之一[22], 比如氨基糖苷类抗生素[23].外排泵机制则可介导抗生素和重金属等多种污染物排出细胞外, 因而也是微生物适应环境胁迫条件的主要途径[22].
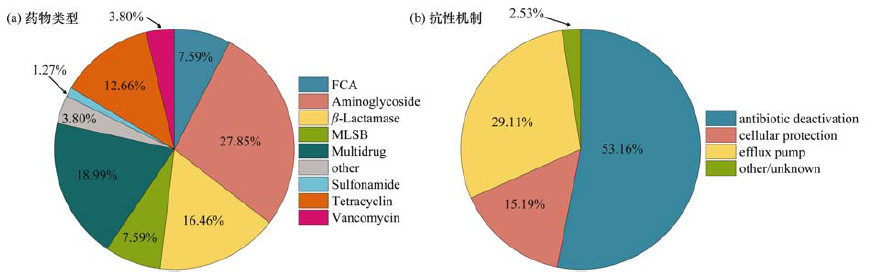
紫色土丘陵区3种养殖场土壤中抗生素抗性基因丰度见图 2.土壤样品中各类型抗性基因包括可移动基因元件的绝对丰度数量级范围为106~109 copies·g-1[图 2(a)].其中, 多种耐药类抗性基因的丰度最高(6.04×107~1.15×109 copies·g-1), 其次是β-内酰胺类抗性基因(5.90×107~8.27×108 copies·g-1), 而万古霉素类抗性基因呈现出最低丰度(0~1.47×107 copies·g-1).根据每个细菌具有的16S rRNA基因数[24], 将检测到的ARGs和MGEs丰度进行归一化[18], 计算ARGs的相对丰度[图 2(b)].与基因绝对丰度类似, 多重耐药类抗性基因具有最高的相对丰度, 这与已有关于施用猪粪紫色土[19]和施用鸡粪温室土[25]的研究结果一致.值得注意的是, 养殖场土壤中可移动基因元件的丰度较高, 仅次于多重耐药类基因(图 2), 说明养殖场土壤中抗性基因发生水平迁移的可能性较高[20].
|
图 2 不同养殖场土壤中抗生素抗性基因丰度 Fig. 2 Abundance of ARGs detected in the soil samples of different livestock feedlots |
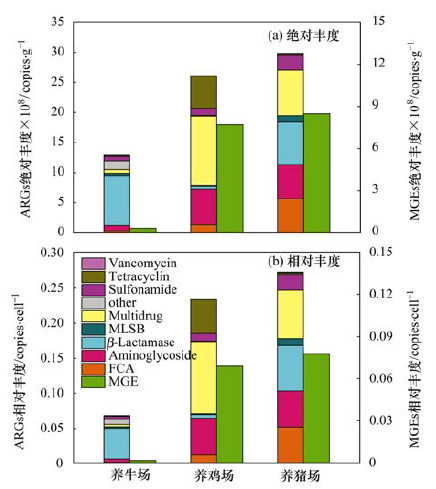
已有研究表明, 动物种类对畜禽粪便和施粪肥土中抗生素抗性基因的多样性和丰度均有影响[13].本研究中, 养牛场、养鸡场和养猪场的土壤样品中分别检测到27、44和36种抗性基因[图 3(a)], 养鸡场和养猪场土壤中耐药基因种类数高于养牛场的样品.相似地, 不同养殖场土壤样品中ARGs的丰度也表现为养猪场和养鸡场高于养牛场(图 2).这与前人的研究结果一致[26], 其原因可能与养殖不同动物所使用的抗生素种类和使用量不同有关[16].相对于养牛场, 猪场和鸡场的养殖密度较大, 出栏时间也更短, 因此养猪场和养鸡场的抗生素使用量一般远大于养牛场[27].这种差别导致猪粪和鸡粪中抗生素残留量[28]和耐药基因丰度和种类[13]均高于牛粪, 从而使猪粪和鸡粪受纳环境中耐药基因多样性一般高于牛粪. Qian等[12]对陕西省12家规模化养殖场的调查结果显示畜禽粪便中抗性基因的多样性和丰度大小顺序为鸡粪>猪粪>牛粪. Peng等[13]分析了长期施用猪粪和牛粪土壤中38种抗性基因检出情况, 发现猪粪和牛粪中分别有32种和23种抗性基因被检出, 而在施肥土中这些数字分别为25种和10种.他们还发现施用猪粪显著提高土壤中抗性基因丰度而施加牛粪并没有导致土壤中抗生素耐药性的扩散.
|
图 3 不同类型畜禽养殖场土壤中抗生素抗性基因种类 Fig. 3 Numbers of ARGs detected in the soil samples collected from different livestock feedlots |
通过韦恩图进一步分析不同养殖场土壤中抗性基因的关系, 结果表明土壤中检测到的抗性基因中3种养殖场共有基因仅5种(aadA2-01、aadA2-03、cphA-01、qacEdelta1-01和sul2), 多数基因属于不同养殖场所独有[图 3(b)].养牛场、养鸡场和养猪场独有的耐药基因分别占其总检出数的37.04%、59.09%和55.56%.不同养殖场土壤中各类抗性基因的丰度占比也不同, 如养牛场土壤的β-内酰胺类抗性基因丰度最高而养猪场和养鸡场均以多重耐药类基因为最高(图 2).上述结果说明不同养殖场土壤中耐药基因分布情况也可能存在差别.采用主坐标分析方法分析3种养殖场土壤中抗性基因的分布特征, 结果表明不同养殖场样品存在明显差异, 第一轴和第二轴共解释了98.39%的抗性基因分异, 说明养殖的动物种类对养殖场土壤中耐药基因分布特征有明显的影响[图 4(a)].聚类分析也得出了相似的结果, 不同养殖场土壤按动物种类分别形成3簇, 且养猪场和养鸡场土壤中抗性基因的差别没有它们与养牛场土壤样品的差别明显[图 4(b)].
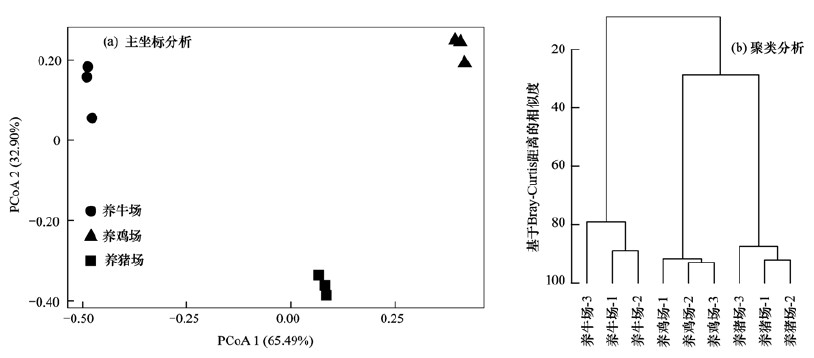
|
图 4 养殖场土壤中抗生素抗性基因的主坐标分析和聚类分析 Fig. 4 PCoA analysis and hierarchical clustering of ARGs in the soil samples of livestock feedlots |
耐药基因的水平迁移是导致抗生素耐药性在环境中快速传播和扩散的主要原因[29].已有的研究发现河水[18]和施粪肥土[13]中抗生素抗性基因与可移动基因元件, 包括整合子和转座子间存在显著的正相关性. Pearson相关分析表明(表 1), 养殖场土壤中氟喹诺酮类/氯霉素类/胺酰醇类抗性基因、氨基糖苷类抗性基因、多重耐药类抗性基因、磺胺类抗性基因的丰度和耐药基因总丰度与可移动基因元件的相对丰度和绝对丰度均显著正相关(P < 0.05).本研究中耐药基因与可移动基因元件之间显著的正相关关系以及样品中较高的可移动基因元件丰度说明养殖场土壤中抗性基因发生水平迁移的可能性较高, 可移动基因元件可能促进了抗生素耐药性在养殖场土壤中的传播和扩散.然而考虑到本研究选取的采样点较少, 且未包含未受污染的对照样品, 上述现象是否具有普遍性, 仍需进一步地以不同动物、不同养殖规模和不同养殖方式的多个畜禽养殖场为考察对象加以系统研究与确认.
|
|
表 1 抗生素抗性基因与可移动基因元件的相关关系1) Table 1 Relationship of between ARGs and MGEs |
3 结论
(1) 动物种类对紫色土丘陵区养殖土壤抗生素抗性基因有明显影响, 表现为养猪场和养鸡场土壤中抗生素抗性基因丰度和多样性均高于养牛场.
(2) 可移动基因元件丰度与抗性基因丰度显著相关, 说明样品中较高的可移动基因元件丰度可能促进了抗性基因在养殖场土壤环境中的传播和扩散.
| [1] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants:studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [2] |
朱永官, 欧阳纬莹, 吴楠, 等. 抗生素耐药性的来源与控制对策[J]. 中国科学院院刊, 2015, 30(4): 509-516. Zhu Y G, Ouyang W Y, Wu N, et al. Antibiotic resistance:sources and mitigation[J]. Bulletin of Chinese Academy of Sciences, 2015, 30(4): 509-516. |
| [3] |
苏建强, 黄福义, 朱永官. 环境抗生素抗性基因研究进展[J]. 生物多样性, 2013, 21(4): 481-487. Su J Q, Huang F Y, Zhu Y G. Antibiotic resistance genes in the environment[J]. Biodiversity Science, 2013, 21(4): 481-487. |
| [4] | D'Costa V M, King C E, Kalan L, et al. Antibiotic resistance is ancient[J]. Nature, 2011, 477(7365): 457-461. DOI:10.1038/nature10388 |
| [5] | Knapp C W, Dolfing J, Ehlert P A I, et al. Evidence of increasing antibiotic resistance gene abundances in archived soils since 1940[J]. Environmental Science & Technology, 2010, 44(2): 580-587. |
| [6] | Looft T, Johnson T A, Allen H K, et al. In-feed antibiotic effects on the swine intestinal microbiome[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(5): 1691-1696. DOI:10.1073/pnas.1120238109 |
| [7] | Chee-Sanford J C, Mackie R I, Koike S, et al. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste[J]. Journal of Environmental Quality, 2009, 38(3): 1086-1108. DOI:10.2134/jeq2008.0128 |
| [8] | Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440. DOI:10.1073/pnas.1222743110 |
| [9] | He L Y, Liu Y S, Su H C, et al. Dissemination of antibiotic resistance genes in representative broiler feedlots environments:identification of indicator ARGs and correlations with environmental variables[J]. Environmental Science & Technology, 2014, 48(22): 13120-13129. |
| [10] | Forsberg K J, Reyes A, Wang B, et al. The shared antibiotic resistome of soil bacteria and human pathogens[J]. Science, 2012, 337(6098): 1107-1111. DOI:10.1126/science.1220761 |
| [11] | Cheng W X, Chen H, Su C, et al. Abundance and persistence of antibiotic resistance genes in livestock farms:a comprehensive investigation in eastern China[J]. Environment International, 2013, 61: 1-7. DOI:10.1016/j.envint.2013.08.023 |
| [12] | Qian X, Gu J, Sun W, et al. Diversity, abundance, and persistence of antibiotic resistance genes in various types of animal manure following industrial composting[J]. Journal of Hazardous Materials, 2018, 344: 716-722. DOI:10.1016/j.jhazmat.2017.11.020 |
| [13] | Peng S, Feng Y Z, Wang Y M, et al. Prevalence of antibiotic resistance genes in soils after continually applied with different manure for 30 years[J]. Journal of Hazardous Materials, 2017, 340: 16-25. DOI:10.1016/j.jhazmat.2017.06.059 |
| [14] | Zhou B R, Wang C, Zhao Q, et al. Prevalence and dissemination of antibiotic resistance genes and coselection of heavy metals in Chinese dairy farms[J]. Journal of Hazardous Materials, 2016, 320: 10-17. DOI:10.1016/j.jhazmat.2016.08.007 |
| [15] |
仇焕广, 廖绍攀, 井月, 等. 我国畜禽粪便污染的区域差异与发展趋势分析[J]. 环境科学, 2013, 34(7): 2766-2774. Qiu H G, Liao S P, Jing Y, et al. Regional differences and development tendency of livestock manure pollution in China[J]. Environmental Science, 2013, 34(7): 2766-2774. |
| [16] | Van Boeckel T P, Brower C, Gilbert M, et al. Global trends in antimicrobial use in food animals[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(18): 5649-5654. DOI:10.1073/pnas.1503141112 |
| [17] |
鲜青松, 唐翔宇, 朱波. 坡耕地薄层紫色土-岩石系统中氮磷的迁移特征[J]. 环境科学, 2017, 38(7): 2843-2849. Xian Q S, Tang X Y, Zhu B. Transport of nitrogen and phosphorus from sloping farmland with thin purple soil overlying rocks[J]. Environmental Science, 2017, 38(7): 2843-2849. |
| [18] |
张丹丹, 郭亚平, 任红云, 等. 福建省敖江下游抗生素抗性基因分布特征[J]. 环境科学, 2018, 39(6): 2600-2606. Zhang D D, Guo Y P, Ren H Y, et al. Characteristics of antibiotic resistance genes in downstream areas of the Aojiang river, Fujian province[J]. Environmental Science, 2018, 39(6): 2600-2606. |
| [19] | Cheng J H, Tang X Y, Cui J F. Effect of long-term manure slurry application on the occurrence of antibiotic resistance genes in arable purple soil (entisol)[J]. Science of the Total Environment, 2019, 647: 853-861. DOI:10.1016/j.scitotenv.2018.08.028 |
| [20] |
黄福义, 安新丽, 陈青林, 等. 梅花鹿养殖场抗生素抗性基因分布特征[J]. 环境科学, 2016, 37(11): 4402-4409. Huang F Y, An X L, Chen Q L, et al. Distribution characteristics of antibiotic resistance genes in sika deer farm[J]. Environmental Science, 2016, 37(11): 4402-4409. |
| [21] |
魏光伟, 魏文康, 余永鹏, 等. 饲料抗生素添加剂的应用技术[J]. 广东饲料, 2007, 16(3): 15-16. Wei G W, Wei W K, Yu Y P, et al. The technique of antibiosis application in Feed[J]. Guangdong Feed, 2007, 16(3): 15-16. DOI:10.3969/j.issn.1005-8613.2007.03.007 |
| [22] | Chen B W, Lin L, Fang L, et al. Complex pollution of antibiotic resistance genes due to beta-lactam and aminoglycoside use in aquaculture farming[J]. Water Research, 2018, 134: 200-208. DOI:10.1016/j.watres.2018.02.003 |
| [23] | Zhang X X, Zhang T, Fang H H P. Antibiotic resistance genes in water environment[J]. Applied Microbiology and Biotechnology, 2009, 82(3): 397-414. DOI:10.1007/s00253-008-1829-z |
| [24] | Klappenbach J A, Saxman P R, Cole J R, et al. rrndb:the ribosomal RNA operon copy number database[J]. Nucleic Acids Research, 2001, 29(1): 181-184. DOI:10.1093/nar/29.1.181 |
| [25] | Fang H, Wang H F, Cai L, et al. Prevalence of antibiotic resistance genes and bacterial pathogens in long-term manured greenhouse soils as revealed by metagenomic survey[J]. Environmental Science & Technology, 2015, 49(2): 1095-1104. |
| [26] |
段曼莉.生物炭对土壤中抗生素及其抗性基因变化的影响研究[D].杨凌: 西北农林科技大学, 2017. 23-24. Duan M L. Effects of biochar application on variation of antibiotic and antibiotic resistance genes in soil[D]. Yangling: Northwest A & F University, 2017. 23-24. http://cdmd.cnki.com.cn/Article/CDMD-10712-1018037666.htm |
| [27] | Zhang Q Q, Ying G G, Pan C G, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China:source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 2015, 49(11): 6772-6782. |
| [28] | Hou J, Wan W N, Mao D Q, et al. Occurrence and distribution of sulfonamides, tetracyclines, quinolones, macrolides, and nitrofurans in livestock manure and amended soils of Northern China[J]. Environmental Science and Pollution Research, 2015, 22(6): 4545-4554. DOI:10.1007/s11356-014-3632-y |
| [29] | Dantas G, Sommer M O A. How to fight back against antibiotic resistance[J]. American Scientist, 2014, 102(1): 42-51. DOI:10.1511/2014.106.42 |
 2019, Vol. 40
2019, Vol. 40


