城市污水处理系统真核微生物群落特性与地域性差异
秦文韬,
张冰,
孙晨翔,
陈湛,
文湘华
环境科学  2019, Vol. 40 2019, Vol. 40 Issue (5): 2368-2374 Issue (5): 2368-2374
|
|
城市污水是人类活动的必然产物, 其产生量随着城市与社会经济的发展不断增加, 城市污水处理厂(wastewater treatment plant, WWTP)作为城市的“肠道”, 是控制水污染, 保证城市与人类可持续发展的重要设施.我国城市污水处理厂普遍采用活性污泥法处理污水, 其中微生物是污染物去除的主体, 因此微生物群落是系统稳定运行的基础.
在群落中, 除原核生物外, 真菌、原生动物、藻类等真核微生物作为不可或缺的一部分, 在保障系统稳定运行过程中发挥着重要的作用.一方面, 原生动物可以摄食处理水中游离细菌, 指示活性污泥性质; 通过优势种群变化、形态变化、繁殖方式变化来指示反应操作环境; 通过种群密度和种类的变化预测出水水质; 通过对细菌、有机颗粒和藻类等的强化和捕食作用强化净化效果[1~3]; 通过监测纤毛类的多样性可以预估污水处理厂的运行状况[4].另一方面, 隐孢子虫和贾第鞭毛虫等病原性原生动物能导致人和动物腹泻, 且具有个体小、潜伏期长、致病性强且易暴发流行的特点, 对下游生物造成极大的威胁[5].
活性污泥中真菌的出现一般也与水质有关.一方面, 丝状真菌是形成活性污泥絮凝体的骨架, 使污泥具有良好的沉淀性能, 保持高的净化效率、低的出水浓度和出水悬浮物浓度[6].另一方面, 丝状真菌, 如Trichosporon属, 若大量异常地增殖则会引发污泥膨胀现象, 常造成处理水质下降和污泥的大量流失, 影响处理系统的稳定运行[7].因此, 深入理解污水处理系统中真核微生物的特性, 对提高出水水质、保障污水处理系统的稳定运行具有重要作用.
传统的微生物培养方法和形态学研究手段因自身存在的不足和问题, 限制了人们对真核微生物的认识[8~10], Meng等[11]利用密度梯度离心结合定量蛋白银染色的方法在黄海48个站位的沉积物样品中鉴定出198种底栖纤毛虫; 代仁海[12]利用同样的方法在春季长江口及东海的39个站位的沉积物样品中检获278种底栖纤毛虫, 但其中约37%的种类由于数量过少等原因导致分类信息不足, 难以准确鉴定.近年来高通量测序技术的研究手段极大地提高了微生物群落研究的深度与水平, 现已被广泛用来解析农田、森林、土壤、海洋等环境中的微生物[13~19].在污水处理系统中, 相关研究大多集中于利用高通量测序技术结合其他生物信息学方法解析系统内细菌群落的结构和功能[20, 21], 采用这一技术研究系统内真核微生物群落的报道还很少见.此外, 我国不同区域的地理环境和饮食结构均存在差异, 且有研究显示自然系统内真核微生物群落和污水处理系统内的细菌群落均具有地域性差异[22~24], 然而不同区域活性污泥系统内的真核微生物群落地域性差异研究尚未开展.
因此, 本研究拟利用高通量测序技术, 结合多种生态学及数理统计相关分析方法研究我国城市污水处理系统中真核微生物的群落特性与地域性差异, 深化对污水处理系统中真核微生物的认识, 以期为指导污水处理系统的优化设计、提高出水水质、保障系统稳定高效运行提供科学依据.
1 材料与方法 1.1 样品采集与DNA提取污泥样品采集于2014年7~8月, 选取了北京, 深圳和无锡的14个污水处理厂, 为保证后续数据统计学分析结果的可靠性, 样品采集遵循每个城市至少采集4个WWTPs, 每个WWTP至少采集3个曝气池活性污泥样品(分别在曝气池前、中、后端)以及一个曝气池上清液样(曝气池中端), 若同一WWTP具有不同的处理工艺, 则在不同处理工艺的曝气池中均进行了活性污泥样品和曝气池上清液样品的采集.同时, 原位测定曝气池混合液的温度、pH、溶解氧浓度和电导率等参数, 收集WWTPs的地理信息、城市气候信息, WWTPs采用的处理工艺、设计参数、进出水水质情况和操作参数等基本信息.
采用统一的方法, 每个样品采集30 mL, 并迅速置于冰上24 h内带回实验室, 充分摇匀后于15 000g离心10 min后弃去上清液, 取沉淀相保存于-80℃用于微生物DNA提取.采用PowerSoil®DNA提取试剂盒提取污泥样品的DNA, 然后用ND-2000分光光度计和NanoDrop测定其浓度和纯度, 保证每个样品满足测序的DNA质量要求.
1.2 18S rDNA高通量测序利用引物TAReuk454FWD1(5′-CCAGCASC YGCGGTAATTCC-3′)和TAReukREV3(5′-ACTTTCG TTCTTGATYRA-3′)[25]通过Illumina MiSeq测序平台完成活性污泥系统内真核微生物18S rDNA测序, 然后在Galaxy平台(http://zhoulab5.rccc.ou.edu:8080/root)[26]上完成原始数据的整合, 物种信息分类, 生成操作分类单元(operational taxonomic unit, OTU)表格, 详细实验步骤和数据预处理过程可参考文献[27, 28].
1.3 数据分析本研究使用了非度量多维尺度法(nonmetric multidimensional scaling, NMDS)和相异性分析非参数检验方法ADONIS(permutational multivariate analysis of variance)、ANOSIM(analysis of similarities)和MRPP(multi-response permutation procedure)检验北京、深圳和无锡这3个城市微生物群落结构的差异, 采用多元回归矩阵分析(multiple regression on distance matrix, MRM)[29]和partial mantel test分析确定影响群落结构的重要因素.以上分析通过R语言vegan、ape、ecodist程序包和Excel完成.
2 结果与讨论 2.1 OTU及其相对丰度的分布特征为了保证样品间数据的均等性, 本研究按照每个样品8 190条序列的标准对测序结果进行随机抽平处理, 61个样品共得到499 590条序列.然后按照97%相似度的标准进行序列的OTU划分, 共获得3 794个OTUs.
数据分析结果表明, OTU的相对丰度分布极其不均匀, 只有少数的OTU相对丰度较高, 其中, 相对丰度大于0.10%的OTU仅有120个[图 1(a)], 这也是常被观测到的一种生态学现象[30, 31].此外, 前84个OTUs(1.07%)的累积相对丰度就已达到总丰度的80%[图 1(b)].同样地, OTU在样品中的分布也十分不均匀[图 1(c)], 只有4个OTUs被所有样品共有, 存在于50或更多个样品中的OTU只有37个, 85.40%的OTU仅存在于不超过10个样品中.另外, 从图 1(c)还可以看出:活性污泥系统真核微生物群落中绝大多数OTU的分布频率与相对丰度都很低, 但分布频率较高的OTU倾向于具有较高的相对丰度, 即存在少量分布广泛且相对丰度较高的OTU, 这与污水处理系统中细菌群落的分布规律相似[21].
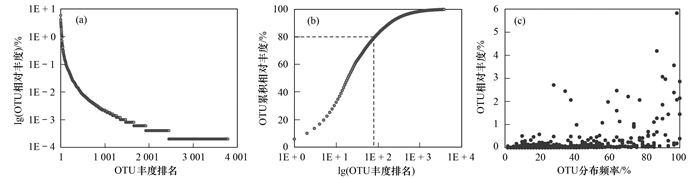
|
(a)OTU相对丰度排名-相对丰度; (b)OTU相对丰度排名-累积相对丰度; (c)OTU分布频率-相对丰度 图 1 OTU丰度-频率分布示意 Fig. 1 Distribution of OTU abundance-frequency |
α多样性能够反映样品内的微生物群落的丰富度和均匀度.本研究中, 活性污泥系统内每个样品中真核微生物群落的OTU数目为212~689, 估算的OTU丰富度(Chao1)为370~1192, 香农-威纳指数为1.58~4.80, 辛普森相关指数为1.8~38.65, 均匀度指数为0.29~0.73.城市水平来看, 3个城市活性污泥系统内真核微生物群落多样性不同(表 1), 其中无锡群落的物种丰富度(实际值和估测值)最高, 深圳次之, 北京群落的物种丰富度则明显低于无锡和深圳, 香农-威纳指数在3个城市间也呈现相同的规律, 各城市群落物种均匀度差异不大.
|
|
表 1 城市样品水平活性污泥系统内真核微生物群落α多样性指数 Table 1 Sample level α diversity indices of eukaryotic microbial community in the AS system of each city |
与传统的研究方法相比, 本研究利用高通量测序技术检获的真核微生物多样性更高. Matsunaga等[32]通过构建18S rRNA基因克隆文库的方法研究了活性污泥中的真核微生物多样性, 在获取的834个克隆中, 60%的克隆很难鉴定, 最终仅获取了80个OTUs; Matsubayashi等[33]利用同样的方法研究了厌氧污泥中的真核微生物多样性, 发现系统内85%的真核微生物都是未知的, 样品中估算的OTU数量(Chao1)仅为34~77个.尽管不同的数据处理软件、参数的选择等均可导致OTU数量的改变, 进而影响分子多样性的评价[34], 且前期PCR扩增和测序等过程均可能导致微生物群落物种多样性的高估, 但目前对其高估程度还难以准确定量[35].
与活性污泥系统内的细菌群落比较发现, 真核微生物多样性较低. Zhang等[22]使用454焦磷酸测序方法获得的活性污泥系统内细菌群落OTU数目为1 183~3 567个, Xia等[36]采用Illumina测序方法分别研究了MBR和OD系统内12周的活性污泥样品内的细菌群落多样性, 结果表明MBR系统和OD系统样品的OTU数目分别为2 297~3 330和1 802~3 517个, 均高于真核微生物多样性.
2.3 不同空间尺度下的活性污泥系统内真核微生物群落物种组成物种组成是微生物群落研究的一项重要内容, 是探究群落优势菌种或核心物种的基础, 但不同空间尺度下活性污泥系统内的微生物群落组成可能有所差异.因此, 本研究主要从区域、城市和污水处理厂这3个空间尺度, Supergroup、Division两个水平对OTU所属的物种信息进行分析, 从而全面地揭示活性污泥系统内真核微生物群落的组成特征.
在区域尺度上, 在Supergroup水平下, 3 794个OTUs分属于10个Supergroup[图 2(a)], 其中, Opisthokonta的相对丰度最高, 为54.06%, Alveolata次之, 为35.90%.在Division水平下, 3 794个OTUs分属于26个Division, 其中, 相对丰度大于1.0%的仅有10个[图 2(b)], 但它们的累积相对丰度已大于98%.具体地, 87.08%的OTUs属于Ciliophora、Fungi和Metazoa, 其中, 相对丰度最高的是Ciliophora, 为34.35%, 其次是Fungi和Metazoa, 相对丰度分别为33.02%和19.71%.
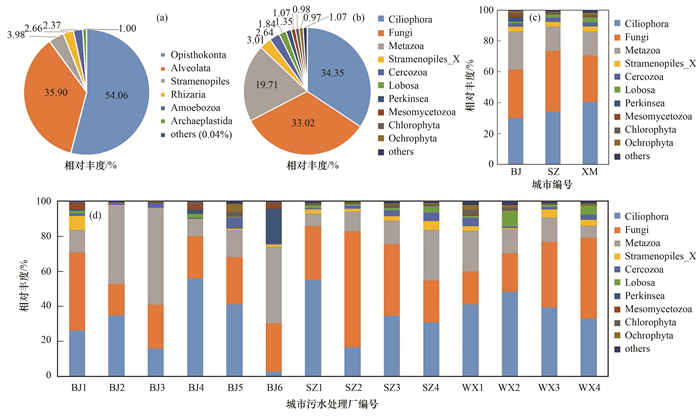
|
(a)整体尺度, Supergroup水平; (b)整体尺度, Division水平; (c)城市尺度, Devision水平; (d)污水处理厂尺度, Devision水平(相对丰度小于1.0%的统一划归为others) 图 2 不同尺度/水平下活性污泥系统内真核微生物群落组成 Fig. 2 Composition of eukaryotic microbial community in the AS system in different scales/levels |
在城市尺度、Division水平下[图 2(c)], 3个城市的活性污泥真核微生物群落均以Ciliophora, Fungi和Metazoa为主要组成成分, 总相对丰度为86.22%~89.40%, 但是北京和无锡群落均以Ciliophora为最主要的组分, 深圳群落则以Fungi为最主要的组分.
在污水处理厂尺度、Division水平下[图 2(d)], 除BJ6外的13个污水处理厂内的Ciliophora、Fungi和Metazoa的总相对丰度为83.21%~97.30%, BJ6较特殊, 三者总相对丰度仅为74.31%, 其他组分相对丰度较高.分析发现Perkinsea在各污水处理厂群落内分布极其不均匀, 在BJ6群落内的相对丰度高达19.99%, 但它们在BJ3和SZ4两个群落内不存在, 在其他厂内的相对丰度也均很低(0.02%~2.53%). Perkinsea主要包括一些双壳类软体动物的寄生虫, 常存活于淡水环境中[37].
2.4 活性污泥系统内真核微生物群落地域分布差异采用基于Bray-Curtis距离的NMDS方法对来自于不同污水处理厂的样品进行了群落结构分析, 结果如图 3所示:整体上, 来自北京、深圳和无锡这3个城市的活性污泥系统内真核微生物群落各自聚为一类, 表明3个城市的活性污泥系统内真核微生物群落结构存在差异.相异性分析非参数检验方法ADONIS、ANOSIM和MRPP的结果也为3个城市真核微生物群落结构存在显著差异提供了佐证(P < 0.01).具体看, 来自某一个城市不同污水处理厂的活性污泥真核微生物群落倾向于聚类在一起, 即来自同一城市的活性污泥系统内真核微生物群落具有较强的相似性, 不同污水处理厂的活性污泥系统内真核微生物群落结构存在差异, 表明活性污泥系统内真核微生物存在地域性差异.这与自然系统内真核微生物群落和污水处理系统内的细菌群落均存在地域性差异的结论一致[22~24].
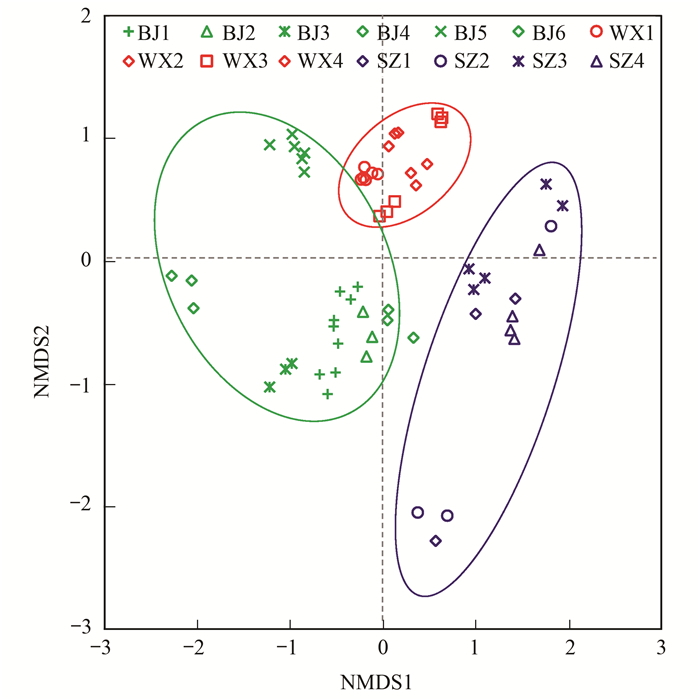
|
图 3 基于NMDS的活性污泥系统内真核微生物群落结构分析 Fig. 3 Structure analysis using NMDS of eukaryotic microbial community in the AS system |
群落多样性和结构主要受确定性因素和随机因素的影响.确定性因素通常基于生态位理论描述[38], 随机因素由中性理论阐述[39], 现有研究主要关注确定性因素中的环境条件对活性污泥系统内微生物群落结构的影响.基于高通量方法的研究表明WWTPs的水质参数、操作条件、地理位置和处理工艺等因素均会对污水处理系统微生物群落结构产生影响[20], 研究对象不同, 影响作用较显著的因素种类不尽相同[40~42].
为了探明城市污水处理厂活性污泥系统内真核微生物群落结构的影响因子, 本研究利用partial mantel test分析了地理位置、进出水水质参数、操作条件等参数与群落结构的相关性, 结果表明, 在参与分析的20个因子中, 10个因子与群落结构显著相关(P<0.05), 其中相关系数最大、相关性最强的因子是出水总氮浓度(r=0.61), 其次是水力停留时间(r=0.50), 污泥龄(r=0.43)和出水总磷浓度(r=0.38).与出水污染物参数相比, 进水污染物参数与群落相关性较弱[进水BOD浓度(r=0.33), 进水总磷浓度(r=0.26)], 此外, 与群落结构具有显著相关性的因素还有地理纬度(r=0.30), 曝气池溶解氧浓度(r=0.22), 年平均气温(r=0.22)和曝气池混合液浓度(r=0.22).
由于partial mantel test只能逐一分析各环境因子与群落结构的相关性或将多个环境因子作为一个整体考察其与群落结构的相关性, 为了探究不同环境因子对群落结构影响的相对重要性, 本研究使用相同的影响因子组合构建了MRM模型.结果表明, 模型显著且拟合度较高(R2=0.92, P<0.01).对于单个因子而言(表 2), 曝气池混合液温度是造成群落存在差异最重要的因素.环境温度影响生物处理构筑物中生物化学过程的速度和趋向, 影响微生物的新陈代谢、进化速率等生理活动, 适宜的温度能够促进、强化微生物的生理活动, 反之, 能够减弱甚至破坏微生物的生理活动, 导致微生物形态和生理特性的改变, 甚至可能导致微生物死亡, 进而影响活性污泥的微生物种群的形成和发展[43].
|
|
表 2 MRM分析各环境因子对活性污泥系统内真核微生物群落结构影响的权重1) Table 2 Influence of enviromental factors on eukaryotic microbial community structure in the AS system |
与进水污染物浓度(进水BOD浓度)相比, 出水污染物浓度(出水总氮浓度、出水总磷浓度)与群落结构具有较强的相关性, 其中出水总氮浓度与群落结构的相关性强于出水总磷浓度(表 2), Wei等[44]基于ITS数据对污水处理厂中真菌群落结构影响因素研究表明, 出水总氮浓度与群落结构有很强的相关性.氮是微生物生长、繁殖、代谢不可缺少的重要元素, 另外, 出水总氮浓度也是出水水质监测的一个重要指标, 因此这也侧面反映了真核微生物作为出水水质的指示生物, 其数量和密度与出水水质有重要关联[3, 45].齐嵘等[46]采用活体镜检计数的方法对无机废水硝化过程中原生动物群落结构进行了研究, 结果发现原生动物优势种类的数量变化与出水NO3--N和NO2--N的浓度存在一定联系, 即随着NO2--N的大量累积, 表壳虫生长繁殖旺盛, 密度最高可达575 ind.·mL-1, 成为系统中绝对优势的原生动物; 但当NO2--N浓度小于10 mg·L-1时, 几乎未见表壳虫的存在, 而系统中的另一种典型的原生动物累枝虫的数量则随着NO3--N浓度的持续增加而逐渐增加.
此外, 地理距离也是影响群落结构的重要因子(表 2), 这与上文3个城市群落结构存在显著差异的结果相一致, 说明微生物种群的确存在地域性差异, 事实上, 地理距离的差异导致了环境的异质性, 如气温、降水量的异质性, 进而导致了群落地域性差异. Bahram等[47]报道了影响表层土壤真菌分布的最主要因素是降水量. Telford等[48]的研究发现自然系统内的真核微生物(硅藻)会因为扩散限制而呈有限的地理分布, 其他学者的研究结果[49~51]也同样证明了微生物地域性差异的存在, 且地理距离造成的群落间的差异可能因空间限制而被长期保留.
此外, 常规污水处理厂操作运行参数曝气池pH、污泥龄和曝气池混合液浓度对群落结构也存在重要影响, 它们也是活性污泥系统内细菌群落的重要影响因子[20, 52].
3 结论(1) 不同空间尺度下, 活性污泥系统内真核微生物群落优势种群组成相似.在Devision水平上, 3个城市真核微生物群落优势种群主要由真菌、原生动物(纤毛亚门)和后生动物组成.
(2) 不同城市污水处理厂活性污泥系统内真核微生物多样性存在差异, 无锡群落的多样性(丰富度和香农-威纳多样性指数)最高, 北京的最低, 且3个城市的活性污泥系统内真核微生物群落结构存在显著的地域性差异.
(3) 利用Partial mantel test和MRM探讨了影响活性污泥系统真核微生物群落结构的因素, 结果表明, 曝气池混合液温度和出水总氮浓度与活性污泥系统内真核微生物群落结构的相关性较强.
| [1] | Madoni P, Davoli D, Chierici E. Comparative analysis of the activated sludge microfauna in several sewage treatment works[J]. Water Research, 1993, 27(9): 1485-1491. DOI:10.1016/0043-1354(93)90029-H |
| [2] | Madoni P. A sludge biotic index (SBI) for the evaluation of the biological performance of activated sludge plants based on the microfauna analysis[J]. Water Research, 1994, 28(1): 67-75. |
| [3] | Madoni P. Protozoa in wastewater treatment processes:a minireview[J]. Italian Journal of Zoology, 2011, 78(1): 3-11. DOI:10.1080/11250000903373797 |
| [4] | Salvadó H, Gracia M P. Determination of organic loading rate of activated sludge plants based on protozoan analysis[J]. Water Research, 1993, 27(5): 891-895. DOI:10.1016/0043-1354(93)90154-A |
| [5] | Hashimoto A, Kunikane S, Hirata T. Prevalence of Cryptosporidium oocysts and Giardia cysts in the drinking water supply in Japan[J]. Water Research, 2002, 36(3): 519-526. DOI:10.1016/S0043-1354(01)00279-2 |
| [6] | Rittmann B E, McCarty P L.环境生物技术原理与应用[M].文湘华, 王建龙, 译.北京: 清华大学出版社, 2012. |
| [7] | Zheng S K, Sun J Y, Han H. Effect of dissolved oxygen changes on activated sludge fungal bulking during lab-scale treatment of acidic industrial wastewater[J]. Environmental Science & Technology, 2011, 45(20): 8928-8934. |
| [8] | Eschenhagen M, Schuppler M, Roske I. Molecular characterization of the microbial community structure in two activated sludge systems for the advanced treatment of domestic effluents[J]. Water Research, 2003, 37(13): 3224-3232. DOI:10.1016/S0043-1354(03)00136-2 |
| [9] | Karayanni H, Christaki U, Van Wambeke F, et al. Evaluation of double formalin-Lugol's fixation in assessing number and biomass of ciliates:an example of estimations at mesoscale in NE Atlantic[J]. Journal of Microbiological Methods, 2004, 56(3): 349-358. DOI:10.1016/j.mimet.2003.11.002 |
| [10] |
郑祥, 刘俊新. 膜生物反应器系统中原生动物的群落特征[J]. 环境科学, 2009, 30(9): 2635-2640. Zheng X, Liu J X. Protozoan community diversity in membrane bioreactor[J]. Environmental Science, 2009, 30(9): 2635-2640. |
| [11] | Meng Z C, Xu K D, Dai R H, et al. Ciliate community structure, diversity and trophic role in offshore sediments from the Yellow Sea[J]. European Journal of Protistology, 2012, 48(1): 73-84. DOI:10.1016/j.ejop.2011.08.001 |
| [12] | 代仁海.黄东海底栖纤毛虫多样性及微型底栖生物群落结构特点[D].青岛: 中国科学院研究生院(海洋研究所), 2012. http://cdmd.cnki.com.cn/Article/CDMD-80068-1012411002.htm |
| [13] | Moll J, Hoppe B, König S, et al. Spatial distribution of fungal communities in an arable soil[J]. PLoS One, 2016, 11(2): e0148130. DOI:10.1371/journal.pone.0148130 |
| [14] | Zhou J Z, Deng Y, Shen L N, et al. Temperature mediates continental-scale diversity of microbes in forest soils[J]. Nature Communications, 2016, 7: 12083. DOI:10.1038/ncomms12083 |
| [15] | Wang Z, Xu G J, Xu H L. Determining β-diversity of protozoa for bioassessment in coastal ecosystems using community-based dispersions[J]. Ecological Indicators, 2017, 72: 47-52. DOI:10.1016/j.ecolind.2016.08.001 |
| [16] | Fuhrman J A, Steele J A, Hewson I, et al. A latitudinal diversity gradient in planktonic marine bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(22): 7774-7778. DOI:10.1073/pnas.0803070105 |
| [17] | Shi S J, Nuccio E, Herman D J, et al. Successional trajectories of rhizosphere bacterial communities over consecutive seasons[J]. mBio, 2015, 6(4): e00746-15. |
| [18] | Wang Y, Sheng H F, He Y, et al. Comparison of the levels of bacterial diversity in freshwater, intertidal wetland, and marine sediments by using millions of Illumina tags[J]. Applied and Environmental Microbiology, 2012, 78(23): 8264-8271. DOI:10.1128/AEM.01821-12 |
| [19] | Nagarkar M, Countway P D, Yoo Y D, et al. Temporal dynamics of eukaryotic microbial diversity at a coastal Pacific site[J]. The ISME Journal, 2018, 12(9): 2278-2291. DOI:10.1038/s41396-018-0172-3 |
| [20] | Xia Y, Wen X H, Zhang B, et al. Diversity and assembly patterns of activated sludge microbial communities:a review[J]. Biotechnology Advances, 2018, 36(4): 1038-1047. DOI:10.1016/j.biotechadv.2018.03.005 |
| [21] | Saunders A M, Albertsen M, Vollertsen J, et al. The activated sludge ecosystem contains a core community of abundant organisms[J]. The ISME Journal, 2016, 10(1): 11-20. DOI:10.1038/ismej.2015.117 |
| [22] | Zhang T, Shao M F, Ye L. 454 Pyrosequencing reveals bacterial diversity of activated sludge from 14 sewage treatment plants[J]. The ISME Journal, 2012, 6(6): 1137-1147. DOI:10.1038/ismej.2011.188 |
| [23] | Schmidt P A, Bálint M, Greshake B, et al. Illumina metabarcoding of a soil fungal community[J]. Soil Biology and Biochemistry, 2013, 65: 128-132. DOI:10.1016/j.soilbio.2013.05.014 |
| [24] | Wang X H, Wen X H, Deng Y, et al. Distance-decay relationship for biological wastewater treatment plants[J]. Applied and Environmental Microbiology, 2016, 82(16): 4860-4866. DOI:10.1128/AEM.01071-16 |
| [25] | Stoeck T, Bass D, Nebel M, et al. Multiple marker parallel tag environmental DNA sequencing reveals a highly complex eukaryotic community in marine anoxic water[J]. Molecular Ecology, 2010, 19(S1): 21-31. |
| [26] | Feng K, Zhang Z J, Cai W W, et al. Biodiversity and species competition regulate the resilience of microbial biofilm community[J]. Molecular Ecology, 2017, 26(21): 6170-6182. DOI:10.1111/mec.14356 |
| [27] | Caporaso J G, Lauber C L, Walters W A, et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. The ISME Journal, 2012, 6(8): 1621-1624. DOI:10.1038/ismej.2012.8 |
| [28] | Li Y, He J Z, He Z L, et al. Phylogenetic and functional gene structure shifts of the oral microbiomes in periodontitis patients[J]. The ISME Journal, 2014, 8(9): 1879-1891. DOI:10.1038/ismej.2014.28 |
| [29] | Legendre P, Lapointe F J, Casgrain P. Modeling brain evolution from behavior:a permutational regression approach[J]. Evolution, 1994, 48(5): 1487-1499. DOI:10.1111/j.1558-5646.1994.tb02191.x |
| [30] | McGill B J, Etienne R S, Gray J S, et al. Species abundance distributions:moving beyond single prediction theories to integration within an ecological framework[J]. Ecology Letters, 2007, 10(10): 995-1015. DOI:10.1111/ele.2007.10.issue-10 |
| [31] | Nemergut D R, Schmidt S K, Fukami T, et al. Patterns and processes of microbial community assembly[J]. Microbiology and Molecular Biology Reviews, 2013, 77(3): 342-356. DOI:10.1128/MMBR.00051-12 |
| [32] | Matsunaga K, Kubota K, Harada H. Molecular diversity of eukaryotes in municipal wastewater treatment processes as revealed by 18S rRNA gene analysis[J]. Microbes and Environments, 2014, 29(4): 401-407. DOI:10.1264/jsme2.ME14112 |
| [33] | Matsubayashi M, Shimada Y, Li Y Y, et al. Phylogenetic diversity and in situ detection of eukaryotes in anaerobic sludge digesters[J]. PLoS One, 2017, 12(3): e0172888. DOI:10.1371/journal.pone.0172888 |
| [34] | Santoferrara L F, Grattepanche J D, Katz L A, et al. Pyrosequencing for assessing diversity of eukaryotic microbes:analysis of data on marine planktonic ciliates and comparison with traditional methods[J]. Environmental Microbiology, 2014, 16(9): 2752-2763. DOI:10.1111/emi.2014.16.issue-9 |
| [35] | Kunin V, Engelbrektson A, Ochman H, et al. Wrinkles in the rare biosphere:pyrosequencing errors can lead to artificial inflation of diversity estimates[J]. Environmental Microbiology, 2010, 12(1): 118-123. DOI:10.1111/emi.2010.12.issue-1 |
| [36] | Xia Y, Hu M, Wen X H, et al. Diversity and interactions of microbial functional genes under differing environmental conditions:insights from a membrane bioreactor and an oxidation ditch[J]. Scientific Reports, 2016, 6: 18509. DOI:10.1038/srep18509 |
| [37] | Bråte J, Logares R, Berney C, et al. Freshwater perkinsea and marine-freshwater colonizations revealed by pyrosequencing and phylogeny of environmental rDNA[J]. The ISME Journal, 2010, 4(9): 1144-1153. DOI:10.1038/ismej.2010.39 |
| [38] | Chesson P. Mechanisms of maintenance of species diversity[J]. Annual Review of Ecology and Systematics, 2000, 31: 343-366. DOI:10.1146/annurev.ecolsys.31.1.343 |
| [39] | Hubbell S P. The unified neutral theory of biodiversity and biogeography[M]. Princeton: Princeton University Press, 2001. |
| [40] | Wang X H, Hu M, Xia Y, et al. Pyrosequencing analysis of bacterial diversity in 14 wastewater treatment systems in China[J]. Applied and Environmental Microbiology, 2012, 78(19): 7042-7047. DOI:10.1128/AEM.01617-12 |
| [41] | Liu J, Yang M, Qi R, et al. Comparative study of protozoan communities in full-scale MWTPs in Beijing related to treatment processes[J]. Water Research, 2008, 42(8-9): 1907-1918. DOI:10.1016/j.watres.2007.11.020 |
| [42] | Hu M, Wang X H, Wen X H, et al. Microbial community structures in different wastewater treatment plants as revealed by 454-pyrosequencing analysis[J]. Bioresource Technology, 2012, 117: 72-79. DOI:10.1016/j.biortech.2012.04.061 |
| [43] | Lepère C, Boucher D, Jardillier L, et al. Succession and regulation factors of small eukaryote community composition in a lacustrine ecosystem (Lake Pavin)[J]. Applied and Environmental Microbiology, 2006, 72(4): 2971-2981. DOI:10.1128/AEM.72.4.2971-2981.2006 |
| [44] | Wei Z Y, Liu Y Y, Feng K, et al. The divergence between fungal and bacterial communities in seasonal and spatial variations of wastewater treatment plants[J]. Science of the Total Environment, 2018, 628-629: 969-978. DOI:10.1016/j.scitotenv.2018.02.003 |
| [45] | Tyagi V K, Subramaniyan S, Kazmi A A, et al. Microbial community in conventional and extended aeration activated sludge plants in India[J]. Ecological Indicators, 2008, 8(5): 550-554. DOI:10.1016/j.ecolind.2007.07.002 |
| [46] |
齐嵘, 刘娟, 杨敏, 等. 无机废水硝化过程中原生动物群落结构特征及其变化规律[J]. 环境科学学报, 2013, 33(1): 53-57. Qi R, Liu J, Yang M, et al. Study on protozoan community structures during the inorganic wastewater nitrification process[J]. Acta Scientiae Circumstantiae, 2013, 33(1): 53-57. |
| [47] | Bahram M, Hildebrand F, Forslund S K, et al. Structure and function of the global topsoil microbiome[J]. Nature, 2018, 560(7717): 233-237. DOI:10.1038/s41586-018-0386-6 |
| [48] | Telford R J, Vandvik V, Birks H J B. Dispersal limitations matter for microbial morphospecies[J]. Science, 2006, 312(5776): 1015. DOI:10.1126/science.1125669 |
| [49] | Whitaker R J, Grogan D W, Taylor J W. Geographic barriers isolate endemic populations of hyperthermophilic archaea[J]. Science, 2003, 301(5635): 976-978. DOI:10.1126/science.1086909 |
| [50] | Cho J C, Tiedje J M. Biogeography and degree of endemicity of fluorescent Pseudomonas strains in soil[J]. Applied and Environmental Microbiology, 2000, 66(12): 5448-5456. DOI:10.1128/AEM.66.12.5448-5456.2000 |
| [51] | Borcard D, Legendre P. Environmental control and spatial structure in ecological communities:an example using oribatid mites (Acari, Oribatei)[J]. Environmental and Ecological Statistics, 1994, 1(1): 37-61. DOI:10.1007/BF00714196 |
| [52] | Vuono D C, Benecke J, Henkel J, et al. Disturbance and temporal partitioning of the activated sludge metacommunity[J]. The ISME Journal, 2015, 9(2): 425-435. DOI:10.1038/ismej.2014.139 |