厌氧氨氧化工艺因其高效、低能耗的优势, 在废水生物脱氮领域具有广阔的应用前景.厌氧氨氧化微生物聚集体是厌氧氨氧化菌和伴生菌的集合体, 解析聚集体的微生物群落结构特征对分析厌氧氨氧化反应器的脱氮性能至关重要.
目前已有一些厌氧氨氧化反应器微生物群落结构研究的报道.汪瑶琪等[1]研究了厌氧氨氧化反应器启动过程中微生物群落结构特征, 分析了厌氧氨氧化菌的变化规律. Costa等[2]研究了接种物和操作条件对厌氧氨氧化反应器运行的影响, 分析了微生物群落结构的差异.曹雁等[3]和陈重军等[4]研究了上流式厌氧过滤床、厌氧折流板厌氧氨氧化反应器中微生物群落结构特征. Pereira等[5]、Isanta[6]等和Leal等[7]分别研究了苯酚、温度和C/N对厌氧氨氧化反应器脱氮性能的影响以及不同情况下微生物群落结构的差异. Yang等[8]研究了底物抑制条件下厌氧氨氧化反应器性能和微生物群落结构的关系.这些研究为厌氧氨氧化反应器运行和应用提供了支撑, 但多关注于厌氧氨氧化菌与脱氮性能的关系, 而对于伴生细菌中与氮转化相关的或起支撑作用的功能微生物关注甚少, 如参与协同脱氮的反硝化微生物、参与有机物质降解的异养微生物等.
厌氧氨氧化工艺应用于工业废水处理是研究的热点, 含盐废水是一种常见工业废水, 盐度造成的高渗环境会造成多种影响.目前已有一些从微生物群落角度分析盐度对厌氧氨氧化工艺影响的研究. Kartal等[9]研究了淡水厌氧氨氧化富集培养物经海水驯化后种属的变化. Yang等[10]研究了常温下处理高盐度废水厌氧氨氧化反应器的脱氮性能, 指出优势厌氧氨氧化菌种属为Candidatus Kuenenia属. Gonzalez-Silva等[11]研究了厌氧氨氧化颗粒污泥适应盐度过程中微生物群落结构的变化规律. Wei等[12]研究了厌氧氨氧化反应器处理不同浓度海水时的脱氮性能, 分析了厌氧氨氧化菌的丰度变化及演替规律.盐度对厌氧氨氧化菌和伴生菌均会造成影响, 这些研究多关注了盐度条件下的微生物群落结构及厌氧氨氧化菌的变化, 并未探讨盐度条件下伴生菌的变化及其对系统稳定性的作用.
因此本文对盐度条件下ANAMMOX-EGSB反应器脱氮性能和颗粒污泥微生物群落结构进行了分析, 探讨了盐度对厌氧氨氧化菌及伴生菌的影响, 并分析了伴生菌对反应器稳定性的作用.
1 材料与方法 1.1 反应器运行 1.1.1 反应器设置采用EGSB反应器实现厌氧氨氧化工艺.反应器由有机玻璃制成, 高1 000 mm, 内径50 mm, 总有效容积1.86 L, 其中反应区容积1.5 L, 沉淀区容积0.35 L.反应器外设水浴保温层, 并缠绕铝箔胶带以避光.反应器进水和回流由蠕动泵从反应器底部泵入.
1.1.2 试验原水和接种污泥采用人工合成的无外加COD的含氮废水, 其中NH4+-N和NO2--N由NH4Cl和NaNO2按量比1:1添加, 浓度约为200 mg·L-1.按照HCO3-与NH4+-N摩尔比为2:1添加KHCO3, 用以维持pH值为7.8~8.0.其他主要成分包括KH2PO4 0.025 g·L-1、CaCl2·2H2O 0.2 g·L-1、MgSO4·7H2O 0.2 g·L-1以及微量元素液Ⅰ、Ⅱ各1.25 mL·L-1[13].所需药品NH4Cl、NaNO2、KHCO3、CaCl2·2H2O、MgSO4·7H2O、KH2PO4为工业纯, 配制微量元素液所需药品为分析纯.在废水中按需求添加氯化钠, 配制成含盐废水.
从实验室现有3个ANAMMOX-EGSB反应器(其中1个反应器曾在盐度条件下运行)各取适量颗粒污泥混合后接种于图 1所示的反应器.反应器在氮容积负荷2.58 kg·(m3·d)-1条件下运行, MLSS和MLVSS分别为68.05 g·L-1和21.80 g·L-1.
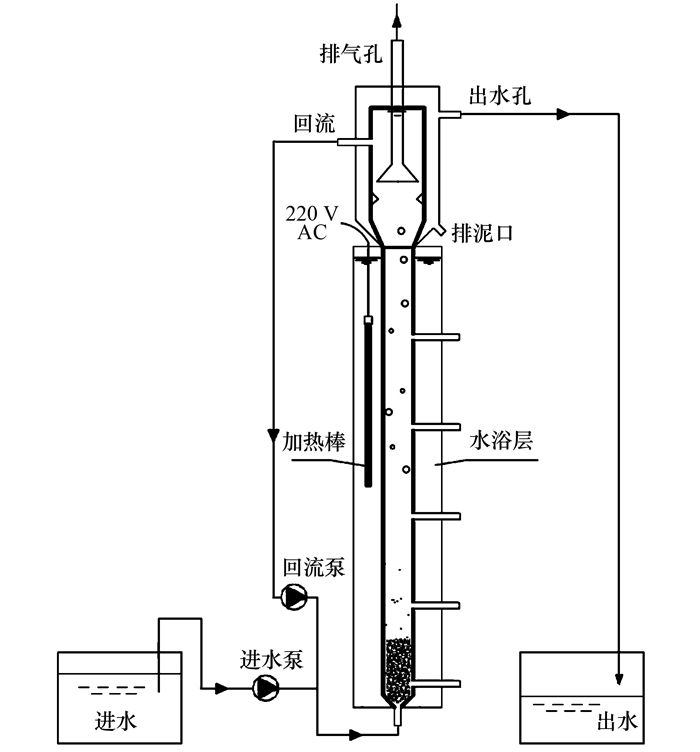
|
图 1 ANAMMOX-EGSB反应器示意 Fig. 1 Diagram of the ANAMMOX-EGSB reactor |
反应器回流比设置为25:1, 上升流速为4.42 m·h-1, HRT为3.72 h, 水浴层温度由加热棒控制在(32±1)℃.经过一段时间的运行, 当反应器氨氮和亚硝氮去除率大于90%时, 认为反应器运行稳定.向合成废水中添加氯化钠, 并以15 g·L-1为梯度分两次提升反应器进水盐度至15 g·L-1和30 g·L-1.
1.2 测试方法 1.2.1 水质检测每两天取反应器进出水, 用0.22 μm针式滤器过滤后测定NH4+-N、NO2--N、NO3--N浓度. NH4+-N采用水杨酸-次氯酸盐分光光度法测定, NO2--N采用N-(1-萘基)-乙二胺光度法测定, NO3--N采用紫外分光光度法测定[14]. TN浓度即为NH4+-N、NO2--N、NO3--N浓度相加.
1.2.2 颗粒污泥表面形貌测定不同盐度下反应器稳定运行后, 取颗粒污泥样品用戊二醛(2.5%)固定, 冷冻干燥并喷金后, 应用FEI Nova400场发射扫描电子显微镜观察表面形貌.
1.2.3 高通量测序不同盐度下反应器稳定运行后, 从反应器底部取颗粒污泥样品0.3 g.委托上海美吉生物医药科技有限公司进行污泥总DNA的提取, 采用细菌16S rRNA基因的V3-V4区通用引物338F/806R(5′-ACTCCTACGGGAGGCAGCA-3′/5′-GGACTACHVGG GTWTCTAAT-3′)对提取的总DNA进行PCR扩增、建库, 于Illumina MiSeq PE 300高通量测序平台进行高通量测序, 每个样品进行了3次生物学重复.
2 结果与分析 2.1 反应器运行性能不同盐度条件下ANAMMOX-EGSB反应器进出水水质和稳定阶段进出水水质分别如图 2和表 1所示.

|
图 2 ANAMMOX-EGSB反应器进出水水质 Fig. 2 Inlet and outlet water quality of the ANAMMOX-EGSB reactor |
|
|
表 1 EGSB反应器稳定阶段进出水水质 Table 1 Inlet and outlet water quality of the ANAMMOX-EGSB reactor at the stable stage |
第1~18 d为反应器0 g·L-1盐度下稳定运行阶段, 反应器氨氮、亚硝氮、硝氮平均去除率分别为95.4%、98.5%和50.2%.第19 d将反应器进水盐度提升至15 g·L-1, 随后反应器去除性能下降, 主要表现为氨氮去除率的下降, 亚硝氮去除率略有波动但相对稳定.第53 d反应器的氨氮去除率恢复至90%以上, 认为反应器性能恢复.第81d继续将反应器进水盐度提升至30 g·L-1, 氨氮和亚硝氮的去除率同样下降并以氨氮为主, 第113 d反应器的氨氮去除率恢复至90%以上, 反应器性能恢复.
第53~81 d为反应器15 g·L-1盐度下稳定运行阶段, 该阶段氨氮、亚硝氮及硝氮的平均去除率分别为88.8%、98.0%和48.5%, 而第113~135 d为反应器30 g·L-1盐度下稳定运行阶段, 该阶段氨氮、亚硝氮及硝氮的平均去除率分别为91.0%、96.9%和51.3%.对比3个稳定阶段可知, 15 g·L-1盐度和30 g·L-1盐度稳定运行阶段反应器的脱氮性能相当, 但都略低于0 g·L-1盐度.表明ANAMMOX-EGSB反应器可耐受不超过30 g·L-1的盐度条件维持脱氮性能稳定.
反应器进水氨氮和亚硝氮浓度按照1:1比例添加, 根据厌氧氨氧化计量关系式[15]可知氨氮和亚硝氮的理论比例为1:1.32, 因此进水氨氮浓度是过量的.假设反应器中仅发生厌氧氨氧化反应, 由亚硝氮浓度即可推算出理论总氮去除率大致介于71.1%~74.0%, 明显低于表 1中反应器的实际总氮去除率.因此除厌氧氨氧化外, 反应器还存在其它脱氮途径, 如进水中过量氨氮的去除, 厌氧氨氧化反应产生硝氮的反硝化等, 这涉及反应器中伴生菌的分析.
2.2 颗粒污泥表面形貌不同盐度条件下反应器颗粒污泥表面形貌如图 3所示.从中可知, 0 g·L-1盐条件下, 颗粒污泥表面主要为大量球菌和少量丝状菌, 表面光滑. 15 g·L-1盐度下, 污泥表面产生一些胞外聚合物, 丝状菌数量增多, 粗糙度增加. 30 g·L-1盐度下, 污泥表面产生大量的胞外聚合物, 出现球状凸起, 丝状菌被胞外聚合物紧密包裹.可见盐度条件下胞外聚合分泌增加, 该结果与王子超等[16]发现盐度导致厌氧颗粒污泥胞外聚合物增加的结论一致.

|
图 3 不同盐度下反应器颗粒污泥表面形貌 Fig. 3 Surface morphology of the granular sludge under different salinities |
图 4为不同盐度下反应器门水平微生物群落柱状图.从中可知, 颗粒污泥中微生物按门水平丰度高低排序依次为浮霉状菌门(Planctomycetes)、绿弯菌门(Chloroflexi)、变形菌门(Proteobacteria)、酸杆菌门(Acidobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、绿菌门(Chlorobi).
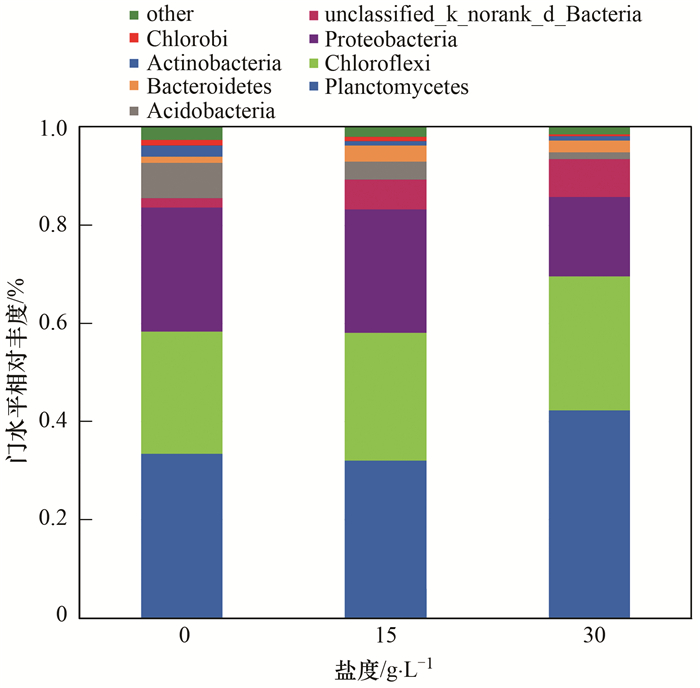
|
图 4 不同盐度下反应器门水平微生物群落柱状图 Fig. 4 Bar chart of the microbial community at the phylum level under different salinities |
有报道表明, 尽管反应器进水成分存在差异, 但在不同的厌氧氨氧化废水处理系统中, 除浮霉状菌门外, 变形菌门、绿弯菌门、绿菌门、拟杆菌门常常被检测到且丰度较高[17~19], 表明这些菌门与厌氧氨氧化菌存在密切的关系.浮霉状菌门、变形菌门、绿弯菌门丰度占总体比例较高且累计丰度超过了80%, 为反应器的优势菌门.
厌氧氨氧化菌属于浮霉状菌门, 反应器中该菌门丰度在盐度条件有所增加, 表明厌氧氨氧化菌可以较好地耐受盐度, 为反应器脱氮性能的稳定提供了支撑.变形菌门是细菌域中菌属最多样化的菌门, 该菌门微生物具有多样的代谢机制、营养类型及形态特征, 包括具备氮素转化功能的氨氧化细菌、亚硝酸盐氧化菌和反硝化菌[20], 该菌门丰度在盐度条件下降低, 可能与所属细菌耐盐能力不同有关.绿弯菌门细菌可降解溶解性有机物、胞外聚合物以及衰亡细菌残体[21], 并具有稳定颗粒污泥结构的作用[22], 该菌门丰度在盐度条件下相对稳定.
拟杆菌门细菌功能广泛, 具备有机物质降解、反硝化、固氮等功能, 对维持颗粒污泥形态也有一定作用[22], 该菌门丰度在盐度条件下有所增加.酸杆菌门细菌也具备降解复杂高分子有机物如木聚糖, 半纤维素, 果胶和几丁质等的能力[23], 该菌门丰度在盐度条件下降低.绿菌门多为光合自养微生物[24], 其丰度在盐度条件下降低.
2.3.2 属水平微生物群落结构图 5为不同盐度下反应器属水平微生物群落Heatmap图(分类水平总丰度前20的物种), 其中标注了不同属所对应的菌门.

|
图 5 不同盐度下反应器属水平微生物群落Heatmap图 Fig. 5 Heatmap of the microbial community at the genus level under different salinities |
如图 5所示, 颗粒污泥在属水平中检测到了归属于浮霉状菌门的Candidatus Jettenia、unclassified Brocadiaceae、Candidatus Kuenenia厌氧氨氧化菌属, 上述3属的丰度在盐度条件下均增加, 3属累计丰度在不含盐条件下为10.33%, 15 g·L-1和30 g·L-1盐度分别为20.90%和35.87%, 其中Candidatus Kuenenia丰度较高.由于厌氧氨氧化菌存在种属间的竞争关系, 故在一个废水处理系统中常以某一种属丰度占据优势[25, 26]. Candidatus Kuenenia对亚硝酸盐的亲和性优于其它厌氧氨氧化菌属[27], 可能是丰度占优的原因. Candidatus Kuenenia在盐度条件下仍可维持较高丰度, 也与Yang等[10]报道30 g·L-1盐度条件下Candidatus Kuenenia为优势属的结论一致.
除厌氧氨氧化菌外, 颗粒污泥在浮霉状菌门还检测到了SM1A02属和I8属, 它们常在厌氧氨氧化系统中被检测到[28, 29], 但功能不明且报道较少.不含盐条件下二者丰度分别为5.1%和17.7%, 盐度条件下处于1.1%~1.9%和4.8%~9.0%范围, 均明显下降.
变形菌门在属水平检测到了反硝化菌, 主要包括Rhodobacteraceae、Myxococcales、Denitratisoma, 累计丰度在不含盐条件下为1.61%, 盐度条件下依次增加至2.88%和3.13%.有报道指出它们均具有一定的耐盐能力[30, 31], 其丰度的提高表明盐度条件下反硝化活动的增强.此外, 在变形菌门中还检测到了Nitrosomonadaceae好氧氨氧化菌, 其丰度在不含盐条件下为1.11%, 盐度条件下介于0.235%~0.511%范围.该属被检出表明系统具有一定的好氧氨氧化能力. Sabumon等[32]曾指出在厌氧氨氧化系统中较低的ORP电位[(-248+25) mV]下氨氮也可被氧化成硝氮, 也表明反应器存在少量的好氧氨氧化细菌. Nitrosomonadaceae丰度的降低一方面表明含盐条件下氨氮氧化的能力降低, 另一方面表明含盐条件下反应器内厌氧水平良好.
变形菌门还存在一些其它功能的微生物, 如γ-Proteobacteria、Limnobacter、Woodsholea等属. γ-Proteobacteria属为主要分布于海洋环境中的自养微生物, 可在厌氧环境下进行生物固氮[33], 其丰度由不含盐条件下的0.895%增加至含盐条件下的7%以上. Limnobacter为甲烷依赖型异养微生物[34], 其丰度由不含盐条件下的2.58%降低至盐度条件的0.5%以下. Woodsholea是常见于海洋或盐碱地中的自养好氧微生物, 可被低浓度有机物抑制[35], 丰度由不含盐条件下的3.74%降低至含盐条件的2.6%以下.
绿弯菌门在属水平主要包括Anaerolineaceae、unclassifed Chloroflexi和Ardenticatenia. Anaerolineaceae具有降解微生物残体的功能[36], 该属丰度在不含盐条件下为9.39%, 在含盐条件下处于5.29%~9.38%范围. unclassifed Chloroflexi和厌氧氨氧化菌关系密切, Kindaichi等[21]通过实验证明unclassifed Chloroflexi可分解厌氧氨氧化菌衰老残体并产生有机物, 该属丰度在不含盐条件下为12.42%, 盐度条件下处于14.93%~16.85%范围. Ardenticatenia具有还原三价铁和硝酸盐的能力[37], 其丰度在不含盐条件下为2.06%, 盐度条件下处于2.57%~3.48%范围.
拟杆菌门在属水平也检测到了反硝化菌, 主要为Saprospiraceae和Cryomorphaceae.拟杆菌门所含反硝化菌丰度不含盐条件下为1.11%, 含盐条件下依次为1.16%和1.18%, 虽然丰度变化不大, 但出现了主要菌属的演替, 由Saprospiraceae转变为了Cryomorphaceae, Cryomorphaceae为丝状反硝化菌[38], 具有较好的耐盐能力.
3 讨论盐度条件下厌氧氨氧化菌丰度的增加体现了其对盐度的适应, 但丰度的提高可能主要与运行时间的延长有关.如有文献报道[22], 在2.67~3.62 kg·(m3·d)-1的负荷条件下厌氧氨氧化菌的丰度随着运行时间的延长, 呈现出1~3倍的增加.
颗粒污泥中, 变形菌门、拟杆菌门、绿弯菌门均检测到了反硝化菌, 其累计丰度由不含盐条件下的4.88%增加至盐度条件下的6.62%和7.79%, 可见盐度条件下反硝化协同脱氮途径是增强的.反应器中污泥的溶解性微生物产物(SMP)是有机物的主要来源, 有报道指出盐度会导致污泥中部分微生物细胞死亡或胞内物质溶出[39, 40], 因此可导致SMP含量增加, 进而使有机物浓度增加.一方面有机物的存在为反硝化脱氮提供了碳源, 支撑协同反硝化脱氮, 但另一方面也有可能对厌氧氨氧化菌产生抑制作用. Molinuevo等[41]认为有机物的存在会引起异养菌的大量繁殖, 与厌氧氨氧化菌竞争电子受体亚硝酸盐形成抑制, 当COD浓度为290 mg·L-1时即可完全抑制厌氧氨氧化.本研究中盐度条件下反硝化菌丰度的提高, 有利于转化厌氧氨氧化反应产生的硝氮, 提高了反应器的脱氮效率, 也在一定程度上解除了有机物可能导致的抑制.该结果也支撑了图 2和表 1中盐度条件下反应器较好的脱氮性能.
颗粒污泥是富集厌氧氨氧化菌的有效方式, 其结构的稳定对厌氧氨氧化活性的发挥具有重要作用.有报道表明绿弯菌门和拟杆菌门与颗粒污泥关系密切[22], Björnsson等[42]的研究表明绿弯菌门细菌在厌氧氨氧化污泥中起到了支撑和骨架作用, 其中所包含的大量丝状菌在颗粒污泥造粒过程中起着重要的作用.本研究中绿弯菌门细菌的丰度在含盐条件下处于26.0%~27.3%范围, 略高于不含盐条件下24.9%的丰度.如图 3所示, 盐度条件下颗粒污泥表面有大量丝状菌, 可能与绿弯菌门增加有关.盐度条件下绿弯菌门丰度的相对稳定表明其对颗粒污泥结构稳定发挥了支撑作用. Fernández-Gomez等[43]的研究表明, 拟杆菌门细菌可分泌大量的胞外多糖, 具有较强的附着能力, 生长于颗粒表面. Cao等[22]的研究也认为该菌门细菌倾向于在颗粒污泥的外层附着生长, 可形成网状结构支撑颗粒的形成.本研究中该菌门的丰度在含盐条件下处于2.4%~3.3%范围, 高于不含盐条件下的1.22%.由图 3也可知, 盐度条件下颗粒污泥表面胞外聚合物分泌明显增多, 30 g·L-1盐度下甚至形成了球状凸起, 推测拟杆菌门丰度的增加支撑了盐度条件下胞外聚合物的分泌的增加.因此, 盐度条件下绿弯菌门和拟杆菌门丰度的提高有利于维持颗粒污泥结构的稳定.
厌氧氨氧化菌为严格厌氧微生物, 由于实际废水处理过程中不可避免存在的少量溶解氧, 将影响厌氧氨氧化菌活性. Jettnia等曾报道[44], 当水中溶解氧大于0.032 mg·L-1即会抑制其活性, 而伴生菌中所含好氧菌及兼性菌可消耗溶液中的溶解氧保证厌氧氨氧化菌的活性.颗粒污泥中检测到的好氧微生物主要为Nitrosomonadaceae和Woodsholea, 它们的累计丰度由不含盐条件下的4.84%降低至盐度下的2.8%~3.0%范围, 由此推测盐度条件下反应器内部厌氧水平有所提高.另外, 反硝化菌在好氧和厌氧条件下都可生存, 故也可消耗微量溶解氧, 盐度条件下反硝化菌丰度的增加, 也有利于维持反应器良好的厌氧条件.
盐度条件下不同伴生菌门优势属的转变反映了微生物对盐度的适应性, 含盐条件下厌氧氨氧化反应器运行稳定时, 这些菌门的优势属在厌氧氨氧污泥中也常被检测到.如Yokota等[45]报道UASB厌氧氨氧化反应器在处理不含盐废水时变形菌门以α-Proteobacteria和β-Proteobacteria为主, 而处理含盐废水时却转变为以γ-Proteobacteria为主. Gonzalez-Silva等[11]也观察到了拟杆菌门丰度在高盐度条件下丰度增加, 并以 Cryomorphaceae和Flavobacteriia为主, 绿弯菌门主要以Anaerolineaceae为主, 这些结果也与本研究结果一致.可见盐度条件下厌氧氨氧化反应器稳定运行时, 伴生菌门所含优势属具有种群相似性.因此处理实际含盐废水时, 选择盐度驯化后且优势菌种群相似的富集培养物作为接种污泥将有利于反应器启动及后续运行.
颗粒态厌氧氨氧化污泥相比于絮状污泥能更好地维持厌氧微区, 有利于伴生菌中的反硝化菌发挥协同脱氮, 有利于绿弯菌门和拟杆菌门等起支撑作用的伴生菌稳定污泥结构, 也有利于好氧和兼性伴生细菌消耗微量溶解氧. Yokota等[45]的研究也表明以颗粒态污泥为接种污泥的UASB厌氧氨氧化反应器处理含盐高氨氮废水时, 厌氧氨氧化菌丰度快速增加且获得了很高的氮去除负荷, 而以非颗粒态污泥为接种污泥时仅在内壁发现红色培养物生长且氮去除负荷极低, 因此颗粒态污泥不仅有利于厌氧氨氧化菌的富集, 也有利于伴生菌与厌氧氨氧化菌协同发挥作用.
4 结论(1) ANAMMOX-EGSB反应器在两次盐度提升后脱氮性能均下降, 随着运行时间的延长又可恢复, 氨氮、亚硝氮平均去除率可分别在88.8%~95.4%、96.9%~98.5%范围内维持稳定, 而且反应器内存在明显的反硝化协同脱氮.厌氧氨氧化菌可适应30 g·L-1的盐度, 其丰度在盐度条件下随运行时间延长而增加. Candidatus Kuenenia为厌氧氨氧化菌的优势属.盐度条件下颗粒污泥表面有大量丝状菌和胞外聚合物.
(2) 盐度条件下反硝化菌属丰度提高, 增强了反硝化协同脱氮, 也避免了有机物对厌氧氨氧化的抑制.绿弯菌门和拟杆菌门丰度的提高有利于维持颗粒污泥结构的稳定.盐度条件下好氧微生物和反硝化菌存在也有利于维持反应器内部厌氧水平.因此, 盐度条件下伴生菌对厌氧氨氧化菌功能的发挥提供了支撑.
| [1] |
汪瑶琪, 张敏, 姜滢, 等. 厌氧氨氧化启动过程及微生物群落结构特征[J]. 环境科学, 2017, 38(12): 5184-5191. Wang Y Q, Zhang M, Jiang Y, et al. Start-up and characteristics of the microbial community structure of ANAMMOX[J]. Environmental Science, 2017, 38(12): 5184-5191. |
| [2] | Costa M C M S, Carvalho L, Leal C D, et al. Impact of inocula and operating conditions on the microbial community structure of two anammox reactors[J]. Environmental Technology, 2014, 35(14): 1811-1822. DOI:10.1080/09593330.2014.883432 |
| [3] |
曹雁, 王桐屿, 秦玉洁, 等. 厌氧氨氧化反应器脱氮性能及细菌群落多样性分析[J]. 环境科学, 2017, 38(4): 1544-1550. Cao Y, Wang T Y, Qin Y J, et al. Nitrogen removal characteristics and diversity of microbial community in ANAMMOX reactor[J]. Environmental Science, 2017, 38(4): 1544-1550. |
| [4] |
陈重军, 张海芹, 汪瑶琪, 等. 基于高通量测序的ABR厌氧氨氧化反应器各隔室细菌群落特征分析[J]. 环境科学, 2016, 37(7): 2652-2658. Chen C J, Zhang H Q, Wang Y Q, et al. Characteristics of microbial community in each compartment of ABR ANAMMOX reactor based on high-throughput sequencing[J]. Environmental Science, 2016, 37(7): 2652-2658. |
| [5] | Pereira A D, Leal C D, Dias M F, et al. Effect of phenol on the nitrogen removal performance and microbial community structure and composition of an anammox reactor[J]. Bioresource Technology, 2014, 166: 103-111. DOI:10.1016/j.biortech.2014.05.043 |
| [6] | Isanta E, Bezerra T, Fernández I, et al. Microbial community shifts on an anammox reactor after a temperature shock using 454-pyrosequencing analysis[J]. Bioresource Technology, 2015, 181: 207-213. DOI:10.1016/j.biortech.2015.01.064 |
| [7] | Leal C D, Pereira A D, Nunes F T, et al. Anammox for nitrogen removal from anaerobically pre-treated municipal wastewater:effect of COD/N ratios on process performance and bacterial community structure[J]. Bioresource Technology, 2016, 211: 257-266. DOI:10.1016/j.biortech.2016.03.107 |
| [8] | Yang W, He S L, Han M, et al. Nitrogen removal performance and microbial community structure in the start-up and substrate inhibition stages of an anammox reactor[J]. Journal of bioscience and bioengineering, 2018, 126(1): 88-95. DOI:10.1016/j.jbiosc.2018.02.004 |
| [9] | Kartal B, Koleva M, Arsov R, et al. Adaptation of a freshwater anammox population to high salinity wastewater[J]. Journal of Biotechnology, 2006, 126(4): 546-553. DOI:10.1016/j.jbiotec.2006.05.012 |
| [10] | Yang J C, Zhang L, Hira D, et al. Anammox treatment of high-salinity wastewater at ambient temperature[J]. Bioresource Technology, 2011, 102(3): 2367-2372. DOI:10.1016/j.biortech.2010.10.101 |
| [11] | Gonzalez-Silva B M, Rønning A J, Andreassen I K, et al. Changes in the microbial community of an anammox consortium during adaptation to marine conditions revealed by 454 pyrosequencing[J]. Applied Microbiology and Biotechnology, 2017, 101(12): 5149-5162. DOI:10.1007/s00253-017-8160-5 |
| [12] | Wei Q Y, Kawagoshi Y, Huang X W, et al. Nitrogen removal properties in a continuous marine anammox bacteria reactor under rapid and extensive salinity changes[J]. Chemosphere, 2016, 148: 444-451. DOI:10.1016/j.chemosphere.2016.01.041 |
| [13] | Van De Graaf A A, De Bruijn P, Robertson L A, et al. Autotrophic growth of anaerobic ammonium-oxidizing micro-organisms in a fluidized bed reactor[J]. Microbiology, 1996, 142(8): 2187-2196. DOI:10.1099/13500872-142-8-2187 |
| [14] | 国家环境保护总局. 水和废水监测分析方法[M]. (第四版). 北京: 中国环境科学出版社, 2002. |
| [15] | Strous M, Heijnen J J, Kuenen J G, et al. The sequencing batch reactor as a powerful tool for the study of slowly growing anaerobic ammonium-oxidizing microorganisms[J]. Applied Microbiology and Biotechnology, 1998, 50(5): 589-596. DOI:10.1007/s002530051340 |
| [16] |
王子超, 高孟春, 魏俊峰, 等. 盐度变化对厌氧污泥胞外聚合物的影响[J]. 环境科学学报, 2016, 36(9): 3273-3281. Wang Z C, Gao M C, Wei J F, et al. Effect of salinity on extracellular polymeric substances of anaerobic sludge[J]. Acta Sctentiae Circumstantiae, 2016, 36(9): 3273-3281. |
| [17] | Li X R, Du B, Fu H X, et al. The bacterial diversity in an anaerobic ammonium-oxidizing (anammox) reactor community[J]. Systematic and Applied Microbiology, 2009, 32(4): 278-289. DOI:10.1016/j.syapm.2009.03.002 |
| [18] | Park H, Rosenthal A, Ramalingam K, et al. Linking community profiles, gene expression and N-removal in anammox bioreactors treating municipal anaerobic digestion reject water[J]. Environmental Science & Technology, 2010, 44(16): 6110-6116. |
| [19] | Cho S, Takahashi Y, Fujii N, et al. Nitrogen removal performance and microbial community analysis of an anaerobic up-flow granular bed anammox reactor[J]. Chemosphere, 2010, 78(9): 1129-1135. DOI:10.1016/j.chemosphere.2009.12.034 |
| [20] | Garrity G, Brenner D J, Krieg N R, et al. Bergey's manual® of systematic bacteriology:Volume 2:the proteobacteria[M]. Berlin: Springer-Verlag, 2005: 735-769. |
| [21] | Kindaichi T, Yuri S, Ozaki N, et al. Ecophysiological role and function of uncultured Chloroflexi in an anammox reactor[J]. Water Science & Technology, 2012, 66(12): 2556-2561. |
| [22] | Cao S B, Du R, Li B K, et al. High-throughput profiling of microbial community structures in an ANAMMOX-UASB reactor treating high-strength wastewater[J]. Applied Microbiology and Biotechnology, 2016, 100(14): 6457-6467. DOI:10.1007/s00253-016-7427-6 |
| [23] | Ward N L, Challacombe J F, Janssen P H, et al. Three genomes from the phylum Acidobacteria provide insight into the lifestyles of these microorganisms in soils[J]. Applied and Environmental Microbiology, 2009, 75(7): 2046-2056. DOI:10.1128/AEM.02294-08 |
| [24] | Bryant D A, Frigaard N U. Prokaryotic photosynthesis and phototrophy illuminated[J]. Trends in microbiology, 2006, 14(11): 488-496. DOI:10.1016/j.tim.2006.09.001 |
| [25] | Kartal B, Rattray J, Van Niftrik sL A, et al. Candidatus "Anammoxoglobus propionicus" a new propionate oxidizing species of anaerobic ammonium oxidizing bacteria[J]. Systematic and Applied Microbiology, 2007, 30(1): 39-49. DOI:10.1016/j.syapm.2006.03.004 |
| [26] | Hu B L, Zheng P, Tang C J, et al. Identification and quantification of anammox bacteria in eight nitrogen removal reactors[J]. Water Research, 2010, 44(17): 5014-5020. DOI:10.1016/j.watres.2010.07.021 |
| [27] | Sun W J, Banihani Q, Sierra-Alvarez R, et al. Stoichiometric and molecular evidence for the enrichment of anaerobic ammonium oxidizing bacteria from wastewater treatment plant sludge samples[J]. Chemosphere, 2011, 84(9): 1262-1269. DOI:10.1016/j.chemosphere.2011.04.079 |
| [28] | Ding S Z, Bao P, Wang B, et al. Long-term stable simultaneous partial nitrification, anammox and denitrification (SNAD) process treating real domestic sewage using suspended activated sludge[J]. Chemical Engineering Journal, 2018, 339: 180-188. DOI:10.1016/j.cej.2018.01.128 |
| [29] | Liu W R, Yang D H, Chen W J, et al. High-throughput sequencing-based microbial characterization of size fractionated biomass in an anoxic anammox reactor for low-strength wastewater at low temperatures[J]. Bioresource Technology, 2017, 231: 45-52. DOI:10.1016/j.biortech.2017.01.050 |
| [30] | Pujalte M J, Lucena T, Ruvira M A, et al. The family Rhodobacteraceae[A]. In: Rosenberg E, DeLong E F, Lory S, et al (Eds.). The Prokaryotes: Alphaproteobacteria and Betaproteobacteria. Berlin: Springer, 2014. 439-512. |
| [31] | Iizuka T, Jojima Y, Fudou R, et al. Isolation of myxobacteria from the marine environment[J]. FEMS Microbiology Letters, 1998, 169(2): 317-322. DOI:10.1111/fml.1998.169.issue-2 |
| [32] | Sabumon P C. Anaerobic ammonia removal in presence of organic matter:a novel route[J]. Journal of Hazardous Materials, 2007, 149(1): 49-59. DOI:10.1016/j.jhazmat.2007.03.052 |
| [33] | Riemann L, Farnelid H, Steward G F. Nitrogenase genes in non-cyanobacterial plankton:prevalence, diversity and regulation in marine waters[J]. Aquatic Microbial Ecology, 2010, 61(3): 235-247. DOI:10.3354/ame01431 |
| [34] | Spring S, Kämpfer P, Schleifer K H. Limnobacter thiooxidans gen. nov., sp. nov., a novel thiosulfate-oxidizing bacterium isolated from freshwater lake sediment[J]. International Journal of Systematic and Evolutionary Microbiology, 2001, 51(4): 1463-1470. DOI:10.1099/00207713-51-4-1463 |
| [35] | Abraham W R, Strömpl C, Vancanneyt M, et al. Woodsholea maritima gen. nov., sp. nov., a marine bacterium with a low diversity of polar lipids[J]. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(4): 1227-1234. DOI:10.1099/ijs.0.02943-0 |
| [36] | Nelson M C, Morrison M, Yu Z T. A meta-analysis of the microbial diversity observed in anaerobic digesters[J]. Bioresource Technology, 2011, 102(4): 3730-3739. DOI:10.1016/j.biortech.2010.11.119 |
| [37] | Kawaichi S, Ito N, Kamikawa R, et al. Ardenticatena maritima gen. nov., sp. nov., a ferric iron-and nitrate-reducing bacterium of the phylum 'Chloroflexi' isolated from an iron-rich coastal hydrothermal field, and description of Ardenticatenia classis nov[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(8): 2992-3002. |
| [38] | Bowman J P. The family cryomorphaceae[M]. In: Rosenberg E, DeLong E F, Lory S, et al (Eds.). Berlin: Springer, 2014. 539-550. |
| [39] | Windey K, De Bo I, Verstraete W. Oxygen-limited autotrophic nitrification-denitrification (OLAND) in a rotating biological contactor treating high-salinity wastewater[J]. Water Research, 2005, 39(18): 4512-4520. DOI:10.1016/j.watres.2005.09.002 |
| [40] | Dinçer A R, Kargi F. Performance of rotating biological disc system treating saline wastewater[J]. Process Biochemistry, 2001, 36(8-9): 901-906. DOI:10.1016/S0032-9592(00)00287-9 |
| [41] | Molinuevo B, García M C, Karakashev D, et al. Anammox for ammonia removal from pig manure effluents:effect of organic matter content on process performance[J]. Bioresource Technology, 2009, 100(7): 2171-2175. DOI:10.1016/j.biortech.2008.10.038 |
| [42] | Björnsson L, Hugenholtz P, Tyson G W, et al. Filamentous Chloroflexi (green non-sulfur bacteria) are abundant in wastewater treatment processes with biological nutrient removal[J]. Microbiology, 2002, 148(8): 2309-2318. DOI:10.1099/00221287-148-8-2309 |
| [43] | Fernández-Gomez B, Richter M, Schüler M, et al. Ecology of marine Bacteroidetes:a comparative genomics approach[J]. The ISME Journal, 2013, 7(5): 1026-1037. DOI:10.1038/ismej.2012.169 |
| [44] | Jetten M, Schmid M, Van De Pas-Schoonen K, et al. Anammox organisms:enrichment, cultivation, and environmental analysis[J]. Methods in Enzymology, 2005, 397: 34-57. DOI:10.1016/S0076-6879(05)97003-1 |
| [45] | Yokota N, Watanabe Y, Tokutomi T, et al. High-rate nitrogen removal from waste brine by marine anammox bacteria in a pilot-scale UASB reactor[J]. Applied Microbiology and Biotechnology, 2018, 102(3): 1501-1512. DOI:10.1007/s00253-017-8663-0 |
 2019, Vol. 40
2019, Vol. 40


