2. 环境保护部南京环境科学研究所, 南京 210042;
3. 南阳师范学院生命科学学院, 南阳 473061
2. Nanjing Institute of Environmental Sciences, Ministry of Environmental Protection, Nanjing 210042, China;
3. School of Life Science and Technology, Nanyang Normal University, Nanyang 473061, China
库滨带或缓冲带(riparian buffer strips)是陆地生态系统和水域生态系统的过渡带, 通过库滨带植物及其土壤微生物拦截、吸收、分解污染物质, 能有效遏制水土流失、防止水体富营养化[1, 2].长期监测表明, 由于存在农业面源、村镇生活污水等污染, 造成丹江口库区承载着较大的氮、磷负荷[3, 4].有研究表明白茅、狗牙根、剌槐、苘麻、蛇床群丛等典型库滨带植物在氮、磷等元素的截留和消减过程中起到重要作用[5~8].微生物是土壤元素生物化学地球循环的主要驱动力, 目前库滨带植物截留和消减氮、磷等元素过程中微生物群落组成和功能研究较少开展[9, 10].王萃等[11]研究了北京密云水库芦苇、杂草等不同植被的库滨带土壤中氨氧化细菌(AOB)的多样性和丰度特征.梁菊等[12]对太湖竺山湾缓冲带人工草林土壤反硝化势和反硝化细菌数量进行了测定.高通量测序技术(high throughput sequencing)能获取大量微生物16S rRNA基因相关的种群信息, 可较全面地了解生物群落组成特征, 目前国内外研究者已采用该方法研究各种土壤微生物群落组成、分布特征及其影响因素等[13~17], 但目前其在库滨带植物土壤微生物群落组成研究中应用较少.同时, 之前高通量测序数据主要关注于微生物群落结构(α和β多样性), 无法开展其功能研究. PICRUSt(phylogenetic investigation of communities by reconstruction of unobserved states)是一款菌群功能和代谢的预测软件, 可以通过16S rRNA基因序列预测对应的细菌代谢功能谱[18]. PICRUSt功能预测分析已在植物根际土壤[19, 20]、湖泊沉积物[21]、海水[22]等多种生境中成功应用, 初步分析了微生物功能.目前, 基于高通量测序的PICRUSt功能预测分析在库滨带微生物研究中鲜见报道, 该方法的运用将为探明库滨带微生物群落组成和功能提供重要帮助.
本研究选取丹江口库区库滨带人工种植的4种典型植物(草本植物香根草、芦苇、乔木植物杜梨和灌木植物假奓包叶), 采用16S rDNA MiSeq高通量测序技术(Illumina公司MiSeq PE300测序平台)研究根际细菌群落多群落组成, 结合PICRUSt分析预测根际细菌功能, 从微生物群落和功能角度分析不同植物截留和消减氮、磷等元素的差异, 以期为丹江口水库库滨带植被构建及其水环境保护提供参考依据.
1 材料与方法 1.1 样品采集与理化指标测定选取构建于丹江口库区库滨带种植资源圃(坐标:32°34′22.85″N, 111°31′32.67″E)的草本植物香根草(Vetiveria zizanioides, 后文图注中缩写为VZ)、芦苇(Phragmites australis, PA)、乔木植物杜梨(Pyrus betulifolia, PB)和灌木植物假奓包叶(Discocleidion rufescens, DR).根据之前研究, 这些植物在丹江口库区库滨带具有良好的生存能力, 并具有净化水质、拦截污染物和泥沙等功能[6, 7].于2017年6月采用抖落法收集植物根际土壤样品, 每样品设置3个生物学重复.将收集的土壤样品置于预先灭菌容器中存于冰盒, 运回实验室用于提取细菌总DNA, 剩余土壤样品用于理化指标测定.所采土壤样品带回实验室于阴凉处室温风干, 测定土壤样品pH值、有机质总量(SOM)、总氮(TN)、总磷(TP)、总钾(TK)、铵态氮(NH4+-N)和硝态氮(NO3--N)等理化指标.
1.2 样品总DNA提取根际细菌总DNA采用美国MP公司FastDNA® Spin Kit for Soil试剂盒提取, 提取步骤参照说明书.将提取得到的根际细菌总DNA通过微量紫外分光光度计(NanoDrop® ND-1000, Wilmington, DE, USA)测定DNA浓度和纯度[14].
1.3 高通量测序采用通用引物(338F/806R)对细菌16S rRNA基因的V3~V4区进行PCR扩增, 扩增体系为5×FastPfu Buffer 4 μL、2.5 mmol·L-1、dNTPs 2 μL、Forward Primer(5 μmol·L-1) 0.8 μL、Reverse Primer(5 μmol·L-1) 0.8 μL、FastPfu Polymerase 0.4 μL、DNA模板10 ng, 补充ddH2O至20 μL. PCR扩增的反应条件为: 94℃, 5 min; 30×(94℃, 30 s; 54℃, 30 s; 72℃, 45 s); 72℃, 10 min[4, 23].修饰后的通用引物含有不同的Tag标签用以区分不同样品, 利用上海美吉生物医药科技有限公司的MiSeq PE300测序仪(Illumina Inc, San Diego, CA, USA)完成序列测定.
1.4 数据分析 1.4.1 高通量数据分析高通量数据的生物信息学分析采用QⅡME(quantitative insights into microbial ecology)进行, 根据序列的相似度, 将序列归为多个OTU(operational taxonomic unit), OTU产出后, 统计各个样品含有OTU情况及每个OTU中含有序列的数目, 得到每个OTU的分类学信息[24].选取相似度在97%条件下的OTU生成预期的稀释曲线[25].利用非度量多维尺度(NMDS)、Correlation聚类法分别进行数据处理、细菌群落分布、聚类分析.采用LEfSe在线工具寻找组与组之间有统计学差异的生物标志物[26].
1.4.2 PICRUSt功能预测分析功能和代谢途径预测采用PICRUSt软件进行分析, 利用QⅡME获得的colsed OTU Table与COG、KEGG数据库进行比对, 获得不同的数据库功能预测信息, 具体分析步骤基于在线分析平台(http://picrust.github.io/picrust/)[18].预测基因丰度采用Heml heat map illustrator program (版本1.0.3)软件进行热图绘制[27].
1.4.3 统计分析土壤理化性质、高通量测序结果及多样性指数等数据分析在SPSS 17.0中进行, 组间差异性检验采用t检验和单因素方差分析.
2 结果与分析 2.1 丹江口库区库库滨带植物根际土壤理化性质对所采集4种人工种植的库滨带植物土壤理化性质进行测定, 结果如表 1所示. 4种植物根际土壤pH值较为相近, 介于7.20~7.91之间.总氮、总磷含量方面芦苇样品最高, 分别为0.65 g·kg-1和0.80 g·kg-1, 显著高于其他样品(P < 0.05), 假奓包叶总氮最低, 为0.24 g·kg-1, 香根草总磷含量最低, 为0.58g·kg-1. 4种土壤样品中除香根草外(95.12 mg·kg-1), 硝态氮含量均超过100 mg·kg-1, 其中假奓包叶样品最高(为129.82 mg·kg-1).铵态氮含量也是假奓包叶样品最高, 为77.38 mg·kg-1, 香根草次之, 为68.19 mg·kg-1.土壤有机质介于20.58~27.46 g·kg-1之间, 杜梨和芦苇样品高于其他样品.
|
|
表 1 各样品土壤主要理化参数1) Table 1 Main physical and chemical properties of different samples |
2.2 不同植物根际细菌群落结构分析 2.2.1 高通量测序结果及多样性的评估
高通量测序结果表明, 样品平均测序序列条数为37344, 样品的文库覆盖率(good's coverage)高于97.35%, 在测序条数达到30000条以上时稀释性曲线趋向平稳, 表明测序数据量合理, 能够代表物种的丰富度(表 2).采用群落丰富度指数Chao1和ACE、群落多样性指数Shannon、Simpson和文库覆盖率等对丹江口库区库滨带植物根际细菌群落进行评估, 结果表明样品具有丰富的群落组成, 其群落多样性均较高(表 2).综合Sobs、丰富度指数Chao1和Ace以及多样性指数Simpson、Shannon、文库覆盖率, 4种植物根际细菌群落多样性排序为:芦苇>香根草>假奓包叶>杜梨.
|
|
表 2 不同样品根际细菌群落多样性评估1) Table 2 Estimation of the rhizobacteria community diversity in different samples |
2.2.2 细菌群落组成及Beta多样性分析
高通量测序结果表明, 4种库滨带植物根际细菌由31个门组成, 包括变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、芽单胞菌门(Gemmatimonadetes)、酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、硝化螺旋菌门(Nitrospirae)、疣微菌门(Verrucomicrobia)等(图 1).属于变形菌门、拟杆菌门、放线菌门、芽单胞菌门、酸杆菌门和绿弯菌门的序列总和占全部序列的77.79%~90.68%, 这些细菌为优势种群(图 1).其中, 变形菌门是所有样品中含量最高的门, 占比为28.01%~48.53%;酸杆菌门为香根草、芦苇和假奓包叶的含量第二高的门, 占比10.82~16.52%, 放线菌门为杜梨含量第二高的门, 占比为19.04%.对其他分类单元细菌进行分析, 香根草样品包含22门、43纲、81目、134科、212属; 芦苇样品包含23门、45纲、89目、156科、229属; 假奓包叶样品包含26门、49纲、97目、163科、229属; 杜梨样品包含23门、47纲、91目、145科、213属(图 2).
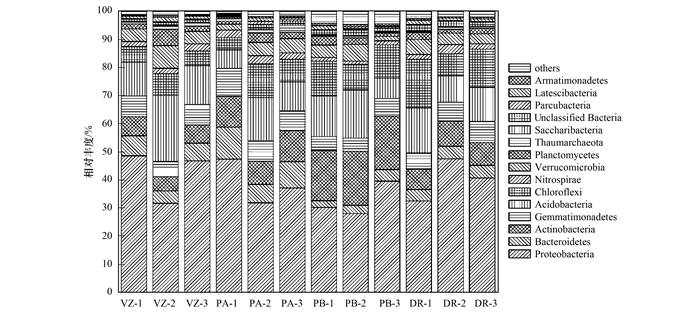
|
图 1 不同样品门水平上物种相对丰度分布 Fig. 1 Relative read abundance of rhizobacteria community structures at the phylum level in different samples |
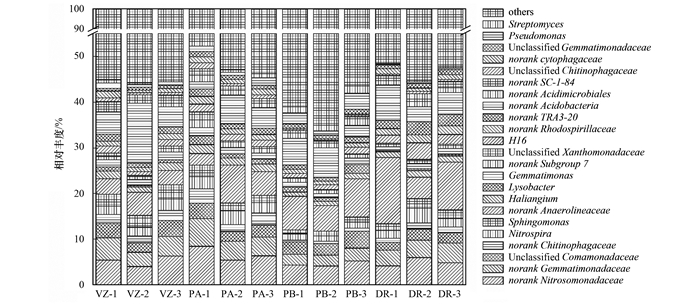
|
图 2 不同样品属水平上物种相对丰度分布 Fig. 2 Relative read abundance of rhizobacteria community structures at the genus level in different samples |
非度量多维尺度(NMDS)和UPGMA聚类分析均能描述不同样品间群落差异, 如果两个样品距离较近, 表示这两个样品的物种组成较相似.丹江口库区库滨带植物根际细菌群落NMDS分析(OTUs水平)结果如图 3所示, NMDS排序的胁强系数(Stress)为0.10, 可用NMDS的二维点图表示.在NMDS图中香根草聚集于图右侧, 芦苇样品聚集于图下侧, 假奓包叶样品聚集于图上侧, 杜梨样品聚集于图左侧, 以上结果表明不同植物类型根际细菌群落结构有所差异.基于unweighted unifrac的UPGMA聚类分析结果与NMDS分析结果类似, 在0.176的相似性水平上, 4组样品分为4大组, 其中香根草和芦苇群落结构在0.215相似性水平上聚集于一起, 表明群落结构较其他样品更为相似(图 4).
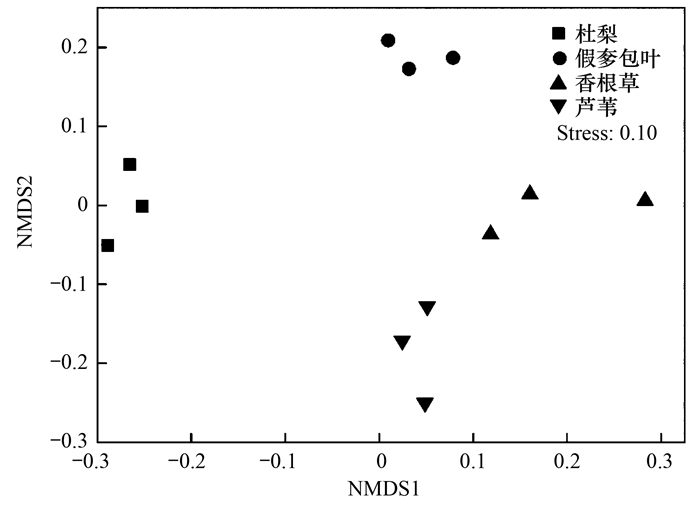
|
图 3 不同植物根际土壤细菌多样性的NMDS分析 Fig. 3 NMDS results for the rhizobacteria community diversity in different plants |
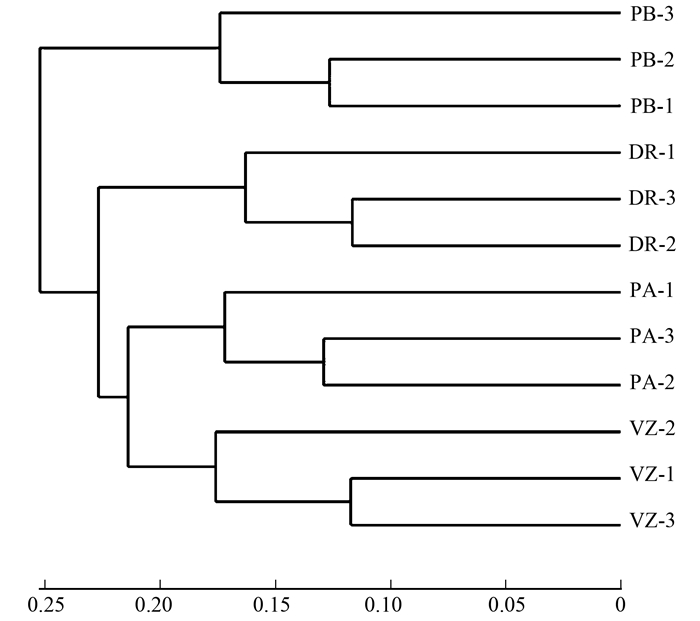
|
图 4 不同样品UPGMA聚类图 Fig. 4 UPGMA clusters of different bacterial communities |
采用在线统计工具LEfSe来寻找宏基因组生物标志物, 结果用饼形图表示(图 5)[18], 其中最内圈表示门水平的差异, 依次往外的圈表示纲、目、科、属, 不同颜色扇形面积表示在此范围内的菌均有差异.在门的水平上, 香根草样品中2个门(Zixibacteria和TM6)的细菌存在显著差异, 芦苇样品中4个门(Candidatus Berkelbacteria、Chloroflexi、Parcubacteria和Saccharibacteria)的细菌存在显著差异, 假奓包叶样品中1个门(Ignavibacteriae)的细菌存在显著差异, 杜梨样品中4个主要的门(Actinobacteria、Firmicutes、Spirochaetae和Synergistetes)的细菌存在显著差异.在属的水平上, 香根草样品中变形菌门的Devosia、Acidocella、Roseomonas、Sphingobium, 酸杆菌门的Acidicapsa, 放线菌门的Frankia, 拟杆菌门的Pedobacter等共34个属的细菌存在显著差异; 芦苇样品中变形菌门的Phenylobacterium、Defluviicoccus、Ferrovibrio、Achromobacter, 酸杆菌门的Bifidobacterium、Actinoplanes、Kibdelosporangium, 拟杆菌门的Adhaeribacter、Sporocytophaga、Flavihumibacter、Niabella, 厚壁菌门的Faecalibaculum, Synergistetes的Thermovirga, 疣微菌门的Alterococcus、Pedosphaera等共59属的细菌存在显著差异; 假奓包叶样品中变形菌门的Rhizorhapis、Magnetospirillum、Sulfurifustis, 酸杆菌门的Bryobacter、Paludibaculum, 拟杆菌门的Emticicia, 厚壁菌门的Domibacillus等共43属的细菌存在显著差异; 杜梨样品中放线菌门Nocardia、Tsukamurella、Jatrophihabitans、Blastococcus, 酸杆菌门的Terriglobus, 拟杆菌门的Lentimicrobium、Solitalea, 蓝细菌门的Synechococcus、Leptolyngbya, 厚壁菌门的Paenibacillus、Staphylococcus、Streptococcus, 变形菌门的Pleomorphomonas、Mesorhizobium、Shinella、Parvibaculum等共81属的细菌存在显著差异(图 5).
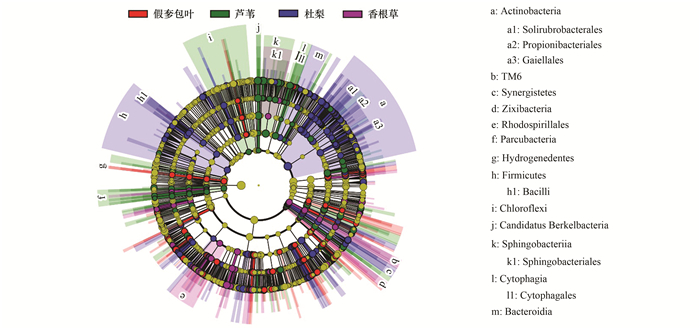
|
图 5 不同样品差异细菌分布 Fig. 5 Taxon with statistical differences between different bacterial communities |
为了获得不同植被土壤细菌的功能, 本研究采用PICRUSt软件进行菌群预测分析.利用PICRUSt预测微生物的功能时, 可以采用weighted nearest sequenced taxon index(weighted NSTI)指数来评估每个给定的样品中的序列与已知数据库中测定基因的加权平均距离[18]. NSTI指数的数值越小, 说明预测样品中的微生物组成与已知的数据库匹配度越高.不同植被土壤细菌功能预测的NSTI指数如图 6所示, 香根草样品NSTI指数为0.18~0.23, 芦苇样品NSTI指数为0.18~0.22, 杜梨样品NSTI指数为0.17~0.19, 假奓包叶样品NSTI指数为0.20~0.22, 该结果与Langille等[18]研究的土壤样品NSTI指数相近(平均NSTI值为0.17).

|
图 6 Picrust功能预测指标NSTI Fig. 6 NSTI of picrust predictive functional profiling |
通过对KEGG数据库(Kyoto encyclopedia of genes and genomes)进行比对, 共获得6类生物代谢通路功能分析(一级功能层):代谢(metabolism)、遗传信息处理(genetic information processing)、环境信息处理(environmental information processing)、细胞过程(cellular processes)、有机系统(organismal systems)和人类疾病(human diseases)(图 7).其中代谢、遗传信息处理和环境信息处理为其主要组成, 占比分别为51.59%~52.20%、15.76%~16.31%和12.58%~13.57%.不同植被土壤细菌群落功能预测比较表明, 代谢、遗传信息处理、环境信息处理和有机系统4个一级功能层预测基因拷贝数顺序为假奓包叶>芦苇>香根草>杜梨, 剩余的2个一级功能层(细胞过程和人类疾病)预测基因拷贝数为假奓包叶>香根草>芦苇>杜梨.同时对预测基因二级功能层进行分析, 发现其由次生产物代谢的生物合成(biosynthesis of other secondary metabolites)、转录(transcription)、多糖生物合成和代谢(glycan biosynthesis and metabolism)、细胞生长和死亡(cell growth and death)等38个子功能组成(图 7).对二级功能层预测基因拷贝数进行分析, 发现碳水化合物代谢(carbohydrate Metabolism)、氨基酸代谢(amino Acid Metabolism)、次生产物代谢的生物合成、维他命及辅因子代谢(metabolism of cofactors and vitamins)、细胞生长和死亡等20个子功能预测基因拷贝数顺序为假奓包叶>芦苇>香根草>杜梨; 多糖生物合成和代谢、信号转导(signal Transduction)等8个子功能预测基因拷贝数顺序为假奓包叶>香根草>芦苇>杜梨; 38个子功能组成中, 除异生素生物降解和代谢(xenobiotics viodegradation and metabolism)、膜运输(membrane transport)、免疫系统疾病(immune system diseases)之外, 杜梨样品预测基因拷贝数均为最低.
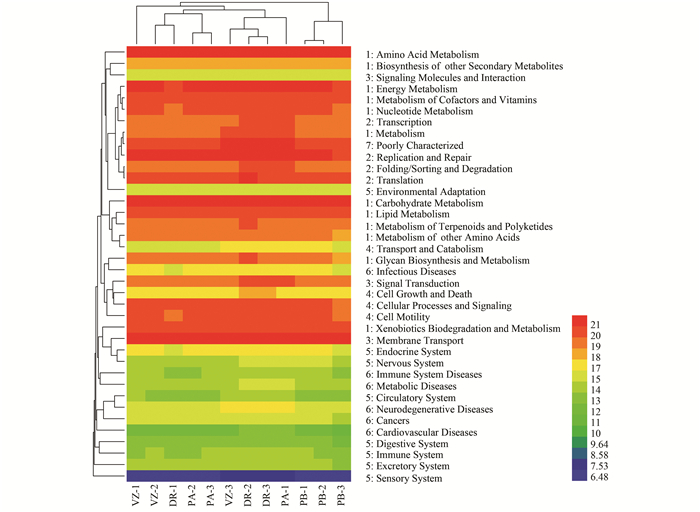
|
图中数字代表一级功能层分布, 1为代谢, 2为遗传信息处理, 3为环境信息处理, 4为细胞过程, 5为有机系统, 6为人类疾病 图 7 不同样品预测功能基因二级功能层热图 Fig. 7 Heat map of predicted functional profiles predicted for different plants (hierarchy level 2) |
目前库滨带植物根际微生物群落组成和功能研究较少开展[9, 10].王萃等[11]采用末端限制性片段长度多态性(T-RFLP)结合定量PCR的方法研究了北京密云水库沉积物和芦苇、杂草等不同植被的库滨带土壤中氨氧化细菌(AOB)的多样性和丰度特征, 发现沉积物与其他土壤样品AOB多样性有明显差异.任庆水等[28]采用T-RFLP技术研究了人工植被(牛鞭草与狗牙根)重建过程对三峡库区消落带土壤细菌群落结构的影响.以上研究, 鉴于实验方法的局限性, 均未对群落组成进行鉴定, 与T-RFLP等方法相比, 高通量测序技术能获取大量微生物16S rRNA基因相关的种群信息, 能更全面地了解生物群落组成特征[13].针对丹江口库区库滨带植物根际土壤微生物群落组成研究较少开展的现状, 本实验采用Illumina公司MiSeq测序平台对4种人工种植的库滨带植物根际土壤细菌群落组成进行了研究, 发现其主要由变形菌门、拟杆菌门、放线菌门、芽单胞菌门等31个门, Nitrospira、Sphingomonas、Haliangium、Lysobacter、Gemmatimonas、Pseudomonas等343个属的细菌组成, 表现出群落组成的丰富性.有研究表明, 植物根系通过产生分泌物和脱落物为根际区土壤微生物提供有效碳源和氮源, 导致根际土壤化学性质和生物学特性显著不同于非根际土壤, 即表现出根际效应[29, 30].本研究对丹江口库区库滨带4种植物根际土壤理化性质和细菌群落进行了分析, 结果表明不同植物类型根际土壤理化性质和生物学特性具有差异.细菌群落组成的NMDS分析表明, 2种草本植物香根草和芦苇细菌群落结构较为相似, 灌木植物假奓包叶细菌群落结构介于乔木植物杜梨与草本植物之间, 乔木植物杜梨细菌群落结构差异最大(表 2、图 3和图 4).研究表明不同植物类型对根际细菌群落组成具有显著影响, 该结果与此结论一致[31~34].综合Sobs、多样性指数和文库覆盖率分析, 4种植物根际细菌群落多样性排序为:芦苇>香根草>假奓包叶>杜梨.通过以上研究, 初步探明了丹江口库区库滨带不同植物根际细菌群落组成和影响因素.
3.2 库滨带植物根际细菌功能预测分析之前关于土壤细菌群落的高通量测序分析主要关注于细菌群落结构(α和β多样性), 对其功能的研究开展较少[10, 14~16].基于高通量测序的PICRUSt功能预测分析相较于宏基因组研究, 更方便快捷, 成本也更低, 同时预测效果可靠性较高[18].目前该方法已在土壤、水体微生物等不同生境中得到良好地应用, 为探明其功能提供了帮助[22, 35~37].董志颖等[22]采用PICRUSt功能预测分析过量氮输入对细菌群落代谢潜力的影响, 发现氮输入在一定程度上影响了微宇宙中的固氮、硝化、异化硝酸盐还原、反硝化、异化硝酸盐还原到铵和同化硝酸盐还原途径的关键基因.目前PICRUSt功能预测分析在库滨带微生物研究中鲜见报道, 为了探明丹江口库区库滨带不同植物根际细菌功能, 本研究将MiSeq高通量测序结果进行PICRUSt功能预测分析.结果表明, 丹江口库区库滨带根际细菌主要涉及代谢、遗传信息处理等6个生物代谢通路, 次生产物代谢的生物合成、转录、多糖生物合成和代谢、细胞生长和死亡等38个子功能, 表现出功能上的丰富性.为了比较不同植物根际细菌功能差异, 对不同植物根际细菌预测基因拷贝数进行分析, 发现一级功能层和二级功能层预测基因拷贝数整体趋势均为假奓包叶>芦苇>香根草>杜梨.该结果初步分析了丹江口库区库滨带不同植物根际细菌功能的差异, 但鉴于PICRUSt功能预测分析的局限性, 后续需要结合宏基因组测序和氮磷等元素循环功能基因分析, 从而准确掌握库滨带植物截留和消减氮、磷等元素过程中微生物群落功能.
4 结论(1) 本研究采用MiSeq测序的方法研究了4种人工种植的库滨带植物根际土壤细菌群落组成, 发现其主要由变形菌门、拟杆菌门、放线菌门等31个门, Nitrospira、Sphingomonas、Haliangium等343个属的细菌组成, 表现出群落组成的丰富性.
(2) 细菌群落分析表明不同植物类型对细菌群落组成具有显著影响, 草本植物香根草和芦苇细菌群落结构较为相似, 灌木植物假奓包叶细菌群落结构介于乔木植物杜梨与草本植物之间, 乔木植物杜梨细菌群落结构差异最大.细菌群落多样性排序为:芦苇>香根草>假奓包叶>杜梨.
(3) PICRUSt功能预测分析表明, 库滨带植物根际细菌主要涉及代谢、遗传信息处理等6个生物代谢通路, 次生产物代谢的生物合成、转录、多糖生物合成和代谢、细胞生长和死亡等38个子功能, 表现出功能上的丰富性.丹江口库区库滨带植物根际细菌代谢能力整体趋势为假奓包叶>芦苇>香根草>杜梨.
| [1] | Stutter M I, Chardon W J, Kronvang B. Riparian buffer strips as a multifunctional management tool in agricultural landscapes:introduction[J]. Journal of Environmental Quality, 2012, 41(2): 297-303. DOI:10.2134/jeq2011.0439 |
| [2] |
王晓锋, 袁兴中, 刘红, 等. 三峡库区消落带4种典型植物根际土壤养分与氮素赋存形态[J]. 环境科学, 2015, 36(10): 3662-3673. Wang X F, Yuan X Z, Liu H, et al. Nutrient characteristics and nitrogen forms of rhizosphere soils under four typical plants in the littoral zone of TGR[J]. Environmental Science, 2015, 36(10): 3662-3673. |
| [3] | Chen P, Li L, Zhang H B. Spatio-temporal variations and source apportionment of water pollution in Danjiangkou Reservoir basin, central China[J]. Water, 2015, 7(6): 2591-2611. |
| [4] |
陈兆进, 丁传雨, 朱静亚, 等. 丹江口水库枯水期浮游细菌群落组成及影响因素研究[J]. 中国环境科学, 2017, 37(1): 336-344. Chen Z J, Ding C Y, Zhu J Y, et al. Community structure and influencing factors of bacterioplankton during low water periods in Danjiangkou Reservoir[J]. China Environmental Science, 2017, 37(1): 336-344. |
| [5] |
占海歌, 蒋娟, 郝好鑫, 等. 丹江口水库库滨带典型植物群落氮矿化特征[J]. 中国水土保持科学, 2017, 15(1): 43-50. Zhan H G, Jiang J, Hao H X, et al. Soil nitrogen mineralization characteristics on typical vegetation community of riparian buffer strip in Danjiangkou Reservoir[J]. Science of Soil and Water Conservation, 2017, 15(1): 43-50. |
| [6] |
刘瑞雪, 陈龙清, 史志华. 丹江口水库水滨带植物群落空间分布及环境解释[J]. 生态学报, 2015, 35(4): 1208-1216. Liu R X, Chen L Q, Shi Z H. Spatial distribution of plant communities and environmental interpretation in the riparian zone of Danjiangkou Reservoir[J]. Acta Ecologica Sinica, 2015, 35(4): 1208-1216. |
| [7] |
曾祉祥, 雷沛, 张洪, 等. 丹江口水库典型消落区土壤氮磷赋存形态及释放特征研究[J]. 环境科学学报, 2015, 35(5): 1383-1392. Zeng Z X, Lei P, Zhang H, et al. Nitrogen and phosphorus fractions and releasing characteristics of the soils from the representative water-level-fluctuating zone of Danjiangkou Reservoir[J]. Acta Scientiae Circumstantiae, 2015, 35(5): 1383-1392. |
| [8] |
滕泽栋, 李敏, 朱静, 等. 野鸭湖湿地芦苇根际微生物多样性与磷素形态关系[J]. 环境科学, 2017, 38(11): 4589-4597. Teng Z D, Li M, Zhu J, et al. Effects of soil microbial diversity on the phosphate fraction in the rhizosphere of phragmites communis in the Yeyahu wetland in Beijing, China[J]. Environmental Science, 2017, 38(11): 4589-4597. |
| [9] | Falkowski P G, Fenchel T, Delong E F. The microbial engines that drive earth's biogeochemical cycles[J]. Science, 2008, 320(5879): 1034-1039. DOI:10.1126/science.1153213 |
| [10] | Rich J J, Myrold D D. Community composition and activities of denitrifying bacteria from adjacent agricultural soil, riparian soil, and creek sediment in Oregon, USA[J]. Soil Biology and Biochemistry, 2004, 36(9): 1431-1441. DOI:10.1016/j.soilbio.2004.03.008 |
| [11] |
王萃, 鲍林林, 王晓燕. 密云水库底泥和库滨区土壤中氨氧化细菌的多样性和丰度[J]. 环境科学学报, 2013, 33(12): 3334-3340. Wang C, Bao L L, Wang X Y. Diversity and abundance of ammonia oxidizing bacteria (AOB) communities in sediment and riparian soil around Miyun Reservoir[J]. Acta Scientiae Circumstantiae, 2013, 33(12): 3334-3340. |
| [12] |
梁菊, 叶春, 李春华, 等. 太湖竺山湾缓冲带两种人工草林土壤反硝化作用比较[J]. 环境科学研究, 2017, 30(5): 744-754. Liang J, Ye C, Li C H, et al. Comparison of soil denitrification in two kinds of planted forests in the buffer zone of Zhushan bay, Taihu lake[J]. Research of Environmental Sciences, 2017, 30(5): 744-754. |
| [13] | van Dijk E L, Auger H, Jaszczyszyn Y, et al. Ten years of next-generation sequencing technology[J]. Trends in Genetics, 2014, 30(9): 418-426. DOI:10.1016/j.tig.2014.07.001 |
| [14] |
丁传雨, 郑远, 任学敏, 等. 能源植物修复土壤镉污染过程中细菌群落分析[J]. 环境科学学报, 2016, 36(8): 3009-3016. Ding C Y, Zheng Y, Ren X M, et al. Changes in bacterial community composition during the remediation of Cd-contaminated soils of bioenergy crops[J]. Acta Scientiae Circumstantiae, 2016, 36(8): 3009-3016. |
| [15] | Liu J J, Sui Y Y, Yu Z H, et al. High throughput sequencing analysis of biogeographical distribution of bacterial communities in the black soils of northeast China[J]. Soil Biology and Biochemistry, 2014, 70: 113-122. DOI:10.1016/j.soilbio.2013.12.014 |
| [16] | Žifčáková L, Větrovsky T, Howe A, et al. Microbial activity in forest soil reflects the changes in ecosystem properties between summer and winter[J]. Environmental Microbiology, 2016, 18(1): 288-301. DOI:10.1111/emi.2016.18.issue-1 |
| [17] | Yang Y, Wang N, Guo X Y, et al. Comparative analysis of bacterial community structure in the rhizosphere of maize by high-throughput pyrosequencing[J]. PLoS One, 2017, 12(5): e0178425. DOI:10.1371/journal.pone.0178425 |
| [18] | Langille M G I, Zaneveld J, Caporaso J G, et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences[J]. Nature Biotechnology, 2013, 31(9): 814-821. DOI:10.1038/nbt.2676 |
| [19] | Pii Y, Borruso L, Brusetti L, et al. The interaction between iron nutrition, plant species and soil type shapes the rhizosphere microbiome[J]. Plant Physiology and Biochemistry, 2016, 99: 39-48. DOI:10.1016/j.plaphy.2015.12.002 |
| [20] | Jiang L F, Song M K, Yang L, et al. Exploring the influence of environmental factors on bacterial communities within the rhizosphere of the Cu-tolerant plant, Elsholtzia splendens[J]. Scientific Reports, 2016, 6: 36302. DOI:10.1038/srep36302 |
| [21] |
阴星望, 田伟, 丁一, 等. 丹江口库区表层沉积物细菌多样性及功能预测分析[J]. 湖泊科学, 2018, 30(4): 1052-1063. Yin X W, Tian W, Ding Y, et al. Composition and predictive functional analysis of bacterial communities in surface sediments of the Danjiangkou Reservoir[J]. Journal of Lake Sciences, 2018, 30(4): 1052-1063. |
| [22] |
董志颖, 洪慢, 胡晗静, 等. 过量氮输入对寡营养海水细菌群落代谢潜力的影响[J]. 环境科学学报, 2018, 38(2): 457-466. Dong Z Y, Hong M, Hu H J, et al. Effect of excess nitrogen loading on the metabolic potential of the bacterial community in oligotrophic coastal water[J]. Acta Scientiae Circumstantiae, 2018, 38(2): 457-466. |
| [23] | Chen Z J, Zheng Y, Ding C Y, et al. Integrated metagenomics and molecular ecological network analysis of bacterial community composition during the phytoremediation of cadmium-contaminated soils by bioenergy crops[J]. Ecotoxicology and Environmental Safety, 2017, 145: 111-118. DOI:10.1016/j.ecoenv.2017.07.019 |
| [24] | Caporaso J G, Kuczynski J, Stombaugh J, et al. QⅡME allows analysis of high-throughput community sequencing data[J]. Nature methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [25] | Schloss P D, Westcott S L, Ryabin T, et al. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [26] | Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biology, 2011, 12(6): R60. DOI:10.1186/gb-2011-12-6-r60 |
| [27] | Deng W K, Wang Y B, Liu Z X, et al. HemI:a toolkit for illustrating heatmaps[J]. PLoS One, 2014, 9(11): e111988. DOI:10.1371/journal.pone.0111988 |
| [28] |
任庆水, 马朋, 李昌晓, 等. 三峡库区消落带两种草本植被土壤细菌群落多样性[J]. 生态学报, 2016, 36(11): 3261-3272. Ren Q S, Ma P, Li C X, et al. Evaluation of bacterial diversity under different herb vegetation types in the hydro-fluctuation zone of the Three Gorges Reservoir in China[J]. Acta Ecologica Sinica, 2016, 36(11): 3261-3272. |
| [29] | Bais H P, Weir T L, Perry L G, et al. The role of root exudates in rhizosphere interactions with plants and other organisms[J]. Annual Review of Plant Biology, 2006, 57: 233-266. DOI:10.1146/annurev.arplant.57.032905.105159 |
| [30] | Sánchez-Cañizares C, Jorrín B, Poole P S, et al. Understanding the holobiont:the interdependence of plants and their microbiome[J]. Current Opinion in Microbiology, 2017, 38: 188-196. DOI:10.1016/j.mib.2017.07.001 |
| [31] |
刘洋, 黄懿梅, 曾全超. 黄土高原不同植被类型下土壤细菌群落特征研究[J]. 环境科学, 2016, 37(10): 3931-3938. Liu Y, Huang Y M, Zeng Q C. Soil bacterial communities under different vegetation types in the Loess Plateau[J]. Environmental Science, 2016, 37(10): 3931-3938. |
| [32] |
朱平, 陈仁升, 宋耀选, 等. 祁连山中部4种典型植被类型土壤细菌群落结构差异[J]. 生态学报, 2017, 37(10): 3505-3514. Zhu P, Chen R S, Song Y X, et al. Soil bacterial community composition and diversity of four representative vegetation types in the middle section of the Qilian Mountains, China[J]. Acta Ecologica Sinica, 2017, 37(10): 3505-3514. |
| [33] | Marschner P, Yang C H, Lieberei R, et al. Soil and plant specific effects on bacterial community composition in the rhizosphere[J]. Soil Biology and Biochemistry, 2001, 33(11): 1437-1445. DOI:10.1016/S0038-0717(01)00052-9 |
| [34] | Tkacz A, Cheema J, Chandra G, et al. Stability and succession of the rhizosphere microbiota depends upon plant type and soil composition[J]. The ISME Journal, 2015, 9(11): 2349-2359. DOI:10.1038/ismej.2015.41 |
| [35] | Zarraonaindia I, Owens S M, Weisenhorn P, et al. The soil microbiome influences grapevine-associated microbiota[J]. mBio, 2015, 6(2): e02527-14. |
| [36] | Sun D Q, Meng J, Xu E G, et al. Microbial community structure and predicted bacterial metabolic functions in biochar pellets aged in soil after 34 months[J]. Applied Soil Ecology, 2016, 100: 135-143. DOI:10.1016/j.apsoil.2015.12.012 |
| [37] | Zhu S S, Vivanco J M, Manter D K. Nitrogen fertilizer rate affects root exudation, the rhizosphere microbiome and nitrogen-use-efficiency of maize[J]. Applied Soil Ecology, 2016, 107: 324-333. DOI:10.1016/j.apsoil.2016.07.009 |
 2019, Vol. 40
2019, Vol. 40


