由于具有稳定的化学结构、简单的生产过程和低成本, 偶氮染料是被广泛应用于印染、造纸和皮革等行业的典型活性染料[1].据统计每年生产的7万t染料中, 偶氮染料约占70%, 且其中10%~15%可能在生产过程中排放至水体[2].偶氮染料具有致癌性, 不仅会导致人体肝脏和肾受损, 也会在水中积累并抑制水生生物的生长[2, 3].因此, 偶氮染料的有效控制是工业废水处理的重要组成部分.
相比化学和物理处理, 偶氮染料的生物处理具有环境友好、高效和污泥产量低等优势[4].好氧生物处理条件下, 氧气比偶氮染料优先作为电子受体, 因此, 厌氧生物处理更适于偶氮染料降解. Li等[5]采用厌氧生物处理染料酸性橙7, 得到脱色率为71.8%和化学需氧量(COD)去除率为50.6%. Reddy等[6]采用缺氧间歇式反应器处理染料酸性黑10B, 得到脱色率为49.5%.生物降解过程中, 偶氮染料利用共基质降解释放的电子断裂—N=N—, 生成无色芳香胺类化合物.芳香胺类化合物需要在好氧条件下利用非特异性酶实现完全矿化[6].因此, 厌氧/好氧(AO)生物工艺常用于处理印染废水. Balapure等[7]采用序批式厌氧/微氧工艺处理印染废水, 脱色率可达到99%, 其中厌氧段脱色率为80%. Frindt等[8]利用厌氧/好氧工艺处理含偶氮染料活性橙107废水, 厌氧脱色率为97%, 而偶氮染料代谢产物芳香胺化合物在好氧条件下得到降解.
偶氮染料的生物降解主要依靠微生物酶作用实现.偶氮还原酶和虫漆酶是偶氮染料降解的主要作用酶, 能产生此两种酶的菌群包括Xenophilus azovorans KF46F、Pigmentiphaga kullae K24、Enterococcus faecalis、Staphylococcus aureus、Escherichia coli、Bacillus sp.OY1-2和Rhodobacter sphaeroides等[9].以上很多菌群可成为偶氮染料降解的优势菌种. Chen等[10]采用填充床生物反应器处理含维多利亚蓝废水时, 得到优势菌种为Aeromonas hydrophila. Xie等[4]采用水解酸化处理染料活性黑5和活性艳蓝19印染废水, 发现Klebsiella、Lactococcus和Desulfovibrio可能是染料降解的优势菌, 且Dysgonomonas sp.和Prevotella sp.可能在染料代谢过程中起重要作用.
印染废水中氨氮(NH4+-N)可通过硝化反硝化去除, 其中硝化反应是限制过程.硝化过程对有毒物质较为敏感, 而偶氮染料的代谢产物苯胺是一种有毒物质, 因此可能会影响硝化效率. Khin等[11]发现苯胺浓度在3~250 mg·L-1之间, 硝化会受到不同程度的抑制, 仅当苯胺浓度低于0.5 mg·L-1时, 才会发生完全硝化. Thanh等[12]发现当苯胺浓度超过20 mg·L-1时, 硝化反应完全受到抑制, 且苯胺抑制亚硝酸盐氮(NO2--N)氧化强于NH4+-N氧化.而Joel等[13]证明苯胺降解和硝化可在污泥龄为7 d的活性污泥反应器中同时发生, 硝化菌会适应苯胺存在的环境.
本研究采用水解酸化/AO工艺处理含有染料活性艳红X-3B(RR2)的废水, 重点考察工艺运行性能.同时, 采用高通量测序方法检测水解酸化/AO工艺中的菌群种类, 以解析其在染料去除或脱氮过程中的作用.此外, 本研究还分析了水解酸化过程中影响色度去除的关键因素以及硝化过程中染料及其代谢产物的影响, 以期为该工艺应用于实际印染废水生物处理提供理论指导和技术支持.
1 材料与方法 1.1 试验装置及其运行水解酸化序批式反应器(SBR)体积为6 L, 运行过程中温度为25℃、挥发性悬浮固体浓度(VSS)为(5.17±0.33)g·L-1.水解酸化SBR运行周期为6 h, 包括5 h厌氧搅拌(含10 min进水), 50 min静置沉淀和10 min出水. AO SBR反应器有效体积为5 L, 运行温度为25℃且VSS为(2.11±0.28)g·L-1. AO SBR运行周期为6 h, 包括2 h缺氧段(含10 min进水), 3 h好氧段, 50 min静置沉淀和10 min出水.反应器进出水通过定时器控制蠕动泵启闭实现.
印染废水以蛋白胨、淀粉和乙酸钠为混合碳源, 其COD浓度为400 mg·L-1(6:3:1);其他进水组分包括114.6 mg·L-1NH4Cl, 30 mg·L-1偶氮染料RR2, 100 mg·L-1 CaCl2·2H2O, 100 mg·L-1 MgSO4·7H2O, 35.2 mg·L-1KH2PO4, 200 mg·L-1NaHCO3以及1 mL·L-1微量元素(1 g·L-1 FeCl2·4H2O, 100 mg·L-1 CoCl2·6H2O, 200 mg·L-1 NiCl2·6H2O, 100 mg·L-1 MnCl2·4H2O, 100 mg·L-1 NaMoO4·2H2O, 100 mg·L-1 H3BO3, 100 mg·L-1 NaWO4·2H2O和100 mg·L-1 NaSe2O3).水解酸化反应器每个运行周期进水3 L, 其出水作为AO反应器进水, AO反应器每个运行周期进水2.5 L.两个反应器的水力停留时间均为12 h.
反应器驯化稳定后, 测定两个反应器在运行周期内染料降解和氮浓度的变化.每隔一定时间, 从水解酸化反应器中取水样测定COD、色度和NH4+-N浓度变化, 从AO反应器中取水样测定NH4+-N、NO2--N和硝酸盐氮(NO3--N)浓度变化.
1.2 碳源与温度对RR2脱色的影响采用批处理试验, 考察了碳源种类、COD浓度和温度对厌氧水解酸化强化RR2色度去除的影响.试验使用250 mL的丝口瓶, 反应体积为200 mL, 取驯化稳定污泥100 mL与100 mL自配水进行混合.碳源种类试验中自配水分别以蛋白胨、淀粉和乙酸钠为碳源, COD浓度均为400 mg·L-1. COD浓度影响试验采用混合碳源, 试验中蛋白胨、淀粉和乙酸钠比例为6:3:1, COD浓度分别为200、400和800 mg·L-1.其他组分均与自配废水组分相同.以上碳源浓度和种类影响试验采用的温度分别为25℃和35℃.
试验开始前, 向反应瓶中通氮气4 min以保证反应处于厌氧状态.试验开始后, 采用摇床振荡, 其速度为170 r·min-1.反应时间为12 h, 分别在反应的0、1、2、3、4、6、8、10和12 h时取水样, 测定反应过程中的COD浓度和色度.每个试验条件设计两组平行试验, 取平均值作为试验结果.
1.3 染料及其中间产物对AO硝化过程的抑制对于硝化试验, 重点考察了染料RR2及其典型中间产物苯胺对AO硝化效率的影响.试验使用250 mL的锥形瓶, 反应体积为200 mL.取驯化稳定污泥100 mL与100 mL不含有碳源的自配水进行混合.染料抑制AO硝化试验中染料浓度分别为0、4、8、16、32和64 mg·L-1, 苯胺抑制AO硝化试验中苯胺浓度分别为0、0.75、1.5、3.0、4.5、6.0和7.5 mg·L-1, 其他组分均与水解酸化反应器出水组分相同.
试验采用磁力搅拌器(上海振荣磁力搅拌器90-1)搅拌, 保证泥水混合均匀.曝气采用可调式气泵(海利双孔9602), 曝气流量为4.0 L·min-1, 保证溶解氧(DO)大于4 mg·L-1.反应时间为120 min, 反应开始后每隔10 min取水样, 40 min后每隔20 min取水样.测定反应过程中DO、NH4+-N、NO2--N和NO3--N浓度的变化.
1.4 分析方法COD、NH4+-N、NO2--N、NO3--N、悬浮固体浓度(SS)和VSS依据国家规定的标准方法测定[14].通过全波长扫描确定染料RR2在512 nm处吸光度最高, 因此用此波长处的吸光度表征色度, 并用来计算色度去除率.苯胺测定方法依照GB 11889-89中N-(1-萘基)乙二胺偶氮分光光度法.分光光度计为哈希DR3900.
取水解酸化反应器和AO反应器中污泥, 采用PowerSoil DNA Isolation Kit (Laboratories Inc., CA, USA)提取DNA.提取后的DNA样品在V4区进行特异性扩增, 利用Illumina Miseq 2500测序平台, 采用双末端测序Paire-end方法进行高通量测序.利用fastqc软件进行质量评价后, 进一步利用perl脚本进行质量控制.利用Flash或Mothur将原始数据拼接成完整的目标片段, 并进行质量评价和控制.在相似性为97%条件下, 将过滤后的目标片段聚类成操作分类单元(OTU), 并选取代表性的序列与数据库比对, 进行OTU物种注释.根据OTU的绝对丰度及注释信息, 统计样品在各分类学水平上序列构成情况.
2 结果与讨论 2.1 水解酸化/AO工艺运行性能分析水解酸化/AO工艺稳定运行条件下水质关键参数见表 1.进水COD为389.5 mg·L-1, 出水COD为30.4 mg·L-1, 其总去除率为92.2%.进水染料浓度为30 mg·L-1, 吸光度为0.445左右, 出水吸光度为0.129, 色度总去除率为71.0%.进水NH4+-N浓度为31.74 mg·L-1, 水解酸化出水中NH4+-N浓度为37.42 mg·L-1; AO反应器出水NH4+-N、NO2--N和NO3--N浓度分别为5.23、11.03和4.39 mg·L-1, 出现了明显的NO2--N积累现象.
|
|
表 1 水解酸化-AO工艺稳定状态运行性能 Table 1 System performance of hydrolysis/acidification and AO process under steady conditions |
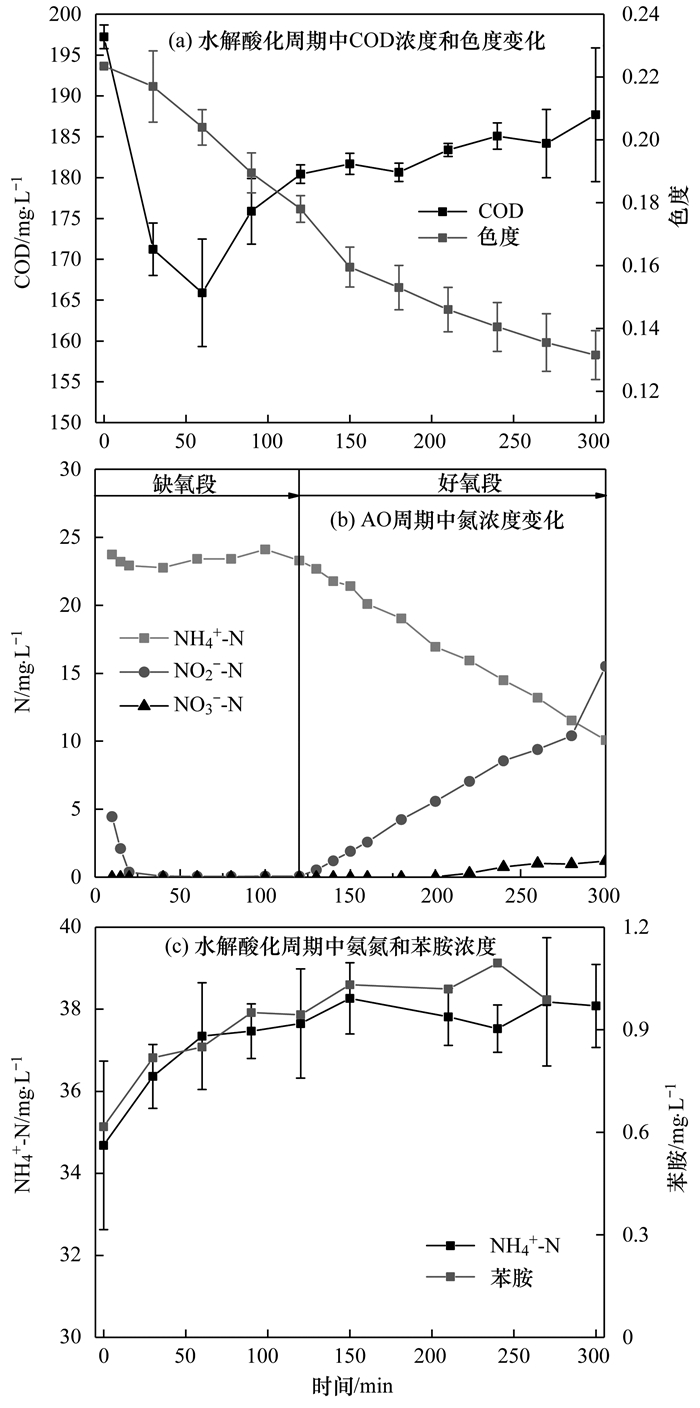
工艺稳定运行条件下, 水解酸化反应器出水COD为195.8 mg·L-1(表 1), COD去除率为49.7%.其他相关研究也得到厌氧段COD去除率为50%左右的结论[15].在水解酸化周期中COD浓度变化不明显[图 1(a)]; AO过程中存在硝化反硝化, 反硝化和好氧过程均能消耗碳源, 因此AO过程是COD去除的主要过程. Tomei等[16]也发现厌氧/好氧工艺处理实际印染废水时, 厌氧阶段COD去除率仅为11%~14%, 好氧阶段结束后COD去除率为59%~75%, 说明COD主要通过好氧过程去除. Baêta等[17]发现厌氧/好氧工艺处理印染废水时, 好氧过程可有效去除厌氧过程中难降解的有机物、厌氧过程中生成的挥发酸(VFAs)和染料代谢产物芳香胺等物质.因此, 好氧过程COD去除率明显高于厌氧过程.
|
图 1 水解酸化和AO周期中COD、色度、氮和苯胺浓度变化 Fig. 1 Variations of the concentrations of COD, color, nitrogen and aniline in the hydrolysis/acidification reactor and nitrogen in the AO reactor during typical reaction cycles |
从表 1得到水解酸化阶段色度去除率为72.3%, 说明水解酸化/AO工艺中水解酸化过程是染料RR2降解的主要过程.水解酸化过程中色度去除符合一级反应动力学[图 1(a)], 染料降解速率约为2.55 mg·(L·h)-1. Jonstrup等[18]也发现印染废水脱色主要发生于厌氧过程, 当温度为37℃, 水力停留时间为3 d, 偶氮染料浓度为100~2 000 mg·L-1时, 厌氧过程脱色率可以达到98%以上.本研究水解酸化过程脱色率为72.3%, 可能由于水解酸化温度为25℃, 水力停留时间为12 h, 由此导致脱色率低于Jonstrup等[18]的98%.
根据图 1(b)中NO2--N和NO3--N浓度变化, AO反应过程中在20 min内完成反硝化; 硝化过程中, NO3--N浓度较低, 出现明显的NO2--N积累, 亚硝化率为73.8%.由图 1(c)可知, 苯胺浓度随染料降解而增加, 因此染料及其代谢产物苯胺等可能对硝化菌活性有抑制作用, 导致NO2--N积累, 总氮的去除率仅为34.9%.根据Singh等[9]的研究, 水解酸化脱色率为72.3%, 可以推测理论上苯胺的生成量为3.28 mg·L-1.在虫漆酶等酶的作用下, 偶氮染料不能完全降解为苯胺, 因此测出的苯胺浓度约为1 mg·L-1.晋玉亮等[19]的研究发现在进水苯胺浓度超过3 mg·L-1时, 活性污泥的硝化能力丧失, 且苯胺对硝化的抑制是可恢复的; 当苯胺浓度低于0.5 mg·L-1时, NH4+-N去除率可以恢复至正常水平.
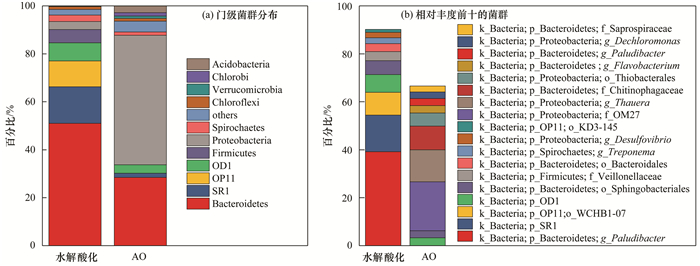
2.2 菌群分析水解酸化反应器中主要的门级菌群, 包括相对丰度为50.98%的拟杆菌门Bacteroidetes、15.27%的SR1、10.78%的OP11和5.57%的厚壁菌门Firmicutes[图 2(a)].厌氧染料降解工艺中, Bacteroidetes和Firmicutes多为优势菌种, 其中Bacteroidetes可降解复杂结构偶氮染料有机物[4, 20]. Bacteroidetes门中的Paludibacter是一种碳水化合物发酵菌[20]. Paludibacter是水解酸化反应器中相对丰度最高的菌种, 其丰度为39.18%[图 2(b)].这说明Paludibacter是水解酸化反应器中主要碳源代谢菌种. Firmicutes门中的Clostridia可利用乙酸、乙醇和丁酸等生成氢气[21], 在水解酸化反应器中相对丰度为3.75%. SR1的碱基序列与Prosthecobacter algae strain EBTL04和Prosthecobacter dejongeii strain FC1相似度为97%, OP11的碱基序列与Victivallis vadensis strain Cello相似度为87%, 但这两类菌群的作用尚不明晰.在水解酸化反应器中相对丰度前10的菌群中, 其他菌种没有研究证明具有偶氮染料降解能力, 只有Proteobacteria门中的Desulfovibrio由于含有偶氮还原酶, 可将偶氮染料降解成芳香胺类物质[4]. Desulfovibrio在水解酸化反应器中相对丰度为2.23%, 这说明Desulfovibrio是水解酸化反应器中主要的偶氮染料降解菌.
|
图 2 水解酸化和AO反应器中的菌群分布 Fig. 2 Microbial community distribution in the hydrolysis acidification and AO reactors |
AO反应器中主要的门级菌群为相对丰度为54.0%的Proteobacteria、28.4%的Bacteroidetes、以及在1%~3.5%之间的OD1、Acidobacteria、SR1、Spirochaetes、Chlorobi、Chloroflexi和Verrucomicrobia. AO反应器中主要的硝化菌为Nitrospira, 相对丰度为0.28%, 是一种常见的硝化菌[22].本研究反硝化菌中相对丰度最高的为Rhodocyclaceae中Thauera, 相对丰度为13.35%. Thauera不仅是常见的反硝化菌, 还具有降解芳香胺类物质的能力[23].在NO2--N积累条件下, Thauera可成为反硝化菌中优势菌种[24]; 这与AO反应器中出现NO2--N积累现象一致.由于AO进水为水解酸化出水, 因此含有RR2代谢产物芳香胺等物质, 可被Thauera降解.同属于Rhodocyclaceae的Dechloromonas相对丰度为2.73%, 也是一种高效的反硝化菌[25]. AO反应器中检测到的相对丰度较高的OD1, 其碱基序列与Lactobacillus paracollinoides strain DSM 15502和Lactobacillus collinoides strain JCM1123有95%的相似度, 因此可能具有乳酸菌Lactobacillus发酵的功能[26], 可将碳源降解用于反硝化.
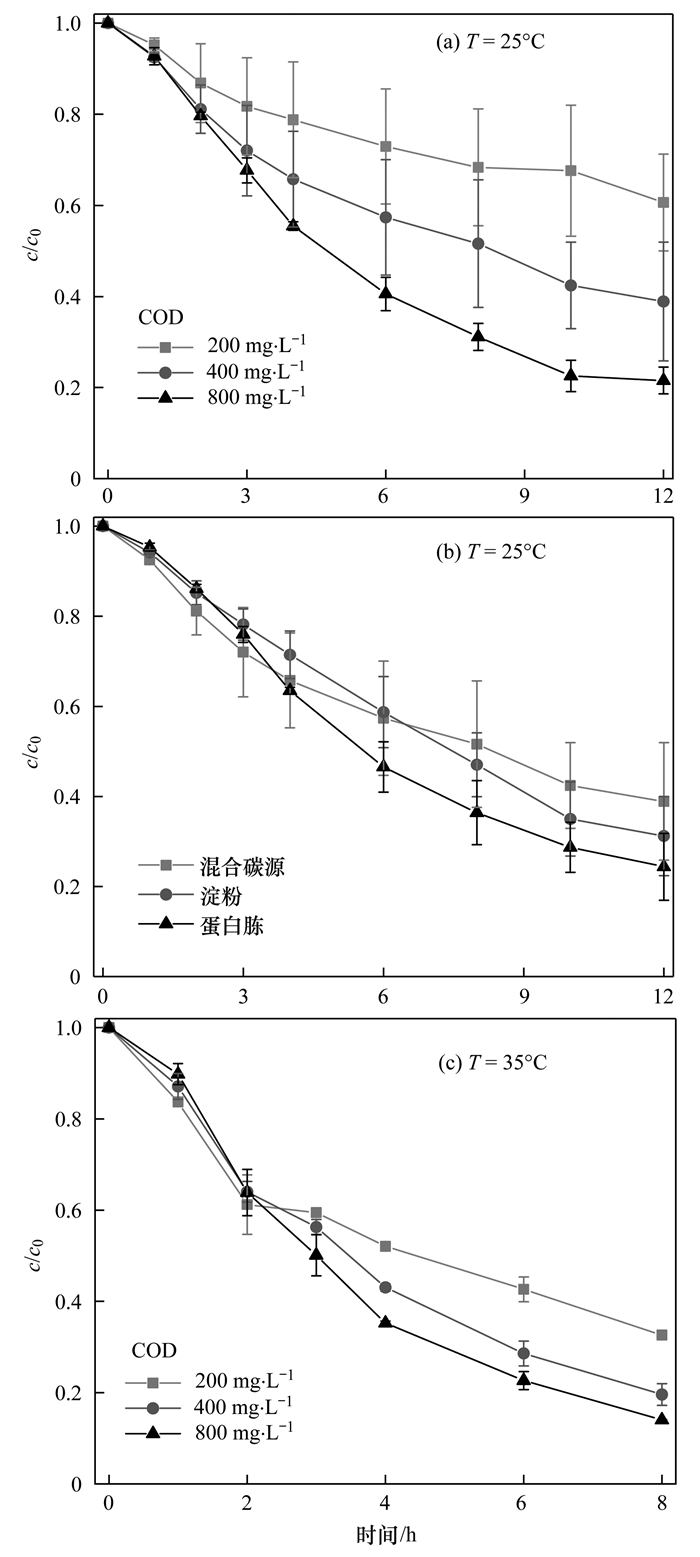
2.3 碳源及温度对RR2脱色的影响偶氮染料的降解是通过—N=N—的断裂实现的.偶氮染料作为电子受体时, 偶氮键—N=N—断裂需要4个电子, 第一步需要2个电子使偶氮键断裂为—N—N—, 第二步需要2个电子使偶氮键完全断裂[12].淀粉和蛋白胨等碳源作为电子供体, 在降解过程中可以提供偶氮染料降解所需电子.经过预试验发现乙酸钠作为碳源时, 染料RR2降解速率最慢, 仅为淀粉降解速率的1/3左右, 表明乙酸钠不是RR2脱色的主要碳源.以蛋白胨、淀粉和混合碳源为碳源, 在不同条件下染料RR2降解试验结果如图 3, 计算得到各条件下RR2的降解一级反应动力学参数如表 2.
|
图 3 不同COD浓度和碳源种类条件下RR2浓度变化 Fig. 3 Concentration of RR2 for different COD contents and carbon sources |
|
|
表 2 染料降解试验一级反应动力学拟合方程及降解速率 Table 2 First kinetic equation of azo dye degradation and its degradation rate |
在COD=400 mg·L-1、25℃条件下, 以蛋白胨为碳源时, RR2的降解速率最快, 为3.63 mg·(L·h)-1, 其次为淀粉2.91 mg·(L·h)-1, 而混合碳源条件下最慢, 仅为2.55 mg·(L·h)-1(图 3).因此, 以蛋白胨和淀粉为单一碳源时染料RR2降解速率高于混合碳源. Rasool等[27]研究了以乳酸、葡萄糖和乙醇为碳源时印染废水处理效果, 结果表明与以葡萄糖为碳源相比, 以乳酸和葡萄糖作为碳源时色度去除率提高. Cirik等[28]处理含有Cr的印染废水时也发现, 乙醇作为碳源时色度去除率比葡萄糖作为碳源时高.因此碳源种类影响偶氮染料降解的效果.
由表 2中RR2降解速率可知在本研究中, 25℃条件下, COD浓度由200 mg·L-1提高至400 mg·L-1和800 mg·L-1, RR2降解速率分别提高了88.9%和208.9%.在35℃条件下, COD浓度由200 mg·L-1提高至400 mg·L-1和800 mg·L-1, RR2降解速率分别提高了37.6%和64.4%. Rasool等[27]处理含有偶氮染料直接红80的废水, 发现分别将碳源乳酸盐、葡萄糖和乙醇COD浓度从3000 mg·L-1降至500 mg·L-1时, 色度去除率从98.23%降至78.46%、63.37%和69.10%, 指出高COD浓度能够提供充足电子, 有利于偶氮染料降解. Cui等[29]也发现降解染料酸性橙7, 当乙酸盐和葡萄糖为碳源时电子供体不足会导致脱色率降低.因此当COD浓度提高时, 电子供体数量充足, 偶氮染料降解速率明显高于电子供体不足条件.
当COD浓度分别为200、400和800 mg·L-1时, 温度由25℃提高到35℃, RR2降解速率分别提高了231.1%、141.2%和76.25%, 高温下染料RR2降解速率提高的比例远高于COD浓度增加时降解速率提高的比例, 说明COD浓度对RR2降解速率的影响没有温度的影响明显.因此, 维持一定温度是强化偶氮染料RR2降解的关键因素之一.
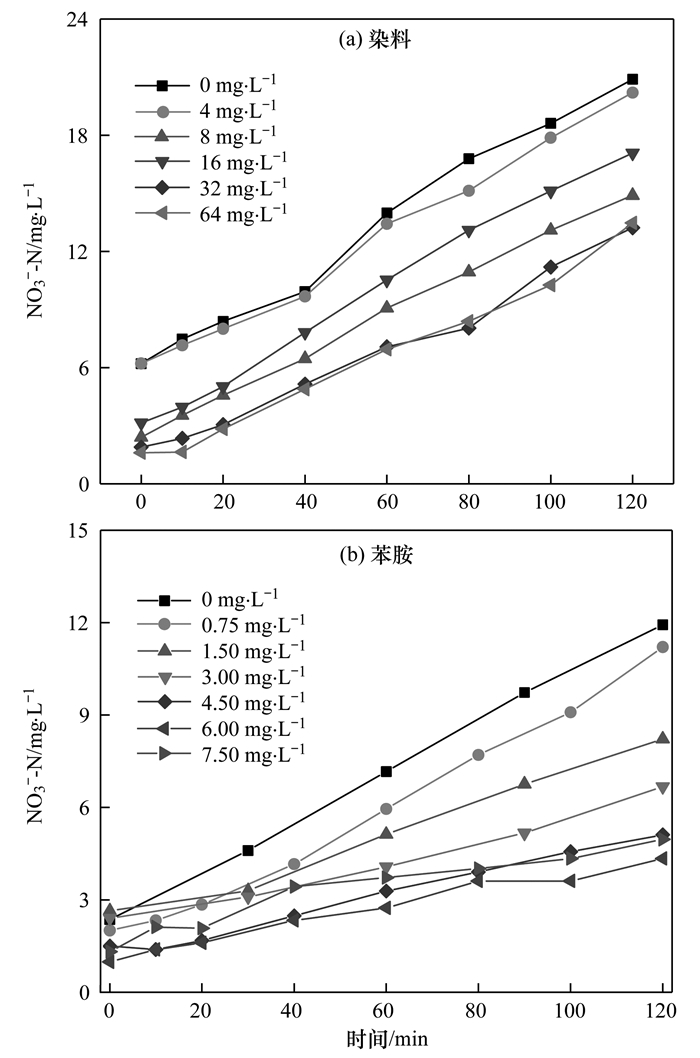
2.4 染料及其中间产物对硝化活性的影响图 4(a)显示染料RR2浓度在0~64 mg·L-1范围内, NO3--N生成速率基本一致, 且随染料浓度增加, NO3--N生成速率没有变化, 说明染料RR2对硝化过程没有影响, 在0~64 mg·L-1浓度范围内不影响硝化菌的活性. Sarvajith等[30]的研究发现相较于好氧条件, 微氧(DO<0.5 mg·L-1)条件下利用颗粒污泥处理含有活性黄15的废水时, 可以有效提高NH4+-N去除率, 且在8 h内出现NO2--N积累.本研究中染料RR2对氮去除率没有影响, 可能是由于试验过程中DO为7 mg·L-1, 因此RR2对硝化过程影响不大.
|
图 4 染料和苯胺抑制硝化试验 Fig. 4 Nitrification inhibition with azo dye RR2 and aniline |
由图 4(b)可知, 苯胺浓度从0 mg·L-1增加到7.5 mg·L-1时, NO3--N生成速率减小, 说明苯胺对硝化过程有抑制作用. Shabbir等[31]的研究表明当进水苯胺浓度高于250 mg·L-1时, 亚硝酸盐氮氧化菌(NOB)活性被完全抑制且不能恢复; 当初始苯胺浓度为100 mg·L-1时, 硝化菌活性会被抑制, 但将苯胺浓度降至4 mg·L-1以下时, 硝化菌活性可以恢复.
根据反应速率计算结果(表 3), 当苯胺浓度从0 mg·L-1增加到6 mg·L-1时, 氨氮氧化速率从1.38 mg·(L·h)-1减小到0.53 mg·(L·h)-1, NO3--N生成速率和氨氮氧化速率趋势一致, 且没有出现NO2--N积累, 说明本试验中苯胺主要对氨氮氧化菌(AOB)活性有抑制作用, 而对NOB活性没有抑制作用.当苯胺浓度增加到7.5 mg·L-1时, 氨氮氧化速率没有进一步降低, 说明苯胺浓度超过6 mg·L-1时, 硝化反应被抑制到最低反应速率, 为0.53 mg·(L·h)-1左右.该结果与长期运行过程中NOB活性被抑制不一致, 可能是由于试验接种污泥为AO反应器长期驯化污泥, NOB活性较低, 因此加入低浓度苯胺后, AOB首先出现了明显的抑制作用.试验中的反应时间仅为2 h, 反应时间较短, 因此没有出现NOB抑制作用. Khin等[11]的研究中也发现周期试验中, 反应8 h左右后抑制NOB活性, 且进水苯胺浓度超过250 mg·L-1后NOB活性不能恢复.
|
|
表 3 苯胺抑制硝化试验反应速率 Table 3 Reaction rate of the nitrification inhibition with aniline |
3 结论
(1) 水解酸化/AO工艺处理含有RR2的印染废水时, 可以有效去除色度、COD和NH4+-N, 去除率分别为71.0%、92.2%和83.5%.
(2) 水解酸化反应器中主要偶氮染料降解菌为Desulfovibrio, AO反应器中硝化菌主要为Nitrospira, 反硝化菌为Thauera和Dechloromonas.
(3) 水解酸化过程中COD浓度为400 mg·L-1时, 温度由25℃提高至35℃, 色度去除率提高141.2%;温度为25℃时, COD浓度从200 mg·L-1提高至800 mg·L-1, 色度去除率提高208.9%.
(4) AO反应器中苯胺浓度为1 mg·L-1时, 出现NO2--N积累, 亚硝化率为73.8%.染料RR2对硝化过程没有抑制作用, 苯胺对硝化过程有抑制作用.苯胺浓度超过6 mg·L-1时氨氮氧化速率达到最低值0.53 mg·(L·h)-1.
| [1] | Khorram A G, Fallah N. Treatment of textile dyeing factory wastewater by electrocoagulation with low sludge settling time:optimization of operating parameters by RSM[J]. Journal of Environmental Chemical Engineering, 2018, 6(1): 635-642. DOI:10.1016/j.jece.2017.12.054 |
| [2] | Zhao T Q, Li P, Tai C, et al. Efficient decolorization of typical azo dyes using low-frequency ultrasound in presence of carbonate and hydrogen peroxide[J]. Journal of Hazardous Materials, 2018, 346: 42-51. DOI:10.1016/j.jhazmat.2017.12.009 |
| [3] | Srinivasan S, Sadasivam S K. Exploring docking and aerobic-microaerophilic biodegradation of textile azo dye by bacterial systems[J]. Journal of Water Process Engineering, 2018, 22: 180-191. DOI:10.1016/j.jwpe.2018.02.004 |
| [4] | Xie X H, Liu N, Ping J, et al. Illumina MiSeq sequencing reveals microbial community in HA process for dyeing wastewater treatment fed with different co-substrates[J]. Chemosphere, 2018, 201: 578-585. DOI:10.1016/j.chemosphere.2018.03.025 |
| [5] | Li S Y, Cao Y, Bi C C, et al. Promoting electron transfer to enhance anaerobic treatment of azo dye wastewater with adding Fe(OH)3[J]. Bioresource Technology, 2018, 245: 138-144. |
| [6] | Reddy C N, Kumar A N, Mohan S V. Metabolic phasing of anoxic-PDBR for high rate treatment of azo dye wastewater[J]. Journal of Hazardous Materials, 2018, 343: 49-58. DOI:10.1016/j.jhazmat.2017.08.065 |
| [7] | Balapure K, Jain K, Bhatt N, et al. Exploring bioremediation strategies to enhance the mineralization of textile industrial wastewater through sequential anaerobic-microaerophilic process[J]. International Biodeterioration & Biodegradation, 2016, 106: 97-105. |
| [8] | Frindt B, Mattusch J, Reemtsma T, et al. Multidimensional monitoring of anaerobic/aerobic azo dye based wastewater treatments by hyphenated UPLC-ICP-MS/ESI-Q-TOF-MS techniques[J]. Environmental Science and Pollution Research, 2017, 24(12): 10929-10938. DOI:10.1007/s11356-016-7075-5 |
| [9] | Singh R L, Singh P K, Singh R P. Enzymatic decolorization and degradation of azo dyes-a review[J]. International Biodeterioration & Biodegradation, 2015, 104: 21-31. |
| [10] | Chen C Y, Wang G H, Tseng I H, et al. Analysis of bacterial diversity and efficiency of continuous removal of Victoria Blue R from wastewater by using packed-bed bioreactor[J]. Chemosphere, 2016, 145: 17-24. DOI:10.1016/j.chemosphere.2015.11.061 |
| [11] | Khin T, Gheewala S H, Annachhatre A P. Modeling of nitrification inhibition with aniline in suspended-growth processes[J]. Water Environment Research, 2002, 74(6): 531-540. DOI:10.2175/106143002X140332 |
| [12] | Thanh N P. An assessment of effect of inhibitor aniline on the biological nitrification process[J]. Asian Journal of Experimental Biological Sciences, 2010, 1(3): 503-512. |
| [13] | Joel A R, Grady C P L Jr. Inhibition of nitrification-effects of aniline after biodegradation[J]. Journal of Water Pollution Control Federation, 1977, 49(5): 778-788. |
| [14] | 国家环境保护总局. 水和废水监测分析方法[M]. 第四版. 北京: 中国环境科学出版社, 2002: 100-124. |
| [15] | Cui D Z, Zhang H, He R B, et al. The comparative study on the rapid decolorization of azo, anthraquinone and triphenylmethane dyes by anaerobic sludge[J]. International Journal of Environmental Research and Public Health, 2016, 13(11): E1053. DOI:10.3390/ijerph13111053 |
| [16] | Tomei M C, Angelucci D M, Daugulis A J. Sequential anaerobic-aerobic decolourization of a real textile wastewater in a two-phase partitioning bioreactor[J]. Science of the Total Environment, 2016, 573: 585-593. DOI:10.1016/j.scitotenv.2016.08.140 |
| [17] | Baêta B E L, Lima D R S, Silva S Q, et al. Evaluation of soluble microbial products and aromatic amines accumulation during a combined anaerobic/aerobic treatment of a model azo dye[J]. Chemical Engineering Journal, 2015, 259: 936-944. DOI:10.1016/j.cej.2014.08.050 |
| [18] | Jonstrup M, Kumar N, Murto M, et al. Sequential anaerobic-aerobic treatment of azo dyes:decolourisation and amine degradability[J]. Desalination, 2011, 280(1-3): 339-346. DOI:10.1016/j.desal.2011.07.022 |
| [19] |
晋玉亮, 王旭, 杨冲, 等. 苯胺对氨氮硝化反应抑制作用的实证研究[J]. 中国给水排水, 2016, 32(23): 85-87. Jin Y L, Wang X, Yang C, et al. Empirical study on aniline inhibition on nitrification of ammonia nitrogen[J]. China Water & Wastewater, 2016, 32(23): 85-87. |
| [20] |
岳秀, 唐嘉丽, 于广平, 等. 双氧水协同生化法强化处理印染废水[J]. 环境科学, 2017, 38(9): 3769-3780. Yue X, Tang J L, Yu G P, et al. Enhanced treatment of printing and dyeing wastewater using H2O2-biochemical method[J]. Environmental Science, 2017, 38(9): 3769-3780. |
| [21] | Chen H B, Chang S. Impact of temperatures on microbial community structures of sewage sludge biological hydrolysis[J]. Bioresource Technology, 2017, 245: 502-510. DOI:10.1016/j.biortech.2017.08.143 |
| [22] |
李卫华, 孙英杰, 刘子梁, 等. 序批式生物反应器填埋场脱氮微生物多样性分析[J]. 环境科学, 2016, 37(1): 342-349. Li W H, Sun Y J, Liu Z L, et al. Analysis on diversity of denitrifying microorganisms in sequential batch bioreactor landfill[J]. Environmental Science, 2016, 37(1): 342-349. |
| [23] | Zhou J, Li H S, Chen X L, et al. Cometabolic degradation of low-strength coking wastewater and the bacterial community revealed by high-throughput sequencing[J]. Bioresource Technology, 2017, 245: 379-385. DOI:10.1016/j.biortech.2017.08.119 |
| [24] | Cao S B, Du R, Li B K, et al. High-throughput profiling of microbial community structures in an anammox-UASB reactor treating high-strength wastewater[J]. Applied Microbiology and Biotechnology, 2016, 100(14): 6457-6467. DOI:10.1007/s00253-016-7427-6 |
| [25] | Zhang Y Q, Frankenberger W T Jr. Supplementing Bacillus sp. RS1 with Dechloromonas sp. HZ for enhancing selenate reduction in agricultural drainag e water[J]. Science of the Total Environment, 2007, 372(2-3): 397-405. DOI:10.1016/j.scitotenv.2006.10.027 |
| [26] | Quijada N M, De Filippis F, Sanz J J, et al. Different Lactobacillus populations dominate in "Chorizo de león" manufacturing performed in different production plants[J]. Food Microbiology, 2018, 70: 94-102. DOI:10.1016/j.fm.2017.09.009 |
| [27] | Rasool K, Mahmoud K A, Lee D S. Influence of co-substrate on textile wastewater treatment and microbial community changes in the anaerobic biological sulfate reduction process[J]. Journal of Hazardous Materials, 2015, 299: 453-461. DOI:10.1016/j.jhazmat.2015.07.044 |
| [28] | Cirik K, Dursun N, Sahinkaya E, et al. Effect of electron donor source on the treatment of Cr(Ⅵ)-containing textile wastewater using sulfate-reducing fluidized bed reactors (FBRs)[J]. Bioresource Technology, 2013, 133: 414-420. DOI:10.1016/j.biortech.2013.01.064 |
| [29] | Cui M H, Cui D, Gao L, et al. Azo dye decolorization in an up-flow bioelectrochemical reactor with domestic wastewater as a cost-effective yet highly efficient electron donor source[J]. Water Research, 2016, 105: 520-526. DOI:10.1016/j.watres.2016.09.027 |
| [30] | Sarvajith M, Reddy G K K, Nancharaiah Y V. Textile dye biodecolourization and ammonium removal over nitrite in aerobic granular sludge sequencing batch reactors[J]. Journal of Hazardous Materials, 2018, 342: 536-543. DOI:10.1016/j.jhazmat.2017.08.064 |
| [31] | Gheewala S H, Annachhatre A P. Biodegradation of aniline[J]. Water Science and Technology, 1997, 36(10): 53-63. DOI:10.2166/wst.1997.0358 |
 2018, Vol. 39
2018, Vol. 39


