2. 中南大学教育部生物冶金重点实验室, 长沙 410083
2. Key Laboratory of Biometallurgy of Ministry of Education, Central South University, Changsha 410083, China
由于采矿、冶炼、污水灌溉等人类活动, 我国土壤重金属污染问题愈发严重[1~3].据统计, 超过10%的耕地已遭受不同程度的重金属污染[4].土壤重金属浓度的增加是土壤退化的重要原因, 高浓度的重金属污染对土壤植被和微生物非常有害[5, 6].土壤微生物参与土壤生物化学过程、有机物分解和维持土壤结构, 对陆地生态系统的能量流动和物质循环至关重要[7~9].土壤为土壤微生物提供生存环境的同时也增加了土壤微生物对有害物质的敏感度[10].在许多研究中, 土壤微生物群落特征已作为一个重要指标反映土壤生态功能和土壤肥力[11~14].国内外学者对重金属污染土壤, 尤其是对工业区周边土壤微生物的研究日益增多.
Stefanowicz等[15]对英国和波兰4个废弃铅锌、铜和镍冶炼厂附近的39个土壤样品中细菌和真菌多样性分析发现, 细菌和真菌的多样性指数均随着重金属浓度升高而减小, 细菌变化幅度更大; 张雪晴等[16]比较安徽铜陵铜矿土壤样品中细菌和真菌的多样性和丰度发现, 重金属污染程度增加, 真菌丰度变化更为显著; Gołębiewski等[17]对波兰南部工业区周边土壤微生物群落分析发现, 在高浓度Zn环境下, 属和种水平上细菌的多样性和丰度均显著减小; He等[18]对重金属Pb、Cd和Zn污染稻田的土壤微生物群落研究, 发现Zn不仅与细菌多样性、丰度负相关, 且显著影响细菌的群落结构; Berg等[19]采用基于16S rRNA高通量测序技术和[3H]标记亮氨酸掺入法研究长期遭受Cu污染的土壤细菌, 发现Cu显著影响细菌群落结构与对Cu的耐受能力, 但与细菌的丰度无显著关系; Marcin等认为[13], 重金属污染对微生物群落的影响会受到其他环境因素的影响而不同.
清水塘工业区位于湖南省株洲市, 是我国著名的老工业基地, 由于产业结构和技术工艺等原因, 园区周边土壤重金属污染严重[20].目前关于该区域土壤微生物群落结构的研究较少[10, 21].本文选择株洲清水塘工业区以西的4块水稻田12个土壤样点作为研究对象, 基于16S rRNA高通量测序技术分析了其微生物群落结构差异, 探讨了微生物多样性和群落结构与重金属的关系, 以期为清水塘工业区周边土壤重金属污染与生态风险的评估提供较为全面的微生物信息参考, 进一步提高对工业区周边土壤重金属污染对微生物影响的认知, 并为该地区或其他相似地区的土壤重金属修复提供理论依据.
1 材料与方法 1.1 样品采集与处理2016年10月土壤样品采集于株洲市清水塘工业区以西5 km范围内的4块稻田, 分别编号为A、B、C、D, 其与工业区(株冶为中心)的距离分别为5、3、1、0.5 km, 如图 1所示.

|
图 1 研究区域及采样地示意 Fig. 1 Research areas and sampling sites |
每块稻田设置3个采样点(距离大于50 m).样品采集方法采用5点法[22], 即挖取5个深度5~10 cm、约100 g、直径为1.5 cm的土块, 混匀作为一个样品, 用2 mm尼龙筛筛去石块, 恒温箱低温保存, 运输至实验室.
采集的土壤样品一部分风干, 用于土壤理化性质分析; 一部分保存于-20℃, 用于后续生物学分析.
1.2 土壤理化参数测定土壤pH(按照水土质量比为2.5:1)采用pH计测定; 土壤总有机碳(TOC)采用重铬酸钾氧化法测定[23], 总氮(TN)采用凯式法测定[23]; Cu、Zn、Pb、Cd、Cr和As含量采用ICP-OES方法测定.
1.3 DNA提取、PCR扩增及MiSeq测序取0.25 g样品, 用土壤DNA提取试剂盒(mobio powersoil dna isolation kit)提取DNA, 测定浓度后置于-80℃冰箱保存.
对16S rRNA基因的V4高变区片段进行PCR扩增, 引物序列为515F(5′-GTGYCAGCMG CCGCGGTA-3′), 806R(5′-GGACTACHVGGGTWTC TAAT-3′). PCR扩增条件为:95℃, 5 min; 95℃, 30 s; 55℃, 30 s; 72℃, 45 s), 30个循环; 72℃, 10 min.每个样本3个重复, 将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测, 使用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)切胶回收PCR产物, Tris_HCl洗脱; 2%琼脂糖电泳检测.根据电泳初步定量结果, 将PCR产物用QuantiFluorTM-ST蓝色荧光定量系统(Promega公司)进行检测定量, 按照每个样本的测序量要求, 进行相应比例的混合.
DNA样品送至上海美吉生物医药科技有限公司Illumina MiSeq PE300平台上机测序.
1.4 原始数据处理MiSeq PE300测序平台获得双端序列数据, 每个样本数据量约为30000条序列.为了保证后续生物信息分析的准确性, 在Trimmomatic和FLASH软件平台上对测序数据进行过滤和拼接:过滤Read尾部质量值20以下的碱基; 根据PE reads之间的overlap关系, 将成对reads拼接(merge)成一条序列, 最小overlap长度为10bp; 拼接序列的overlap区允许的最大错配比率为0.2, 筛选不符合序列; 根据序列首尾两端的barcode和引物区分样品, 并调整序列方向, barcode允许的错配数为0, 最大引物错配数为2.
1.5 数据分析基于97%相似度水平, 在Usearch(vsesion 7.0)平台上进行OTU聚类, 所获得的结果与Silva 16S rRNA数据库相似性比对, 采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析, 统计并分析各分类学水平上各样品的群落组成; 根据OTU分析结果分类学信息, 利用R语言工具, 作图并分析样品稀释性曲线(rarefaction curve), 并计算丰度指数(ACE和Chao)、多样性指数(Shannon)和覆盖率; 利用韦恩图和距离热图比较样本间OTU相似性; 通过Spearman相关性热图研究重金属与微生物群落结构的关系.
2 结果与分析 2.1 土壤理化特性参数及各区域样品重金属污染状况土壤理化特性见表 1.如表所示, 4块稻田土壤重金属含量差异较大, 主要重金属的分布如下:Cd(4.17~16 mg·kg-1), Cu(38.5~136 mg·kg-1), Pb(105~465 mg·kg-1), Zn(330~1 620 mg·kg-1), Cr(84.2~156 mg·kg-1), As(29.3~64.3 mg·kg-1); 其中样地D土壤中的Cd、Cu、Pb、Zn浓度均最高.
|
|
表 1 土壤理化参数和综合污染指数 Table 1 Physical and chemical properties of the soil and pollution appraisal indexes |
重金属污染程度, 可用内梅罗指数法来评估.内梅罗指数法是当前国内外进行综合污染指数计算的最常用的方法之一[24, 25].
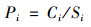
|
(1) |
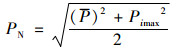
|
(2) |
式中, PN为综合污染指数; Pi为重金属i的污染指数; Ci为重金属含量实测值; Si为土壤环境质量标准值(国家二级标准值); P为各污染物种污染指数的平均值; Pimax为各污染物中污染指数最大值.
如表 1所示, 4个样地的PN均大于3, 根据土壤综合污染程度分级标准(表 2), 均为重污染, 污染程度高低为:样地D(38.62)>样地C(32.77)>样地B(11.02)>样地A(10.09).
|
|
表 2 土壤重金属污染分级标准[25] Table 2 Criteria for classification of soil heavy metal pollution |
2.2 测序结果
12个土壤样品一共检测到471 008条16S rRNA有效序列, 平均序列数为39 250条序列, 平均长度为273bp, 以97%相似性聚类分析, 共得到3 273个OTUs, 各样本的覆盖率范围为0.981 0~0.983 3, 说明土样中基因被检测概率较高.
稀释性曲线(如图 2)走向趋于平缓, 说明测序数量趋于饱和, 测序所获得的数据充足.
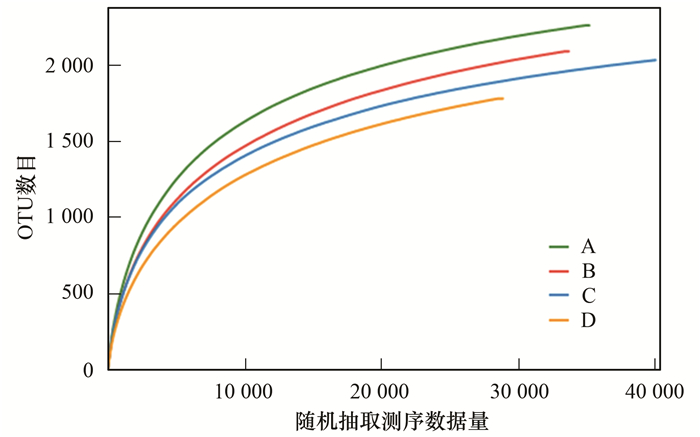
|
图 2 稀释性曲线 Fig. 2 Rarefaction curves |
微生物α多样性指数如表 3所示. Sobs为实测OTU数目; Chao、ACE指数为群落丰度指数, Shannon指数为群落多样性指数.样本微生物群落丰度和多样性指数趋向规律与重金属污染程度趋向一致:样地A>样地B>样地C>样地D, 重金属污染程度越高, 土壤微生物群落丰度和多样性均越低.
|
|
表 3 群落丰度和多样性指数1) Table 3 Species richness and diversity estimators |
2.4 微生物群落结构
4组土壤样品共检测出3 273个OTUs, 分属于细菌和古菌, 共48个门, 119个纲, 230个目, 379个科, 562个属和1 061个种.
以门作为分类学水平分析4组土壤样品的微生物群落结构, 如图 3所示. 4组样品中平均相对丰度最高的门是变形菌门(Proteobacteria), 为49.56%, 其相对丰度的分布为样地D(54.56%)>样地A(50.8%)>样地C(48.01%)>样地B(44.78%), 随着污染程度的增加先减后增; 平均相对丰度仅次于变形菌门(Proteobacteria)的是绿弯菌门(Chloroflexi), 平均相对丰度为13.07%, 其相对丰度的分布为样地C(15.28%)>样地B(14.69%)>样地D(13.17%)>样地A(9.13%), 随着污染程度增加无明显变化趋势, 但在样地A中最低; 酸杆菌门(Acidobacteria)的平均相对丰度为8.77%, 其相对丰度的分布为样地A(8.75%)>样地B(7.9%)>样地C(7.1%)>样地D(6.56%), 随着污染程度增加递减; 疣微菌门(Verrucomicrobia)的平均相对丰度为4.82%, 其相对丰度的分布为样地B(6.09%)>样地A(4.59%)>样地C(4.51%)>样地D(4.10%), 随着污染程度增加无明显变化趋势, 在样地B中最高; 硝化螺旋菌门(Nitrospirae)的平均相对丰度为4.04%, 其相对丰度的分布为样地A(6.26%)>样地B(5.42%)>样地C(2.52%)>样地D(1.97%), 随着污染程度的增加递减.

|
Total为门分类水平上所有样本的微生物相对丰度的平均值 图 3 土壤微生物门分类水平组成 Fig. 3 Microbial community structure in samples at phylum level |
在纲分类学水平上, 变形菌门的组成在4组土壤样品中差异最为明显. 图 4为4组土壤样品变形菌门群落组成. β-变形菌纲(β-Proteobacteria)的平均相对丰度最高, 为20.35%, 其相对丰度的分布为样地D(22.97%)>样地B(21.21%)>样地A(19.65%)>样地C(17.56%), 随着污染程度的增加呈递减趋势, 在样地C中最低; δ-变形菌纲(δ-Proteobacteria)的平均相对丰度为17.33%, 其相对丰度的分布为样地C(19.58%)>样地D(19.48%)>样地A(19.34%)>样地B(10.92%), 随着污染程度增加无明显变化趋势; γ-变形菌纲(γ-Proteobacteria)的平均相对丰度为7.53%, 其相对丰度的分布为样地B(9.24%)>样地D(8.99%)>样地A(6.37%)>样地C(5.54%), 随着污染程度增加呈递减趋势, 在样地C中最低; α-变形菌纲(α-Proteobacteria)的平均相对丰度最低, 为4.27%, 其相对丰度的分布为样地A(5.36%)>样地C(5.27%)>样地B(3.39%)>样地D(3.07%), 随着污染程度增加呈递减趋势.
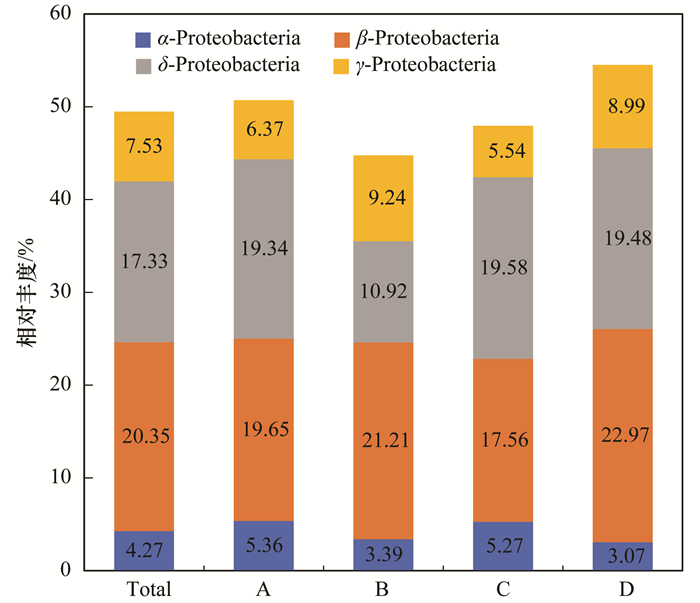
|
Total为纲分类水平上所有样本的微生物相对丰度的平均值 图 4 变形菌门群落及结构 Fig. 4 Communities and distribution of Proteobacteria |
以属作为分类学水平分析4组土壤样品的微生物群落结构, 如图 5所示.将平均丰度低于1%的部分合并为others, 剩余36个属水平分类微生物包括已分类的13个、未科学命名(norank)19个和未分类(unclassified)3个. Anaerolineaceae_norank属于厌氧绳菌科(Anaerolineaceae), 平均相对丰度为8.8%, 占绿弯菌门67.3%, 是4组土壤样品相对丰度均最高的属, 其相对丰度的分布为样地D(10.33%)>样地C(9.22%)>样地B(8.96%)>样地A(6.70%), 随着污染程度增加而递减; 氧化亚铁硫杆菌属(Thiobacillus)的平均相对丰度为5.3%, 占变形菌门10.69%, 其相对丰度的分布为样地B(8.04%)>样地D(6.88%)>样地C(4.05%)>样地A(2.23%), 随着污染程度增加呈递减趋势, 在样地B中最高; 硝化螺旋菌属(Nitrospira)的平均相对丰度为4.04%, 是硝化螺旋菌门唯一的属, 其相对丰度的分布为样地A(6.26%)>样地B(5.42%)>样地C(2.52%)>样地D(1.97%), 随着污染程度增加递减; Sulfurifustis的平均相对丰度为3.69%, 占变形菌门的7.44%, 其相对丰度的分布为样地D(6.39%)>样地C(3.32%)>样地B(3.05%)>样地A(2.00%), 随着重金属污染程度增加而递增.
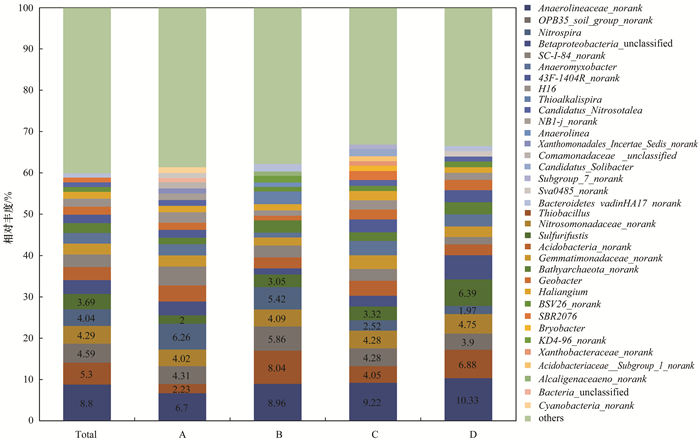
|
Total为属分类水平上所有样本的微生物相对丰度的平均值 图 5 土壤微生物属水平组成 Fig. 5 Microbial community structure in samples at genus level |
以上从门、纲、属这3个水平对4个样地群落组成的分析表明重金属污染是影响微生物群落结构的重要因素.
2.5 样本间的相似性分析Morisita-Horn距离热图反映丰度较高物种的相似性, Bray-Curtis距离热图反映丰度较低物种的相似性, 样本间距离热图颜色的深浅反映了其相似性的高低, 颜色越深, 相似性越低[17].以97%的聚类水平, Morisita-Horn距离热图的颜色明显比Bray-Curtis距离热图的更浅, 说明Morisita-Horn相似性比Bray-Curtis相似性更高, 即样本间丰度较高的OTUs子群落比丰度较低的OTUs子群落更为相似(图 6).
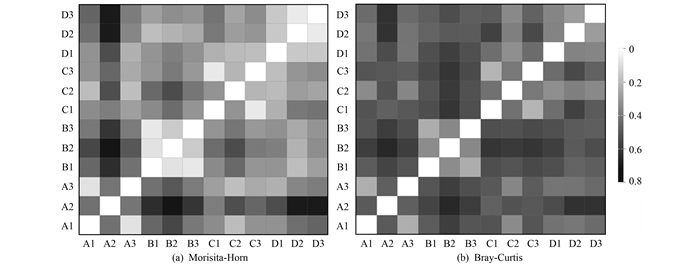
|
图 6 Morisita-Horn和Bray-Curtis的距离热图 Fig. 6 Community distance heatmaps based on Morisita-Horn and Bray-Curtis dissimilarities |
OTU分布韦恩图如图 7所示. 4组样本共有OTU数目为1723, 占总OTU个数的52.64%, 说明不同重金属污染程度下稻田土壤微生物群落结构相似性很高.样地A、样地B、样地C、样地D特有的OTU数目分别为101、85、46、53, 其特有的OTU数目随重金属污染程度增加而减少.
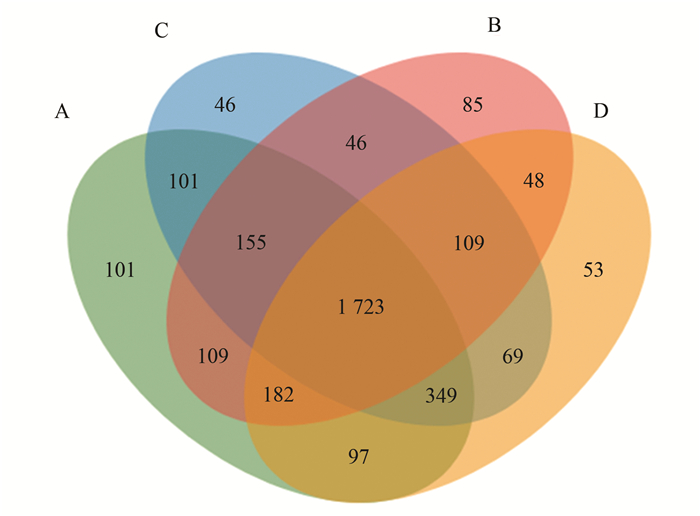
|
图 7 OTU分布韦恩图 Fig. 7 Venn diagrams of shared OTUs |
不同重金属对微生物多样性指数的影响用非线性回归分析来表征[17, 18].如图 8所示, 重金属Cd、Zn与多样性指数Sobs、Shannon、Chao均有显著负相关关系(P≤0.05), 即随着重金属Zn和Cd含量的升高, 微生物群落实测OTUs(Sobs)、物种多样性指数(Shannon)和物种丰度指数(Chao)显著降低, 而其它重金属与微生物群落多样性指数间无显著相关性.
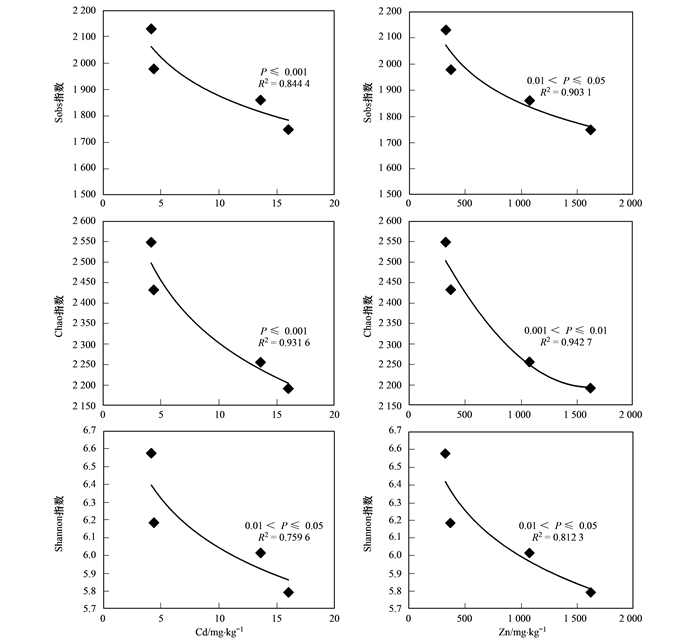
|
图 8 微生物多样性指数与重金属 Cd、Zn含量的关系 Fig. 8 Correlations between microbial diversity indices (Sobs, Chao, and Shannon) and Cd and Zn concentrations |
微生物与重金属含量的Spearman相关性, 可反映重金属对微生物群落结构的影响[27].本研究选择相对丰度高于0.05%的404个OTU, 分析其与重金属含量的Spearman相关性关系, 发现共有159个OTU具有显著相关, 其中正相关73个, 负相关89个.
不同丰度区间的OTU和重金属显著相关不同(如表 4).微生物群落相对丰度在0.05%~0.1%之间, 与重金属显著正/负相关的OTU数目最多, 为85个; 其次是在0.1%~0.5%之间, 为66个; 高于0.5%最少, 为8个.这反映了相对丰度在0.05%~0.1%之间的微生物受重金属影响最大, 高于0.5%受重金属影响较小.与重金属Cd、Cr、Cu、Pb、Zn、As显著相关的OTU数目分别为69、61、61、52、59、14, 说明重金属Cd对相对丰度高于0.05%的微生物群落影响最大.
|
|
表 4 不同丰度区间的OTU和重金属的显著相关性个数 Table 4 Significant relation numbers of heavy metals with different richness threshold values at OTU level |
如表 5所示, 159个与重金属显著相关的OTU序列属于13个门.相对丰度最高的变形菌门与重金属显著相关的OTU数目最多, 为85个.其中δ-变形菌纲与重金属Cd、Cu、Pb、Zn主要为显著正相关, β-变形菌纲与6种重金属均为显著正相关.硝化螺旋菌门与6种重金属均为显著负相关; 绿弯菌门与6种重金属主要为显著正相关; 其他门类微生物与重金属也具有显著的相关性.
|
|
表 5 重金属对微生物门类Spearman相关性统计1) Table 5 Spearman's correlation of heavy metals at phylum level |
将159个与重金属显著相关的OTU序列比对到属水平, 如表 6所示.发现60个OTU序列能比对到属水平, 分属于25个属.鞘脂单胞菌属、H16、硫碱螺旋菌属、硝化螺菌属、芽单胞菌属和多种重金属有显著的负相关, 说明这几类微生物对重金属的耐受能力较弱; 厌氧黏细菌属、地杆菌属、Haliangium、螺旋体属和多种重金属有显著的正相关, 说明这几类微生物对重金属的耐受能力较强.
|
|
表 6 基于OTU分类学水平Spearman相关性分析1) Table 6 Spearman's correlation at OTU level |
3 讨论 3.1 稻田土壤微生物群落的结构组成
4个样地土壤样品的优势菌门均为变形菌门、绿弯菌门、酸杆菌门、疣微菌门、硝化螺旋菌门, 表明这些优势菌群能在重金属污染环境下生存, 是清水塘工业区周边稻田土壤的优势微生物群落. Chen等[2]利用DGGE技术研究发现, 中国江苏省某冶炼厂周边长期遭受重金属Pb、Cd、Zn和Cu污染的稻田土壤中主要为变形菌门、酸杆菌门、绿弯菌门; He等[18]利用Illumina高通量测序技术发现中国广东省几个矿区周边长期遭受重金属Cd、Pb和Zn污染的稻田土壤中主要为变形菌、放线菌、厚壁菌门、酸杆菌门.本研究中变形菌门的相对丰度最高, 得到与上述研究相近的微生物优势菌群, 说明在不同地理环境下受重金属污染的稻田土壤都有相似的微生物组成.
从韦恩图(如图 7)结果看出, 尽管4个样地土壤的重金属污染程度不同, OTU的重叠率仍很高, 这说明在相近地理环境下, 重金属污染程度不同的稻田土壤微生物组成也趋于相近.
3.2 重金属对稻田土壤微生物群落丰度和多样性的影响有研究发现, 重金属污染是影响微生物群落丰度和多样性的重要因素, 高重金属污染环境下, 土壤微生物群落丰度和多样性均显著减小[28~32]. 4个样地土壤样品中, 重金属污染程度越高, 其微生物群落丰度和多样性越低, 并与重金属Cd、Zn显著负相关(如图 8), 这与Chen等[3]和He等[18]的研究结果相近. Berg等[19]认为微生物群落丰度和多样性与重金属Cu并无显著关系, 本研究也得出与其类似的结果. Marcin等认为[13]高浓度Zn对微生物群落的均匀度也有显著负面影响, 但本研究中各组样品的均匀度无差异(文中并无显示), 这可能是由于本研究的土壤类型和重金属浓度不同, 其研究的土壤类型是沙质土且重金属Zn、Cr含量更高.
3.3 重金属对稻田土壤微生物群落结构的影响不同重金属污染对不同微生物群落的影响不同[33].通过样本距离热图(如图 6)发现, 重金属污染程度不同的样品中, 相对丰度较高的微生物群落比相对丰度较低的微生物群落之间更为相似, 说明重金属污染对优势微生物群落的影响更小.比较不同丰度区间OTU和重金属相关性(如表 4)也得到类似的结果:相对丰度高于0.5%的微生物和重金属主要为显著正相关, 相对丰度在0.1%~0.5%之间的微生物和重金属显著正/负相关的OTU数目相近, 相对丰度在0.05%~0.1%之间的微生物和重金属主要为显著负相关.说明相对丰度更高的微生物群落对重金属的耐受能力更强.
在重金属污染更高的环境下, 变形菌门类微生物的相对丰度更高[17, 34, 35].但在4个样地土壤中, 变形菌门相对丰度的高低为:样地D>样地A>样地C>样地B, 随着重金属污染程度增加先减后增.可能是由于样地A土壤样品的重金属Cr浓度明显低于其他3组.重金属Cr与微生物群落结构、物种多样性和物种丰度有显著的负相关关系[36].从门水平Spearman相关性分析发现(如表 5), 变形菌门和重金属Cr显著负相关的OTU数最多, Cr是影响变形菌门丰度的重要因素.绿弯菌门是4组样品中平均相对丰度第二高的微生物群落, 在重金属污染程度更高的样地B、C、D中相对丰度更高, 与重金属Cr主要为显著正相关(如表 5).酸杆菌门相对丰度的分布为:样地A>样地B>样地C>样地D, 随着重金属污染程度增加递减, 与Chen等[3]所研究的相似, 从门水平Spearman相关性分析发现(如表 5), 酸杆菌门与Cd、Cu、Pb、Zn、As显著负相关, 说明重金属对酸杆菌门主要为负面影响; 硝化螺旋菌门相对丰度的分布为:样地A>样地B>样地C>样地D, 随着重金属污染程度增加递减, 从门水平Spearman相关性分析发现(如表 5), 硝化螺旋菌门有14个OTU与Cd、Cr、Cu、Pb、Zn显著负相关, 说明重金属污染不利于硝化螺旋菌门生存. Li等[37]认为古菌对重金属污染有一定的抵抗能力, 本研究也发现广古菌门和深古菌门均与重金属有正相关关系.
δ-变形菌纲与Cd、Cr、Cu、Pb、Zn主要为显著正相关, 其中厌氧黏细菌属、地杆菌属、Haliangium均和多种重金属显著正相关.有研究发现, 厌氧黏细菌属、地杆菌属参与变价重金属的氧化还原和有机卤代物的生物代谢过程, 可作为环境修复的理想菌群[38~41]; Hemme等[42]发现γ-变形菌纲的微生物有多种重金属抗性机制, 重金属耐性能力较强.本研究中发现γ-变形菌纲的Sulfurifustis和Cd、Cr、Cu、Pb、Zn均有显著正相关, 说明Sulfurifustis对重金属耐受能力较强.硝化螺菌属为硝化螺旋菌门唯一属, 与多种重金属均为显著负相关, 说明硝化螺菌属对重金属污染敏感度较高, 在高重金属污染的环境下相对丰度显著降低.
4 结论(1) 清水塘工业区周边4块稻田土壤微生物群落主要有:变形菌门、绿弯菌门、酸杆菌门、疣微菌门、硝化螺旋菌门, 其平均相对丰度分别为49.56%、13.07%、8.77%、4.82%、4.04%.
(2) 不同重金属污染程度下样本间相似性较高, 相对丰度较高的微生物群落比相对丰度较低的微生物群落更趋向于相似.
(3) 重金属Cd、Zn的含量和稻田土壤微生物群落丰度和多样性指数有显著负相关关系.
(4) 相对丰度在0.05%~0.1%之间的微生物群落和重金属显著相关的OTU数目最多, 相对丰度高于0.05%的微生物群落和重金属Cd显著相关的OTU数目最多.
(5) 相对丰度最高的变形菌门和重金属显著相关的OTU数目最多, 其中δ-变形菌纲的微生物群落和Cd、Cr、Cu、Pb、Zn主要为显著正相关; 绿弯菌门和重金属Cr为显著正相关; 酸杆菌门、硝化螺旋菌门和多种重金属均为显著负相关.
| [1] | Wei B G, Yang L S. A review of heavy metal contaminations in urban soils, urban road dusts and agricultural soils from China[J]. Microchemical Journal, 2010, 94(2): 99-107. DOI:10.1016/j.microc.2009.09.014 |
| [2] | Chen H Y, Teng Y G, Lu S J, et al. Contamination features and health risk of soil heavy metals in China[J]. Science of the Total Environment, 2015, 512-513: 143-153. DOI:10.1016/j.scitotenv.2015.01.025 |
| [3] | Chen J H, He F, Zhang X H, et al. Heavy metal pollution decreases microbial abundance, diversity and activity within particle-size fractions of a paddy soil[J]. FEMS Microbiology Ecology, 2014, 87(1): 164-181. DOI:10.1111/1574-6941.12212 |
| [4] | Zhang X Y, Zhong T Y, Liu L, et al. Impact of soil heavy metal pollution on food safety in China[J]. PLoS One, 2015, 10(8): e0135182. DOI:10.1371/journal.pone.0135182 |
| [5] | Xu X Y, Schierz A, Xu N, et al. Comparison of the characteristics and mechanisms of Hg(Ⅱ) sorption by biochars and activated carbon[J]. Journal of Colloid and Interface Science, 2015, 463: 55-60. |
| [6] | Wang N, Xue X M, Juhasz A L, et al. Biochar increases arsenic release from an anaerobic paddy soil due to enhanced microbial reduction of iron and arsenic[J]. Environmental Pollution, 2017, 220: 514-522. DOI:10.1016/j.envpol.2016.09.095 |
| [7] | Giller K E, Witter E, Mcgrath S P. Toxicity of heavy metals to microorganisms and microbial processes in agricultural soils:a review[J]. Soil Biology and Biochemistry, 1998, 30(10-11): 1389-1414. |
| [8] | Kowalchuk G A, Stephen J R. Ammonia-oxidizing bacteria:a model for molecular microbial ecology[J]. Annual Review of Microbiology, 2001, 55: 485-529. DOI:10.1146/annurev.micro.55.1.485 |
| [9] | Lens P N, Kuenen J G. The biological sulfur cycle:novel opportunities for environmental biotechnology[J]. Water Science and Technology, 2001, 44(8): 57-66. DOI:10.2166/wst.2001.0464 |
| [10] | Deng L J, Zeng G M, Fan C Z, et al. Response of rhizosphere microbial community structure and diversity to heavy metal co-pollution in arable soil[J]. Applied Microbiology and Biotechnology, 2015, 99(19): 8259-8269. DOI:10.1007/s00253-015-6662-6 |
| [11] | Wang Y P, Shi J Y, Wang H, et al. The influence of soil heavy metals pollution on soil microbial biomass, enzyme activity, and community composition near a copper smelter[J]. Ecotoxicology and Environmental Safety, 2007, 67(1): 75-81. DOI:10.1016/j.ecoenv.2006.03.007 |
| [12] | Antunes S C, Pereira R, Marques S M, et al. Impaired microbial activity caused by metal pollution:A field study in a deactivated uranium mining area[J]. Science of the Total Environment, 2011, 410-411: 87-95. DOI:10.1016/j.scitotenv.2011.09.003 |
| [13] | Marcin C, Marcin G, Justyna M P, et al. Diversity of microorganisms from forest soils differently polluted with heavy metals[J]. Applied Soil Ecology, 2013, 64: 7-14. DOI:10.1016/j.apsoil.2012.11.004 |
| [14] | Elzobair K A, Stromberger M E, Ippolito J A, et al. Contrasting effects of biochar versus manure on soil microbial communities and enzyme activities in an Aridisol[J]. Chemosphere, 2015, 142: 145-152. |
| [15] | Stefanowicz A M, Niklińska M, Laskowski R. Metals affect soil bacterial and fungal functional diversity differently[J]. Environmental Toxicology and Chemistry, 2008, 27(3): 591-598. DOI:10.1897/07-288.1 |
| [16] |
张雪晴, 张琴, 程园园, 等. 铜矿重金属污染对土壤微生物群落多样性和酶活力的影响[J]. 生态环境学报, 2016, 25(3): 517-522. Zhang X Q, Zhang Q, Cheng Y Y, et al. The impact of heavy metal contamination on soil microbial diversity and enzyme activities in a copper mine[J]. Ecology and Environmental Sciences, 2016, 25(3): 517-522. |
| [17] | Gołębiewski M, Deja-sikora E, Cichosz M, et al. 16S rDNA pyrosequencing analysis of bacterial community in heavy metals polluted soils[J]. Microbial Ecology, 2014, 67(3): 635-647. DOI:10.1007/s00248-013-0344-7 |
| [18] | He H D, Li W C, YU R Q, et al. Illumina-based analysis of bulk and rhizosphere soil bacterial communities in paddy fields under mixed heavy metal contamination[J]. Pedosphere, 2017, 27(3): 569-578. DOI:10.1016/S1002-0160(17)60352-7 |
| [19] | Berg J, Brandt K K, Al-Soud W A, et al. Selection for Cu-tolerant bacterial communities with altered composition, but unaltered richness, via long-term Cu exposure[J]. Applied and Environmental Microbiology, 2012, 78(20): 7438-7446. DOI:10.1128/AEM.01071-12 |
| [20] | Jiang M, Zeng G M, Zhang C, et al. Assessment of heavy metal contamination in the surrounding soils and surface sediments in Xiawangang River, Qingshuitang District[J]. PLoS One, 2013, 8(8): e71176. DOI:10.1371/journal.pone.0071176 |
| [21] |
王振中, 胡觉莲, 张友梅, 等. 湖南省清水塘工业区重金属污染对土壤动物群落生态影响的研究[J]. 地理科学, 1994, 14(1): 64-72. Wang Z Z, Hu J L, Zhang Y M, et al. Influence of heavy metal pollution on soil animal community ecology in Qingshuitang area in Hunan[J]. Scientia Geographica Sinica, 1994, 14(1): 64-72. |
| [22] | Yang Y F, Gao Y, Wang S P, et al. The microbial gene diversity along an elevation gradient of the Tibetan grassland[J]. The ISME Journal Multidisciplinary Journal of Microbial Ecology, 2014, 8(2): 430-440. |
| [23] | 鲁如坤. 土壤农业化学分析方法[M]. 北京: 中国农业科技出版社, 2000. |
| [24] |
范拴喜, 甘卓亭, 李美娟, 等. 土壤重金属污染评价方法进展[J]. 中国农学通报, 2010, 26(17): 310-315. Fan S X, Gan Z T, Li M J, et al. Progress of assessment methods of heavy metal pollution in soil[J]. Chinese Agricultural Science Bulletin, 2010, 26(17): 310-315. |
| [25] |
郭伟, 赵仁鑫, 张君, 等. 内蒙古包头铁矿区土壤重金属污染特征及其评价[J]. 环境科学, 2011, 32(10): 3099-3105. Guo W, Zhao R X, Zhang J, et al. Distribution characteristic and assessment of soil heavy metal pollution in the iron mining of Baotou in Inner Mongolia[J]. Environmental Science, 2011, 32(10): 3099-3105. |
| [26] |
GB 15618-1995, 土壤环境质量标准[S]. GB 15618-1995, Environmental quality STANDARD for soils[S]. |
| [27] | Guo H, Nasir M, Lv J, et al. Understanding the variation of microbial community in heavy metals contaminated soil using high throughput sequencing[J]. Ecotoxicology and Environmental Safety, 2017, 144: 300-306. DOI:10.1016/j.ecoenv.2017.06.048 |
| [28] | Xie Y, Fan J B, Zhu W X, et al. Effect of heavy metals pollution on soil microbial diversity and bermudagrass genetic variation[J]. Frontiers in Plant Science, 2016, 7: 755. |
| [29] | Yang Y R, Dong M, Cao Y P, et al. Comparisons of soil properties, enzyme activities and microbial communities in heavy metal contaminated bulk and rhizosphere soils of Robinia pseudoacacia L. in the northern foot of Qinling Mountain[J]. Forests, 2017, 8(11): 430. DOI:10.3390/f8110430 |
| [30] | Oliveira A, Pampulha M E. Effects of long-term heavy metal contamination on soil microbial characteristics[J]. Journal of Bioscience and Bioengineering, 2006, 102(3): 157-161. DOI:10.1263/jbb.102.157 |
| [31] | Khan S, Hesham A E L, Qiao M, et al. Effects of Cd and Pb on soil microbial community structure and activities[J]. Environmental Science and Pollution Research, 2010, 17(2): 288-296. DOI:10.1007/s11356-009-0134-4 |
| [32] | Ding Z L, Wu J P, You A Q, et al. Effects of heavy metals on soil microbial community structure and diversity in the rice (Oryza sativa L. subsp. japonica, food crops institute of Jiangsu academy of agricultural sciences) rhizosphere[J]. Soil Science and Plant Nutrition, 2017, 63(1): 75-83. DOI:10.1080/00380768.2016.1247385 |
| [33] | Prasad M N V. Metals in the environment, analysis by biodiversity[J]. Journal of Organometallic Chemistry, 2001, 669(1): 207. |
| [34] | Bourceret A, Cébron A, Tisserant E, et al. The bacterial and fungal diversity of an aged PAH-and heavy metal-contaminated soil is affected by plant cover and edaphic parameters[J]. Microbial Ecology, 2016, 71(3): 711-724. DOI:10.1007/s00248-015-0682-8 |
| [35] | Liu Y R, Wang J J, Zheng Y M, et al. Patterns of bacterial diversity along a long-term mercury-contaminated gradient in the paddy soils[J]. Microbial Ecology, 2014, 68(3): 575-583. DOI:10.1007/s00248-014-0430-5 |
| [36] | Sheik C S, Mitchell T W, Rizvi F Z, et al. Exposure of soil microbial communities to chromium and arsenic alters their diversity and structure[J]. PLoS One, 2012, 7(6): e40059. DOI:10.1371/journal.pone.0040059 |
| [37] | Li X Q, Meng D L, Li J, et al. Response of soil microbial communities and microbial interactions to long-term heavy metal contamination[J]. Environmental Pollution, 2017, 231: 908-917. DOI:10.1016/j.envpol.2017.08.057 |
| [38] | Shelobolina E S, Vrionis H A, Findlay2 R H, et al. Geobacter uraniireducens sp. nov., isolated from subsurface sediment undergoing uranium bioremediation[J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58: 1075-1078. DOI:10.1099/ijs.0.65377-0 |
| [39] | Prakash O, Gihring T M, Dalton D D, et al. Geobacter daltonii sp. nov., an Fe(Ⅲ)-and uranium(Ⅵ)-reducing bacterium isolated from a shallow subsurface exposed to mixed heavy metal and hydrocarbon contamination[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60: 546-553. DOI:10.1099/ijs.0.010843-0 |
| [40] | Hwang C, Copeland A, Lucas S, et al. Complete genome sequence of Anaeromyxobacter sp. Fw109-5, an anaerobic, metal-reducing bacterium isolated from a contaminated subsurface environment[J]. Genome Announcements, 2015, 3(1): e01449-14. |
| [41] | Sun M, Xiao T F, Ning Z P, et al. Microbial community analysis in rice paddy soils irrigated by acid mine drainage contaminated water[J]. Applied Microbiology and Biotechnology, 2015, 99(6): 2911-2922. DOI:10.1007/s00253-014-6194-5 |
| [42] | Hemme C L, Deng Y, Gentry T J, et al. Metagenomic insights into evolution of a heavy metal-contaminated groundwater microbial community[J]. The ISME Journal Multidisciplinary Journal of Microbial Ecology, 2010, 4(5): 660-672. |
 2018, Vol. 39
2018, Vol. 39


