2. 寒旱地区水资源综合利用教育部工程研究中心, 兰州 730070;
3. 甘肃农业大学资源与环境学院, 兰州 730070
2. Engineering Research Center for Cold and Arid Regions Water Resource Comprehensive Utilization, Ministry of Education, Lanzhou 730070, China;
3. College of Natural Resources and Environment, Gansu Agricultural University, Lanzhou 730070, China
污染物进入水体后, 由于水体的自净作用, 污染物浓度会发生衰减.自净过程中污染物浓度衰减主要是由于发生了物理反应(如沉降、稀释等), 化学反应(如氧化还原反应、水解反应等)及生物反应(如微生物的新陈代谢、浮游植物的吸收等).其中生化反应能使污染物实现真正降解[1], 有助于减少水体中的污染物总量, 生物降解过程是生化反应的主要方面[2].在水生态系统中, 作为污染物生物降解的主要承担者, 微生物群落通过新陈代谢的方式, 降解水体中的各种污染物.
然而, 微生物生态系统是一个比单一物种复杂得多的系统, 大部分的微生物只参加一种或两种反应, 表现出一种或两种生理功能, 少数微生物则参与多种反应, 表现出多种生理功能[3].除此之外, 在微生物生态系统中, 虽然优势菌是影响群落功能的主要因素, 但是近年来诸多研究表明低丰度菌也对群落功能有着巨大贡献, 起到关键作用的并不仅仅是丰度较高的优势菌, 还包括大量的低丰度菌[4, 5].因此, 如何从众多的微生物菌群中筛选出参与生物降解的关键类群及了解其生态互作关系, 是理解污染物微生物降解机理的关键问题之一.目前, 将16S rRNA的测序技术获得的大量微生物组学数据与种间相互作用推断模型相结合, 通过构建微生物之间的共现性网络, 已经成为研究和预测土壤、海洋及活性污泥等环境中微生物群落的关键物种[6, 7]、演替[8~10]、生态互作关系[3, 11, 12]等的主流方法.
经研究, 在窖水的自净过程中, 表征污染物的各项水质指标均出现下降趋势, 影响窖水水质变化的主要因素有泥沙物理沉降和微生物的生物降解作用, 其中微生物的生物降解发挥了重要的作用.本文拟通过构建窖水微生物类群的共现性关联网络, 筛选参与生物降解的关键类群并预测其生态互作关系, 以期为揭示窖水水质变化机制提供依据.
1 材料与方法 1.1 水样采集及处理窖水样品取自我国干旱半干旱典型集雨地区同一个自然村的农家庭院, 针对不同集流面和水窖类型组合现场取样, 水窖结构及采样点布设情况如图 1所示.其中, 1、2、3号为黄土地集流面+红黏土水窖模式(A), 4、5、6号为黄土地集流面+混凝土水窖模式(B), 7、8、9号为混凝土集流面+混凝土水窖模式(C), 10、11、12号为混凝土集流面+红黏土水窖模式(D).水样置于冰盒中带回实验室, 取400 mL的水样经0.22 μm聚碳酸酯膜负压过滤, 收集滤膜, 用事先灭菌且经过75%的酒精消毒的医用剪刀剪碎后, 置于2 mL的无菌离心管中, 于-80℃的超低温冰箱中保存至DNA提取.

|
图 1 水窖结构及采样点布设示意 Fig. 1 Structure of cellar water and sample layout |
窖水水样的细菌基因组DNA采用Water DNA Isolation Kit试剂盒提取.待DNA完全溶解后, 使用超微量紫外可见分光光度计(美国Quawell Q5000)检测其浓度, 并根据D260/D280的值检测其纯度, 在凝胶成像系统上拍照观察分析DNA样品的完整性.
以12个DNA原液作为PCR模板, 利用细菌16S rDNA通用引物进行V4区扩增, 反应结束后配制2%琼脂糖凝胶电泳检测PCR产物.经检测, PCR扩增图谱条带清晰明显, 且条带位置一致, 背景干净, 符合后续分析要求.委托上海派森诺生物科技有限公司, 使用Illumina MiSeq对V4区进行高通量测序.
1.3 生物信息学分析使用Qiime对原始数据进行聚类分析, 主要步骤包括双末端序列装配、去除低质量序列、去重复, 运用Mothur软件去嵌合体, 得到优质序列.对优质序列按序列相似度0.97进行聚类, 选取每个类中最长的序列为代表序列, 使用Greengene注释数据库对获得的OTUs代表序列进行分类学鉴定, 获得每个OTU代表序列的分类学信息.之后对OTU进行精简处理, 去掉丰度值小于总序列条数0.001%的OTU, 得到精简后的OTU列表.利用Mothur软件中的summary. single命令, 计算菌群Chao、ACE、Shannon、Simpson多样性指数.计算各OTUs在各样品中的相对丰度并分别在纲、属水平上进行汇总, 使用R语言, 对丰度前50位的属进行聚类分析并绘制热图.基于16S rRNA基因序列, 利用预测宏基因组功能基因的生物信息学软件包(PICRUSt), 进行窖水细菌群落基因功能预测, 并统计分析与新陈代谢相关的功能基因.基于OTUs相对丰度, 使用Mothur软件, 计算相对丰度前50位的优势菌属之间的Spearman等级相关系数, 对rho>0.6且P值<0.01的相关优势菌属构建关联网络, 并导入Cytoscape软件进行可视化.
2 结果与分析 2.1 窖水水质特性分析及评价水窖各水样水质指标有所不同(表 1).集雨窖水作为生活饮用水水源地, 属地表水, 应根据《地表水环境质量标准》(GB 3838-2002)中的Ⅲ类水质标准进行评价.窖水水质类别主要以Ⅱ类(高锰酸盐指数、NH4+-N)和Ⅲ类(TP、高锰酸盐指数)为主, 其中超标水质指标为TN, 总体达到Ⅳ~Ⅴ类水质标准, 超标率为75%.
|
|
表 1 窖水物理化学指标 Table 1 Physico-chemical parameters of cellar water |
水质评价过程中往往涉及众多水质指标, 且各指标之间存在着紧密的联系.在涉及水质多指标综合评价的过程中, 可能会因为指标间存在共线性问题而无法客观地对水质污染状况进行评价, 具有一定的局限性.为了进一步辨识反映窖水水质污染状况的关键因子, 解析污染成因, 对窖水水质指标数据进行主成分分析(表 2).
|
|
表 2 窖水水质主成分荷载矩阵 Table 2 Water quality of principal component loading matrix of cellar water |
通过降维(特征值>1.0)提取了3个主成分, 其累积贡献率达到了93.559%, 体现了水质指标的绝大多数信息(表 2).第一主成分PC1的贡献率为51.413%, 反应的信息量最大, NO2--N(0.923)、NO3--N(0.744)、TN(0.896)、TP(0.909)正相关程度较高, 浊度(-0.716)、高锰酸盐指数(-0.928)、UV254(-0.713)负相关程度较高, 主要反映窖水水质受氮、磷污染的状况; 第二主成分PC2的贡献率为26.846%, 反映的信息量较大, 浊度(0.604)、BOD5(0.748)、NH4+-N(0.797)正相关程度较大, 反映泥沙、可生物降解有机物、氨氮等对窖水水质的影响; 第三主成分PC3的贡献率为15.300%, 反映的信息量较小, BOD5(0.636)正相关程度较高, 温度(-0.619)负相关程度较高, 反映可生物降解有机物对窖水水质的影响.
2.2 窖水微生物多样性及群落组成 2.2.1 微生物多样性经过质控和筛选, 12个样品共获得201240条序列, 优质率为92.99%;在97%的相似水平上共获得1605条OTUs.各样品OTU稀释性曲线见图 2, 趋于平坦, 但仍未达到饱和.但香农指数稀释曲线(图 3)平坦, 代表覆盖率水平的Goods coverage指数均大于96%, 说明测序获取了绝大多数样本信息, 能够反映窖水的微生物群落的多样性.从OTU稀释曲线可以直观地看出样品间微生物α多样性的差异.

|
图 2 OTU稀释曲线 Fig. 2 Rarefaction curve of OTUs |

|
图 3 香农指数稀释曲线 Fig. 3 Rarefaction curve of Shannon |
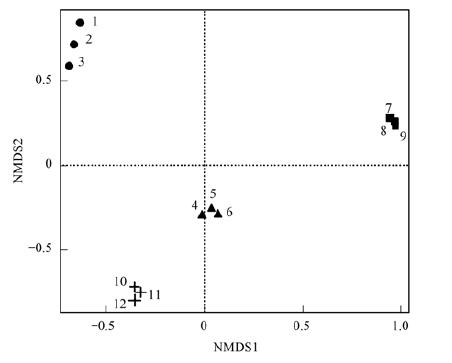
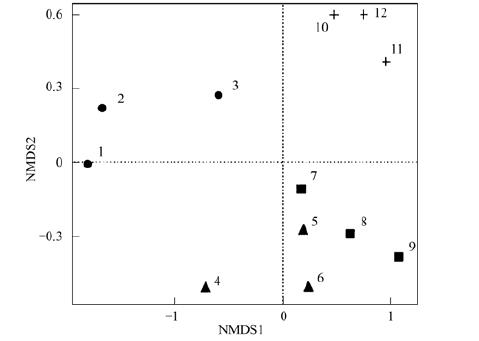
根据样品间微生物的进化信息及丰度信息进行Unifrac分析, 得到样品差异的距离矩阵, 并在距离矩阵的基础上采用无度量多维尺度指数(NMDS)进行β多样性分析.基于Unifrac的非加权NMDS(图 4)分析显示, 同类型的集流面和水窖组合模式的不同深度窖水样品的点与点间的距离较小, 能聚为一类, 表明不同深度处窖水的微生物类群结构在进化关系上相似度高; 而不同类型的集流面和水窖组合模式的各窖水样品的点与点距离较大, 表明不同集流面与水窖组合模式间窖水微生物群落结构在进化关系上差异较大.基于Unifrac的加权NMDS(图 5)分析, 由于同时计算了微生物的进化信息及丰度信息, 各样品点虽然仍能实现组内聚类, 但各样品点的距离明显较只计算进化关系要大, 尤其是组间的5、6、7、8号样品点出现交叠现象, 表明窖水各样品微生物菌落相对丰度信息在组内及组间存在较为明显的差异.
|
图 4 样品群落结构Unifrac非加权无度量多维尺度分析图 Fig. 4 Unweighted Unifrac non-metric multidimensional scaling of sample |
|
图 5 样品群落结构Unifrac加权无度量多维尺度分析图 Fig. 5 Weighted Unifrac non-metric multidimensional scaling of sample |
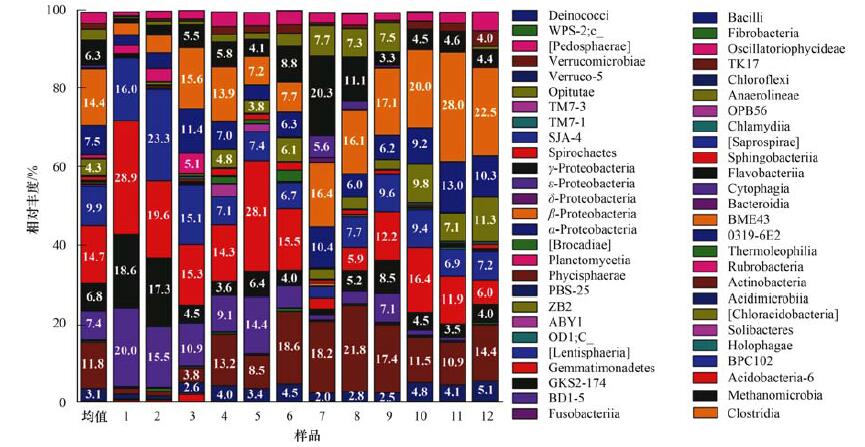
窖水样品中微生物在纲水平上的群落结构及分布特征如下:酸微菌纲(Acidimicrobiia)、放线菌纲(Actinobacteria)、噬纤维菌纲(Cytophagia)、黄杆菌纲(Flavobacteriia)、鞘脂杆菌纲(Sphingobacteriia)、[Saprospirae]、ZB2、α-变形菌纲(α-Proteobacteria)、β-变形菌纲(β-Proteobacteria)、δ-变形菌纲(δ-Proteobacteria)、ε-变形菌纲(ε-Proteobacteria)、γ-变形菌纲(γ-Proteobacteria)、丰收神菌纲(Opitutae)、疣微菌纲(Verrucomicrobiae)、[Pedosphaerae]、芽单胞菌纲(Gemmatimonadetes)、浮霉状菌纲(Planctomycetia)是各样品中相对丰度较大的优势菌(相对丰度≥1.00%), 构成了纲层次上的微生物基本群落结构, 其差异主要体现在优势菌群地位及其相对丰度的变化(图 6);而数量众多的相对丰度较小(相对丰度<1.00%)的35种非优势菌在各样品中的组成及相对丰度各不相同, 个别非优势菌出现在特定的窖水水样中.
|
图 6 纲水平的细菌群落结构及分布 Fig. 6 Bacterial communities and their distributions at class level |
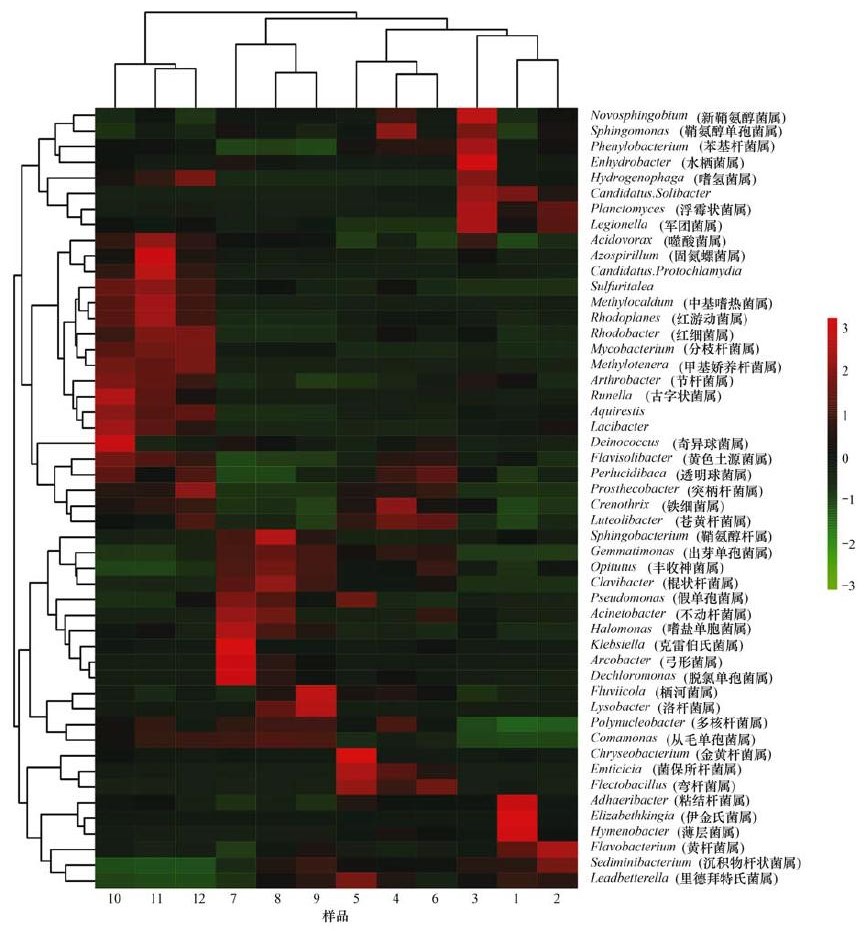
属水平上, 将相对丰度前50位的微生物类群进行相似性聚类分析, 并绘制热图(图 7), 通过颜色梯度及相似程度来反映窖水各样品在属分类水平上群落组成的相似性和差异性.从热图纵向表示的各样品的聚类情况显示, 特定集流面和水窖组合模式收集和贮存的窖水, 其样品虽然取样深度不同, 但能聚为一类, 而不同的集流面和水窖组合模式收集和贮存的窖水样品无法聚类, 表明窖水属分类水平上群落组成的组内相似性及组间差异性.这种相似性与差异性主要表现在各样品优势菌属的组成及丰度: A模式的水样(1、2、3)主要以Flavobacterium(12.2%、17.2%、4.50%)、Sediminibacterium(8.10%、12.6%、7.90%)、Hydrogenophaga(0.80%、1.10%、5.80%)、Planctomyces(1.80%、3.20%、5.00%)、Acidovorax(1.00%、2.10%、6.00%)、Elizabethkingia(6.10%、0.00%、0.00%)等13种菌属为主; B模式的水样(4、5、6)则主要以Emticicia(5.00%、8.00%、2.90%)、Prosthecobacter(1.00%、0.90%、1.20%)、Crenothrix(4.50%、2.50%、2.30%)、Chryseobacterium(0.00%、2.40%、0.00%)等9种菌属为主; C模式的水样(7、8、9)主要以Opitutus(2.30%、2.80%、2.20%)、Acinetobacter(9.40%、7.50%、0.10%)、Klebsiella(8.10%、0.50%、0.30%)、Arcobacter(5.40%、1.90%、0.60%)、Polynucleobacter(5.70%、6.20%、6.30%)、Comamonas (1.80%、2.00%、1.90%)等13种菌属为主; D模式的水样(10、11、12)主要以Acidovorax(5.70%、8.60%、5.60%)、Rhodobacter(3.40%、5.00%、4.90%)、Mycobacterium(2.40%、2.80%、3.00%)、Runella(0.70%、0.40%、0.20%)、Aquirestis(7.90%、5.90%、6.30%)、Lacibacter(0.60%、0.40%、0.30%)、Prosthecobacter(1.00%、1.00%、2.00%)、Crenothrix(2.40%、2.90%、3.00%)等17种菌属为主.
|
图 7 属水平上的热图和聚类分析 Fig. 7 Heatmap and clustering tree analysis of dominant genera |
环境样品中微生物功能基因具有高度的多样性, 通过分析KO level 2层次上与新陈代谢相关的基因功能分类(图 8), 表明窖水中微生物新陈代谢相关的功能主要表现为氨基酸代谢(20.95%)、碳水化合物代谢(20.20%)、能量代谢(11.04%)及脂类代谢(8.02%).而聚类热图(图 8)颜色梯度的变化, 则反映了窖水各样品中微生物新陈代谢功能基因数量在不同功能分类水平上的相似性和差异性.
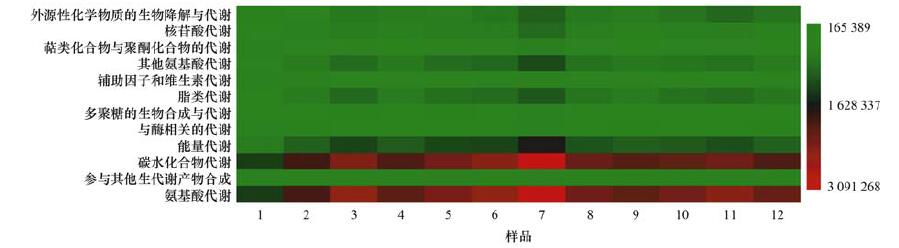
|
图 8 微生物新陈代谢功能基因聚类热图 Fig. 8 Heat map and clustering analysis of metabolism functional genes |
微生物对环境的作用主要是通过微生物群落代谢功能特性的差异来实现, 了解窖水中细菌群落的与新陈代谢相关的功能特性与水环境因子的相关性, 对更好地理解细菌群落对水质因子的响应具有重要作用.根据窖水中表征细菌群落的、与新陈代谢相关的基因功能类别与水质因子的RDA分析(图 9)表明, 反映窖水氮、磷水平的指标TN、NO2--N、NO3--N、TP与基因功能类别M-1~M-12呈正相关关系; NH4+-N、BOD5与基因功能类别M-1~M-9正相关, 与M-10~M-12负相关; UV254与基因功能类别M-1~M-12呈负相关关系.
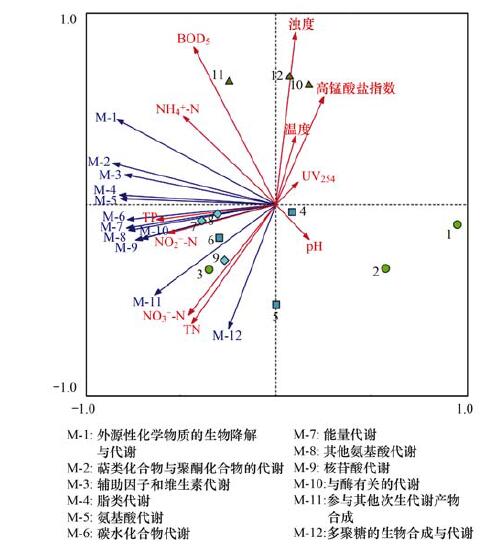
|
图 9 微生物与新陈代谢相关的功能基因和水质因子的RDA分析 Fig. 9 RDA analysis of water parameters and microbial metabolism functional genes |
表征细菌群落的、与新陈代谢相关的基因功能类别, 是窖水中具备相应特定生态功能的菌群分布特征在基因层面上的阐释, 表明在窖水中存在大量的、具有相对特定生态功能的细菌, 进行着诸多活跃的与新陈代谢相关活动.根据相对丰度前50位的细菌菌群和水质因子的RDA分析, 可以把主要菌群分为3组(Ⅰ、Ⅱ、Ⅲ), 见图 10.第Ⅰ组中的Comamonas、Polynucleobacter、Dechloromonas、Clavibacter、Gemmatimonas、Acinetobacter、Sphingobacterium、Deinococcus等21种细菌属与反映窖水氮、磷水平的指标TN、NO2--N、NO3--N、TP、NH4+-N及pH值呈显著性正相关; 第Ⅱ组中的Novosphingobium、Methylocaldum、Rhodobacter、Methylotenera、Arthrobacter、Azospirillum、Rhodoplanes等20种细菌属与反映窖水有机物污染的指标高锰酸盐指数、BOD5、UV254及浊度呈显著性正相关; 而第Ⅲ组中的Sphingomonas、Leadbetterella、Sediminibacterium、Hymenobacter、Elizabethkingia、Candidatus. Solibacter、Legionella、Planctomyces、Flavobacterium共9种细菌与TN、NO2--N、NO3--N、TP、NH4+-N、pH、高锰酸盐指数、BOD5、UV254、浊度呈负相关关系.
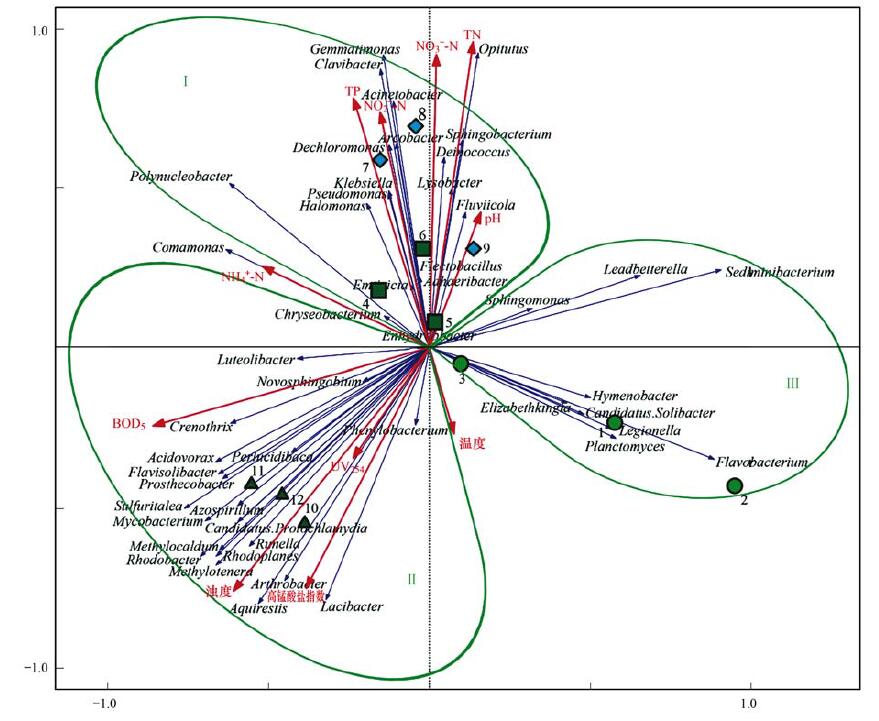
|
图 10 微生物群落与水质因子的RDA分析 Fig. 10 RDA analysis of water parameters and microbial community |
为进一步从具有相对特定生态功能的细菌中, 筛选出参与窖水中污染物微生物降解的关键类群, 了解其生态互作关系, 本研究借助窖水中相对丰度前50位的优势菌属的丰度数据, 基于菌属间的Spearman等级相关系数, 通过共现性网络分析, 构建了7个共现性网络(Ⅰ~Ⅶ), 见图 11.
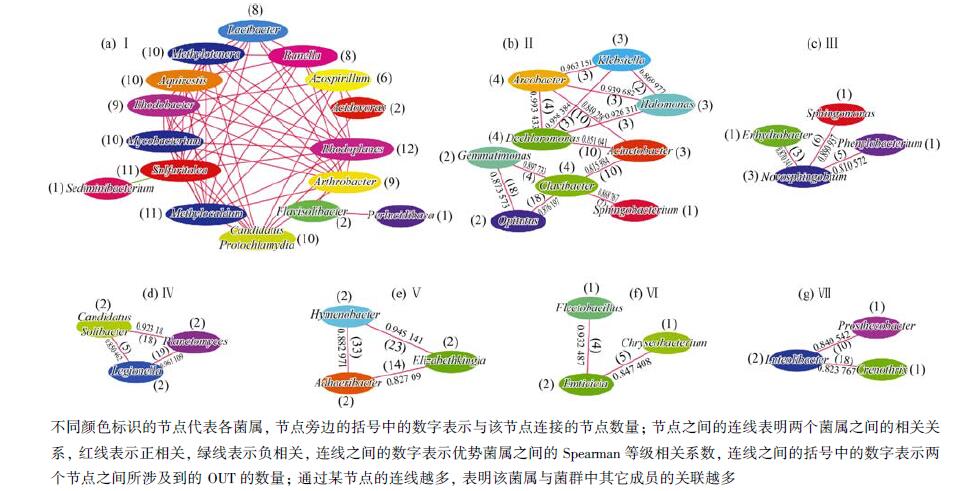
|
图 11 优势菌属的共现性网络 Fig. 11 Correlation network of co-occurring microbial genera |
相关研究表明, 通过不同微生物之间的共现性网络可以推测微生物之间的相互作用[13~15], 具有共生关系的微生物在丰度上有一定的相关性, 譬如处于互利共生关系的微生物的丰度会呈正相关关系[16], 而处于竞争关系的微生物的丰度会呈现负相关关系[17].分析所构建的窖水中优势菌属之间的共现性网络各节点的菌属间的Spearman等级相关系数(图 11、表 3)可知, 除Sediminibacterium与Sulfuritalea呈显著负相关(-0.812125)外, 其余菌属均呈现显著正相关(Spearman系数>0.800000)关系, 说明窖水中Sediminibacterium与Sulfuritalea营竞争关系, 其余节点的菌属营互利共生关系.
|
|
表 3 关联网络(a)相关参数1) Table 3 Parameters of association network about(a) |
除此之外, 可以通过微生物之间的共现性网络的拓扑属性推测关键的微生物类群[18]及不同分类微生物类群之间的生态互作关系[13, 15].微生物共现性网络的拓扑属性主要指点度中心度, 即与单个网络节点直接相连的其他节点的数量, 具有较高点度中心度的节点定义为大于网络总节点数的1/2的节点数(引入HN值评价).共现性网络中两个节点之间的连线(即边)来代表微生物类群间的生态互作关系, 其互作强度可以用一条边连接的两个微生物类群之间存在的相关OTUs的数量[19], 较强的互作强度定义为大于网络中的边涉及的OTUs总数与总边数的比值的OTUs数目(引入SE值衡量).共现性网络Ⅰ[图 11(a)]共得到16个有效的节点和60个边, HN及SE均为8.则具有较高点度中心度的菌属(HN)为: Rhodoplanes(12)、Arthrobacter(9)、Candidatus Protochlamydia(10)、Methylocaldum(11)、Sulfuritalea(11)、Mycobacterium(10)、Aquirestis(10)、Rhodobacter(9)、Methylotenera(10);互作强度较高的菌属的边(SE)为: Sulfuritalea-Sediminibacterium(13)、Sulfuritalea-Aquirestis(9)、Sulfuritalea-Rhodobacter(20)、Azospirillum-Rhodobacter(18)、Methylocaldum-Rhodobacter(17)、Arthrobacter-Rhodobacter(18)、Rhodoplanes-Aquirestis(10)、Rhodoplanes-Rhodobacter(21)、Rhodoplanes-Mycobacterium(9)、Rhodoplanes-Methylocaldum(9)、Perlucidibaca-Flavisolibacter(13)、Candidatus Protochlamydia-Rhodobacter(17)、Methylotenera-Aquirestis(10)、Methylotenera-Rhodobacter(21)、Methylotenera-Mycobacterium(9)、Methylotenera-Sulfuritalea(9)、Methylotenera-Rhodoplanes(10)、Rhodobacter-Aquirestis(21)、Mycobacterium-Aquirestis(9)、Mycobacterium-Rhodobacter(20)、Flavisolibacter-Rhodoplanes(16);共现性网络Ⅱ[图 11(b)]共得到9个有效的节点和13个边, HN及SE分别为5、7.网络中菌属的点度中心度较低(HN<5), 互作强度较高的菌属的边(SE)为: Acinetobacter-Arcobacter(10)、Acinetobacter-Dechloromonas(10)、Acinetobacter-Clavibacter(10)、Optitutus-Clavibacter(18)、Optitutus-Gemmatimonas(18);共现性网络Ⅲ[图 11(c)]共得到4个有效的节点和3个边, HN及SE分别为2、5.具有较高点度中心度的菌属(HN)为Novosphingobium(3), 互作强度较高的菌属的边(SE)为Novosphingobium-不同颜色标识的节点代表各菌属, 节点旁边的括号中的数字表示与该节点连接的节点数量; 节点之间的连线表明两个菌属之间的相关关系, 红线表示正相关, 绿线表示负相关, 连线之间的数字表示优势菌属之间的Spearman等级相关系数, 连线之间的括号中的数字表示两个节点之间所涉及到的OUT的数量; 通过某节点的连线越多, 表明该菌属与菌群中其它成员的关联越多Sphingobium(6);共现性网络Ⅳ[图 11(d)]共得到3个有效的节点和3个边, HN及SE分别为2、14.网络中菌属的点度中心度较低(HN=2), 互作强度较高的菌属的边(SE)为Planctomyces-Candidatus Solibacter(18)、Planctomyces-Legionella(19);共现性网络Ⅴ[图 11(e)]共得到3个有效的节点和3个边, HN及SE分别为2、23.网络中菌属的点度中心度较低(HN=2), 互作强度较高的菌属的边(SE)为Hymenobacter-Adhaeribacter(33);共现性网络Ⅵ[图 11(f)]共得到3个有效的节点和2个边, HN及SE分别为2、5.网络中菌属的点度中心度(HN=2)及互作强度(SE≤5)均较低; 共现性网络Ⅶ[图 11(g)]共得到3个有效的节点和2个边, HN及SE分别为2、14.网络中菌属的点度中心度较低(HN≤2), 互作强度较高的菌属的边(SE)为Luteolibacter-Crenothrix(18).
综合分析窖水共现性网络的节点微生物相关性、点度中心度及菌属间的互作强度, 认为窖水中的关键的微生物类群为Rhodobacter、Methylocaldum、Methylotenera、Acinetobacter、Novosphingobium、Planctomyces、Hymenobacter、Luteolibacter, 其中最为重要的微生物类群为Rhodobacter.
3 讨论 3.1 细菌群落及功能基因多样性与水质因子的相关性阐释微生物群落中微生物多样性与功能之间的联系是微生物生态学领域研究的难点之一, 主要体现在以下两个层面: ①虽然环境中的微生物分类学多样性与功能多样性有密切的联系, 但由于细菌微生物群落中广泛存在的功能冗余现象, 致使二者在大多数情况下并未呈现显著相关关系.即在水环境污染胁迫作用下, 水环境某特定位点及某区域范围内的微生物群落发生了较大幅度改变, 而其功能却未发生显著变化或表现出响应滞后现象[20~22]; ②尽管有多种方法, 如可利用美国Biolog公司的Biolog-Eco板来表征微生物局部代谢多样性、利用宏基因组和宏转录组测序等高通量技术来全面了解微生物群落功能的潜力和活力、或是利用基于16S rRNA基因序列的PICRUSt生物信息学工具来预测对应细菌和古菌的代谢功能图谱, 可以对微生物群落功能进行表征和研究.但是, 目前分子生物学方法还无法就基于微生物遗传多样性的指标与基于微生物活动的功能多样性的指标之间建立联系, 甚至对于如何描述微生物的功能多样性也不是很清楚[23].
虽然存在诸多困难, 使得人们无法就基于微生物遗传多样性的指标与基于微生物活动的功能多样性的指标之间建立有效、直接的联系, 但这并不妨碍从另一个角度来探讨两者之间潜在的密切联系.通过细菌与新陈代谢相关的功能基因和水质因子间的相关性分析(图 9)发现, 表征窖水氮、磷、有机物污染的多数指标均和细菌与新陈代谢相关的功能基因呈正相关关系, 进一步通过细菌群落和水质因子间的相关性分析(图 10)显示, 窖水不同水质因子对细菌菌落的影响存在明显的分组, 即组内菌属生态功能的相似性及组间菌属生态功能的差异性, 说明在窖水中存在具有相对特定生态功能的细菌, 进行着诸多活跃的与新陈代谢功能基因相关的代谢活动.
3.2 优势微生物类群的关联网络分析本研究基于16S rRNA基因的序列分析和数据统计学分析发现, 细菌群落组成(图 6、7)及具有相对特定生态功能的微生物基因序列(图 8)多样性丰富.绝大多数的微生物类群具备脱氮、除磷、降解有机物的相对特定生态功能, 其中15种是具有脱氮、除磷功能的异养硝化-好氧反硝化菌[24], 说明微生物是窖水中氮、磷及有机物等主要污染物降解的主要驱动者.由于微生物主要以群落的水平在特定的环境下承担特定的生态功能, 群落中的微生物之间存在复杂相互作用.因此借助共现性关联网络分析, 筛选参与窖水中污染物微生物降解的关键类群并预测窖水中微生物之间的生态互作关系, 对加深窖水中污染物微生物降解机制的认识至关重要.
通过综合分析窖水微生物类群共现性关联网络的节点微生物相关性、点度中心度及菌属间的互作强度, 获悉窖水微生物类群共现性关联网络中大部分节点的菌属营互利共生类型的生态关系, 认为: Rhodobacter、Methylocaldum、Methylotenera、Acinetobacter、Novosphingobium、Planctomyces、Hymenobacter、Luteolibacter的点度中心度较高, 与其他菌属的互作强度较高, 为参与窖水污染物微生物降解的关键类群, 且以Rhodobacter最为重要.值得注意的是, 研究通过高通量测序截面数据, 基于Spearman等级相关系数推断得到的菌属生态互作关系, 并不是物种间相互作用的本质, 不能有效地区分窖水中微生物类群之间的关系是直接相关(功能互补)还是间接相关(有相似的环境要求), 增加了分析的复杂性并给分析网络的拓扑属性带来了较大的误差.虽研究结果很难区分出以氮、磷及有机物污染为主要特征的窖水中微生物菌属之间相互作用的本质, 但仍然在很大程度上阐释了潜在的菌属相互作用.由于时间序列数据能更好地捕捉到物种间相互作用导致的种群数量的动态变化, 更有利于推断物种间相互作用的方向和强度[25], 因此本研究为进一步通过时间序列数据研究窖水菌种之间的相互作用、更准确地筛选关键微生物类群提供了依据.
4 结论本研究基于16S rRNA的高通量测序微生物组学截面数据与微生物物种的同现或相关种间作用推断模型, 构建了以氮、磷及有机物污染为主要特征的窖水中微生物菌属间的共现性关联网络, 并依据所构建的共现性关联网络的拓扑属性筛选出了关键的微生物类群, 推断与预测了窖水中微生物菌属间潜在的生态互作关系.表明窖水微生物类群共现性关联网络中大部分节点的菌属营互利共生类型的生态关系, 关键的微生物菌属为: Rhodobacter、Methylocaldum、Methylotenera、Acinetobacter、Novosphingobium、Planctomyces、Hymenobacter、Luteolibacter. Rhodobacter为关键细菌的代表属.
| [1] |
冯帅, 李叙勇, 邓建才. 平原河网典型污染物生物降解系数的研究[J]. 环境科学, 2016, 37(5): 1724-1733. Feng S, Li X Y, Deng J C. Biodegradation coefficients of typical pollutants in the plain rivers network[J]. Environmental Science, 2016, 37(5): 1724-1733. |
| [2] | Ostroumov S A. Polyfunctional role of biodiversity in processes leading to water purification:current conceptualizations and concluding remarks[J]. Hydrobiologia, 2002, 469(1-3): 203-204. |
| [3] | Lupatini M, Suleiman A K A, Jacques R J S, et al. Network topology reveals high connectance levels and few key microbial genera within soils[J]. Frontiers in Environmental Science, 2014, 2: 1-11. |
| [4] | Adam I K, Duarte M, Pathmanathan J, et al. Microbial communities in pyrene amended soil-compost mixture and fertilized soil[J]. AMB Express, 2017, 7: 7. DOI:10.1186/s13568-016-0306-9 |
| [5] |
王斌, 陈庆彩, 胡晓珂. 微生物降解芘过程中的关键细菌[J]. 微生物学报, 2017, 57(6): 856-866. Wang B, Chen Q C, Hu X K. Key bacteria during microbial degradation of pyrene[J]. Acta Microbiologica Sinica, 2017, 57(6): 856-866. |
| [6] | Trosvik P, de Muinck E J. Ecology of bacteria in the human gastrointestinal tract-identification of keystone and foundation taxa[J]. Microbiome, 2015, 3: 44. DOI:10.1186/s40168-015-0107-4 |
| [7] | Fisher C K, Methta P. Identifying keystone species in the human gut microbiome from metagenomic timeseries using sparse linear regression[J]. PLoS One, 2014, 9(7): e102451. DOI:10.1371/journal.pone.0102451 |
| [8] | Shi S J, Nuccio E E, Shi Z J, et al. The interconnected rhizosphere:high network complexity dominates rhizosphere assemblages[J]. Ecology Letters, 2016, 19(8): 926-936. DOI:10.1111/ele.12630 |
| [9] | Dethlefsen L, Eckburg P B, Bik E M, et al. Assembly of the human intestinal microbiota[J]. Trends in Ecology & Evolution, 2006, 21(9): 517-523. |
| [10] | Marino S, Baxter N T, Huffnagle G B, et al. Mathematical modeling of primary succession of murine intestinal microbiota[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(1): 439-444. DOI:10.1073/pnas.1311322111 |
| [11] | Fuhrman J A. Microbial community structure and its functional implications[J]. Nature, 2009, 459(7244): 193-199. DOI:10.1038/nature08058 |
| [12] | Ju F, Xia Y, Guo F, et al. Taxonomic relatedness shapes bacterial assembly in activated sludge of globally distributed wastewater treatment plants[J]. Environmental Microbiology, 2014, 16(8): 2421-2432. DOI:10.1111/1462-2920.12355 |
| [13] | Faust K, Raes J. Microbial interactions:from networks to models[J]. Nature Reviews Microbiology, 2012, 10(8): 538-550. DOI:10.1038/nrmicro2832 |
| [14] | Barberán A, Bates S T, Casamayor E O, et al. Using network analysis to explore co-occurrence patterns in soil microbial communities[J]. The ISME Journal, 2012, 6(2): 343-351. DOI:10.1038/ismej.2011.119 |
| [15] | Fuhrman J A, Cram J A, Needham D M. Marine microbial community dynamics and their ecological interpretation[J]. Nature Reviews Microbiology, 2015, 13(3): 133-146. DOI:10.1038/nrmicro3417 |
| [16] | Molloy S. Environmental microbiology:disentangling syntrophy[J]. Nature Reviews Microbiology, 2014, 12(1): 7. |
| [17] | Hibbing M E, Fuqua C, Parsek M R, et al. Bacterial competition:surviving and thriving in the microbial jungle[J]. Nature Reviews Microbiology, 2010, 8(1): 15-25. DOI:10.1038/nrmicro2259 |
| [18] | Zhou J Z, Deng Y, Luo F, et al. Phylogenetic molecular ecological network of soil microbial communities in response to elevated CO2[J]. MBio, 2011, 2(4): e00122-11. |
| [19] | Vályi K, Mardhiah U, Rillig M C, et al. Community assembly and coexistence in communities of Arbuscular mycorrhizal fungi[J]. The ISME Journal, 2016, 10(10): 2341-2351. DOI:10.1038/ismej.2016.46 |
| [20] | Comte J, del Giorgio P A. Linking the patterns of change in composition and function in bacterioplankton successions along environmental gradients[J]. Ecology, 2010, 91(5): 1466-1476. DOI:10.1890/09-0848.1 |
| [21] | Baho D L, Peter H, Tranvik L J. Resistance and resilience of microbial communities-temporal and spatial insurance against Perturbations[J]. Environmental Microbiology, 2012, 14(9): 2283-2292. DOI:10.1111/emi.2012.14.issue-9 |
| [22] | Berga M, Székely A J, Langenheder S. Effects of disturbance intensity and frequency on bacterial community composition and function[J]. PLoS One, 2012, 7(5): e36959. DOI:10.1371/journal.pone.0036959 |
| [23] |
李晶, 刘玉荣, 贺纪正, 等. 土壤微生物对华宁胁迫的响应机制[J]. 环境科学学报, 2013, 4(33): 959-967. Li J, Liu Y R, He J Z, et al. Insights into the responses of soil microbial community to the environmental disturbances[J]. Acta Scientiae Circumstantiae, 2013, 4(33): 959-967. |
| [24] |
杨浩, 张国珍, 杨晓妮, 等. 16S rRNA高通量测序研究集雨窖水中微生物群落结构及多样性[J]. 环境科学, 2017, 4(38): 1704-1716. Yang H, Zhang G Z, Yang X Y, et al. Microbial community structure and diversity in cellar water by 16s rrna high-throughput sequencing[J]. Environmental Science, 2017, 4(38): 1704-1716. |
| [25] | Faust K, Lahti L, Gonze D, et al. Metagenomics meets time series analysis:unraveling microbial community dynamics[J]. Current Opinion in Microbiology, 2015, 25: 56-66. DOI:10.1016/j.mib.2015.04.004 |
 2018, Vol. 39
2018, Vol. 39


