硝化作用作为自然界中氮素循环调控的重要环节, 普遍发生于土壤环境中, 对污水处理中的生物学过程及饮用水生产也起着重要作用.而农业土壤中氮肥的施用会进一步促进硝化作用的发生[1, 2], 致硝酸盐淋溶损失, 造成氮肥的浪费[3~6]及地下水的硝酸盐污染[7].
自19世纪末[8], 一直认为硝化作用分两步进行——首先是氨氧化细菌(ammonia-oxidizing bacteria, AOB)[9]和/或氨氧化古菌(ammonia-oxidizing archeae, AOA)[10]氧化氨为亚硝酸盐, 其次亚硝酸盐氧化细菌(nitrite-oxidizing bacteria, NOB)[9]氧化亚硝酸盐为硝酸盐.然而早前有学者[11, 12]曾根据动力学理论上的可行性, 提出单步硝化作用的理论即由单一微生物独自完成氨氧化为硝态氮的整个过程.但直到2015年底, 有学者相继在不同环境中发现能进行单步硝化作用的微生物——已经纯培养的细菌(Candidatus Nitrospira nitrosa、Candidatus Nitrospira nitrificans)[13]、(Candidatus Nitrospira inopinata)[14]和未经纯培养的细菌(类Nitrospira)[15], 从而证实了单步硝化作用的存在.这些微生物被定义为全程氨氧化微生物(Comammox), 均属于亚硝酸盐氧化细菌中的硝化螺菌属(Nitrospira).根据已发表的宏基因组数据库中基因序列的比对发现全程氨氧化细菌广泛分布于农业土壤、森林土壤、稻田土壤、淡水环境及工程系统[13, 14].全程氨氧化细菌的发现使学者需要有更多的工作去重新评估土壤中AOA、AOB以及Comammox在生态系统中的分布及相对贡献.
氮肥施用会促进硝化作用的发生, 且与施肥量成正相关[16, 17].佟德利等[18]对分别施用尿素、硫酸铵、碳酸氢铵这3种氮肥及分别不同施用量的试验中, 其结果显示这3种氮肥的施用均促进土壤硝化作用, 且净硝化速率随施用量增加而增加.本研究选取不施肥的对照组(blank control)、常规施肥(NPK)、高量施肥(1.5NPKS)这3种不同施肥量的农业利用土壤, 通过实时荧光定量PCR技术分析氨氧化细菌、氨氧化古菌及全程氨氧化细菌丰度对不同施肥量的响应, 以期为进一步在土壤环境中展开单步硝化研究奠定基础.
1 材料与方法 1.1 供试土壤土样均采自西南大学长期定位试验田(106°24′33″E, 29°48′36″N), 海拔266.3 m, 该土壤是由侏罗系沙溪庙组灰棕紫色沙泥岩母质上发育而成的中性紫色水稻土.
本研究选择的3个试验处理为(表 1): blank control(不施肥对照组)、NPK、1.5NPKS(1.5 NPK+水稻秸秆), 其中N肥为尿素, P肥为过磷酸钙, K肥为氯化钾. 1991~1996年每季施肥量为: N 150 kg·hm-2、P2O5 75 kg·hm-2、K2O 75 kg·hm-2, 水稻秸秆7.5 t·hm)-2. 1996年秋季之后, 每季P、K肥用量由原来的75 kg·hm-2改为60 kg·hm-2; 小麦氮肥用量改为135 kg·hm-2, 水稻氮肥用量不变.水稻秸秆于每年小麦播种前撒入.水稻和小麦均使用60%的N肥及全部P、K肥作为基肥, 40% N肥作为分蘖肥. 1991~2017年一直采用稻-麦轮茬制.
|
|
表 1 基本理化性质 Table 1 Basic physical and chemical properties of fertilizer treatments |
本试验点自1991年始, 没有设立重复小区, 但小区面积较大(120 m2), 故将小区均分为3个区域即3次假重复, 每区域选取4 m×5 m样方的表层土以保障数据的准确性.
1.2 研究方法采样时间为2017年12月, 选取0~20 cm表层土壤作为研究对象, 土样采回后, 去掉植物根系等杂质, 分两部分保存:一部分土样采用鲜土过2 cm筛后立即进行土壤硝化势的测定和土壤DNA的提取, 另一部分则风干研磨过0.25 mm、1 mm筛用于测定其基本理化性质.
1.2.1 化学分析方法土壤pH值于1:2.5土水比条件下, 采用梅特勒LE 438复合电极(梅特勒-托利多仪器有限公司, 上海, 中国)进行测定; 土壤有机质采用重铬酸钾容重法测定; 全氮采用改进的凯氏法测定; 土壤硝态氮和铵态氮均采用流动分析仪测定(AA3全自动连续流动分析仪, SEAL Analitical, 德国).所有样品均设置3次重复.
硝化势测定按鲁如坤所述(悬浮液培养法)稍作修改[19]:称取15 g鲜土于250 mL三角瓶中, 加入100 mL液体培养基(1.5 mL 0.2 mol·L-1 KH2PO4, 3.5 mL 0.2 mol·L-1 K2HPO4与15 mL 0.05 mol·L-1(NH4)2SO4到1 L容量瓶, 混匀并用H2SO4/NaOH调至pH 7.2), 用带气孔的橡皮塞塞紧后置于振荡器上振荡24 h(200 r·min-1, 25℃), 并于2、4、22、24 h取样测定NO3--N含量; 另按上述土:水比测定原始土壤中NO3--N含量, 均2次重复.以培养时间为横坐标, NO3--N含量为纵坐标, 求出斜率, 即土壤硝化势[mg·(kg·d)-1].
1.2.2 定量PCR分析方法首先, 采用FastDNA® SPIN Kit for Soil (MP Biomedicals, LLC)试剂盒、并按试剂盒操作说明提取土壤微生物总DNA, 并于-20℃保存待测.随后, 采用实时荧光定量PCR法在QuantStudioTM6 Flex定量PCR仪(Thermo Fisher Scientific, Singapore)上测定氨氧化细菌(AOB)、氨氧化古菌(AOA)及全程氨氧化菌Comammox Clade A和Clade B amoA基因的拷贝数. AOB、AOA、Comammox Clade A、Comammox Clade B的amoA基因定量PCR所用扩增引物见表 2. AOB、AOA、Comammox Clade A、Comammox Clade B的amoA基因定量PCR所用反应体系均为20 μL, 主要包含:待扩增的模板DNA 1 μL、上下引物及ROX染料各0.4 μL, Taq DNA聚合酶10 μL、灭菌水7.8 μL. AOA amoA基因定量PCR扩增条件为: 95℃, 1.5 min; 40×(95℃, 30 s; 55℃, 45 s; 72℃, 45 s with plate read); Melt curve 60.0℃ to 95.0℃, increment 0.5℃, 0: 05+plate read. AOB扩增条件为: 95℃, 3.0 min; 38×(95℃, 30 s; 60℃, 1.5 min with plate read); Melt curve 60.0℃ to 95.0℃, increment 0.5℃, 0: 05+plate read. Comammox Clade A、Comammox Clade B的amoA基因定量PCR扩增条件均为: 95℃, 3.0 min; 45×(95℃, 30 s; 52℃, 45 s; 72℃, 1 min with plate read); Melt curve 60.0℃ to 95.0℃, increment 0.5℃, 0: 05+plate read.
|
|
表 2 AOB、AOA、Comammox Clade A、Comammox Clade B amoA基因的引物序列 Table 2 Primers for AOB, AOA, Comammox Clade A, and Comammox Clade B amoA genes |
通过液体LB培养基培养的含有目的基因的克隆子, 按试剂盒(MiniBEST Plasmid Purification Kit)说明书提取、纯化质粒后, 首先对质粒进行测序验证, 然后在NanoDrop®ND-1000 UV-Vis分光光度计上测定质粒的浓度, 并计算出目的基因的拷贝数.最后用TE缓冲溶液将质粒连续稀释6~8个梯度以制作定量PCR的标准曲线.其中, 所用的Comammox Clade A质粒片段为: TTATAACTGG GTGAACTATCGACAACCGTTTGGAGCAACCATAAC C ATTCTGGCACTCCTCGCAGGAAAGTGGGTCACGGTTA TTGCCGCTTGGTGGTGGTGGTCCAACTATCCGTATAA CTTCGTCATGCCCGCCACTTTGCTCCCCAGCGCATTG GTCATGGACATCGTTCTGTTGCTGACCCGAAGCTGGG TGACCACAGCGGTGGTTGGAGCATGGCTGTTCGCAG CGCTGTTCTACCCGACCAACTGGGCTTTATTCGGGTA CAGCAAGACACCGATCGTCGTGGATGGGACACTGCT CTCTTGGGCCGACTATATGGGCTTCGTGTATGTCCGT ACCGGAACCCCTGAATATATCCGGCTGATCGAGGTG GGGTCACTTCGCACGTTCGGCGGACACAGCACGATG ATCT; 所用的Comammox Clade B质粒片段为: AAATCCAGACGGTGTGTATGCCGAACGTACGAAGCG ATCCCACCTCGATGATCCGGATATACTCAGGCGTCC CGGTACGAACATACGTAAACCCCATGTAGTCGGCCA GCGAGAGAAGCTGACCGTCGACCACCAACGGTTGG TGGCTATATCCGAAGAGAGCATATTGCGTTGGATTA AACATCATTGCGAATGCCCAGACCCCGAATATCGCA GTGAGCATCCAGCTTCGAGTCAGTAACAGGGTACAG TCGAGGATCAATGCGCTGGAGACCATGGTGGCTGGC ATAACGAAGTTCATCGGGAAATTGGCCCACCAATAC CAAGCAAATACAATCGTGATCCACTTCCCGACGAGG AGCCCCGTGATGCTCAACGTTGCACCAAACGGTTGC CTGTAGAACGTCCAGAAGTA.
1.3 数据处理与统计分析所有数据均采用Microsoft Excel 2016和SPSS 18进行统计分析.采用单因素(one-way ANOVA)、Duncan法进行方差分析及多重比较(α =0.05).利用Origin 8.5软件作图.图表中数据为平均值±标准差.
2 结果与分析 2.1 不同施肥量处理下全程氨氧化细菌(Comammox Nitrospira)丰度全程氨氧化细菌分支A(Comammox Clade A)和分支B(Comammox Clade B)丰度随施肥量的变化情况如图 1所示.如图 1(a), 与对照组相比, NPK和1.5NPKS处理土壤Comammox Clade A amoA基因拷贝数均显著增加, 分别为对照组基因拷贝数(以干土计, 下同)(9.0×107 copies·g-1)的1.9和8.0倍, 说明施肥能显著提高Comammox Clade A amoA基因拷贝数, 且随施肥量增加, 其丰度增加显著.而Comammox Clade B amoA基因拷贝数在对照组、NPK、1.5NPKS这3种不同施肥量中差异不显著[图 1(b)], 且比Comammox Clade A低一个数量级, 3种处理中Clade B amoA基因拷贝数分别为1.5×107、1.2×107、1.7×107 copies·g-1, 说明施肥处理对Clade B amoA基因拷贝数无显著影响.
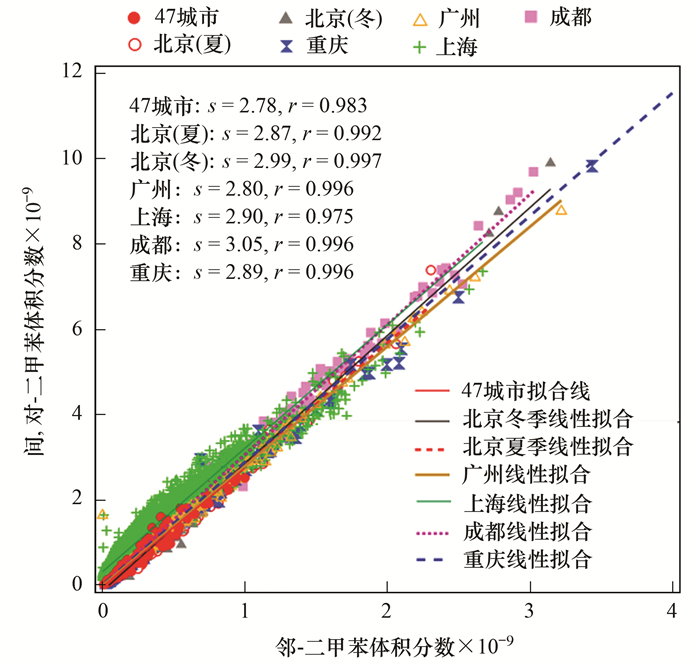
|
误差线表示标准差, 不同小写字母表示不同施肥处理显著性差异(n=3, P < 0.05)下同 图 1 不同施肥量管理下Comammox Clade A、Comammox Clade B amoA基因拷贝数 Fig. 1 Comammox Clade A and Comammox Clade B amoA gene copies under different fertilizer treatments |
由图 2可以看出, NPK、1.5NPKS这2种施肥处理土壤均显著增加AOA amoA基因拷贝数, 分别增加到对照组的3.2、7.2倍[图 2(a)]; AOB由对照组的2.0×105 copies·g-1分别增加到6.1×107 copies·g-1、9.3×107 copies·g-1, 均提高两个数量级[图 2(b)]; 其次, 1.5 NPKS处理AOA、AOB丰度均显著高于NPK处理土壤.说明AOA、AOB受施肥影响显著, 随施肥量增加而显著增加.

|
图 2 不同施肥量管理下AOA、AOB amoA基因拷贝数 Fig. 2 Ammonia-oxidizing bacteria (AOB) and archaea (AOA) amoA gene copies under different fertilizer treatments |
如图 3所示, 不同施肥量处理间其硝化势差异显著(P<0.05). 1.5NPKS处理硝化势(以干土中含有硝态氮的氮含量计, 下同)达到对照组[(6.82±1.20)mg·(kg·d)-1]的9.1倍, 是NPK处理硝化势[(44.87±5.63) mg·(kg·d)-1]的1.4倍.可见, 施肥处理硝化势显著高于不施肥处理土壤, 且随着施肥量增加而硝化势显著增强, 说明施肥显著提高耕作土壤的硝化作用, 与前人的研究报道相似[16~18].
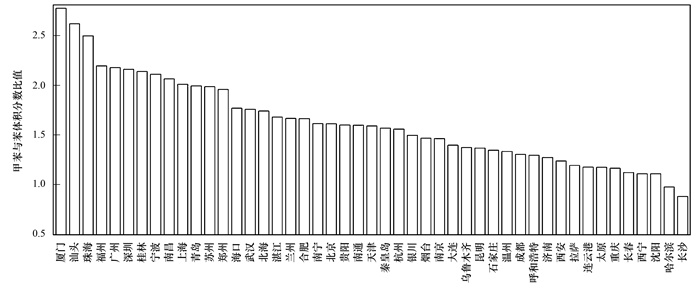
|
图 3 不同施肥量管理下土壤硝化势 Fig. 3 Soil nitrification potential under different fertilizer treatments |
有学者根据已发表的宏基因数据库中DNA序列比对后推测, 全程氨氧化微生物可能广泛分布于土壤、水等生态环境中[13~15, 23].本研究在对照组、常规施肥(NPK)、高量施肥(1.5NPKS)这3种不同施肥量的稻田土壤中, 都检测出高数量级的全程氨氧化细菌(Comammox)amoA基因拷贝数, 分支A(Comammox Clade A)与分支B(Comammox Clade B)amoA基因拷贝数分别为9.0×107~7.2×108 copies·g-1、1.2×107~1.7×107 copies·g-1, 进一步证实全程氨氧化细菌与氨氧化细菌(AOA)和氨氧化古菌(AOB)一样, 广泛分布于农田土壤中.其次, 3种施肥量土壤中Comammox amoA基因拷贝数(分支A与分支B之和)与AOA、AOB的比值及AOA与AOB的比值均大于1(表 3), 表明在3种不同施肥量水稻土中均是全程氨氧化细菌amoA基因拷贝数最高, 其次是氨氧化古菌, 而氨氧化细菌最低.表明全程氨氧化细菌在中性紫色水稻土的硝化微生物中占有比AOA、AOB更大的比例.
|
|
表 3 不同施肥管理下各硝化菌amoA基因拷贝数之比情况 Table 3 Nitrifying microorganisms and amoA gene copy ratios under different fertilizer treatments |
氨氧化古菌曾被证明在低氮的贫营养条件下, 对硝化作用贡献更大, 而氨氧化细菌在高氮量条件下对硝化作用起主导作用[24~26].有研究发现在pH为5.6的酸性水稻土中, 氨氧化古菌在硝化作用起主导作用[27], 中性紫色土中硝化作用则是由氨氧化细菌和氨氧化古菌共同推动[28], 而在pH为8.2的石灰性水稻土中硝化作用由氨氧化细菌起主导作用[27].然而最近的研究发现全程氨氧化细菌Candidatus Nitrospira inopinata比可培养的大多数氨氧化古菌对氨具有更高的亲和力. Kits等[29]通过氨氧化的动力学分析发现Nitrospira inopinata可在氨浓度为0.049 μmol·L-1时进行生长, 而培养的3种氨氧化古菌Nitrososphaera gargensis、Nitrososphaera viennensis、Candidatus Nitrosotenuis uzonensis可适应的极低氨浓度分别为0.7、0.36、4.4 μmol·L-1.此外, 也有研究表明Comammox Nitrospira在低氧、磷、铜等贫瘠的低营养条件下亦能缓慢生长. Palomo等[23]发现全程氨氧化细菌具有β-AOB不具备的能编码允许在低浓度氧气下高效生长的基因; 还有研究发现其基因组含有可在磷不足和饥饿下高度表达的碱性磷酸酶(phoD)[30, 31], 而这是AOA基因组中未检测到的且β-AOB中也不普遍存在的[27].本研究结果显示, 在不施肥的对照组中Comammox amoA基因拷贝数(分支A与分支B之和)与AOA、AOB的比值分别为7.2、524.4(表 3), 说明在不施肥的耕作土壤中全程氨氧化细菌(Comammox)丰度比耐贫营养条件的氨氧化古菌更高, 比氨氧化细菌高甚至两个数量级, 表明全程氨氧化细菌比AOA、AOB更适应低氮条件.
Pjevac等[22]在水稻土(采自意大利维切里)中检测到全程氨氧化细菌分支A(Comammox Clade A)、分支B (Comammox Clade B)丰度数量级(以DNA计, 下同)均为102 ng-1, 相当于1011g-1, 但分支A丰度略高于分支B.本研究中Comammox Clade A丰度与Comammox Clade B的比值在3种施肥量土壤中均远大于1, 且由对照组的6.1增加到1.5NPKS的43.1(表 3); 同时结合图 1, 即结果显示为Comammox Clade A对施肥更敏感, 随施肥量增加而增加, 而Comammox Clade B却无显著变化.表明在中性紫色水稻土中全程氨氧化细菌以分支A(Comammox Clade A)为主.
土壤微生物受不同施肥管理[32, 33]的影响, 而土壤硝化作用是土壤中仅由微生物完成的生物学过程, 因此土壤硝化作用严格受施肥管理的影响, 随施肥而土壤硝化势增强[34, 35].氮肥施用可增加氨氧化微生物丰度, Zhou等[36]对不同施肥处理的水稻土的研究表明, 与对照组(不施肥)或无N素的肥料处理相比, 施用N肥处理能显著增加氨氧化细菌的基因拷贝数.通过DNA-SIP微域培养试验发现, 长期施肥不仅改变氨氧化细菌(AOB)和氨氧化古菌(AOA)的相对丰度, 对其中“活跃的”AOA、AOB类群也产生较大影响[37].本试验结果显示与对照组相比, 长期施肥后氨氧化细菌、氨氧化古菌及全程氨氧化细菌分支A的丰度(图 1和图 2)均显著增加; 对比常规施肥(NPK)和高量施肥(1.5NPKS)处理土壤(表 3), 结果显示1.5NPKS处理土壤中Comammox丰度与AOA、AOB的比值及AOA与AOB的比值分别为NPK处理中比值的2.0、2.6、1.3倍.虽然高量施肥处理中施用了秸秆, 但有研究发现cry1Ac/cpti双价抗虫基因水稻的秸秆还田降解对土壤氮素转化关键微生物群落丰度与组成均无明显影响[38], 故表明随施肥量增加其丰度增加更显著, 硝化势变化趋势也与此一致(图 3).故推测全程氨氧化细菌很可能在中性紫色水稻土的硝化作用中起作用, 尤其是Comammox Clade A.这或许可以解释以前有学者将水稻土在适宜硝化作用的条件下用13CO2处理后, 从硝化螺菌属(Nitrospira)中提取到的DNA被标记的程度要大于AOA和AOB的原因[39].
4 结论(1) 本试验在3种施肥量的水稻土中均检测到高数量级的全程氨氧化细菌(Comammox Nitrospira), 表明全程氨氧化细菌广泛分布于中性紫色水稻土中.
(2) 在中性紫色水稻土中, 全程氨氧化细菌有比氨氧化古菌(AOA)和氨氧化细菌(AOB)更高的丰度, 尤其是在不施肥的贫营养环境中, 其丰度甚至是耐贫营养条件的氨氧化古菌丰度的7.2倍.
(3) 全程氨氧化细菌分支A(Comammox Clade A)随施肥量增加有显著增加趋势, 与硝化势、AOA、AOB变化趋势一致, 而全程氨氧化细菌分支B(Comammox Clade B)受施肥影响不显著.
(4) 中性紫色水稻土中全程氨氧化细菌以Clade A为主.
| [1] |
蔡祖聪, 赵维. 土地利用方式对湿润亚热带土壤硝化作用的影响[J]. 土壤学报, 2009, 46(5): 795-801. Cai Z C, Zhao W. Effects of land use types on nitrification in humid subtropical soils of China[J]. Acta Pedologica Sinica, 2009, 46(5): 795-801. DOI:10.3321/j.issn:0564-3929.2009.05.006 |
| [2] |
于涌杰. 土地利用方式对中国东南部红壤微生物特性及氮转化作用的影响[D]. 南京: 南京师范大学, 2012. Yu Y J. Effect of land-use types on microbial properties and nitrogen transformation in red soils of Southeastern China[D]. Nanjing: Nanjing Normal University, 2012. http: //cdmd. cnki. com. cn/Article/CDMD-10319-1013105169. htm |
| [3] | Oenema O, van Liere L, Schoumans O. Effects of lowering nitrogen and phosphorus surpluses in agriculture on the quality of groundwater and surface water in the Netherlands[J]. Journal of Hydrology, 2005, 304(1-4): 289-301. DOI:10.1016/j.jhydrol.2004.07.044 |
| [4] | Zhang W L, Tian Z X, Zhang N, et al. Nitrate pollution of groundwater in northern China[J]. Agriculture, Ecosystems & Environment, 1996, 59(3): 223-231. |
| [5] | Galloway J N, Dentener F J, Capone D G, et al. Nitrogen cycles:past, present, and future[J]. Biogeochemistry, 2004, 70(2): 153-226. DOI:10.1007/s10533-004-0370-0 |
| [6] | Randall G W, Vetsch J A, Huffman J R. Nitrate losses in subsurface drainage from a corn-soybean rotation as affected by time of nitrogen application and use of nitrapyrin[J]. Journal of Environmental Quality, 2003, 32(5): 1764-1772. DOI:10.2134/jeq2003.1764 |
| [7] |
朱兆良, David N, 孙波. 中国农业面源污染控制对策[M]. 北京: 中国环境科学出版社, 2006. Zhu Z L, David N, Sun B. Policy for reducing non-point pollution from crop production in China[M]. Beijing: China Environmental Science Press, 2006. |
| [8] | Winogradsky S. The morphology of the contributions of nitrification system[J]. Archives of Biological Sciences, 1890, 4: 257-275. |
| [9] | Bock E, Wagner M. Oxidation of inorganic nitrogen compounds as an energy source[A]. In: Dworkin M, Falkow S, Rosenberg E, et al (eds. ). The Prokaryotes[C]. New York: Springer, 2006: 457-495. https: //link. springer. com/referenceworkentry/10. 1007%2F0-387-30742-7_16 |
| [10] | Könneke M, Bernhard A E, de la Torre J R, et al. Isolation of an autotrophic ammonia-oxidizing marine archaeon[J]. Nature, 2005, 437(7058): 543-546. DOI:10.1038/nature03911 |
| [11] |
郑平, 冯孝善. 硝化作用的生化原理[J]. 微生物学通报, 1999, 26(3): 215-217. Zheng P, Feng X S. Biochemical principles of nitrification[J]. Microbiology China, 1999, 26(3): 215-217. |
| [12] | Costa E, Pérez J, Kreft J U. Why is metabolic labour divided in nitrification?[J]. Trends in Microbiology, 2006, 14(5): 213-219. DOI:10.1016/j.tim.2006.03.006 |
| [13] | van Kessel M A H J, Speth D R, Albertsen M, et al. Complete nitrification by a single microorganism[J]. Nature, 2015, 528(7583): 555-559. DOI:10.1038/nature16459 |
| [14] | Daims H, Lebedeva E V, Pjevac P, et al. Complete nitrification by Nitrospira bacteria[J]. Nature, 2015, 528(7583): 504-509. DOI:10.1038/nature16461 |
| [15] | Pinto A J, Marcus D N, Ijaz U Z, et al. Metagenomic evidence for the Presence of Comammox Nitrospira-like bacteria in a drinking water system[J]. Msphere, 2016, 1(1): e00054-15. |
| [16] | Martikainen P J. Nitrification in two coniferous forest soils after different fertilization treatments[J]. Soil Biology and Biochemistry, 1984, 16(6): 577-582. DOI:10.1016/0038-0717(84)90075-0 |
| [17] | Martikainen P J. Nitrification in forest soil of different pH as affected by urea, ammonium sulphate and potassium sulphate[J]. Soil Biology and Biochemistry, 1985, 17(3): 363-367. DOI:10.1016/0038-0717(85)90074-4 |
| [18] |
佟德利, 徐仁扣. 三种氮肥对红壤硝化作用及酸化过程影响的研究[J]. 植物营养与肥料学报, 2012, 18(4): 853-859. Tong D L, Xu R K. Effects of urea, (NH4)2SO4 and NH4HCO3 on nitrification and acidification of a red soil[J]. Plant Nutrition and Fertilizer Science, 2012, 18(4): 853-859. |
| [19] | 鲁如坤. 土壤农业化学分析方法[M]. 北京: 中国农业科技出版社, 2000. |
| [20] | Beman J M, Francis C A. Diversity of ammonia-oxidizing archaea and bacteria in the sediments of a hypernutrified subtropical estuary:Bahía del Tóbari, Mexico[J]. Applied and Environmental Microbiology, 2006, 72(12): 7767-7777. DOI:10.1128/AEM.00946-06 |
| [21] | Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker:molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied & Environmental Microbiology, 1997, 63: 4704-4712. |
| [22] | Pjevac P, Schauberger C, Poghosyan L, et al. AmoA-targeted polymerase chain reaction primers for the specific detection and quantification of comammox Nitrospira in the environment[J]. Frontiers in Microbiology, 2017, 8: 1508. DOI:10.3389/fmicb.2017.01508 |
| [23] | Palomo A, Pedersen A G, Fowler S J, et al. Comparative genomics sheds light on niche differentiation and the evolutionary history of comammox Nitrospira[J]. The ISME Journal, 2018, 12: 1779-1793. DOI:10.1038/s41396-018-0083-3 |
| [24] | Zhang L M, Hu H W, Shen J P, et al. Ammonia-oxidizing archaea have more important role than ammonia-oxidizing bacteria in ammonia oxidation of strongly acidic soils[J]. The ISME Journal, 2012, 6(5): 1032-1045. DOI:10.1038/ismej.2011.168 |
| [25] | Xia W W, Zhang C X, Zeng X W, et al. Autotrophic growth of nitrifying community in an agricultural soil[J]. The ISME Journal, 2011, 5(7): 1226-1236. DOI:10.1038/ismej.2011.5 |
| [26] | Verhamme D T, Prosser J I, Nicol G W. Ammonia concentration determines differential growth of ammonia-oxidising archaea and bacteria in soil microcosms[J]. The ISME Journal, 2011, 5(6): 1067-1071. DOI:10.1038/ismej.2010.191 |
| [27] | Jiang X J, Hou X Y, Zhou X, et al. pH regulates key players of nitrification in paddy soils[J]. Soil Biology and Biochemistry, 2015, 81: 9-16. DOI:10.1016/j.soilbio.2014.10.025 |
| [28] |
闫小娟. 三种紫色土硝化作用及其硝化微生物的研究[D]. 重庆: 西南大学, 2016. Yan X J. Research on nitrification and nitrifying microoganisms of three purple soils[D]. Chongqing: Southwest University, 2016. http: //cdmd. cnki. com. cn/Article/CDMD-10635-1016767218. htm |
| [29] | Kits K D, Sedlacek C J, Lebedeva E V, et al. Kinetic analysis of a complete nitrifier reveals an oligotrophic lifestyle[J]. Nature, 2017, 549(7671): 269-272. DOI:10.1038/nature23679 |
| [30] | Kageyama H, Tripathi K, Rai A K, et al. An alkaline phosphatase/phosphodiesterase, PhoD, induced by salt stress and secreted out of the cells of Aphanothece halophytica, a halotolerant cyanobacterium[J]. Applied and Environmental Microbiology, 2011, 77(15): 5178-5183. DOI:10.1128/AEM.00667-11 |
| [31] | Shen Y C, Hu Y N, Shaw G C. Expressions of alkaline phosphatase genes during phosphate starvation are under positive influences of multiple cell wall hydrolase genes in Bacillus subtilis[J]. Journal of General and Applied Microbiology, 2016, 62(2): 106-109. DOI:10.2323/jgam.62.106 |
| [32] | Lupwayi N Z, Lafond G P, Ziadi N, et al. Soil microbial response to nitrogen fertilizer and tillage in barley and corn[J]. Soil and Tillage Research, 2012, 118: 139-146. DOI:10.1016/j.still.2011.11.006 |
| [33] | Sradnick A, Murugan R, Oltmanns M, et al. Changes in functional diversity of the soil microbial community in a heterogeneous sandy soil after long-term fertilization with cattle manure and mineral fertilizer[J]. Applied Soil Ecology, 2013, 63: 23-28. DOI:10.1016/j.apsoil.2012.09.011 |
| [34] | Chu H Y, Fujii T, Morimoto S, et al. Community structure of ammonia-oxidizing bacteria under long-term application of mineral fertilizer and organic manure in a sandy loam soil[J]. Applied and Environmental Microbiology, 2007, 73(2): 485-491. DOI:10.1128/AEM.01536-06 |
| [35] | Wertz S, Leigh A K, Grayston S J. Effects of long-term fertilization of forest soils on potential nitrification and on the abundance and community structure of ammonia oxidizers and nitrite oxidizers[J]. FEMS Microbiology Ecology, 2012, 79(1): 142-154. DOI:10.1111/j.1574-6941.2011.01204.x |
| [36] | Zhou Z F, Shi X J, Zheng Y, et al. Abundance and community structure of ammonia-oxidizing bacteria and archaea in purple soil under long-term fertilization[J]. European Journal of Soil Biology, 2014, 60: 24-33. DOI:10.1016/j.ejsobi.2013.10.003 |
| [37] | 王欣丽. 酸性红壤氨氧化微生物群落分布和功能及其驱动因子[D]. 南京: 南京师范大学, 2014. http: //cdmd. cnki. com. cn/Article/CDMD-10319-1015423749. htm |
| [38] |
王沛譞, 徐焱, 宋亚娜. 转基因水稻秸秆还田对土壤硝化反硝化微生物群落的影响[J]. 中国生态农业学报, 2018, 26(1): 8-15. Wang P X, Xu Y, Song Y N. Effect of transgenic rice straw return to soil on nitrification and denitrification microbial community[J]. Chinese Journal of Eco-Agriculture, 2018, 26(1): 8-15. |
| [39] | Wang B Z, Zhao J, Guo Z Y, et al. Differential contributions of ammonia oxidizers and nitrite oxidizers to nitrification in four paddy soils[J]. The ISME Journal, 2015, 9(5): 1062-1075. DOI:10.1038/ismej.2014.194 |
 2018, Vol. 39
2018, Vol. 39


