2. 北京大学环境科学与工程学院, 北京 100871;
3. 浙江清华长三角研究院生态环境研究所, 浙江省水质科学与技术重点实验室, 嘉兴 314006;
4. 江南大学环境与土木工程学院, 无锡 214122
2. College of Environmental Sciences and Engineering, Peking University, Beijing 100871, China;
3. Zhejiang Provincial Key Laboratory of Water Science and Technology, Department of Environmental Technology and Ecology, Yangtze Delta Region Institute of Tsinghua University, Jiaxing 314006, China;
4. School of Environment and Civil Engineering, Jiangnan University, Wuxi 214122, China
由于抗生素的广泛使用乃至滥用, 环境中残留的抗生素对自然环境中的微生物群落产生了选择性压力, 使得抗生素抗性基因(antibiotic resistance genes, ARGs)在环境中持久存在并广泛传播.目前ARGs已经成为了学术界的研究热点[1~3], 大量研究表明ARGs广泛存在于自然水环境中, 如河流[4, 5]、湖泊[6, 7]乃至海洋[8, 9].废水处理系统(wastewater treatment plants, WWTPs)是污染治理体系中的一个重要环节, 它汇集了人类生活和生产产生的大量生活污水、医疗污水和工业废水等, 被认为是自然水环境中ARGs污染的重要点源[10], 且促进了ARGs的诱导和迁移[11], 因此, ARGs在不同类型的废水处理系统中的分布特征得到了广泛关注.制药废水处理系统(pharmaceutical WWTPs, PWWTPs)的进水中往往含有高浓度的抗生素及生产原料和中间体[12], 对生物处理单元的微生物群落形成高强度选择性压力, 为ARGs在污水中的持久存在和传播扩散提供了有利条件, 一些研究证实制药废水处理系统的出水中ARGs的浓度高于市政污水处理系统的出水[13, 14].制药废水处理后往往排入下水道, 与其他污水(主要包括未经处理的生活污水和经过处理的其它工业废水)合并后再进入综合性废水处理系统(integrated WWTPs, IWWTPs), 进行统一处理后再排入水环境中.因此, 为了保护自然水生态安全, ARGs在制药废水和综合性两级废水处理系统中的分布特征和去除效率应给予高度关注.
本研究选取了浙江省某精细化工园区内一家药业集团的制药废水处理系统和接纳该制药废水处理系统出水的综合性废水处理系统, 沿两级废水处理工艺流程采集污水和污泥样品, 以8类共28种ARGs作为目标基因进行定性检测, 并对高频率检出的7种ARGs和16S rRNA进行定量检测, 分析ARGs在具有上、下游关系的两座废水处理系统中的分布特征和去除效果, 探索ARGs的去除机制.
1 材料与方法 1.1 两级废水处理系统简介选取浙江省某精细化工园区的两座废水处理系统为研究对象, 它们分别是药业集团的废水处理系统(PWWTP)和园区综合性废水处理系统(IWWTP).该药业集团是化学合成医药原料和兽药原料的生产商, 主导产品为喹诺酮类抗菌药、大环内酯类抗菌药等抗生素类药物, 其中盐酸环丙沙星、恩诺沙星、阿奇霉素、克拉霉素、罗红霉素等产品在全球市场上占据优势地位.该公司产生的废水是典型的制药废水, 有机污染物浓度高(COD可达5000 mg ·L-1), 水质波动大, 可生化性较差.该公司的废水处理系统采用A2O工艺, 设计处理能力为2400 m3 ·d-1, 实际处理水量约为1000 m3 ·d-1.制药废水经处理后排入园区下水道, 与园区其它工业废水及周边生活污水混合(工业废水和生活污水的比例约为6 :4), 再进入园区综合性废水处理系统, 该处理系统主体工艺为氧化沟, 分厌氧段、缺氧段和好氧段运行, 设计处理能力为22.5万m3 ·d-1, 目前实际处理水量约为11.5万m3 ·d-1, 处理后出水排入临近海域.
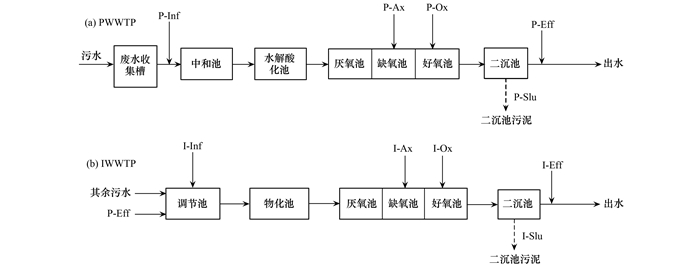
1.2 样品采集及预处理本研究的采样时间是2015年4月, 两座废水处理系统的工艺流程示意图和采样点见图 1, 采样点具体包括:进水(P-Inf和I-Inf)、缺氧池出水(P-Ax和I-Ax)、好氧池出水(P-Ox和I-Ox)和二沉池出水(P-Eff和I-Eff).厌氧池出水由于取样条件限制未取得.此外, 还采集了二沉池污泥样品(P-Slu和I-Slu).每个采样点采集了1 L水样, 并存放于预先清洗干净的无菌聚乙烯塑料瓶中, 冷藏运输回实验室后置于4℃保存.污泥样品存放于无菌的塑料袋中, 冷藏运输回实验室后置于-20℃保存.
|
图 1 两座废水处理系统工艺流程和采样点示意 Fig. 1 Treatment processes and sampling sites of the two WWTPs |
水样中DNA的收集采用0.22 μm混合纤维素酯滤膜(Millipore, 德国)过滤截留方法, 污泥样品则称取约0.2 g转移到洁净的收集管中, 随后二者均使用PowerSoil DNA Isolation Kit (Mo Bio, 美国)试剂盒进行DNA提取.提取完成后, 使用质量浓度为1%的琼脂糖凝胶电泳和Nanodrop 2000 (Thermo Fisher, 美国)对DNA浓度和纯度进行检测.
1.4 普通PCR检测本研究选择了8类共27种ARGs进行定性检测, 具体包括7种四环素类ARGs(tetA、tetB、tetC、tetM、tetO、tetQ、tetW), 4种磺胺类ARGs(sul Ⅰ 、sul Ⅱ 、sul Ⅲ 、sulA), 3种甲氧苄啶ARGs(dfrA 1 、dfrA 12 、dfrA 13 ), 3种β-内酰胺类ARGs(ampC、blaSHV、blaPSE- 1 ), 2种大环内酯类ARGs(ermA、ermB), 3种氯霉素类ARGs(cat Ⅰ 、cat Ⅱ 、floR), 2种氨基糖苷类ARGs[aac(3)-Ⅱ a、aac(3)-Ⅳ ]和3种万古霉素ARGs(vanA、vanB、vanC).所用的引物序列和片段大小见表 1.本实验所使用的引物均由生工生物工程(上海)股份有限公司合成.普通PCR采用25 μL体系, 包含12.5 μL Premix Ex TaqTM Hot Start Version (TaKaRa, 中国); 各1 μL的上、下游引物; 1 μL的DNA模板; 9.5 μL ddH2O. PCR反应程序为: 95℃预变性3 min; 94℃变性30 s, 55℃退火30 s, 72℃延伸60 s, 共35个循环; 最后于72℃延伸10 min. PCR产物置于4℃保存, 并使用质量浓度为1%~2%的琼脂糖凝胶电泳进行检测.
|
|
表 1 PCR引物序列 Table 1 Sequences of premiers in PCR reaction |
1.5 实时荧光定量PCR检测
根据普通PCR定性检测的结果, 本研究对tetO、tetQ、tetW、sul Ⅰ 、sul Ⅱ 、dfrA 13 、floR共7种ARGs及16S rRNA进行定量测定, 所用引物序列同表 1.采用SYBR Green Ⅰ方法、使用实时荧光定量PCR仪(BioRad CFX96 Touch, 美国)对各目标基因进行定量测定.定量PCR采用20 μL体系, 包含10.0 μL SYBR Premix Ex TaqTM (TaKaRa, 中国), 引物各0.4 μL, DNA模板1 μL, ddH2O 8.2 μL.反应程序为: 95℃预变性1~2 min; 95℃变性30 s, 退火30 s, 72℃延伸30 s, 共40个循环.溶解曲线程序为55~95℃, 每0.5℃读数一次, 期间停留30 s.测定时, 将标准质粒以10倍梯度稀释, 再根据质粒浓度计算得到标准质粒的拷贝数, 与通过实时荧光定量PCR测定得到的Ct值对应可做出标准曲线, 标准质粒的构建方法参照张衍等[24]的研究.各目标基因的标准曲线的线性相关系数在0.988~0.999之间.每个样品均设置3个平行样, 以减少误差.
1.6 数据分析使用Excel 2016和Past3.0对数据进行分析, 相关性分析采用Pearson相关方法, P < 0.05(95%置信区间)被认为是显著相关.
2 结果与讨论 2.1 两座废水处理系统进水中ARGs存在情况在27种ARGs中, P-Inf中共检出了10种ARGs, I-Inf中共检出了15种ARGs, 结果如表 2所示.与P-Inf相比, I-Inf中的ARGs种类更为丰富.在检出的ARGs中, 四环素类和磺胺类ARGs的检出较为普遍, 甲氧苄啶类和大环内酯类ARGs有少量检出, 而β-内酰胺类ARGs和万古霉素类ARGs基本没有检出.检测的7种四环素类ARGs包含3种抗性机制为编码外排泵蛋白的tetA、tetB、tetC基因和4种编码核糖体保护蛋白的tetM、tetO、tetQ、tetW基因, 而检出的基因都属于后者, 这说明编码核糖体保护蛋白的四环素类ARGs更有可能存在于废水处理系统中.
|
|
表 2 两座废水处理系统的进水中ARGs检出情况1) Table 2 Presence of ARGs in the influents of two WWTPs |
2.2 两座废水处理系统中ARGs的分布特征
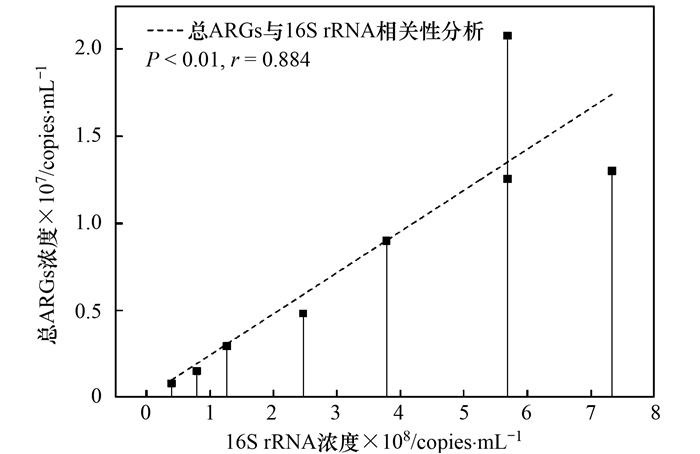
两座废水处理系统采集的样品中目标基因的绝对丰度分布情况如图 2所示.在所有水样中, 16S rRNA浓度为3.94×107~7.34×108 copies ·mL-1, 高出ARGs浓度0.55~4.58个数量级; 除P-Ax以外, 其余生物单元水样中16S rRNA的浓度均高于进水和出水, 说明微生物在生物处理单元有效地增殖.在泥样中, 两座废水处理系统的二沉池污泥中16S rRNA浓度均在1011 copies ·g-1水平, 远高于水样中16S rRNA的浓度水平.有报道指出6个PWWTP水样中16S rRNA主要在105~108 copies ·mL-1的浓度水平[14], 与本研究结果一致.另有研究显示, 在我国杭州、哈尔滨等地的市政污水处理厂的水样中, 16S rRNA在108~1012 copies ·mL-1的浓度水平[25~28], 明显高于制药废水处理系统水样中的浓度水平.这意味着与市政污水相比, 制药废水和工业废水对微生物群落产生了更高的选择性压力, 因此两个废水处理系统中微生物浓度下降.相关性分析结果显示(图 3), 两座废水处理系统水样中16S rRNA浓度与7种ARGs总浓度显著正相关(P < 0.01, r=0.884), 并且与sul Ⅰ (P < 0.05, r=0.818)和sul Ⅱ (P < 0.05, r=0.756)两种磺胺类抗性基因显著正相关, 说明废水水样中ARGs浓度受到微生物浓度变化的影响.

|
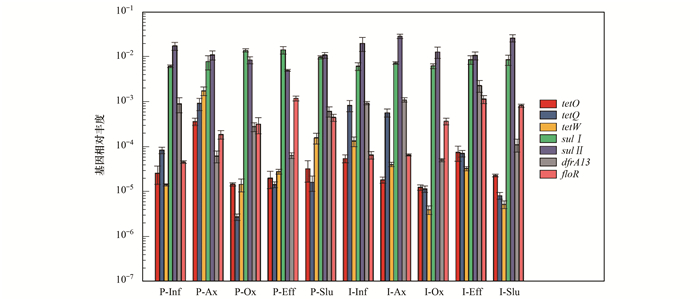
图中P-Slu和I-Slu样点单位为copies ·g-1, 余下样点为copies ·mL-1 图 2 两座废水处理系统中ARGs和16S rRNA的分布 Fig. 2 Concentrations of ARGs and 16S rRNA in the two WWTPs |
|
图 3 废水处理系统水样中16S rRNA与总ARGs相关性 Fig. 3 Correlation between 16S rRNA and the total ARGs in water samples from the two WWTPs |
有研究指出在PWWTP中, 相比于抗生素浓度, 微生物浓度是影响ARGs的更为重要的因素[14].在两座废水处理系统的进水中, ARGs浓度在1.79×103 copies ·mL-1(tetW, P-Inf)~6.14×103 copies ·mL-1(sul Ⅱ , I-Inf)之间, 其中, sul Ⅰ 和sul Ⅱ 基因绝对丰度最高, 随后依次是dfrA 13 、tetQ、floR、tetO和tetW基因.两座系统进水中ARGs相对丰度的分布顺序与绝对丰度一致(图 4), 仍旧是sul Ⅰ 和sul Ⅱ 基因最高, 随后依次是dfrA 13 、tetQ、floR、tetO和tetW基因.有研究显示, sul Ⅰ 和sul Ⅱ 基因以105~107 copies ·mL-1的绝对丰度存在于不同的市政污水处理系统的进水中, 浓度水平高于四环素类ARGs[25, 29, 30], 在制药废水处理系统中也有同样的情况[31, 32], 这与本研究的结果一致.四环素类ARGs中, tetO、tetQ和tetW基因在废水处理系统中的分布较早地得到了关注, 在制药废水处理系统中也有研究[31, 32].本研究对这3种四环素类ARGs的相关性分析显示, tetO基因与tetW基因显著正相关(P < 0.01, r=0.915), 而tetQ基因与tetO(P=0.27, r=0.442)、tetW(P=0.56, r=0.246)基因正相关但不显著, 说明这3种四环素类ARGs在两级废水处理系统中的浓度水平具备相似性, 这可能与它们具备相同的抗性机制有关.与四环素类和磺胺类ARGs不同, 甲氧苄啶抗性基因dfrA 13 尚未有文献报道存在于污水处理系统中.在本研究中, dfrA 13 的绝对丰度水平高于已有研究中检出频率较高的3种四环素类ARGs, 这表明仅着眼于四环素类和磺胺类ARGs等高频率被检出的ARGs类别, 对废水处理系统中ARGs污染状况的评估将是不全面的, 未来建议引入高通量qPCR、宏基因组等新技术进行更加细致全面的研究.
|
图 4 两座废水处理系统中ARGs的相对丰度 Fig. 4 Relative abundances of ARGs in the two WWTPs |
P-Eff汇入I-Inf中, 再由IWWTP进行处理. IWWTP需处理多种来源的生活污水和工业废水, 不同废水对I-Inf中ARGs的贡献不同.每天P-Eff和I-Inf水样的ARGs总量如表 3所示(ARGs总量估算方法:水样中ARGs单位体积浓度系统每日处理水量), 可以看到:每天P-Eff对I-Inf中总ARGs的贡献率为5.05%, 而每天P-Eff的水量仅占I-Inf的0.87%, 说明PWWTP对IWWTP中ARGs污染贡献较大.更进一步地, P-Eff对I-Inf中不同ARGs的贡献率有较大差异, 贡献率最高的为sul Ⅰ 基因, 达9.12%, 其后依次是floR、tetW、sul Ⅱ 、tetO、tetQ, 贡献率最低的是dfrA 13 基因, 仅为0.17%.通过对水样中ARGs物质流的分析, 可以厘清IWWTP中不同来源废水的ARGs贡献率, 为未来有针对性地控制ARGs污染源提供指导.
|
|
表 3 ARGs在两座废水处理系统中的传输 Table 3 ARGs transportation from the PWWTP to IWWTP |
2.3 两座废水处理系统对ARGs的作用
在两座废水处理系统中, 总ARGs浓度在生物处理单元中升高, 经过二沉池的处理后, 出水中的总ARGs浓度下降, 其中ARGs浓度在1.27×103 copies ·mL-1(tetW, P-Eff)~3.31×106 copies ·mL-1(sul Ⅰ , P-Eff)之间.而二沉池污泥中, ARGs浓度在1.24×106 copies ·g-1(tetW, I-Slu)~6.19×109 copies ·g-1(sul Ⅱ , I-Slu)之间, ARGs浓度高于出水3~4个数量级(图 2), 这与文献[25, 33]结果一致.表明生物处理单元有可能促进ARGs的扩增和传播; 大量的ARGs从水中转移到了泥中, 表明二沉池的污泥沉降作用可能是去除污水中ARGs的主要机制.
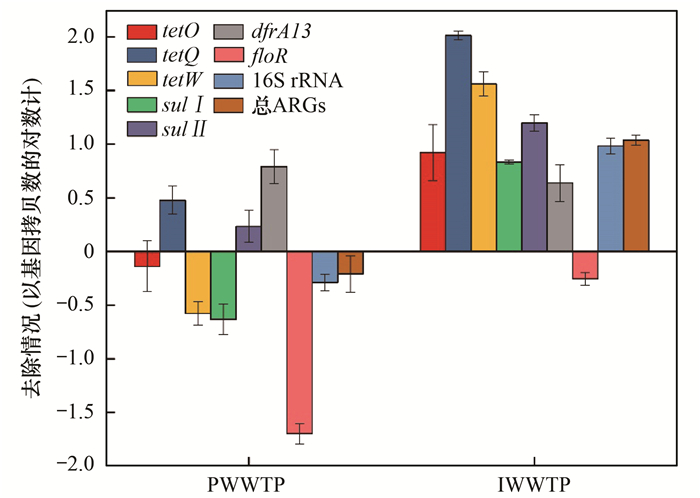
废水处理系统对ARGs能否有效去除目前尚有争议.在中国北方的两座污水处理厂中, ARGs被证实得到了明显的扩增和传播[26], 而在意大利的3座污水处理厂中, 大多数ARGs能够被显著地去除[34].在本研究中, 两座废水处理系统对ARGs的去除同样表现出了差异(图 5).总体来看, 经PWWTP处理后ARGs浓度上升了0.21个数量级, 未表现出对ARGs的去除效果, 经IWWTP处理后总ARGs浓度下降了1.03个数量级, 表明PWWTP对ARGs的去除效果相对较弱.进一步分析不同基因, PWWTP仅对tetQ、sul Ⅱ 和dfrA 13 基因有去除作用, 分别去除了0.48、0.23和0.79个数量级; 而IWWTP对除了floR外的基因均表现出了去除效果, 对tetO、tetQ、tetW、sul Ⅰ 、sul Ⅱ 、dfrA 13 以及16S rRNA分别去除了0.92、2.01、1.56、0.83、1.20、0.64及0.98个数量级. IWWTP对四环素类ARGs表现出较好的去除效果, 这与浙江省[27]、哈尔滨市[25]、江苏省[30]多地的市政污水处理厂中的情况类似.在3种四环素类ARGs中, PWWTP和IWWTP对tetQ的去除效果最好, 这可能与tetQ基因在水环境中的迁移机制有关.有研究表明在这3种四环素类ARGs中, tetQ的迁移率最高[35], 推测其能够快速地迁移到活性污泥中, 并随着污泥沉降作用离开水体而得到去除.相比较之下, 磺胺类ARGs较难被废水处理系统去除, 推测可能与这两种基因均位于高效的水平转移元件上有关[36].经PWWTP和IWWTP处理后, 氯霉素类抗性基因floR的浓度分别上升了1.70和0.25个数量级, 由图 2可以看出, 相较于进水, floR基因浓度在好氧池出水中分别上升了1.12和1.05个数量级, 显示其能够在好氧环境中广泛地扩增和传播, 因此, floR基因在废水处理系统中的分布值得重点关注.有研究关注了浙江省四座市政污水处理系统对ARGs的去除情况, 发现其对tetO、tetQ、tetW基因去除了2.0~3.5个数量级, 对sul Ⅰ 、sul Ⅱ 去除了1~2.5个数量级[27], 去除效果优于本研究中的两座废水处理系统, 这意味着与市政污水处理系统相比, 如何去除制药废水处理系统中的ARGs需要更多的关注.在作者团队后期对该药业集团废水处理系统的调研中, 分别在系统进水、水解酸化池出水和好氧池出水中检出了浓度为5.06、4.88和3.23 mg ·L-1的盐酸环丙沙星[37], 表明废水处理系统中存在高浓度的抗生素, 对微生物群落造成较大的抗生素选择压力, 很可能导致系统中出现高丰度的ARGs, 并使得系统中的ARGs较难被去除.
|
图 5 两座废水处理系统对ARGs和16S rRNA的去除 Fig. 5 Removal of ARGs and 16S rRNA by the two WWTPs |
本研究中的制药废水经PWWTP和IWWTP两级处理后, 最终出水(I-Eff)将排入临近海域, 可能对受纳水体的微生物群落产生影响, 引起抗生素抗性的蔓延和传播, 进而对人类健康产生潜在风险.此外, 高浓度的ARGs进入二沉池污泥中, 可能通过污泥处理和处置过程而在土壤环境中引起ARGs的蔓延和传播.废水处理系统中常见的A2O、氧化沟等工艺是为去除废水中的常规污染物而设计, 对ARGs没有良好的去除效果在情理之中.为减少经废水处理系统排放至自然环境的ARGs的总量, 在保证常规污染物去除效率的基础上, 强化ARGs的高效去除, 将是未来废水处理技术发展需要应对的挑战.
3 结论(1) 在浙江省某精细化工园区的两座废水处理系统进水中, 分别检出了10种和15种ARGs, 其中高频率检出的是四环素类和磺胺类ARGs; 磺胺类ARGs的绝对丰度最高, 为105~106 copies ·mL-1, 随后浓度由高到低依次为dfrA 13 、tetQ、floR、tetO和tetW基因; 首次检出dfrA 13 抗性基因, 且其绝对丰度高于四环素类ARGs.
(2) 两座废水处理系统的水样中16S rRNA浓度低于市政污水处理系统的浓度水平, 并与7种ARGs的总浓度、sul Ⅰ 、sul Ⅱ 基因浓度显著正相关.
(3) 制药废水处理系统出水仅占园区综合性废水处理系统总水量的0.87%, 而总ARGs的贡献率为5.05%, 传播输出最高的3种ARGs为sul Ⅰ 、floR和tetW基因.
(4) 制药废水处理系统使废水中总ARGs浓度上升0.21个数量级, 综合性废水处理系统使废水中总ARGs浓度下降1.03个数量级; 最终出水直接排海, 可能对近海微生物群落产生影响, 应进一步评估ARGs通过废水处理系统进入环境的浓度水平和健康效应.
| [1] | Ferri M, Ranucci E, Romagnoli P, et al. Antimicrobial resistance:a global emerging threat to public health systems[J]. Critical Reviews in Food Science and Nutrition, 2017, 57(13): 2857-2876. DOI:10.1080/10408398.2015.1077192 |
| [2] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants:studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [3] |
黄福义, 李虎, 安新丽, 等. 城市生活污水和生活垃圾渗滤液抗生素抗性基因污染的比较研究[J]. 环境科学, 2016, 37(10): 3949-3954. Huang F Y, Li H, An X L, et al. Comparative investigation of antibiotic resistance genes between wastewater and landfill leachate[J]. Environmental Science, 2016, 37(10): 3949-3954. |
| [4] | Jiang L, Hu X L, Xu T, et al. Prevalence of antibiotic resistance genes and their relationship with antibiotics in the Huangpu River and the drinking water sources, Shanghai, China[J]. Science of the Total Environment, 2013, 458-460: 267-272. DOI:10.1016/j.scitotenv.2013.04.038 |
| [5] | Ouyang W Y, Huang F Y, Zhao Y, et al. Increased levels of antibiotic resistance in urban stream of Jiulongjiang River, China[J]. Applied Microbiology and Biotechnology, 2015, 99(13): 5697-5707. DOI:10.1007/s00253-015-6416-5 |
| [6] | Czekalski N, Sigdel R, Birtel J, et al. Does human activity impact the natural antibiotic resistance background? Abundance of antibiotic resistance genes in 21 Swiss lakes[J]. Environment International, 2015, 81: 45-55. DOI:10.1016/j.envint.2015.04.005 |
| [7] | Yang Y Y, Cao X H, Lin H, et al. Antibiotics and antibiotic resistance genes in sediment of Honghu Lake and East Dongting Lake, China[J]. Microbial Ecology, 2016, 72(4): 791-801. DOI:10.1007/s00248-016-0814-9 |
| [8] | Na G S, Fang X D, Cai Y Q, et al. Occurrence, distribution, and bioaccumulation of antibiotics in coastal environment of Dalian, China[J]. Marine Pollution Bulletin, 2013, 69(1-2): 233-237. DOI:10.1016/j.marpolbul.2012.12.028 |
| [9] | Niu Z G, Zhang K, Zhang Y. Occurrence and distribution of antibiotic resistance genes in the coastal area of the Bohai Bay, China[J]. Marine Pollution Bulletin, 2016, 107(1): 245-250. DOI:10.1016/j.marpolbul.2016.03.064 |
| [10] | Czekalski N, Díez E G, Burgmann H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake[J]. The ISME Journal, 2014, 8(7): 1381-1390. DOI:10.1038/ismej.2014.8 |
| [11] | Rizzo L, Manaia C, Merlin C, et al. Urban wastewater treatment plants as hotspots for antibiotic resistant bacteria and genes spread into the environment:a review[J]. Science of the Total Environment, 2013, 447: 345-360. DOI:10.1016/j.scitotenv.2013.01.032 |
| [12] | Aydin S, Ince B, Ince O. Development of antibiotic resistance genes in microbial communities during long-term operation of anaerobic reactors in the treatment of pharmaceutical wastewater[J]. Water Research, 2015, 83: 337-344. DOI:10.1016/j.watres.2015.07.007 |
| [13] | Liu M M, Zhang Y, Yang M, et al. Abundance and distribution of tetracycline resistance genes and mobile elements in an oxytetracycline production wastewater treatment system[J]. Environmental Science & Technology, 2012, 46(14): 7551-7557. |
| [14] | Guo X Y, Yan Z, Zhang Y, et al. Behavior of antibiotic resistance genes under extremely high-level antibiotic selection pressures in pharmaceutical wastewater treatment plants[J]. Science of the Total Environment, 2018, 612: 119-128. DOI:10.1016/j.scitotenv.2017.08.229 |
| [15] | Maynard C, Fairbrother J M, Bekal S, et al. Antimicrobial resistance genes in enterotoxigenic Escherichia coli O149:K91 isolates obtained over a 23-year period from pigs[J]. Antimicrobial Agents and Chemotherapy, 2003, 47(10): 3214-3221. DOI:10.1128/AAC.47.10.3214-3221.2003 |
| [16] | Aminov R I, Garrigues-Jeanjean N, Mackie R I. Molecular ecology of tetracycline resistance:development and validation of primers for detection of tetracycline resistance genes encoding ribosomal protection proteins[J]. Applied and Environmental Microbiology, 2001, 67(1): 22-32. |
| [17] | Luo Y, Mao D Q, Rysz M, et al. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environmental Science & Technology, 2010, 44(19): 7220-7225. |
| [18] | Ji X L, Shen Q H, Liu F, et al. Antibiotic resistance gene abundances associated with antibiotics and heavy metals in animal manures and agricultural soils adjacent to feedlots in Shanghai; China[J]. Journal of Hazardous Materials, 2012, 235-236: 178-185. DOI:10.1016/j.jhazmat.2012.07.040 |
| [19] | Chen S, Zhao S H, White D G, et al. Characterization of multiple-antimicrobial-resistant Salmonella serovars isolated from retail meats[J]. Applied and Environmental Microbiology, 2004, 70(1): 1-7. |
| [20] | Schwartz T, Kohnen W, Jansen B, et al. Detection of antibiotic-resistant bacteria and their resistance genes in wastewater, surface water, and drinking water biofilms[J]. FEMS Microbiology Ecology, 2003, 43(3): 325-335. DOI:10.1111/fem.2003.43.issue-3 |
| [21] | Strommenger B, Kettlitz C, Werner G, et al. Multiplex PCR assay for simultaneous detection of nine clinically relevant antibiotic resistance genes in Staphylococcus aureus[J]. Journal of Clinical Microbiology, 2003, 41(9): 4089-4094. DOI:10.1128/JCM.41.9.4089-4094.2003 |
| [22] | Stoll C, Sidhu J P S, Tiehm A, et al. Prevalence of clinically relevant antibiotic resistance genes in surface water samples collected from Germany and Australia[J]. Environmental Science & Technology, 2012, 46(17): 9716-9726. |
| [23] | Zhang Y, Chen L J, Sun R H, et al. Effect of wastewater disposal on the bacterial and archaeal community of sea sediment in an industrial area in China[J]. FEMS Microbiology Ecology, 2014, 88(2): 320-332. DOI:10.1111/fem.2014.88.issue-2 |
| [24] |
张衍, 陈吕军, 谢辉, 等. 两座污水处理系统中细胞态和游离态抗生素抗性基因的丰度特征[J]. 环境科学, 2017, 38(9): 3823-3830. Zhang Y, Chen L J, Xie H, et al. Abundance of cell-associated and cell-free antibiotic resistance genes in two wastewater treatment plants[J]. Environmental Science, 2017, 38(9): 3823-3830. |
| [25] | Wen Q X, Yang L, Duan R, et al. Monitoring and evaluation of antibiotic resistance genes in four municipal wastewater treatment plants in Harbin, Northeast China[J]. Environmental Pollution, 2016, 212: 34-40. DOI:10.1016/j.envpol.2016.01.043 |
| [26] | Mao D Q, Yu S, Rysz M, et al. Prevalence and proliferation of antibiotic resistance genes in two municipal wastewater treatment plants[J]. Water Research, 2015, 85: 458-466. DOI:10.1016/j.watres.2015.09.010 |
| [27] | Chen H, Zhang M M. Occurrence and removal of antibiotic resistance genes in municipal wastewater and rural domestic sewage treatment systems in eastern China[J]. Environment International, 2013, 55: 9-14. DOI:10.1016/j.envint.2013.01.019 |
| [28] | Chen H, Zhang M M. Effects of advanced treatment systems on the removal of antibiotic resistance genes in wastewater treatment plants from Hangzhou, China[J]. Environmental Science & Technology, 2013, 47(15): 8157-8163. |
| [29] | Rodríguez-Mozaz S, Chamorro S, Marti E, et al. Occurrence of antibiotics and antibiotic resistance genes in hospital and urban wastewaters and their impact on the receiving river[J]. Water Research, 2015, 69: 234-242. DOI:10.1016/j.watres.2014.11.021 |
| [30] | Du J, Ren H Q, Geng J J, et al. Occurrence and abundance of tetracycline, sulfonamide resistance genes, and class 1 integron in five wastewater treatment plants[J]. Environmental Science and Pollution Research, 2014, 21(12): 7276-7284. DOI:10.1007/s11356-014-2613-5 |
| [31] | Zhai W C, Yang F X, Mao D Q, et al. Fate and removal of various antibiotic resistance genes in typical pharmaceutical wastewater treatment systems[J]. Environmental Science and Pollution Research, 2016, 23(12): 12030-12038. DOI:10.1007/s11356-016-6350-9 |
| [32] | Wang J L, Mao D Q, Mu Q H, et al. Fate and proliferation of typical antibiotic resistance genes in five full-scale pharmaceutical wastewater treatment plants[J]. Science of the Total Environment, 2015, 526: 366-373. DOI:10.1016/j.scitotenv.2015.05.046 |
| [33] | Lee J, Jeon J H, Shin J, et al. Quantitative and qualitative changes in antibiotic resistance genes after passing through treatment processes in municipal wastewater treatment plants[J]. Science of the Total Environment, 2017, 605-606: 906-914. DOI:10.1016/j.scitotenv.2017.06.250 |
| [34] | Cesare A D, Eckert E M, D'Urso S, et al. Co-occurrence of integrase 1, antibiotic and heavy metal resistance genes in municipal wastewater treatment plants[J]. Water Research, 2016, 94: 208-214. DOI:10.1016/j.watres.2016.02.049 |
| [35] | Engemann C A, Keen P L, Knapp C W, et al. Fate of tetracycline resistance genes in aquatic systems:migration from the water column to peripheral biofilms[J]. Environmental Science & Technology, 2008, 42(14): 5131-5136. |
| [36] | Skold O. Resistance to trimethoprim and sulfonamides[J]. Veterinary Research, 2001, 32(3-4): 261-273. |
| [37] | Xie Y W, Chen L J, Liu R. AOX contamination status and genotoxicity of AOX-bearing pharmaceutical wastewater[J]. Journal of Environmental Sciences, 2017, 52: 170-177. DOI:10.1016/j.jes.2016.04.014 |
 2018, Vol. 39
2018, Vol. 39


