2. 中国科学院南京地理与湖泊研究所湖泊与环境国家重点实验室, 南京 210008;
3. 江苏省太湖渔业管理委员会办公室, 苏州 215004
2. State Key Laboratory of Lake Science and Environment, Nanjing Institute of Geography and Limnology, Chinese Academy of Sciences, Nanjing 210008, China;
3. Taihu Fishery Management Committee Office, Suzhou 215004, China
氨氧化作用是在氨氧化微生物的催化下将NH3转化为NO2-的生物过程, 是氮素循环中硝化作用的限速步骤[1, 2].目前发现的能够催化氨氧化作用的微生物主要是氨氧化古菌(ammonia-oxidizing archaea, AOA)和氨氧化细菌(ammonia-oxidizing bacteria, AOB).大量的研究发现氨氧化微生物分布广泛[3, 4], 自然环境中的pH[5]、温度[6]、盐碱度[7]、氨氮浓度[8]和植物根际[9]等都会对氨氧化微生物的群落结构、多样性和丰度造成影响, 并且存在多种因素协同作用的现象[10].
养殖活动会积累大量的氮、磷等营养盐[11], 影响沉积物中的氨氮含量、温度等环境条件, 从而对氨氧化微生物造成影响.海洋渔场的污染会干扰沉积物中的氮循环[12]; 循环养殖系统的生物过滤器[13]中AOA和AOB序列分别归属于Nitrosopumilus和Nitrosospira; 鲑鱼养殖塘表层沉积物中[14]AOB的多样性和丰度显著高于AOA, AOB的优势类群也是Nitrosospira; 上海养殖塘表层沉积物中[15], AOA在数量上更占优势, AOA和AOB的优势类群分别为Nitrososphaera和Nitrosomonas; 湖北公安的淡水养殖塘沉积物[16]中AOA是优势的氨氧化微生物, 关键因素是溶解氧.
目前对淡水养殖塘中氨氧化微生物的研究仍然较少, 涉及的养殖塘面积较小且较为封闭, 不同的养殖塘中的氨氧化微生物的群落特征也不尽相同.氨氧化微生物因其特殊功能对养殖系统生态环境有着重要意义, 而位于太湖东南部面积较大的湖湾东太湖, 长期以来都是太湖的主水产养殖区[17], 因此本研究通过实时荧光定量PCR和克隆文库技术, 以amoA基因为标记, 分析养殖区和对照区沉积物中氨氧化微生物群落特征差异, 结合沉积物的理化性质, 探究渔业养殖对湖泊沉积物中氨氧化微生物的影响, 以期为阐明水产养殖对湖泊生态系统中氮素生物地球化学循环的影响提供基础数据.
1 材料与方法 1.1 研究区域选择和样品采集2016年8月, 在东太湖进行采样.研究区域为东太湖实施了鱼虾蟹混养模式的混养区, 对照区设置在距离养殖区3~5 km的范围内, 是历史上从未进行过水产养殖的区域.养殖区和对照区各设置了3个采样点(图 1).使用柱状采泥器(德国HYDRO-BIOS)采集沉积物样品, 每个样点采集3个平行样品, 共计18个样品, 现场切取0~1 cm深度的沉积物装入无菌袋中置于冰上冷藏, 运回实验室后在-20℃下冷冻保存.

|
养殖区样点:HY1、HY3、HY8;对照区样点:DZ1、DZ2、DZ3 图 1 东太湖区域采样点示意 Fig. 1 Sampling sites in Eastern Lake Taihu |
所有沉积物样品经冷冻干燥机(ALPHA1-2, 德国CHRIST)干燥后研磨并保存在-20℃中, pH使用pH计(pHB-4, 上海雷磁)进行测定, 沉积物经2 mol·L-1 KCl溶液浸提后, 利用连续流动分析仪(San++, SKALER, 荷兰)测定亚硝氮(NO2--N)、硝态氮(NO3--N)和氨氮(NH4+-N).总氮(TN)和总磷(TP)在沉积物样品经消解后再进行测定, 有机质(OM)含量应用标准方法进行测定.
1.3 DNA提取沉积物基因组DNA提取自0.25 g沉积物, 使用强力DNA提取试剂盒(MoBio Laboratory, Solana Beach, CA)进行提取, 具体操作依照试剂盒的操作说明书; 提取出的DNA利用0.8%琼脂糖凝胶进行检验后, 再利用生物分光光度计(Eppendorf, Hamburg, German)测定其浓度.
1.4 实时定量PCR扩增实验所用的扩增仪器为IQ5 Thermocycler(RG65HD, Corbett, Australia). AOA和AOB的amoA基因PCR扩增的引物对分别是Arch-amoAF/Arch-amoAR[18]和amoA-1F/amoA-2R[19], 标准曲线利用连续稀释10倍已知浓度的质粒来建立. AOA和AOB的amoA基因的PCR扩增体系均为20 μL, 其中包括10 μL SYBR Premix Ex TaqTM缓冲液(Takara, Japan)、7.4 μL ddH2O、上下游引物各0.8 μL和1 μL DNA模板. AOA和AOB的扩增程序相同, 扩增程序为:95℃预变性3 min; 95℃ 30 s, 53℃ 1 min, 72℃ 20 s, 循环40次; 最后72℃延伸7 min.
1.5 克隆建库与系统发育分析将PCR产物纯化后(Axygen PCR cleanup purification kit, Hangzhou, China), 连接pGEM-T载体(Promega, Madison, WI, USA)转移至大肠杆菌感受态细胞中(DH5, Takara, Japan)进行克隆.将宿主细胞转移至100 μg·mL-1氨苄西林、40 μg·mL-1 X-Gal和24 μg·mL-1 IPTG的LB培养基上培养过夜, 筛选阳性克隆的转化株, 使用载体引物(T7和SP6)鉴定阳性克隆并寄送到上海美吉生物科技有限公司进行DNA测序, 总共构建了12个文库, AOA的6个文库中得到阳性克隆子180个, 得到序列180条, AOB的6个文库中得到阳性克隆子300个, 序列299条.
测序结果先用DNAStar软件包删除载体序列, 并用ClustalX2[20]软件进行多重序列对比, 使用DOTUR软件包划分OTU并计算多样性指数(SChao1和香农多样性指数H′)和文库覆盖率, 用BLAST在GenBank中与已知序列进行比对, 使用MEGA 7.0软件包计算邻接法系统发育树[21].
1.6 数据分析不同区域沉积物的理化指标和氨氧化细菌与古菌的amoA基因拷贝数使用SPSS 22.0进行One-Way ANOVA分析以检验显著性差异.
1.7 序列上传本研究中所获得的所有amoA基因序列均已上传到GenBank基因库中, 获得的古菌amoA基因序列的登录号为MF154410-MF154469和MG700694-MG700783;获得的细菌amoA基因序列的登录号为MF154470-MF154569和MG700784-MG701012.
2 结果与分析 2.1 理化性质根据表 1可知, 养殖区和对照区的pH值相近呈弱碱性; 养殖区表层沉积物中NO2--N、NH4+-N、TN、TP和有机质的含量均显著高于对照区, NO3--N的含量在两个区域的沉积物中无显著性差异; 总体来说, 东太湖的沉积物中营养负荷均比较高, 水产养殖活动会增加沉积物中的营养负荷.
|
|
表 1 不同区域的沉积物理化指标1) Table 1 Characteristics of sediments in different zones |
2.2 沉积物中氨氧化微生物的丰度
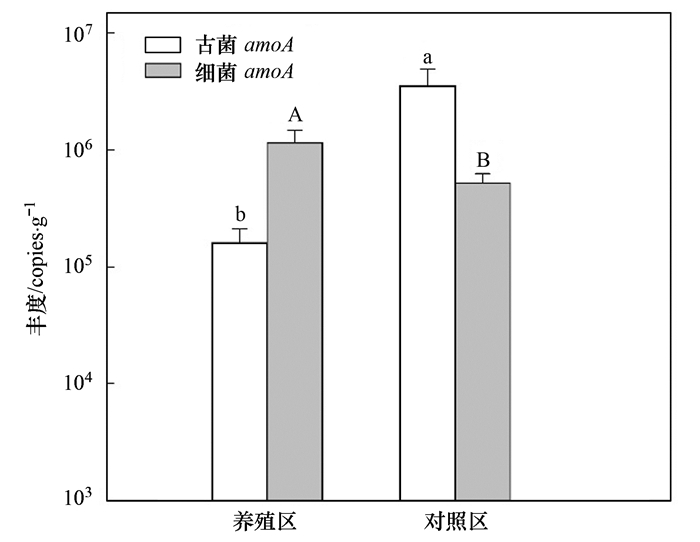
两个区域间氨氧化古菌和细菌的amoA基因拷贝数差异显著(P < 0.01), 养殖区中AOA的丰度低于对照区而AOB的丰度高于对照区; 此外, 养殖区中AOB的丰度高于AOA而对照区中AOB的丰度低于AOA(图 2).
|
图中不同字母表示不同区域同一种氨氧化菌之间amoA基因丰度差异显著(P < 0.01, n=3) 图 2 不同区域沉积物中氨氧化古菌和氨氧化细菌amoA基因丰度 Fig. 2 Abundance of archaeal and bacteria amoA genes in sediments of different zones |
如表 2所示, 所得到的AOA的序列共有180条, AOB的序列共有299条, 每个文库中古菌amoA基因的序列有30条, 细菌amoA基因的序列为50条或49条.养殖区中AOA的OTU数在5~13之间, AOB的OTU数也在5~13之间与AOA相近, 对照区中AOA的OTU数位3~6, AOB的OTU数为9~15之间远大于AOA.联系其他多样性指数(SChao1和香农多样性指数H′)和文库覆盖率的计算结果进行分析可以发现, 一方面, AOA在养殖区的多样性更高而AOB在对照区的多样性更高; 另一方面, 养殖区中AOA和AOB的多样性相近, 而对照区中AOA多样性低于AOB.
|
|
表 2 不同区域沉积物中氨氧化古菌和氨氧化细菌的amoA基因多样性 Table 2 Diversity of archaeal and bacterial amoA sequences in different zones |
2.4 沉积物中氨氧化微生物的群落结构
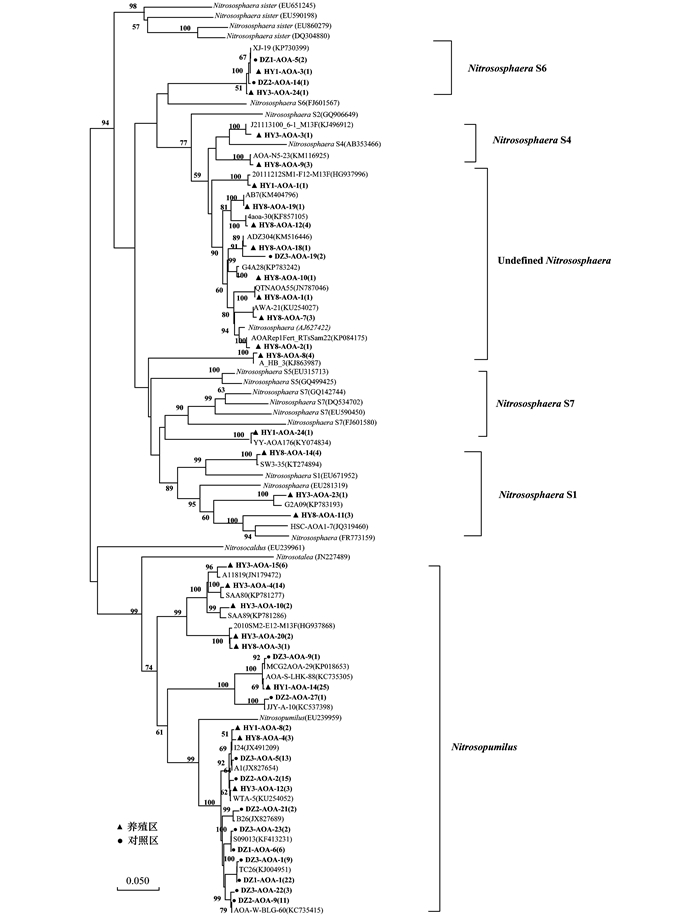
本研究中所获得的AOA与AOB的amoA基因序列所构建系统发育树如图 3和图 4所示.沉积物中古菌的amoA序列划分出较多的类群, 即Nitrososphaera的一些子集群和Nitrosopumilus类群[图 5(a)], 养殖区和对照区中的优势类群均为Nitrosopumilus, 所占比例分别为64.4%和94.4%, 养殖区中Nitrososphaera类群相对丰度更高且分支更具多样性.
|
本研究中所获得的序列加粗显示, 序列名括号中的数字为属同一OTU的序列数, 下同 图 3 不同区域沉积物中氨氧化古菌amoA基因系统发育树 Fig. 3 Phylogenetic analysis of archaeal amoA gene sequences in sediments of different zones |
|
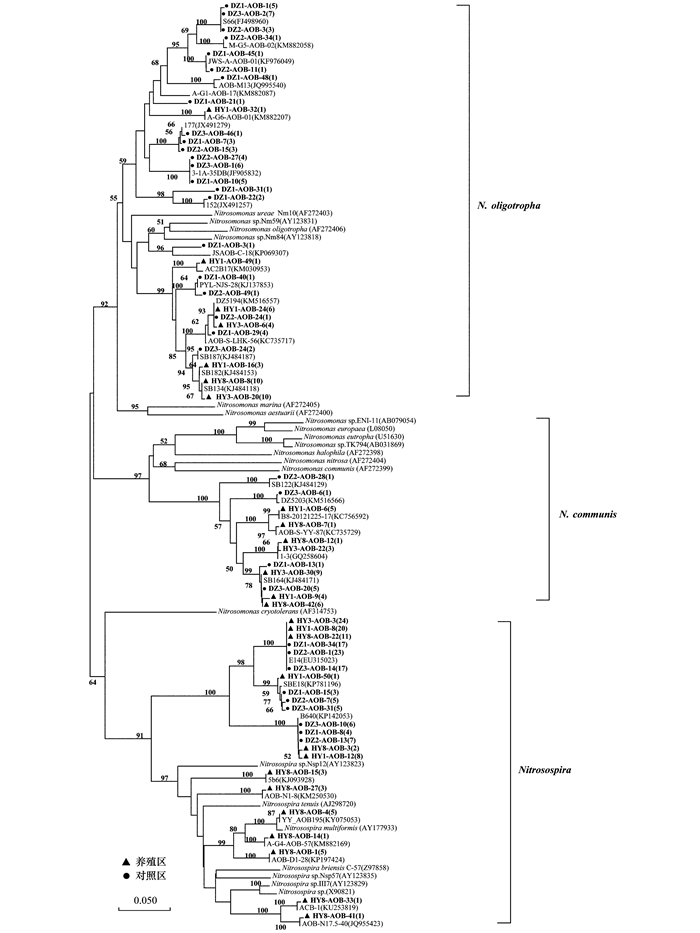
图 4 不同区域沉积物中氨氧化细菌amoA基因系统发育树 Fig. 4 Phylogenetic analysis of bacterial amoA gene sequences in sediments of different zones |

|
图 5 不同区域沉积物中氨氧化微生物类群的相对丰度 Fig. 5 Relative abundance of different archaeal and bacterial amoA clusters in sediments of different zones |
沉积物中细菌的amoA序列划分出3个类群即Nitrosospira、N. communis和N. oligotropha [图 5(b)], 文中所涉及的AOB类群均属于β亚纲(β-Proteobacteria), 均是常见的陆地生态系统中的AOB类群.养殖区和对照区中优势类群均为Nitrosospira, 其在养殖区和对照区所占比例分别为57.06%和58%, 3个类群在两个区域中均有发现且相对关系均为N. oligotropha< N. communis<Nitrosospira [图 5(b)].因此, AOB在养殖区和对照区中有相似的群落组成, 水产养殖对AOB的群落结构没有明显的影响.
3 讨论 3.1 氨氧化微生物的丰度氨氮是氨氧化反应的底物, 一定范围内氨氮含量的增加会促进氨氧化反应和氨氧化微生物的生长, 已有充足的证据表明, 氨氮的含量是划分AOA与AOB生态位的重要因素.有研究表明AOA适合在氨氮含量较低时生长, 高氨氮环境中生长会受到抑制, 而AOB会占优势地位[22~24], 同时, Wu等[25]也证明了太湖沉积物中氨氧化古菌amoA的拷贝数与氨氮含量有着显著性的负相关关系.养殖区的沉积物中丰富的有机质在被异养微生物分解时会消耗大量氧气, 而AOA耐低氧环境, 海洋沉积物AOA的富集培养物对氧气的半饱常数为2.01 μmol·L-1[26, 27]; 对AOB淡水沉积物富集培养发现, 当氧气浓度为0.2~0.8 mg·L-1时, AOB的培养物随着DO浓度降低生长速率下降[8], 在瑞士的寡营养湖泊中, AOA和AOB数量的比值随着深度的增加而增加[28], 因此溶解氧的可利用性也是划分AOA和AOB生态位的重要因素.然而东太湖的平均水深不到1.2 m[17], 沉积物与水的交界面受风浪影响物质交换频繁, 因此, 东太湖沉积物并不是典型的低氧环境, 溶解氧不会对氨氧化微生物的生态位造成决定性的影响.此外, 鲑鱼养殖塘沉积物中AOA的丰度与多样性、间隙水中的氨氮浓度有显著的负相关关系[14], 因此, 东太湖沉积物中氨氧化微生物的丰度在不同区域的差异主要是由氨氮含量的差异造成的.
3.2 氨氧化微生物的多样性一方面不同区域中关于同种氨氧化微生物的比较表明, 养殖区中AOA的多样性更高, 而对照区中AOB的多样性更高.类似的情况在不同营养状态的的苏必利尔湖和伊利湖之间也有发现[29], 富营养化的伊利湖沉积物中AOA的多样性要高于寡营养的苏必利尔湖, 并且AOA的多样性和丰度之间有负相关关系.因此, 养殖区的AOA的多样性更高可能是由于渔业养殖活动导致的湖泊营养程度增加.此外, AOB在两个区域之间的多样性差异则与AOA完全相反, 这可能由于不同的氨氧化微生物对不同环境的适应性差异.另一方面, 通过对比同一区域中不同氨氧化微生物发现, 养殖区中AOA与AOB的多样性相近, 而对照区中AOA多样性低于AOB.在北运河沉积物中[30]和太湖梅梁湾和胥口湾的沉积物中[31]也发现AOA的群落多样性低于AOB, 这与本文对照区中所得到的结果相似; 而在鲑鱼养殖塘沉积物[14]的研究发现, AOB的多样性明显高于AOA, 这样的结果与本研究中养殖区的结果并不相同, 不同的养殖水体环境的沉积物中的氨氧化微生物多样性相对关系可能都有差异, 因此, 关于养殖区沉积物中AOA和AOB多样性仍需更多的后续研究.
3.3 氨氧化微生物的群落结构研究区域沉积物中所获得的AOA序列均属于Nitrososphaera和Nitrosopumilus两个主要类群, Nitrososphaera类群也即是比较熟知的group1.1b分支, 是陆地生态系统中最为常见的类群, Nitrosopumilus是比较熟知的group1.1a类群, 在海洋生态系统中更为常见[32], 而在太湖的竺山湾表层沉积物中[33]和太湖底泥中[25]所获得的序列也均属于Nitrososphaera和Nitrosopumilus两个主要类群.此外, 对照区和养殖区中优势类群均为Nitrosopumilus, Sun等[34]对江苏省8个湖泊沉积物中氨氧化微生物的空间分布进行调查时发现, 所调查的沉积物中的优势类群均为Nitrosopumilus类群, 这一点与本研究中所得结果是相同的, 此外他们只在富营养化湖泊中分离出了归类于Nitrososphaera类群的amoA基因序列, 并且发现间隙水TOC与AOA的群落结构有相关性.因此可能是由于水产养殖导致了养殖区TOC浓度的增加, 从而造成养殖区中Nitrososphaera类群相对比例的增加.
两个区域沉积物中AOB的群落组成很相似, 所有的序列均归类于Nitrosospira、N. communis和N. oligotropha这3个类群, 优势种群均为Nitrosospira, 这与一些养殖塘沉积物[15, 16]中Nitrosomonas类群占优势的情况并不相同, 而Nitrosospira更多是在农田土壤中AOB群落中占优势, 在不同类型、不同利用方式和不同季风带的土壤环境中均有过验证[35], 在淡水湖泊环境中, Nitrosospira占主导的AOB群落结构较为少见, 但在布吉河的水样[36]、浙江省瓯江沉积物[37]和武汉鲑鱼养殖塘沉积物[14]中, 所得到AOB序列大部分或者全部归属于Nitrosospira类群; 主要是环境中物理、化学因素的改变影响了AOB的群落结构分布.
4 结论(1) 渔业养殖区与对照区的沉积物中pH相近呈弱碱性, 渔业养殖区中各组分氮均高于对照区, 养殖区中沉积物营养盐更丰富.
(2) 养殖区中AOB的丰度更高, 对照区中AOA丰度更高, 主要是由于养殖区和对照区的氨氮含量差异, AOA适合在氨氮含量较低时生长, 而在氨氮含量过高的环境中生长会受到抑制, 高氨氮环境中AOB会占优势地位.
(3) AOA在养殖区中的多样性高于对照区, AOB在养殖区中的多样性低于对照区; 养殖区AOA的多样性高于AOB, 对照区中AOA的多样性则低于AOB.
(4) 渔业养殖区和对照区沉积物中AOA的优势类群均为Nitrosopumilus, 养殖区中Nitrososphaera的相对比例大大增加; AOB在两个区域沉积物中群落组成很相似, 优势种群均为Nitrosospira.水产养殖会影响AOA的群落结构, 但对AOB的群落结构则没有明显影响.
| [1] | Beman J M, Francis C A. Diversity of ammonia-oxidizing archaea and bacteria in the sediments of a hypernutrified subtropical estuary:Bahía del Tóbari, Mexico[J]. Applied and Environmental Microbiology, 2006, 72(12): 7767-7777. DOI:10.1128/AEM.00946-06 |
| [2] | Hermansson A, Lindgren P E. Quantification of ammonia-oxidizing bacteria in arable soil by real-time PCR[J]. Applied and Environmental Microbiology, 2001, 67(2): 972-976. |
| [3] | Erguder T H, Boon N, Wittebolle L, et al. Environmental factors shaping the ecological niches of ammonia-oxidizing archaea[J]. FEMS Microbiology Reviews, 2009, 33(5): 855-869. DOI:10.1111/j.1574-6976.2009.00179.x |
| [4] | Wang X Y, Cui W, Bao L L, et al. Abundance and community structure of ammonia-oxidizing microorganisms in reservoir sediment and adjacent soils[J]. Applied Microbiology and Biotechnology, 2014, 98(4): 1883-1892. DOI:10.1007/s00253-013-5174-5 |
| [5] | Nicol G W, Leininger S, Schleper C, et al. The influence of soil pH on the diversity, abundance and transcriptional activity of ammonia oxidizing archaea and bacteria[J]. Environmental Microbiology, 2008, 10(11): 2966-2978. DOI:10.1111/emi.2008.10.issue-11 |
| [6] | Zeng J, Zhao D Y, Yu Z B, et al. Temperature responses of ammonia-oxidizing prokaryotes in freshwater sediment microcosms[J]. PLoS One, 2014, 9(6): e100653. DOI:10.1371/journal.pone.0100653 |
| [7] | Mosier A C, Francis C A. Relative abundance and diversity of ammonia-oxidizing archaea and bacteria in the San Francisco Bay estuary[J]. Environmental Microbiology, 2008, 10(11): 3002-3016. DOI:10.1111/emi.2008.10.issue-11 |
| [8] | French E, Kozlowski J A, Mukherjee M, et al. Ecophysiological characterization of ammonia-oxidizing archaea and bacteria from freshwater[J]. Applied and Environmental Microbiology, 2012, 78(16): 5773-5780. DOI:10.1128/AEM.00432-12 |
| [9] | Huang R, Zhao D Y, Zeng J, et al. Abundance and community composition of ammonia oxidizers in rhizosphere sediment of two submerged macrophytes[J]. Journal of Freshwater Ecology, 2016, 31(3): 407-419. DOI:10.1080/02705060.2016.1187208 |
| [10] | Gubry-Rangin C, Novotnik B, MandiDč-Mulec I, et al. Temperature responses of soil ammonia-oxidising archaea depend on pH[J]. Soil Biology and Biochemistry, 2017, 106: 61-68. DOI:10.1016/j.soilbio.2016.12.007 |
| [11] |
何肖微, 储瑜, 曾巾, 等. 东太湖渔业养殖对沉积物营养盐的影响[J]. 环境科学, 2017, 38(11): 4562-4569. He X W, Chu Y, Zeng J, et al. Effects of optimized fish farming on the sediment nutrients of Eastern Lake Taihu[J]. Environmental Science, 2017, 38(11): 4562-4569. |
| [12] | McCaig A E, Phillips C J, Stephen J R, et al. Nitrogen cycling and community structure of proteobacterial β-subgroup ammonia-oxidizing bacteria within polluted marine fish farm sediments[J]. Applied and Environmental Microbiology, 1999, 65(1): 213-220. |
| [13] | Sakami T, Andoh T, Morita T, et al. Phylogenetic diversity of ammonia-oxidizing archaea and bacteria in biofilters of recirculating aquaculture systems[J]. Marine Genomics, 2012, 7: 27-31. DOI:10.1016/j.margen.2012.04.006 |
| [14] | Zhou Z J, Li H, Song C L, et al. Prevalence of ammonia-oxidizing bacteria over ammonia-oxidizing archaea in sediments as related to nutrient loading in Chinese aquaculture ponds[J]. Journal of Soils and Sediments, 2017, 17(7): 1928-1938. DOI:10.1007/s11368-017-1651-2 |
| [15] | Lu S M, Liu X G, Ma Z J, et al. Vertical segregation and phylogenetic characterization of ammonia-oxidizing bacteria and archaea in the sediment of a freshwater aquaculture pond[J]. Frontiers in Microbiology, 2016, 6: 1539. |
| [16] | Lu S M, Liao M J, Xie C X, et al. Seasonal dynamics of ammonia-oxidizing microorganisms in freshwater aquaculture ponds[J]. Annals of Microbiology, 2015, 65(2): 651-657. DOI:10.1007/s13213-014-0903-2 |
| [17] |
谷孝鸿, 王晓蓉, 胡维平. 东太湖渔业发展对水环境的影响及其生态对策[J]. 上海环境科学, 2003, 22(10): 702-704, 711. Gu X H, Wang X R, Hu W P. Effect of fishery development on water environment and its eco-countermeasure in East lake Taihu[J]. Shanghai Environmental Sciences, 2003, 22(10): 702-704. |
| [18] | Francis C A, Roberts K J, Beman J M, et al. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(41): 14683-14688. DOI:10.1073/pnas.0506625102 |
| [19] | Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker:molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712. |
| [20] | Thompson J D, Gibson T J, Plewniak F, et al. The Clustal_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 1997, 25(24): 4876-4882. DOI:10.1093/nar/25.24.4876 |
| [21] | Altschul S F, Madden T L, Schäffer A A, et al. Gapped blast and PSI-BLAST:a new generation of protein database search programs[J]. Nucleic Acids Research, 1997, 25(17): 3389-3402. DOI:10.1093/nar/25.17.3389 |
| [22] | Hatzenpichler R, Lebedeva E V, Spieck E, et al. A moderately thermophilic ammonia-oxidizing crenarchaeote from a hot spring[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(6): 2134-2139. DOI:10.1073/pnas.0708857105 |
| [23] | Di H J, Cameron K C, Sherlock R R, et al. Nitrous oxide emissions from grazed grassland as affected by a nitrification inhibitor, dicyandiamide, and relationships with ammonia-oxidizing bacteria and archaea[J]. Journal of Soils and Sediments, 2010, 10(5): 943-954. DOI:10.1007/s11368-009-0174-x |
| [24] | Verhamme D T, Prosser J I, Nicol G W. Ammonia concentration determines differential growth of ammonia-oxidising archaea and bacteria in soil microcosms[J]. The ISME Journal, 2011, 5(6): 1067-1071. DOI:10.1038/ismej.2010.191 |
| [25] | Wu Y C, Xiang Y, Wang J J, et al. Heterogeneity of archaeal and bacterial ammonia-oxidizing communities in Lake Taihu, China[J]. Environmental Microbiology Reports, 2010, 2(4): 569-576. DOI:10.1111/emi4.2010.2.issue-4 |
| [26] | Park B J, Park S J, Yoon D N, et al. Cultivation of autotrophic ammonia-oxidizing archaea from marine sediments in coculture with sulfur-oxidizing bacteria[J]. Applied and Environmental Microbiology, 2010, 76(22): 7575-7587. DOI:10.1128/AEM.01478-10 |
| [27] | Lam P, Lavik G, Jensen M M, et al. Revising the nitrogen cycle in the Peruvian oxygen minimum zone[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(12): 4752-4757. DOI:10.1073/pnas.0812444106 |
| [28] | Vissers E W, Anselmetti F S, Bodelier P L E, et al. Temporal and spatial coexistence of archaeal and bacterial amoA genes and gene transcripts in Lake Lucerne[J]. Archaea, 2013, 2013: 289478. |
| [29] | Bollmann A, Bullerjahn G S, McKay R M. Abundance and diversity of ammonia-oxidizing archaea and bacteria in sediments of trophic end members of the Laurentian Great Lakes, Erie and Superior[J]. PLoS One, 2014, 9(5): e97068. DOI:10.1371/journal.pone.0097068 |
| [30] |
鲍林林, 陈永娟, 王晓燕. 北运河沉积物中氨氧化微生物的群落特征[J]. 中国环境科学, 2015, 35(1): 179-189. Bao L L, Chen Y J, Wang X Y. Diversity and abundance of ammonia-oxidizing prokaryotes in surface sediments in Beiyun River[J]. China Environmental Science, 2015, 35(1): 179-189. |
| [31] | Zhao D Y, Zeng J, Wan W H, et al. Vertical distribution of ammonia-oxidizing archaea and bacteria in sediments of a eutrophic lake[J]. Current Microbiology, 2013, 67(3): 327-332. DOI:10.1007/s00284-013-0369-7 |
| [32] | Pester M, Rattei T, Flechl S, et al. amoA-based consensus phylogeny of ammonia-oxidizing archaea and deep sequencing of amoA genes from soils of four different geographic regions[J]. Environmental Microbiology, 2012, 14(2): 525-539. DOI:10.1111/j.1462-2920.2011.02666.x |
| [33] |
向燕, 吴宇澄, 刘国锋, 等. 太湖竺山湾沉积物中氨氧化原核生物的垂直分布与多样性[J]. 生态学报, 2010, 30(6): 1423-1430. Xiang Y, Wu Y C, Liu G F, et al. Vertical distribution and diversity of ammonia-oxidizing prokaryotes in a sediment core from Zhushan Bay, Lake Taihu[J]. Acta Ecologica Sinica, 2010, 30(6): 1423-1430. |
| [34] | Sun X, Wang A L, Yang L Y, et al. Spatial distribution of ammonia-oxidizing archaea and bacteria across eight freshwater lakes in sediments from Jiangsu of China[J]. Journal of Limnology, 2014, 73(2): 312-324. |
| [35] |
汪峰, 曲浩丽, 丁玉芳, 等. 三种农田土壤中氨氧化细菌amoA基因多样性比较分析[J]. 土壤学报, 2012, 49(2): 347-353. Wang F, Qu H L, Ding Y F, et al. Comparative analysis of ammonia monooxygenase (amoA) gene diversity in three cultivated soils[J]. Acta Pedologica Sinica, 2012, 49(2): 347-353. |
| [36] |
孙海美, 白姣姣, 孙卫玲, 等. 布吉河丰水期总细菌和氨氧化细菌的定性和定量研究[J]. 环境科学, 2012, 33(8): 2691-2700. Sun H M, Bai J J, Sun W L, et al. Quantitative and qualitative analysis of total bacteria and ammonia-oxidizing bacteria in Buji River in Wet Season[J]. Environmental Science, 2012, 33(8): 2691-2700. |
| [37] |
李虎, 黄福义, 苏建强, 等. 浙江省瓯江氨氧化古菌和氨氧化细菌分布及多样性特征[J]. 环境科学, 2015, 36(12): 4659-4666. Li H, Huang F Y, Su J Q, et al. Distribution and diversity of ammonium-oxidizing archaea and ammonium-oxidizing bacteria in surface sediments of Oujiang River[J]. Environmental Science, 2015, 36(12): 4659-4666. |
 2018, Vol. 39
2018, Vol. 39


