2. 湖南大学环境科学与工程学院, 长沙 410082;
3. 深圳市罗湖区环境保护与水务局, 深圳 518000;
4. 北京清控人居环境研究院有限公司, 北京 100000
2. College of Environmental Science and Engineering, Hunan University, Changsha 410082, China;
3. Environmental Protection and Water Bureau of Shenzhen Luohu District, Shenzhen 518000, China;
4. Tsinghua Holdings Human Settlements Environment Institute, Beijing 100000, China
CH4是一种导致全球变暖的大气痕量气体.相关研究表明大气中甲烷的体积分数已从工业化前的7.15×10-7, 达到了2008年的1.787×10-6[1], 虽然这远低于大气中CO2的体积分数3.86×10-4, 但相同物质的量的CH4和CO2, 前者造成的温室效应是后者的20~30倍[2], 因此对全球变暖有着重大的影响.研究表明自然界存在一种反硝化厌氧甲烷氧化(nitrite-dependent anaerobic methane oxidation, N-DAMO)过程, 厌氧环境下以NO2-和NO3-为电子受体将CH4氧化为CO2, 同步完成脱氮除CH4过程[3]. Ettwig等[4]的研究中发现在N-DAMO过程中起主要作用的微生物是一类新型细菌, 隶属于NC10门细菌, 并将其命名为“Candidatus Methylomirabilis oxyfera”(M. oxyfera).
目前有关N-DAMO的研究主要集中在用分子克隆技术等生物学手段检测生态环境中M. oxyfera的存在, 根据M. oxyfera或其他NC10门细菌设计特异性探针和引物, 利用荧光定量PCR比较不同环境系统中NC10细菌的数量和丰度情况, 利用分子生物学手段跟踪监测整个N-DAMO过程微生物群落的演变等[5].
近年来, 在海洋[6]、稻田[7, 8]、湖泊[9~12]和其他湿地[13, 14]等自然生态环境中均陆续发现了M. oxyfera的存在, 但是对有关河流环境中M. oxyfera的研究并不多见[15].我国很多河流受到了NOx的严重污染, 这有利于N-DAMO过程的发生, 这些污染物沉积形成的底泥环境成为微生物与生存环境之间进行物质交换的载体[16].
16S rRNA基因携带着一定量的特殊序列信息, 这些序列信息可作为目标引物的设计模板, 是到现在为止能够实际运用的最为广泛的标记基因.功能基因可以标记具有特定生理和代谢功能的微生物, 是多样性分子检测中另一类极为重要的标记基因, 而pmoA功能基因几乎存在所有甲烷氧化菌中, 它编码关键酶pMMO的一个亚基, 已成为甲烷氧化菌生态学研究中广为采用的生物标记物[17].
综上, 本文以浑河为研究对象, 以16S rRNA基因和pmoA功能基因作为标记基因, 通过克隆文库技术考察了浑河底泥M. oxyfera的群落多样性, 调查了不同区域水质和底泥的理化性质, 分析了M. oxyfera的群落多样性和与环境因子(水质和底泥的理化性质)的关系, 以此考察环境因子对M.oxfera的群落多样性的影响, 旨在为研究反硝化厌氧甲烷氧化过程减小自然环境CH4的排放提供理论基础.
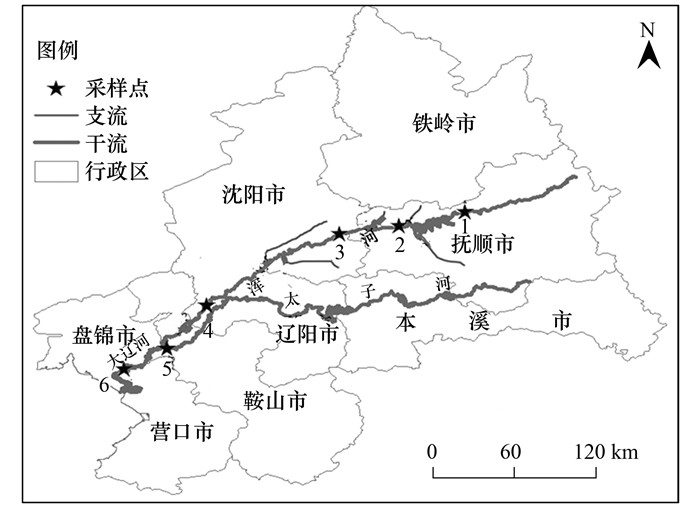
1 材料与方法 1.1 研究区域与采样点的设置浑河地处辽宁省中部(122°20′~125°20′E, 40°00′~42°20′N), 南邻太子河, 北邻辽河, 东邻浑江, 河流自东北向西南, 流经抚顺、沈阳、辽阳、鞍山这4个市, 全长415.4 km.曾有研究表明沈阳和抚顺两个重工业城市曾向浑河中排入了大量的生活污水对浑河水体造成了严重的污染[18].于2014年9月中旬在浑河部分区域采集水样和底泥样品, 2015年3月对部分区域的底泥样品又进行了采集.根据浑河的自然条件和地理位置从东至西共布设了6个采样点, 上游采样点编号为1、2, 中游为3、4, 下游为5、6, 具体位置见图 1.
|
1.南杂木; 2.章党大桥; 3.葛布大桥; 4.黄腊坨; 5.对坨; 6.田庄台 图 1 采样地点示意 Fig. 1 Descriptions of sampling sites |
使用不锈钢采水器采集水样, 在距离底泥上方2~3 cm处采用便携式水质测定仪(Thermo Orion Star, 德国)现场测定河流表层水体(0~10 cm)的pH值.水样(底泥上方2~3cm处)的理化特性分析按照文献[19]中介绍的方法测定, 主要的测定指标有五日生化需氧量(BOD5)、总磷(TP)、氨氮(NH4+-N)、硝氮(NO3--N)、亚硝氮(NO2--N)和总有机碳(TOC).用已灭菌的柱状采泥器采集自底泥表层至10cm深度的底泥, 泥样的理化性质如总氮(TN)、底泥pH等按照文献[20]中介绍的方法测定.
1.3 DNA提取与基因扩增使用Power Soil DNA Kit (Mo Bio Laboratories, Carlsbad, CA)对底泥样品进行总DNA的提取, 操作按照说明书进行. DNA的提取结果用浓度为1%的琼脂糖凝胶电泳检测.采用巢式PCR方法对底泥样品中反硝化厌氧甲烷氧化菌16S rRNA和pmoA功能基因进行扩增. PCR扩增产物用2%琼脂糖凝胶电泳进行检测.采用的PCR扩增体为2×PCR Go Taq® Green Master Mix(Promega, USA)12.5μL, 上下引物各1 μL(10 μmol·L-1), DNA模板2 μL (1~10 ng), 最后用ddH2O补足至25 μL.
对于反硝化厌氧甲烷氧化菌的16S rRNA基因, 第一轮使用的引物为202F[21]和1545R[22], 第二轮使用的引物为qP1F和qP2R[21].反应条件为:94℃预变性4 min, 94℃变性1 min, 57℃(第二轮为65℃)退火1 min, 72℃延伸2 min 30 s, 循环次数为30次, 最后72℃延伸2 min 30 s[23].对于反硝化厌氧甲烷氧化菌的pmoA功能基因, 第一轮使用的引物为A189-b和cmo682[24], 第二轮使用的引物为cmo182和cmo568[25].反应条件为:94℃预变性4 min, 94℃变性1 min, 温度梯度为52~62℃退火1min, 72℃延伸1 min 30 s, 循环次数为30次, 最后72℃延伸10 min[24].将所有的一轮PCR产物混合均匀后作为第二轮PCR的DNA模板.以上两轮PCR扩增产物均经2%的琼脂糖凝胶电泳检测.
1.4 测序与多样性指数分析将经2%的琼脂糖电泳检验确认正确后的M.oxyfera的16S rRNA和其 pmoA功能基因的二轮PCR产物连接到pGM-T(Promega, USA)载体上, 然后转化到感受态细胞并在E.coli DH5α (Promega, USA)中进行涂板, 筛选白斑.每个样品随机挑选20~30个克隆子进行测序, 将所得序列BlastN在GenBank数据库中进行对比, 确认为目标序列后利用DOTUR软件分别在3%和7%的差异性水平下对M. oxyfera的16S rRNA和pmoA功能基因进行OTU分类并计算每个样品的Shannon-Wiener多样性指数和Chao1丰富度指数.克隆库覆盖率(coverage, %)的计算公式[26]:
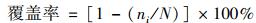
|
式中, ni为在该试验构建的文库中只出现1次的OTU数, N为克隆总数.
1.5 多元直接梯度分析运用Canoco5.0软件将M.oxyfera的群落多样性指数和环境因子进行多元直接梯度分析, 以此揭示环境因子对M.oxyfera的群落多样性产生的影响.
2 结果与分析 2.1 M. oxyfera的群落多样性指数分析分别对9月在浑河采集的6个底泥样品中M. oxyfera的16S rRNA基因和pmoA基因进行特异性扩增, 其群落多样性指数见表 1.对3月采集的3、6号这2个采样点处M. oxyfera的16S rRNA和pmoA功能基因进行特异性扩增, 并和相同采样点9月采集的样品中的M. oxyfera的基因多样性指数对比, 结果见表 2.
|
|
表 1 浑河底泥不同采样点处M.oxyfera的群落多样性指数 Table 1 Community diversity index of M.oxyfera from different sediment samples of the Hunhe River |
|
|
表 2 3月和9月浑河底泥M.oxyfera的群落多样性指数 Table 2 Community diversity index of the M. oxyfera from sediment samples of the Hunhe River collected in September and March |
M. oxyfera的群落多样性指数中Shannon-Wiener指数是用来估算菌群的多样性, 其值越大说明群落多样性越高; OTU数代表这类细菌中种的个数; Chao1指数是用Chao1算法估计群落中含有的OTU数目, 和OTU数目总是呈正相关的变化规律; 因此, 在讨论群落多样性时空分布特征时只讨论OTU数和Shannon-Wiener指数的变化.
2.2 16S rRNA和pmoA基因序列的系统发育分析将9月采集的样品中M. oxyfera测序所得序列经处理后, 在NCBI(Nation Center for Biotechnology Information)网站上进行序列比对, 利用DOTUR软件对16S rRNA基因和pmoA功能基因进行OTU分类, 分别划分为7个OTU和13个OTU.每个OTU的比对结果见表 3和表 4.对于16S rRNA来说, 可以看出每个OTU的序列与NCBI网站上的已知序列的相似度都高于97%, 特别指出7个测序的OTU中有5个属于不可培养的反硝化厌氧甲烷氧化菌的16S rRNA基因序列, 这从一个方面也反映了浑河底泥中存在大量的不可培养的反硝化厌氧甲烷氧化菌.从表 3可以看出pmoA功能基因每个OTU的序列与NCBI网站上的已知序列的相似度都高于96%, 全部的OTU也都是不可培养的pmoA功能基因序列.
|
|
表 3 9月M.oxyfer的16S rRNA基因序列对比信息1) Table 3 The 16S rRNA gene sequence comparison information of M. oxyfera in from samples collected in September |
|
|
表 4 9月M. oxyfer的pmoA基因序列对比信息 Table 4 The pmoA gene sequence comparison information of M. oxyfera from samples collected in September |
2.3 水样和底泥样品的理化指标
9月浑河6个采样点的各项水质指标和底泥理化指标的测定结果如表 5所示.可以看出大部分采样点的pH值范围为7.6~9.2之间, 总体上浑河水质呈弱碱性.所有采样点的水中BOD5值均大于4 mg·L-1, 特别是4、6号采样点水中BOD5值均大于10 mg·L-1属于劣Ⅴ类水, 受有机污染较严重.浑河各采样点TP的平均浓度值分别为0.11 mg·L-1, 根据《地表水环境质量标准(GB 3838-2002)》, 划分为Ⅲ类水.
|
|
表 5 各采样点水质和底泥的理化指标 Table 5 Physicochemical properties of water samples and sediment samples |
浑河底泥的pH值在7.9~8.2之间, 这和浑河河水pH值相一致; 底泥电导率在0.5~0.8 mS·cm-1之间, 均值为0.69 mS·cm-1, 波动不大; 总氮(TN)含量介于0.53~0.69 mg·kg-1之间, 均值为0.58 mg·kg-1.溶解性有机碳(DOC)含量介于23.11~25.13 mg·kg-1之间, 均值为24.20 mg·kg-1.
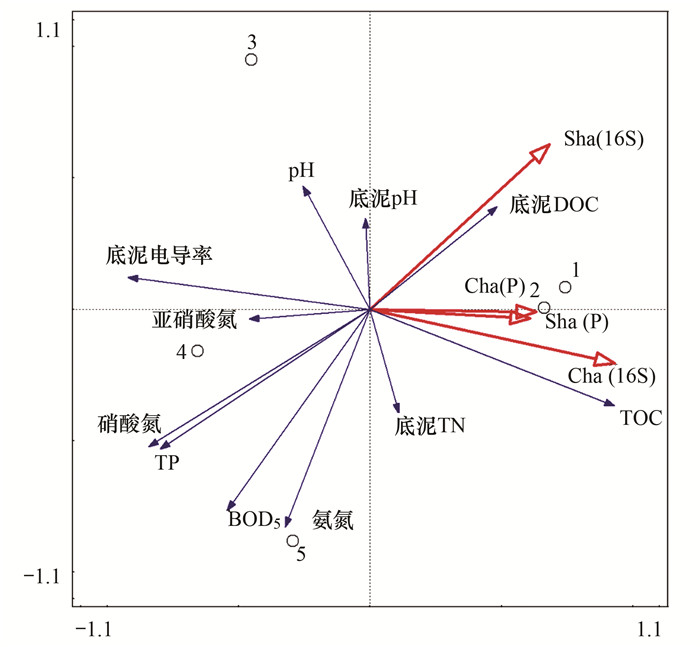
2.4 多元直接梯度分析9月浑河1、2、3、5和6共5个采样点处底泥样品中M.oxfera 的群落多样性指数(Shannon-Wiener指数和Chao1指数)和环境因子(水样和底泥的理化性质)的多元直接梯度分析结果如图 2所示.根据基因多样性指标与环境因子的夹角的余弦值的绝对值的大小可以判断两者的相关性大小情况, 余弦值的正负分别表示正相关性和负相关性; 与多样性指数相关的环境因子的箭线长度代表其对该多样性指数的影响程度.
|
Sha(16S)表示16S rRNA基因的Shannon-Wiener指数; Cha(16S)表示16S rRNA基因的Chao1指数; Sha(P)表示pmoA基因的Shannon-Wiener指数; Cha(P)表示pmoA基因的Chao1指数 图 2 多元直接梯度分析 Fig. 2 Multivariate direct gradient analysis |
由图 2可知16S rRNA的Shannon-Wiener指数和底泥中DOC的浓度呈明显的正相关, 和水样中硝酸氮浓度、TP浓度呈明显的负相关; Chao1指数和水体中TOC的浓度呈现出显著的正相关, 而且TOC的浓度对Chao1指数有较大的影响, 因为从图 2中可以看出TOC指标的箭线比较长, 与底泥电导率和水体中亚硝酸浓度呈负相关.
pmoA基因的Shannon-Wiener指数和Chao1指数之间的夹角比较小, 水体中亚硝酸氮浓度和底泥电导率与它们呈现出较为明显的负相关, 但亚硝酸氮的影响较小; 底泥中的DOC含量和水体中TOC浓度与它们有一定的相关性.
3 讨论 3.1 M. oxyfera的群落多样性区域分布特征从表 1可以得出浑河上、中、下游的6个采样点16S rRNA基因的OTU数在2~5之间, 呈现出先增大后减小的变化趋势, 但变化幅度不大; Shannon-Wiener指数变化范围为0.21~1.4, 中游到下游变化幅度较大, 呈现出中游>上游>下游的分布特征. pmoA功能基因上游的OTU数和Shannon-Wiener指数明显高于中游和下游, 上游的Shannon-Wiener指数分别是中游和下游的3.5倍和2.3倍, 这说明了浑河从上游到下游M. oxyfera的pmoA功能基因种类和多样性水平有较大幅度减少.综上M. oxyfera的多样性呈现出明显的地域性分布特征.
3.2 M. oxyfera的群落多样性季节分布特征由表 2可知, 在3号采样点3月采集的底泥样品中16S rRNA和pmoA的OTU数目分别是6和5, 明显高于9月对应的OTU数, Shannon-Wiener指数也明显比9月高(1.4>0.68; 1.57>0.00).因此, 3号采样点M.oxyfera的16S rRNA和pmoA的基因多样性存在明显的时间性差异.观察6号采样点样3月和9月M.oxyfera的pmoA的OTU数和Shannon-Wiene指数值也有显著的差异.综上, M. oxyfera的群落多样性随着季节的变化呈现出不同的特征.
3.3 环境因子对M. oxyfera的群落多样性的影响对多元直接梯度分析的结果进行分析可以发现底泥中DOC浓度、水体中TOC含量和两种基因的Shannon-Wiener指数和Chao1指数均呈现出显著的正相关; 底泥电导率和水体中亚硝酸氮浓度和pmoA的Shannon-Wiener指数、Chao1指数、16S rRNA的Chao1指数呈现出明显的负相关, 亚硝酸氮浓度还与16S rRNA的Shannon-Wiener指数负相关.这4种环境因子与M. oxyfera的群落多样性指数都有显著的关联, 因此认为底泥中DOC浓度、水体中TOC含量、底泥电导率和水体中亚硝酸氮浓度是影响M. oxyfera群落多样性的主要环境因子.
值得注意的是亚硝酸氮是M. oxyfera进行反硝化厌氧甲烷氧化过程的电子受体, 在本研究中却与两种基因的4个多样性指数均呈明显的负相关, 因此可以推断一定浓度的亚硝酸氮浓度对M. oxyfera细菌的生存起促进作用, 但过高浓度的亚硝酸氮会抑制M. oxyfera的生存, 表现为群落多样性指数降低, 在此之前已有文献对此进行了讨论, 认为过高负荷的亚硝酸盐会对M. oxyfera的生存有一定的抑制作用[27].
4 结论同一时期, 从浑河上游到下游各采样点底泥中M. oxyfera的pmoA功能基因种类和多样性水平有大幅度减少, M. oxyfera的群落多样性呈现出明显的地域差异.在3号采样点3月采集的底泥样品中M. oxyfera的16S rRNA和pmoA的OTU数目和Shannon-Wiener指数也明显比9月高, 同一采样点中M. oxyfera的群落多样性存在明显的季节性差异.多元直接梯度分析结果表明底泥中DOC浓度、底泥电导率和水体中亚硝酸氮浓度、TOC含量是影响M. oxyfera群落多样性的主要环境因子.
| [1] | Dlugokencky E J, Bruhwiler L, White J W C, et al. Observational constraints on recent increases in the atmospheric CH4 burden[J]. Geophysical Research Letters, 2009, 36(18): L18803. DOI:10.1029/2009GL039780 |
| [2] | Hofmann D J, Butler J H, Tans P P. A new look at atmospheric carbon dioxide[J]. Atmospheric Environment, 2009, 43(12): 2084-2086. DOI:10.1016/j.atmosenv.2008.12.028 |
| [3] | Raghoebarsing A A, Pol A, Van de Pas-Schoonen K T, et al. A microbial consortium couples anaerobic methane oxidation to denitrification[J]. Nature, 2006, 440(7086): 918-921. DOI:10.1038/nature04617 |
| [4] | Ettwig K F, Butler M K, Le Paslier D, et al. Nitrite-driven anaerobic methane oxidation by oxygenic bacteria[J]. Nature, 2010, 464(7288): 543-548. DOI:10.1038/nature08883 |
| [5] |
沈李东. 亚硝酸盐型甲烷厌氧氧化微生物特性研究进展[J]. 环境科学, 2015, 36(3): 1133-1140. Shen L D. Research progress on microbial properties of nitrite-dependent anaerobic methane-oxidising bacteria[J]. Environmental Science, 2015, 36(3): 1133-1140. |
| [6] | Chen J, Jiang X W, Gu J D. Existence of novel phylotypes of nitrite-dependent anaerobic methane-oxidizing bacteria in surface and subsurface sediments of the South China Sea[J]. Geomicrobiology Journal, 2015, 32(1): 1-10. DOI:10.1080/01490451.2014.917742 |
| [7] | Zhou L L, Wang Y, Long X E, et al. High abundance and diversity of nitrite-dependent anaerobic methane-oxidizing bacteria in a paddy field profile[J]. FEMS Microbiology Letters, 2014, 360(1): 33-41. DOI:10.1111/fml.2014.360.issue-1 |
| [8] | Wang Y, Zhu G B, Harhangi H R, et al. Co-occurrence and distribution of nitrite-dependent anaerobic ammonium and methane-oxidizing bacteria in a paddy soil[J]. FEMS Microbiology Letters, 2012, 336(2): 79-88. DOI:10.1111/fml.2012.336.issue-2 |
| [9] | Liu Y, Zhang J X, Zhao L, et al. Aerobic and nitrite-dependent methane-oxidizing microorganisms in sediments of freshwater lakes on the Yunnan Plateau[J]. Applied Microbiology and Biotechnology, 2015, 99(5): 2371-2381. DOI:10.1007/s00253-014-6141-5 |
| [10] | Deutzmann J S, Stief P, Brandes J, et al. Anaerobic methane oxidation coupled to denitrification is the dominant methane sink in a deep lake[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(51): 18273-18278. DOI:10.1073/pnas.1411617111 |
| [11] | Kojima, Tsutsumi M, Ishikawa K, et al. Distribution of putative denitrifying methane oxidizing bacteria in sediment of a freshwater lake, Lake Biwa[J]. Systematic and Applied Microbiology, 2012, 35(4): 233-238. DOI:10.1016/j.syapm.2012.03.005 |
| [12] | Deutzmann J S, Schink B. Anaerobic oxidation of methane in sediments of Lake Constance, an oligotrophic freshwater lake[J]. Applied and Environmental Microbiology, 2011, 77(13): 4429-4436. DOI:10.1128/AEM.00340-11 |
| [13] | Shen L D, Huang Q, He Z F, et al. Vertical distribution of nitrite-dependent anaerobic methane-oxidising bacteria in natural freshwater wetland soils[J]. Applied Microbiology and Biotechnology, 2015, 99(1): 349-357. DOI:10.1007/s00253-014-6031-x |
| [14] | Hu B L, Shen L D, Lian X, et al. Evidence for nitrite-dependent anaerobic methane oxidation as a previously overlooked microbial methane sink in wetlands[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(12): 4495-4500. DOI:10.1073/pnas.1318393111 |
| [15] |
周蓉, 祝贵兵, 周磊榴, 等. 稻田土壤反硝化厌氧甲烷氧化菌的时空分布与群落结构分析[J]. 环境科学学报, 2015, 35(3): 729-737. Zhou R, Zhu G B, Zhou L L, et al. Spatial-temporal distribution and community structure analysis of nitrite-dependent anaerobic methane-oxidizing bacteria in paddy soil[J]. Acta Scientiae Circumstantiae, 2015, 35(3): 729-737. |
| [16] |
王佳, 宋永会, 彭剑峰, 等. 浑河底泥反硝化厌氧甲烷氧化菌群落结构时空特征[J]. 环境科学研究, 2015, 28(11): 1670-1676. Wang J, Song Y H, Peng J F, et al. Spatial-temporal distribution and community structure analysis of nitrite-dependent anaerobic methane-oxidizing bacteria in the sediments of the Hun River[J]. Research of Environmental Sciences, 2015, 28(11): 1670-1676. |
| [17] |
邓勇翠, 车荣晓, 吴伊波, 等. 好氧甲烷氧化菌生理生态特征及其在自然湿地中的群落多样性研究进展[J]. 生态学报, 2015, 35(14): 4579-4591. Deng Y C, Che R X, Wu Y B, et al. The physiological and ecological characteristics of aerobic methane oxidizing bacteria and their community diversity in natural wetlands[J]. Acta Ecologica Sinica, 2015, 35(14): 4579-4591. |
| [18] |
杨楠, 于会彬, 宋永会, 等. 白塔堡河上覆水与沉积物间隙水N、P分布特征[J]. 环境科学研究, 2013, 26(7): 728-735. Yang N, Yu H B, Song Y H, et al. Distribution of nitrogen and phosphorus in overlying water and pore water of sediment in Baitabuhe River[J]. Research of Environmental Sciences, 2013, 26(7): 728-735. |
| [19] | 国家环境保护总局. 水和废水监测分析方法[M]. 第四版. 北京: 中国环境科学出版社, 2002. |
| [20] | 张甘霖, 龚子同. 土壤调查实验室分析方法[M]. 北京: 科学出版社, 2012. |
| [21] | Ettwing K F, Van Alen T, Van De Pas-schoonen K T, et al. Enrichment and molecular detection of denitrifying methanotrophic bacteria of the NC10 phylum[J]. Applied and Environmental Microbiology, 2009, 75(11): 3656-3662. DOI:10.1128/AEM.00067-09 |
| [22] | Juretschko S, Timmermann G, Schmid M, et al. Combined molecular and conventional analyses of nitrifying bacterium diversity in activated sludge:Nitrosococcus mobilis and Nitrospira-like bacteria as dominant populations[J]. Applied and Environmental Microbiology, 1998, 64(8): 3042-3051. |
| [23] | Luesken F A, Zhu B L, Van Alen T A, et al. PmoA primers for detection of anaerobic methanotrophs[J]. Applied and Environmental Microbiology, 2011, 77(11): 3877-3880. DOI:10.1128/AEM.02960-10 |
| [24] | Schloss P D, Handelsman J. Introducing DOTUR, a computer program for defining operational taxonomic units and estimating species richness[J]. Applied and Environmental Microbiology, 2005, 71(3): 1501-1506. DOI:10.1128/AEM.71.3.1501-1506.2005 |
| [25] | Luesken F A, Van Alen T A, Van Der Biezen E, et al. Diversity and enrichment of nitrite-dependent anaerobic methane oxidizing bacteria from wastewater sludge[J]. Applied Microbiology and Biotechnology, 2011, 92(4): 845-854. DOI:10.1007/s00253-011-3361-9 |
| [26] |
侯梅锋, 何士龙, 李栋, 等. 连云港海底底泥及青海湖底泥细菌多样性研究[J]. 环境科学, 2011, 32(9): 2681-2688. Hou M F, He S L, Li D, et al. Bacterial diversity in Lianyungang marine sediment and Qinghai Lake sediment[J]. Environmental Science, 2011, 32(9): 2681-2688. |
| [27] |
袁梦冬, 朱静, 吴伟祥. 微生物厌氧甲烷氧化反硝化研究进展[J]. 微生物学报, 2014, 54(2): 129-138. Yuan M D, Zhu J, Wu W X. Research progress in microbial anaerobic methane oxidation coupled to denitrification-A review[J]. Acta Microbiologica Sinica, 2014, 54(2): 129-138. |
 2018, Vol. 39
2018, Vol. 39


