2. 大连市碧流河水库管理局, 大连 116221
2. Management Bureau of Biliuhe Reservoir of Dalian City, Dalian 116221, China
淡水资源是人类赖以生存和发展的基础, 水中细菌状况对水体质量起着决定性作用, 并且微生物菌群能够对水体污染物有净化作用[1].淡水生态系统中细菌数量庞大且种类丰富, 是水域生态系统中食物链和食物网的重要组成部分, 在物质循环和能量流动中起重要作用[2].微生物敏感且易受环境因子影响, 它的代谢产物可以对环境产生指示作用[3].传统分离培养方法仅能从环境中获取不到1%的细菌类群, 不能准确认知环境中微生物的状况[4], 近年来, 利用分子生物学的方法研究环境微生物越来越普及[5], 目前研究对象大部分集中在滨海地区、海洋和湖泊, 例如, 我国的南海南沙海域[6]、青岛近岸海区[7]及云南高原洱海[8], 国外一些地区如西北太平洋深海[9]、巴西里约热内卢州热带沿海泻湖[10]、南极洲弗里克塞尔湖等[11], 而对河流和饮用水源水库的研究较少.
碧流河水库位于辽宁省庄河市、普兰店区和盖州市三市区交界处, 处于大连市最大河流碧流河的干流上, 是大连市居民生活、工业用水、农业灌溉、水库养鱼等主要水源地.碧流河水库是南北狭长的河川库型, 该区域为温暖带大陆性季风气候, 四季分明, 温差较大, 控制水域流量丰富, 平均降水量多年可达到780 mm, 降雨多在7~9月[12].碧流河水库蓄水位正常为69.0 m, 库区为70.0 m[13].近几年碧流河水库蓄水位降低, 水库周边大量开荒种地引发水土流失日趋严重, 淤积量加大, 种植农作物使用的农药化肥流入库区, 污染了水库水质[14], 为了保障饮用水和使用水的安全, 改善水体的质量状况十分重要.实现“东水西调”可以有效改善碧流河水库生态环境, 确保其水资源和土地资源的互补, 水中多种微生物共同作用形成微生物链, 在水体修复的过程中把有机污染物转化为简单的化合物, 保证水质正常功能, 从而改善水体环境质量[15], 本研究采用16S rDNA高通量测序技术分析碧流河水库细菌群落结构特征及其关键驱动因子, 旨在为保障水质安全和优化水质提供科学依据.
1 材料与方法 1.1 样品采集及处理在2016年10月~2017年6月对碧流河水库进行为期一年的水样采集.选取9个采样点, 分别是下游的坝前1, 坝前2和坝前3;中游的刘店1, 刘店2和刘店3;上游八家河河口附近的钟岭, 蛤蜊河河口的桂云花和碧流河河口的大堡.冬季大堡样品因河道无水而缺失.使用有机玻璃采水器采集水样, 根据实际水深分表中底3层采混合样, 由于河水深度变化较大, 表层采集深度小于2 m, 中层采集深度在4~5 m, 底层采集深度8 m左右, 表中底三层分别打水3、4、3次, 将这10次打的水混合, 取1.5 L水样经过0.45 μm微孔滤膜过滤, 过滤后的滤膜加DNA保存液置于冻存管中, -80℃冰箱保存.调查范围及采样点位置如图 1所示.
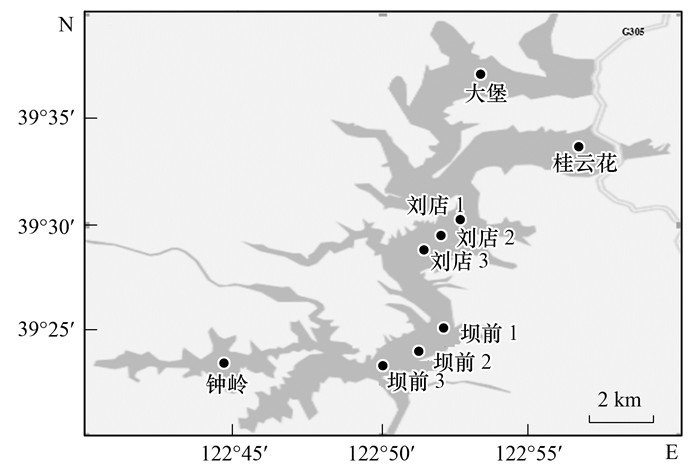
|
图 1 碧流河水库采样点示意 Fig. 1 Sampling point settings in the Biliuhe Reservoir |
采用CTAB法对样本基因组DNA进行提取.取出用DNA保存液保存的滤膜, 将其剪碎于2.0 mL EP管中, 加入1 000 μL CTAB裂解液和20 μL溶菌酶, 65℃振荡水浴2 h, 进行充分裂解.使用离心机HeraeusPico 17(ThermoScientific, USA)12 000 r·min-1离心15 min, 取950 μL上清液, 加入与上清等体积的酚(pH 8.0):氯仿:异戊醇(25:24:1), 颠倒混匀, 12 000 r·min-1离心10 min.取上清液, 加入等体积的氯仿:异戊醇(24:1), 颠倒混匀, 12 000 r·min-1离心10 min.吸取上清至1.5 mL离心管, 加入上清液3/4体积的异丙醇, 上下摇晃, -20℃沉淀. 12 000 r·min-1离心10 min, 倒出液体.用75%乙醇1 mL洗涤2次, 沉淀经超净工作台吹干后加入51 μL ddH2O溶解DNA样品, 于55~60℃下孵育10 min助溶.加RNase A 1 μL消化RNA, 37℃放置15 min.
之后利用琼脂糖凝胶电泳检测DNA的纯度和浓度, 取适量的样品于离心管中, 使用无菌水稀释样品至1 ng·μL-1.以稀释后的基因组DNA为模板, 使用带Barcode的特异引物515F:5′-GTGCC AGCMGCCGCGGTAA-3′和806R:5′-GGACTACHVG GGTWTCTAAT-3′扩增宏基因组16S rDNA, 使用New England Biolabs公司的Phusion ® High-Fidelity PCR Master Mix with GC Buffer, 和高效高保真酶进行PCR扩增, 扩增条件:98℃预变性1 min; 30个循环包括: 98℃, 10 s; 50℃, 30 s; 72℃, 30 s; 72℃, 5 min.得到PCR产物.
根据PCR产物浓度进行等量混样, 充分混匀后使用2%的琼脂糖凝胶电泳检测PCR产物, 对目的条带使用qiagen公司提供的胶回收试剂盒纯化回收产物.
使用TruSeq ® DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建, 构建好的文库经过Qubit和Q-PCR定量, 文库合格后, 使用HiSeq2500 PE250进行上机测序.
1.3 水体理化指标测定测定的水体理化指标有pH、水温(T)、总溶解固体(TSS)、溶解氧(DO)、透明度(SD)、总氮(TN)、总磷(TP)、氨氮(NH4+-N)、硝酸盐(NO3--N)、亚硝酸盐(NO2--N)、活性磷(ADP)、化学耗氧量(COD)、叶绿素a(Chla)等十几项指标, 其中现场使用pH计、温度计、透明度盘测定水样pH、温度和透明度, 其余理化指标在大连海洋大学水生生物学实验室测定, 测定方法按照文献[16]中规定的方法进行.
1.4 测序数据处理根据Barcode序列和PCR扩增引物序列从下机数据中拆分出各样品数据, 截去Barcode和引物序列后使用FLASH[17], 对每个样品的reads进行拼接, 得到的拼接序列为原始Tags数据(Raw Tags); 拼接得到的Raw Tags, 需要经过严格的过滤处理[18]得到高质量的Tags数据(Clean Tags).参照Qiime[19]的Tags质量控制流程, 进行Tags截取, Tags长度过滤, 将处理得到的Tags去除嵌合体序列[20, 21].
利用Uparse软件[22], 对所有样品的全部Effective Tags进行聚类, 默认以97%的一致性(Identity)将序列聚类成为OTUs(operational taxonomic units).同时会选取OTUs的代表性序列, 依据其算法原则, 筛选的是OTUs中出现频数最高的序列作为OTUs的代表序列.对OTUs代表序列进行物种注释, 用Mothur软件与SILVA[23]的SSUrRNA数据库[24]进行物种注释分析(设定阈值为0.8~1.0), 获得分类学信息并分别在各个分类水平:kingdom(界)、phylum(门)、class(纲)、order(目)、family(科)、genus(属)、species(种)统计各样本的群落组成.使用MUSCLE[25]软件进行快速多序列比对, 得到所有OTUs代表序列的系统发生关系.最后对各样品的数据进行均一化处理, 以样品中数据量最少的为标准进行均一化处理, 后续的α多样性分析和β多样性分析都是基于均一化处理后的数据.
根据所有样品在属水平的物种注释及丰度信息, 选取丰度排名前35的属, 根据其在每个样品中的丰度信息, 从物种和样品两个层面进行聚类, 绘制成热图, 便于发现哪些物种在哪些样品中聚集较多或含量较低.利用OTUs数值和注释种类到属水平上计算各采样点的香农-威纳指数(Shannon-Wiener index), 分析比较细菌群落多样性.
1.5 统计分析利用Excel分析计算水体指标参数.使用Qiime软件(Version 1.9.1)计算Shannon指数, 对OTUs进行丰度分析; 使用CANOCOforwindow4.5进行RDA分析, 挖掘样品间的物种组成差异, 并结合环境因素进行关联分析.
2 结果与分析 2.1 碧流河水库细菌多样性分析35个样品总共得到平均有效数据70 045个, 构建OTUs并且获得注释信息的Tags数目平均为67 583个, OTUs平均有1 255个.利用香农-维纳指数比较分析9个样点的细菌多样性(表 1). 表 1显示, 碧流河水库全年细菌群落多样性指数平均达6.921, 从季节分布上看, 冬季 < 夏季 < 春季 < 秋季.碧流河水库细菌多样性水平分布上存在季节差异, 在秋季, 中游采样点的细菌多样性 < 下游采样点 < 上游采样点, 在冬季, 上游采样点的细菌多样性 < 中游采样点 < 下游采样点, 在春季和夏季, 下游采样点的细菌多样性 < 上游采样点 < 中游采样点.根据物种注释结果, 选取每个样品分组(分别为秋季下游FADb、秋季中游FAML、秋季上游FAU、冬季下游WIDb、冬季中游WIML、冬季上游WIU、春季下游SPDb、春季中游SPML、春季上游SPU、夏季下游SUDb、夏季中游SUML、夏季上游SUU)在各分类水平上最大丰度前10的门水平物种相对丰度柱状图(图 2), 同时分析各个水平上的菌群结构.结果显示, 11个主要门类分别是变形菌门Proteobacteria 34.16%、蓝菌门Cyanobacteria 24.35%、放线菌门Actinobacteria 13.35%、拟杆菌门Bacteroidetes 12.42%、浮霉菌门Planctomycetes 6.04%、疣微菌门Verrucomicrobia 3.42%、厚壁菌门Firmicutes 2.51%、酸杆菌门Acidobacteria 1.37%、绿菌门Chlorobi 0.39%、装甲菌门Armatimonadetes 0.17%以及还有一些分类地位不明确的细菌类群和稀有类群Others 1.82%.优势菌门变形菌门包含:β-变形菌纲β-Proteobacteria 18.34%、α-变形菌纲α-Proteobacteria 9.14%、γ-变形菌纲γ-Proteobacteria 5.3%. β-变形菌纲菌群主要集中于伯克氏菌目Burkholderiales 15.75%, 碧流河水库9个采样点菌群变化主要集中在蓝菌门、放线菌门和拟杆菌门上, 共发现细菌44门115纲184目368科865属, 其中, 优势菌属为unidentified_Chloroplast 17.73%, 伦黑墨氏菌属Rheinheimera 1.74%, CL500-3 4.64%, hgcI_clade 5.95%, 黄杆菌属Flavobacterium 2.59%, CL500-29_marine_group 2.67%, 鞘脂单胞菌属Sphingomonas 1.32%, 聚球藻属Synechococcus 1.08%, β-变形菌属Limnohabitans 2.45%.
|
|
表 1 碧流河水库细菌多样性(Shannon)统计 Table 1 Shannon statistical table of bacteria biodiversity in Biliuhe Reservoir |
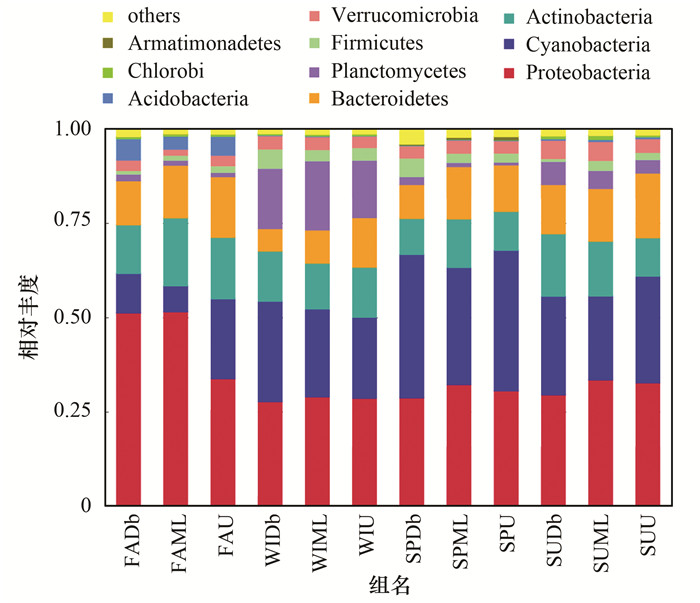
|
图 2 碧流河水库微生物门水平上的物种相对丰度 Fig. 2 Relative abundance of microbial species in the Biliuhe Reservoir at the phylum level |
根据所有样品在属水平的物种注释和丰度信息, 选取丰度排名前35的属, 从物种注释信息(横向)和样品信息(纵向)两个层面进行聚类, 绘制热图(图 3), 根据Z值便于发现哪些物种在哪些样品中聚集较多或含量较低. 图 3显示, 样点在菌群组成上冬春季和夏秋季有显著差异, 其中冬季和春季各个点群落组成相似, 秋季和夏季上中下游采样点群落组成有差异.比较各个采样点菌群结构在一年内的变化, 上游采样点菌群组成在不同季节差异最大, 不同季节主要菌群结构差异明显.秋季CL500-29_marine_group、Rheinheimera、Prochlorothrix、Pedobacter、Sphingomonas、Massilia、Limnobacter聚集较多, 春季Methylobacterium、unidentified_Chloroplast、Enterococcus聚集多, 夏季Phyllobacterium、Roseomonas、Planctomyces、Synechococcus、hgcI_clade、Brevundimonas、Acidibacter、Terrimonas、Acinetobacter聚集多, 冬季Staphylococcus、Marivita、CL500-3、Lactobacillus、Candidatus_Aquirestis、Flavobacterium、Candidatus_Methylopumilus聚集多.
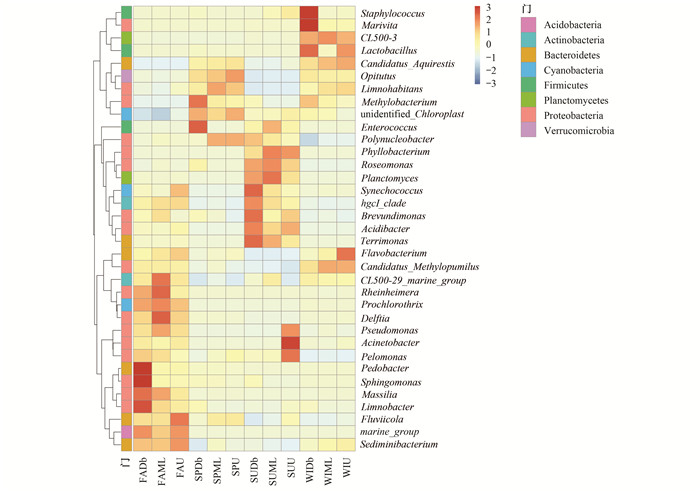
|
图 3 碧流河水库细菌物种丰度聚类 Fig. 3 Cluster analysis of bacterial species abundance in the Biliuhe Reservoir |
VPA分析结果表明(图 4), TSS、TP、COD、T、pH、NH4+-N、Chla这些环境因子的单独解释量为10.64%, TN、ADP、DO、NO2--N、NO3--N、SD、经度、纬度、海拔这些环境因子的单独解释量为18.35%, 这两组环境变量的共同解释量为55.86%, 这16个环境因子的总解释量为84.85%, 可以看出, 这些环境因子对碧流河水库细菌种类分布影响很大, 其中第二组环境因子对细菌群落分布的影响高于第一组.说明地理、化学因子均对微生物群落分布差异作出贡献.
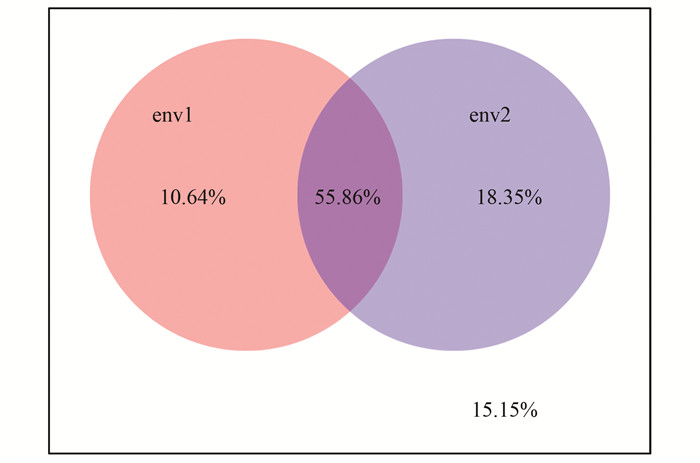
|
env1为TSS、TP、COD、T、pH、NH4+-N、Chla这些环境因子的解释量, env2为TN、ADP、DO、NO2--N、NO3--N、SD、经度、纬度, 海拔这些环境因子的解释量, 圆圈外为不能的解释量 图 4 碧流河水库细菌VPA结果 Fig. 4 VPA results of bacteria in the Biliuhe Reservoir |
反映菌群与水体环境因子之间关系的RDA分析结果见图 5.从中可知, 菌群分布与环境因子之间的关系较复杂, 总之与微生物菌群相关的环境因子很多, 这些环境因子大多是自相关, 经过VIF分析, NO2--N、TP、SD、ADP、NO3--N、T、pH、Chla、TSS和DO这10个环境因子均能影响碧流河水库9个点位的微生物群落分布.其中根据箭头长度可以看出, SD、TSS、pH、NO2--N与微生物种类分布相关性较大, 秋季样品与SD显著正相关, 夏季样品与TSS, TP、ADP和NO2--N显著正相关, 与pH、DO、Chla和T呈显著负相关.春季和冬季样品与SD、NO3--N呈显著负相关.采用envfit函数检验每个环境因子的显著性, 如表 2, 其中r2越小, 表示该环境因子对物种分布影响越小, 环境因子对物种分布的影响大小依次为TSS、SD、pH、ADP、DO、TP(P=0.000 5)
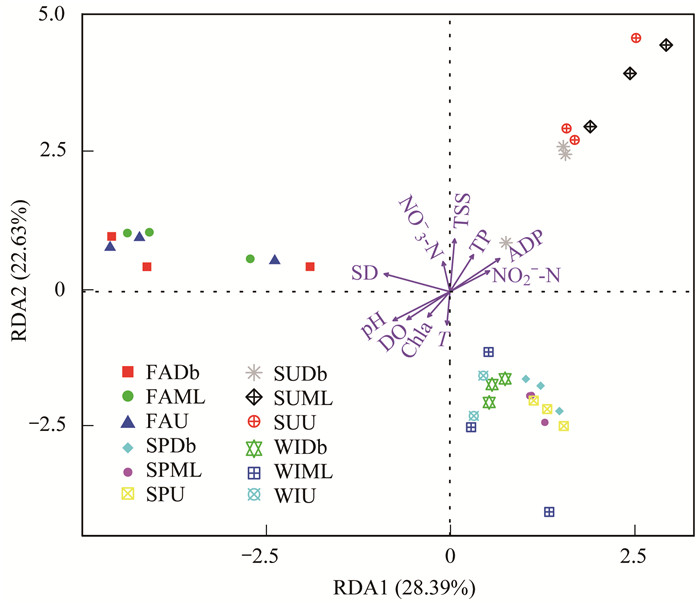
|
图 5 碧流河水库细菌菌群与水体理化因子的RDA分析 Fig. 5 RDA analysis for the correlation between bacteria flora and ecological factors in the Biliuhe Reservoir |
|
|
表 2 碧流河水库细菌与环境因子的RDA envfit分析1) Table 2 RDA envfit analysis for the correlation of bacteria and ecological factors in the Biliuhe Reservoir |
2.3 环境因子与微生物种丰富度之间的关系
用Spearman秩相关来研究碧流河水库环境因子与微生物种丰富度(α-多样性)之间关系, 及环境因子与物种之间的相互变化关系, 得到两两之间的相关性和显著性P值, 相互变化关系总结于图 6.优势菌属受水体环境因子的影响大于受到地形因子的影响, 其中Acidibacter、Brevundimonas、Pelomonas、Planctomyces、Synechococcus、hgcI_clade、Rheinheimera与温度、DO、NO2--N、COD显著正相关, Candidatus_Methylopumilus、Sediminibacterium、Prochlorothrix、Limnobacter、Massilia、Opitutus、Pedobacter、Delftia、marine_group、Pseudomonas、Sphingomonas、CL500-29_marine_group、Flavobacterium、CL500-3、Rheinheimera与SD、TN、TP、NO3--N、ADP显著正相关, 与TSS、Chla显著负相关.各菌群与经度、纬度和深度无显著性差异.
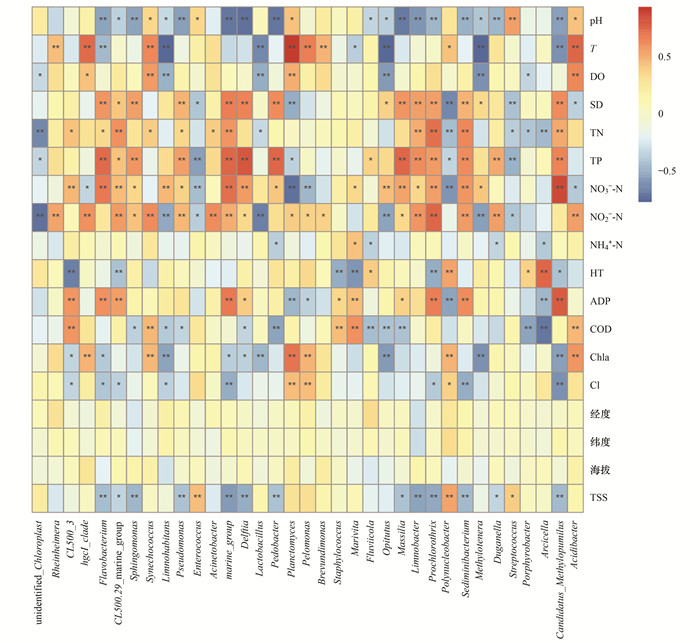
|
纵向为环境因子信息, 横向为物种信息, 中间热图对应的值为Spearman相关系数r, 介于-1~1之间, r < 0为负相关, r>0为正相关, *表示显著性检验P值< 0.05有显著性差异, **表示有极显著差异 图 6 碧流河水库细菌Spearman相关性分析 Fig. 6 Spearman correlation analysis of bacteria in the Biliuhe Reservoir |
众所周知, 多样性指数(如Shannon)是反映生态系统群落结构特征的重要指标, 本文通过16S rDNA高通量测序技术阐明了碧流河水库浮游细菌群落的季节性变化, Shannon多样性指数分析表明, 不同季节碧流河水库浮游细菌群落的多样性是不同的, 其多样性指数平均为6.9, 与其它地方水体相比较, 处于中间水平, 而应用16S rDNA高通量测序技术分析得到的细菌多样性指数与应用PCR-DGGE和T-RFLP技术分析得到的多样性指数相比, 数值更高, 因此能够更好说明碧流河水库细菌的群落结构.细菌门的相对丰度与他们的多样性有关, Møller等[26]进一步验证了Cohan[27]提出的假说, 一个细菌谱系在生境中的生态成功不是因为其中少数竞争力非常强的物种, 而是取决于它的多样性, 在细菌“种”内存在着不同的生态型, 造成生态型不同的原因则是环境的变化, 不同生态型之间的基因交换使物种的竞争力得到增强.这个假说可以解释能否发现更多环境中的优势物种, 多样性指数的大小尤为重要.高通量测序16S rDNA Ⅴ4区研究能较好地研究分析水体细菌群落多样性.碧流河水库9个采样点的浮游细菌多样性指数存在季节性变化, 冬季最低, 秋季最高, 每个样在菌群组成上冬春季与夏秋季差异显著.这与张雅洁等[28]和沈烽等[29]的研究结果类似.这可能是因为冬春季水体温度低, 细菌的新陈代谢能力与生长速率减慢, 夏秋季水体温度高, 细菌新陈代谢旺盛生长速率加快.碧流河水库上中下游细菌多样性空间差异不明显, 这与唐婧等[30]对南明河城区河段、李玉华等[31]对松花江水体、白洁等[32]对南海南部海域、王鹏等[33]对赣江南昌段、白洁等[34]对北黄海海域浮游细菌群落多样性的研究结果均存在空间差异.不同程度的人类活动对于河流细菌群落结构具有不同的影响, 流经城区的河流环境因子变化显著, 细菌丰度和多样性空间差异显著, 碧流河水库远离闹市, 远离现代工业区, 地域差异不明显, 受人类活动影响较小, 因此碧流河水库浮游细菌群落的空间差异不明显.
本研究通过碧流河水库水体中细菌16S rDNA Ⅴ4区高通量测序发现, 库水中细菌除了部分分类地位不明确的菌群和稀有菌群外, 共发现了细菌44门115纲184目368科865属, 表现出丰富的菌群结构.目前研究报道的不同水域水体中[28~37], 出现过23个门类细菌, 有蓝藻门、变形菌门、拟杆菌门、放线菌门、厚壁菌门、疣微菌门、浮霉菌门、绿弯菌门、黏胶球形菌门、候选门OP10、酸杆菌门、绿菌门、泉古菌门、芽单胞菌门、纤维杆菌门(Fibrobacteres)、梭杆菌门(Fusobacteria)、螺旋体门(Spirochaetes)、脱铁杆菌门(Deferribacteres)BRC1、OD1、OP10、SR1和TM7等.变形菌门, 蓝菌门, 放线菌门, 拟杆菌门是碧流河水库水体中的主要菌门, 这与大量研究水体细菌群落结构相似.
微生物群落的功能特征是其生境环境因子与环境理化性质紧密相关并相互限制影响[38~41].本研究RDA分析发现, TSS、SD、pH、ADP、DO、TP、NO2--N与微生物菌群多样性具有相关性, 是其主要的影响因子.近年来, 对于部分研究水库、河流、湖泊、海洋的浮游细菌群落结构及其与环境因子的关系总结如表 3.环境影响因子有温度、pH、溶解氧、氨氮、透明度、总氮、总磷、水深、DOC、磷酸盐等[28~34, 42].自然界氮素循环是各种元素循环的中心, 而微生物参与的氮循环是生态系统中氮循环的主要初级生产力.在各种水生态环境中, 溶解无机氮是异养细菌重要的N源, 但是过量的氮可能导致水体富营养化, 造成水质恶化. Haukka等[43]报道在淡水生态系统中添加总氮和磷酸盐后, 浮游细菌的群落结构发生变化.刘婷婷[44]在水源水库微生物种群结构和功能分析中得出亚硝酸盐氮对硫酸盐还原菌种群垂向分布影响显著.有研究表明, 河流水体细菌群落在不同时期的关键环境驱动因子不同, 其中悬浮颗粒物(TSS)浓度在不同水文时期是主要影响因子[45].曹新益等[46]的研究表明, 不同梯度的pH值会不同程度地影响各类细菌的生长.光照、溶氧和温度随着水深的增加而下降, 微生物群落的分布也会随之变化[2].因此, 水库功能微生物结构特征受到不同水质指标的综合调控.
|
|
表 3 不同水体微生物群落结构特征与关键驱动因子的比较 Table 3 Comparison of the features of the microbial community structure and its key driving factors in different water bodies |
促进微生物生长的环境因子各不相同, 本研究实验结果详细展示了各个环境因子与各微生物群落丰富度之间的关系. Acidibacter等随着温度、DO、NO2--N、COD等的升高而其丰富度增加; Candidatus_Methylopumilus等与SD、TN、TP、NO3--N、ADP正相关, 随着营养物质的增多而丰富度增加; 与TSS、Chla显著负相关, 随着悬浮颗粒的增加透明度降低, 叶绿素a增加水体富营养化加剧, 导致其丰富度降低.陈梦齐等[47]在象山港电厂温排水对浮游细菌的影响的研究中发现, 部分γ-变形菌分支的丰富度随水温升高而升高.陈兆进等[42]在丹江口水库的研究表明T、pH、NH4+-N、SD等环境因子丹江口浮游细菌群落有较大影响, 不同环境因子对不同菌群具有不同影响.微生物种丰富度与环境因子紧密相关, 其中温度、TSS、SD、pH、ADP、DO、TP等为影响微生物群落主要环境因子.
本研究解释了碧流河水库细菌群落构成与环境因子的相关性, 为饮用水源水库水质管理提供了重要数据, 但如何将这些信息有效用于监控水质有待进一步研究.
4 结论(1) 碧流河水库浮游细菌群落结构存在季节性变化, 细菌的群落多样性在冬季时最低, 秋季最高, 菌群组成上冬春季与夏秋季存在差异.
(2) 碧流河水库细菌主要菌门为变形菌门(Proteobacteria 34.16%)、蓝菌门(Cyanobacteria 24.35%)、放线菌门(Actinobacteria13.35%)和拟杆菌门(Bacteroidetes 12.42%).优势菌属为unidentified_Chloroplast 17.73%、hgcI_clade 5.95%和CL500-3 4.64%.
(3) 悬浮物(TSS)、透明度(SD)、酸碱度(pH)、活性磷(ADP)、溶氧(DO)、总磷(TP)、亚硝酸氮(NO2--N)是影响碧流河水库细菌群落结构的主要环境因子.
| [1] | 冯泽晖. 浅谈微生物技术在城市污水处理中的应用[J]. 科技创新与应用, 2017(12): 174. |
| [2] |
任丽娟, 何聃, 邢鹏, 等. 湖泊水体细菌多样性及其生态功能研究进展[J]. 生物多样性, 2013, 21(4): 421-432. Ren L J, He D, Xing P, et al. Bacterial diversity and ecological function in lake water bodies[J]. Biodiversity Science, 2013, 21(4): 421-432. |
| [3] |
郭非, 耿绪云, 李翔, 等. 天津近海可培养细菌多样性的初步调查[J]. 海洋科学, 2009, 33(6): 35-40. Guo F, Geng X Y, Li X, et al. Preliminary investigation of the culturable bacteria diversity in Tianjin coastal waters[J]. Marine Sciences, 2009, 33(6): 35-40. |
| [4] | Amann R I, Ludwig W, Schleifer K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J]. Microbiological Reviews, 1995, 59(1): 143-169. |
| [5] | Spring S, Schulze R, Overmann J, et al. Identification and characterization of ecologically significant prokaryotes in the sediment of freshwater lakes:molecular and cultivation studies[J]. FEMS Microbiology Reviews, 2000, 24(5): 573-590. DOI:10.1111/j.1574-6976.2000.tb00559.x |
| [6] |
戴欣, 周惠, 陈月琴, 等. 中国南海南沙海区沉积物中细菌16S rDNA多样性的初步研究[J]. 自然科学进展, 2002, 12(5): 479-484. Dai X, Zhou H, Chen Y Q, et al. A preliminary study on 16S rDNA diversity of bacteria in the Nansha marine sediment, the South China Sea[J]. Progress in Nature Science, 2002, 12(5): 479-484. |
| [7] |
李筠, 周宏霞, 刘佳琳, 等. 青岛近岸特征环境中海洋异养细菌的分布规律及其分子鉴定[J]. 中国海洋大学学报, 2006, 36(6): 965-970. Li J, Zhou H X, Liu J L, et al. Distribution and identification of marine heterotrophic bacteria in different sea areas close to the shore of Qingdao[J]. Periodical of Ocean University of China, 2006, 36(6): 965-970. |
| [8] | Yang Y Y, Dai Y, Wu Z, et al. Temporal and spatial dynamics of archaeal communities in two freshwater lakes at different trophic status[J]. Frontiers in Microbiology, 2016, 7: 451. |
| [9] |
穆春华, 包振民, 陈刚, 等. 西北太平洋沉积物中细菌多样性的研究[J]. 海洋学报, 2006, 28(4): 121-128. Mu C H, Bao Z M, Chen G, et al. Bacterial diversity in the sediments collected from the Northwest Pacific[J]. Acta Oceanologica Sinica, 2006, 28(4): 121-128. |
| [10] | Laque T, Farjalla V F, Rodado A S, et al. Spatiotemporal variation of bacterial community composition and possible controlling factors in tropical shallow lagoons[J]. Microbial Ecology, 2010, 59(4): 819-829. DOI:10.1007/s00248-010-9642-5 |
| [11] | Brambilla E, Hippe H, Hagelstein A, et al. 16S rDNA Diversity of cultured and uncultured prokaryotes of a mat sample from Lake Fryxell, McMurdo Dry Valleys, Antarctica[J]. Extremophiles, 2001, 5(1): 23-33. DOI:10.1007/s007920000169 |
| [12] |
谢在刚. 碧流河水库水体富营养化控制对策探讨[J]. 吉林水利, 2009(1): 38-40. Xie Z G. Discussion on the eutrophication control Countermeasures in Biliuhe reservoir[J]. Jilin Water Resources, 2009(1): 38-40. |
| [13] | 大连市碧流河水库管理局. 碧流河水库志[M]. 大连: 大连出版社, 1996. |
| [14] |
王广森. 大连市碧流河水库水源涵养林工程设计浅析[J]. 水资源开发与管理, 2016, 7(6): 78-80. Wang G S. On design of water source conservation forest project in Dalian Biliuhe Reservoir[J]. Water Resources Development and Management, 2016, 7(6): 78-80. |
| [15] |
郑焕春, 周青. 微生物在富营养化水体生物修复中的作用[J]. 中国生态农业学报, 2009, 17(1): 197-202. Zheng H C, Zhou Q. Function of microorganism in bioremediation of eutrophic water[J]. Chinese Journal of Eco-Agriculture, 2009, 17(1): 197-202. |
| [16] | 国家环境保护总局. 水和废水监测分析方法[M]. 第四版. 北京: 中国环境科学出版社, 2002. |
| [17] | Anderson M J. A new method for non-parametric multivariate analysis of variance[J]. Austral Ecology, 2001, 26(1): 32-46. |
| [18] | McArdle B H, Anderson M J. Fitting multivariate models to community data:A comment on distance-based redundancy analysis[J]. Ecology, 2001, 82(1): 290-297. DOI:10.1890/0012-9658(2001)082[0290:FMMTCD]2.0.CO;2 |
| [19] | Caporaso J G, Kuczynski J, Stombauqh J, et al. QⅡME allows analysis of high-throughput community sequencing data[J]. Nature methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [20] | Edgar R C, Haas B J, Clemente J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381 |
| [21] | Haas B J, Gevers D, Earl A M, et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J]. Genome Research, 2011, 21(3): 494-504. DOI:10.1101/gr.112730.110 |
| [22] | Edgar R C. UPARSE:highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [23] | Wang Q, Garrity G M, Tiedje J M, et al. Na?ve Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. DOI:10.1128/AEM.00062-07 |
| [24] | Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41(D1): 590-596. |
| [25] | Edgar R C. MUSCLE:multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Research, 2004, 32(5): 1792-1797. DOI:10.1093/nar/gkh340 |
| [26] | Møller A K, Søborg D A, Al-Soud W A, et al. Bacterial community structure in High-Arctic snow and freshwater as revealed by pyrosequencing of 16S rRNA genes and cultivation[J]. Polar Research, 2013, 32(1): 17390. DOI:10.3402/polar.v32i0.17390 |
| [27] | Cohan F M. What are bacterial species?[J]. Annual Review of Microbiology, 2002, 56: 457-487. DOI:10.1146/annurev.micro.56.012302.160634 |
| [28] |
张雅洁, 李珂, 朱浩然, 等. 北海湖微生物群落结构随季节变化特征[J]. 环境科学, 2017, 38(8): 3319-3329. Zhang Y J, Li K, Zhu H R, et al. Community structure of microorganisms and its seasonal variation in Beihai lake[J]. Environmental Science, 2017, 38(8): 3319-3329. |
| [29] |
沈烽, 赵大勇, 黄睿, 等. 南京玄武湖浮游细菌群落结构的季节变化及其与环境因子的关系[J]. 湖泊科学, 2017, 29(3): 662-669. Shen F, Zhao D Y, Huang R, et al. Seasonal variation of bacterioplankton community structure in Xuanwu Lake (Nanjing) and its relationship with environmental factors[J]. Journal of Lake Sciences, 2017, 29(3): 662-669. DOI:10.18307/2017.0315 |
| [30] |
唐婧, 徐小蓉, 商传禹, 等. 南明河城区河段细菌多样性与环境因子的关系[J]. 微生物学报, 2015, 55(8): 1050-1059. Tang J, Xu X R, Shang C Y, et al. Association of bacterial diversity in city area of Nanming river with environmental factors[J]. Acta Microbiologica Sinica, 2015, 55(8): 1050-1059. |
| [31] |
李玉华, 许其功, 赵越, 等. 松花湖水体中不同空间分布的细菌群落结构分析[J]. 农业环境科学学报, 2013, 32(4): 764-770. Li Y H, Xu Q G, Zhao Y, et al. Bacterial community structure in different spatial distribution of Songhua lake[J]. Journal of Agro-Environment Science, 2013, 32(4): 764-770. |
| [32] |
白洁, 刘小沙, 侯瑞, 等. 南海南部海域浮游细菌群落特征及影响因素研究[J]. 中国环境科学, 2014, 34(11): 2950-2957. Bai J, Liu X S, Hou R, et al. Community structure and influencing factors of bacterioplankton in the southern South China Sea[J]. China Environmental Science, 2014, 34(11): 2950-2957. |
| [33] |
王鹏, 陈波, 李传琼, 等. 赣江南昌段丰水期细菌群落特征[J]. 中国环境科学, 2016, 36(8): 2453-2462. Wang P, Chen B, Li C Q, et al. Bacterial communities in Nanchang section of the Ganjiang River in wet seaon[J]. China Environmental Science, 2016, 36(8): 2453-2462. |
| [34] |
白洁, 李海燕, 赵阳国. 黄海北部不同站位海洋细菌群落分布特征[J]. 微生物学报, 2009, 49(3): 343-350. Bai J, Li H Y, Zhao Y G. Bacterial distribution at different stations in the Northern Yellow Sea[J]. Acta Microbiologica Sinica, 2009, 49(3): 343-350. |
| [35] | Dong Y, Zhao Y, Zhang W Y, et al. Bacterial diversity and community structure in the East China Sea by 454 sequencing of the 16S rRNA gene[J]. Chinese Journal of Oceanology and Limnology, 2014, 32(3): 527-541. DOI:10.1007/s00343-014-3215-2 |
| [36] | Newton R J, Jones S E, Eiler A, et al. A guide to the natural history of freshwater lake bacteria[J]. Microbiology and Molecular Biology Reviews, 2011, 75(1): 14-49. DOI:10.1128/MMBR.00028-10 |
| [37] | Zwart G, Crump B C, Kamst-van Agterveld M P, et al. Typical freshwater bacteria:an analysis of available 16S rRNA gene sequences from plankton of lakes and rivers[J]. Aquatic Microbial Ecology, 2002, 28(2): 141-155. |
| [38] |
米亮, 王光华, 金剑, 等. 黑土微生物呼吸及群落功能多样性对温度的响应[J]. 应用生态学报, 2010, 21(6): 1485-1491. Mi L, Wang G H, Jin J, et al. Responses of black soil's microbial respiration and community functional diversity to temperature[J]. Chinese Journal of Applied Ecology, 2010, 21(6): 1485-1491. |
| [39] | Gomez E, Ferreras L, Toresani S. Soil bacterial functional diversity as influenced by organic amendment application[J]. Bioresource Technology, 2006, 97(13): 1484-1489. DOI:10.1016/j.biortech.2005.06.021 |
| [40] |
黎宁, 李华兴, 朱凤娇, 等. 菜园土壤微生物生态特征与土壤理化性质的关系[J]. 应用生态学报, 2006, 17(2): 285-290. Li N, Li H X, Zhu F J, et al. Relationships between soil microbial ecological characteristics and physical-chemical properties of vegetable garden soil[J]. Chinese Journal of Applied Ecology, 2006, 17(2): 285-290. |
| [41] | Manzoni S, Jackson R B, Trofymow J A, et al. The global stoichiometry of litter nitrogen mineralization[J]. Science, 2008, 321(5889): 684-686. DOI:10.1126/science.1159792 |
| [42] |
陈兆进, 丁传雨, 朱静亚, 等. 丹江口水库枯水期浮游细菌群落组成及影响因素研究[J]. 中国环境科学, 2017, 37(1): 336-344. Chen Z J, Ding C Y, Zhu J Y, et al. Community structure and influencing factors of bacterioplankton during low water periods in Danjiangkou Reservoir[J]. China Environmental Science, 2017, 37(1): 336-344. |
| [43] | Haukka K, Kolmonen E, Hyder R, et al. Effect of nutrient loading on bacterioplankton community composition in Lake Mesocosms[J]. Microbial Ecology, 2006, 51(2): 137-146. DOI:10.1007/s00248-005-0049-7 |
| [44] |
刘婷婷. 水源水库微生物种群结构与功能研究[D]. 西安: 西安建筑科技大学, 2013. Liu T T. Microbial community structure and function of drinking water reservoir[D]. Xi'an: Xi'an University of Architecture and Technology, 2013. |
| [45] |
刘睿, 吴巍, 周孝德, 等. 渭河浮游细菌群落结构特征及其关键驱动因子[J]. 环境科学学报, 2017, 37(3): 934-944. Liu R, Wu W, Zhou X D, et al. Bacterioplankton community structure in Weihe river and its relationship with environmental factors[J]. Acta Scientiae Circumstantiae, 2017, 37(3): 934-944. |
| [46] |
曹新益, 徐慧敏, 王司辰, 等. 南京莫愁湖与紫霞湖浮游细菌群落结构的季节性变化及其与环境因子的关系[J]. 化学与生物工程, 2016, 33(12): 19-26, 30. Cao X Y, Xu M H, Wang S C, et al. Seasonal variation of bacterioplankton community structure and its relationship with environmental factors of Mochou Lake and Zixia Lake in Nanjing[J]. Chemistry & Bioengineering, 2016, 33(12): 19-26, 30. DOI:10.3969/j.issn.1672-5425.2016.12.004 |
| [47] |
陈梦齐, 刘紫丹, 戴文芳, 等. 象山港电厂温排水增温对浮游细菌群落空间分布的影响[J]. 生态学报, 2016, 36(20): 6574-6582. Chen M Q, Liu Z D, Dai W F, et al. Effects of thermal discharge on the spatial distribution of bacterioplankton community near a power plant in Xiangshan Bay[J]. Acta Ecologica Sinica, 2016, 36(20): 6574-6582. |
 2018, Vol. 39
2018, Vol. 39


