我国是全球抗生素药物生产和使用大国[1].据统计[2], 我国2013年抗生素使用量高达16.2万t.由于抗生素的大规模使用甚至滥用, 造成大量残留物及其代谢产物进入生态环境[3~6].越来越多的研究报道[7~9], 抗生素是造成细菌抗药性迅速演变和散播的主要原因, 而抗生素抗性基因(ARGs)作为一种新型污染物[10, 11], 已在不同环境介质中被频繁检出[1, 12~16].污水处理剩余污泥是ARGs的一个重要储存库[17], 近年来发现在污泥资源化土地利用过程中会造成ARGs的广泛散播[18].因此, 如何有效地削减污泥中ARGs存在水平, 是控制其在污泥土地利用过程中散播的关键问题.
厌氧消化是目前国内外污泥安全处置的主流工艺[19], 已有研究报道针对其过程参数对ARGs的去除变化影响进行了相关探讨[20~22].前期研究结果表明[23], 在剩余污泥和餐厨废物组成的联合厌氧消化反应体系中, 零价铁(Fe0)的加入不仅可以加快反应进程, 且能够促进四环素抗性基因(TC-ARGs)和第一类整合子intI1基因的削减.尽管如此, 研究同时发现[24], 核糖体保护基因tetM、tetO和tetW丰度在厌氧消化过程中出现反弹现象, 推测可能是由于基因水平转移过程的发生所造成的, 但具体原因还有待进一步研究.
目前, 质粒DNA接合作用、胞外DNA转换作用和噬菌体DNA转导作用是ARGs在细菌微生物之间水平转移的主要机制, 其中质粒DNA接合作用被认为对ARGs的散播起着最为关键的作用[25].因此, 本研究考察了不同Fe0投加量对污泥厌氧消化过程中TC-ARGs和intI1基因丰度的消长影响, 分析了质粒DNA接合作用对TC-ARGs水平转移过程的作用影响, 并探讨了intI1基因与TC-ARGs之间的相关关系, 通过掌握TC-ARGs在污泥厌氧消化过程中的内在演变过程, 以期为其强化削减提供理论基础和技术支撑.
1 材料与方法 1.1 实验药品本研究所使用的实验药品均为分析纯, 除厌氧指示剂购自美国Sigma-Aldrich公司外, 其余购自国药集团化学试剂有限公司.
厌氧培养液组成如下(g·L-1):KCl 1.3, KH2PO4 0.2, CaCl2·2H2O 0.1, MgCl2·6H2O 0.18, NaCl 1.17, NH4Cl 0.5, NaHCO3 2.5, 以及1 mL质量浓度为1 g·L-1的厌氧指示剂溶液和1 mL微量盐溶液.
厌氧指示剂溶液的配置:称取1 g厌氧指示剂溶于1 L超纯水.
微量盐溶液组成如下(g·L-1):MnCl2·4H2O 0.5, CoCl2·6H2O 0.03, NiCl2·6H2O 0.01, ZnCl2 0.05, H3BO3 0.05, CuCl2 0.5, NaMoO4·2H2O 0.05.
1.2 污泥厌氧消化体系的建立及运行接种污泥取自上海松江污水处理厂二沉池剩余污泥, VSS质量浓度约为8.00 g·L-1±0.26 g·L-1.污泥厌氧消化反应器采用容积为250 mL血清瓶, 接种污泥加入量为100 mL, 同时加入100 mL厌氧培养液, 然后使用高纯氮气吹脱5 min.厌氧反应器共设两批平行装置, 每批共有5组, 分别标记为AD-0、AD-1、AD-2、AD-3和AD-4, 每组设置6个血清瓶, 其中AD-0为对照组, 其余4组投加Fe0(10~40 μm), 加入量(以Fe0/VSS计, 下同)分别为0.15 g·g-1(AD-1)、0.25 g·g-1(AD-2)、1.00 g·g-1(AD-3)和3.75 g·g-1(AD-4).将所有厌氧反应器置于恒温摇床内(SPH-1102C, 上海世平实验设备有限公司), 避光, 控制摇速为120 r·min-1, 反应温度为55℃±1℃, pH为7.2~7.8(S220, Mettler Toledo).污泥厌氧消化反应周期为35 d, 每隔7 d进行采样检测分析.
1.3 DNA提取基因组DNA提取:按照预设采样时间, 采集一定量的泥水混合物在4℃和6 000 r·min-1下离心10 min, 倒去上清液, 收集沉淀物.采用TIANamp Soil DNA Kit(TIANGEN)对基因组DNA进行提取, 提取步骤参照试剂盒操作说明书, 所提取的DNA使用1%琼脂糖凝胶电泳和Qubit 2.0核酸蛋白测定仪(Invitrogen)检测其纯度和浓度.
质粒组DNA提取:采用QIAprep Spin Miniprep Kit(Qiagen)从基因组DNA中提取质粒组DNA, 并使用Plasmid-SafeTM ATP-Dependent DNase(Epicentre)对其进行纯化, 以确保其纯度.
1.4 目标ARGs定量检测采用qPCR(LightCycler® 96, Roche)对目标基因进行定量检测分析, 各目标基因所用扩增引物序列、扩增子大小、退火温度、反应体系及反应程序详见文献[23, 24].利用熔解曲线(65~95℃, 每0.5℃读数)分析扩增产物的特异性, 从而优化qPCR反应条件和引物浓度, 消除引物二聚体和非特异性扩增的干扰影响.每组样品设3个平行样, 并使用无菌水作为阴性对照.
1.5 数据分析采用Origin 8.5软件进行数据处理, 并利用SPSS 22.0软件进行统计学分析, 设定统计检验显著性水平P=0.05, 若P < 0.05认为具有显著相关性, 反之则认为相关性不显著.
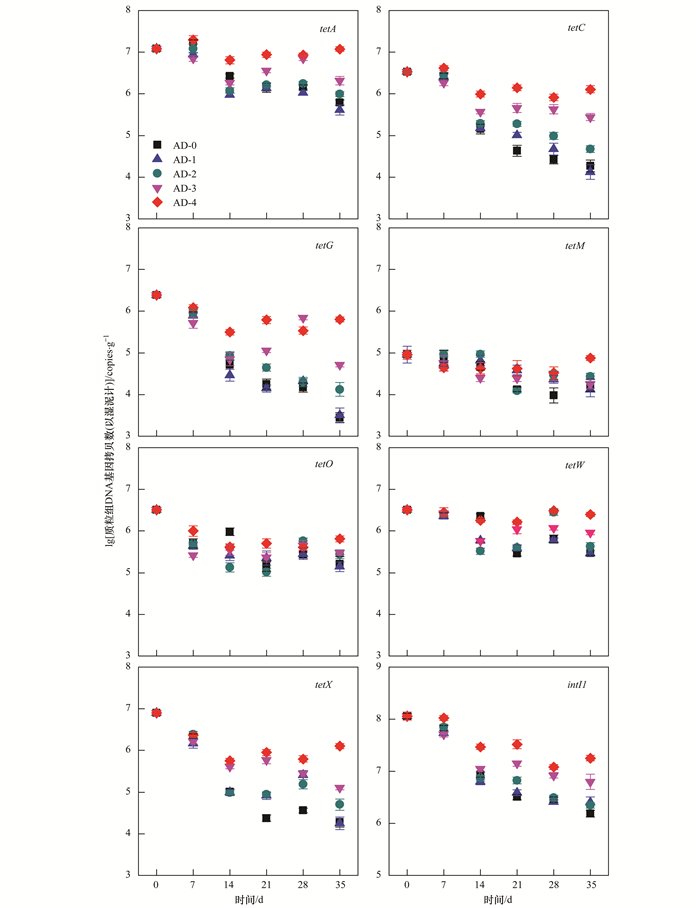
2 结果及讨论 2.1 污泥厌氧消化过程中TC-ARGs和intI1基因的变化特征图 1为目标TC-ARGs和intI1基因丰度在污泥厌氧消化过程中的变化情况.从中可以看出, 在整个反应周期35 d内, AD-0反应器中所有TC-ARGs丰度均得到一定程度的削减, 其中tetX基因降幅最大, 达2.4个数量级, 而tetM基因降幅最小, 基本无去除效果.此外, 不同抗性机制的TC-ARGs在污泥厌氧消化过程中的变化特征各有差异, 其中外排泵基因(tetA、tetC和tetG)和酶修饰基因(tetX)降幅较大, 达1.0~2.4个数量级; 相比之下, 核糖体保护基因(tetM、tetO和tetW)削减效果较差, 最大降幅约为0.8个数量级(tetW), 这与前期研究结果相似[23], 且tetM和tetO基因丰度在整个反应周期内呈现出先降低后升高的变化趋势, 表明污泥厌氧反应时间的延长不利于其有效削减. Ma等[26]的研究同样发现, tetC、tetG和tetX基因丰度随着污泥停留时间的延长而显著降低, 而tetW基因丰度相比初始时更高.与此同时, 除tetO和tetX基因外, 其余TC-ARGs丰度在厌氧消化反应初期(7 d)先升高(图 1), 而后呈现逐渐降低的趋势, 这可能是由于TC-ARGs发生水平转移, 或携带有TC-ARGs的细菌微生物在消化反应初期处于增长期所造成的[27], 这在后续章节将进一步探讨.随着厌氧反应过程的继续进行, TC-ARGs(除tetC和tetG外)到第21 d时已基本降至最低并维持稳定.前期研究结果显示[23], TC-ARGs在污泥厌氧消化周期8 d内呈现逐渐降低的趋势, 这与本研究结果有所不同, 分析原因推测可能是因为厌氧反应底物的不同所导致的, 前期研究中采用的反应底物为餐厨垃圾和剩余污泥的混合体系, 餐厨垃圾一定程度上可以稀释剩余污泥中的有毒成分, 均衡厌氧微生物对营养物质的需求, 提高消化效率, 促进消化反应快速达到稳定[28, 29].

|
图 1 污泥厌氧消化过程中TC-ARGs和intI1基因丰度变化特征 Fig. 1 Quantities of TC-ARGs and intI1 genes during anaerobic sludge digestion process |
此外, 由于整合子基因能够捕获外源性基因, 通过转座子或接合性质粒进行ARGs的水平转移传播[30], 因此本研究同样检测了intI1基因在污泥厌氧消化过程中的变化行为.由图 1可知, intI1基因在接种污泥中的丰度高于TC-ARGs, 达7.43×108 copies·g-1, 与大部分TC-ARGs变化特征类似, intI1基因丰度在反应初期先升高, 而后逐渐降低, 尽管在整个反应周期内降幅达1.8个数量级, 但其削减过程并不显著(P=0.15), 表明intI1基因在污泥厌氧消化过程中相对稳定, 对ARGs水平转移的发生可能具有潜在的促进作用.
2.2 Fe0对污泥厌氧消化过程中TC-ARGs和intI1基因的消长影响Fe0是一种性能优异的还原物质, 近年来已被尝试用于强化污泥厌氧消化进程[31], 并已证实其能够促进有机物水解、原位产氢和产甲烷等有益效果[32].有研究报道[33], Fe0暴露能够导致小鼠肠道细菌微生物群落中tetM、tetO和tetQ基因丰度的变化.前期研究结果表明[23], 一定投加量的Fe0能够增强TC-ARGs和intI1基因的削减效果.然而, 本研究结果发现, 随着Fe0投加量的增大, 尽管污泥厌氧消化过程产物VFAs产量相应升高(数据未列出), 但TC-ARGs和intI1基因丰度降幅却相应减小, 尤其当Fe0投加量为3.75 g·g-1时, TC-ARGs中外排泵基因、核糖体保护基因和酶修饰基因丰度的降幅分别仅为0.09~0.31、0.08~0.45和0.77个数量级, intI1基因降幅也仅为0.60个数量级(图 1).此外, Fe0的加入除在厌氧消化反应初期(7 d)对TC-ARGs和intI1基因丰度具有小幅度削减效果外, 随着厌氧反应的持续进行, TC-ARGs和intI1基因丰度整体上呈现增长的趋势, 推测可能是因为Fe0在促进污泥厌氧反应进程的同时能够强化TC-ARGs的水平转移, 且总体上随着Fe0加入量的增大而呈现加强的趋势.与此同时, 厌氧消化接种底物类型和反应周期的差异同样会影响TC-ARGs丰度的变化行为, 这可能也是导致本研究与前期研究结果有所不同的重要因素.
2.3 Fe0对污泥厌氧消化过程中TC-ARGs水平转移机制过程作用影响根据2.2节结果分析推测, Fe0能够促进污泥厌氧反应过程中TC-ARGs的水平转移, 因此本研究检测了质粒组DNA中TC-ARGs和intI1基因丰度在污泥厌氧消化过程中的变化情况, 如图 2所示.
|
图 2 污泥厌氧消化过程质粒组DNA中TC-ARGs和intI1基因丰度变化特征 Fig. 2 Quantities of TC-ARGs and intI1 genes carried by plasmid DNA during anaerobic sludge digestion process |
从图 2中可以看出, 质粒组DNA中TC-ARGs和intI1基因丰度变化趋势与基因组DNA相类似.在整个反应周期35 d内, AD-0反应器中所有TC-ARGs和intI1基因丰度均得到不同程度削减, 其中外排泵基因、核糖体保护基因和酶修饰基因丰度降幅分别为1.3~2.9、0.7~1.2和2.6个数量级, 而intI1基因降幅约为1.9个数量级, 表明污泥厌氧消化过程能够有效地削减质粒组DNA中TC-ARGs和intI1基因丰度, 这可能与质粒组DNA本身质量浓度的显著削减有关(降幅达82.5%).此外, 不同Fe0投加量对质粒组DNA中TC-ARGs和intI1基因丰度的变化影响不尽相同.随着Fe0投加量的增大, TC-ARGs中外排泵基因和酶修饰基因, 以及intI1基因丰度削减幅度基本呈现出逐渐降低的趋势, 特别是在Fe0投加量为3.75 g·g-1时, 其降幅分别约为0.02~0.6、0.8和0.8个数量级.相比之下, Fe0的加入对核糖体保护基因丰度的作用影响较小, 表明其在污泥厌氧消化过程中存在较为稳定.
由上述分析可知, Fe0对污泥厌氧消化过程质粒组DNA中TC-ARGs的散播和转移具有促进作用, 分析原因推测可能是因为Fe0在厌氧环境中会转化为Fe(OH)2[32], 其良好的絮凝性能可以促使污泥絮体结合更加紧密, 增强污泥微生物细胞之间的相互接触, 从而强化TC-ARGs的水平转移.此外, Fe0的加入能够显著提高VFAs的产生量[23, 31], 降低反应体系pH值, 同样可能会促进污泥厌氧反应过程中TC-ARGs的水平转移[20].
2.4 相关性分析由于整合子基因能够促进外源性基因的获得和表达, 并通过可移动遗传因子、质粒和转座子等在细菌群落之间进行传播[29].从表 1数据分析可知, 在AD-0反应器中, intI1基因与TC-ARGs之间均存在显著正相关性(P < 0.05), 表明污泥厌氧消化过程中intI1基因与TC-ARGs的变化行为密切相关.此外, intI1与tetG基因在所有反应器中均具有显著正相关性(P < 0.05), 表明intI1基因对tetG基因的演变特征具有重要影响, 这与前期研究结果是一致的[24]. Zhang等[22]的研究同样发现, 污泥与食品垃圾联合厌氧消化过程中intI1和tetG基因之间显著相关.相比之下, 在Fe0存在时, intI1与tetM基因之间相关性较差, 这可能是因为除质粒DNA转移作用外, 胞外DNA转换作用和噬菌体DNA转导作用对污泥消化过程中tetM基因的水平转移具有重要影响, 这有待后期进一步深入研究.
|
|
表 1 污泥厌氧消化过程中TC-ARGs与intI1基因相关性分析1) Table 1 Correlation analysis between quantities of TC-ARGs and intI1 gene in anaerobic sludge digestion process |
3 结论
(1) 目标TC-ARGs和intI1基因丰度在污泥厌氧消化过程中均呈现出不同程度的降低趋势, 但不同抗性机制的TC-ARGs变化特征有所差异, 其中核糖体保护基因tetM、tetO和tetW的存在相对稳定.
(2) Fe0的加入虽然能够强化污泥厌氧消化反应进程, 但对TC-ARGs和intI1基因丰度削减影响不显著, 且随着Fe0投加量的增加, 目标基因丰度反而出现升高的趋势.
(3) Fe0对污泥厌氧消化过程质粒组DNA中TC-ARGs和intI1基因丰度消长变化特征与基因组DNA相类似, 表明Fe0对质粒DNA接合转移TC-ARGs具有一定促进作用.
(4) 由相关性分析结果可知, 所有反应器中tetG和intI1基因之间均具有显著正相关关系, 表明intI1基因在污泥厌氧消化过程中对tetG基因的演变行为具有重要影响.
| [1] | Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440. DOI:10.1073/pnas.1222743110 |
| [2] | Zhang Q Q, Ying G G, Pan C G, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China:source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 2015, 49(11): 6772-6782. |
| [3] | Rizzo L, Manaia C, Merlin C, et al. Urban wastewater treatment plants as hotspots for antibiotic resistant bacteria and genes spread into the environment:a review[J]. Science of the Total Environment, 2013, 447: 345-360. DOI:10.1016/j.scitotenv.2013.01.032 |
| [4] | Du J, Zhao H X, Liu S S, et al. Antibiotics in the coastal water of the South Yellow Sea in China:occurrence, distribution and ecological risks[J]. Science of the Total Environment, 2017, 595: 521-527. DOI:10.1016/j.scitotenv.2017.03.281 |
| [5] | Xu Y, Guo C S, Luo Y, et al. Occurrence and distribution of antibiotics, antibiotic resistance genes in the urban rivers in Beijing, China[J]. Environmental Pollution, 2016, 213: 833-840. DOI:10.1016/j.envpol.2016.03.054 |
| [6] |
金磊, 姜蕾, 韩琪, 等. 华东地区某水源水中13种磺胺类抗生素的分布特征及人体健康风险评价[J]. 环境科学, 2016, 37(7): 2515-2521. Jin L, Jiang L, Han Q, et al. Distribution characteristics and health risk assessment of thirteen sulfonamides antibiotics in a drinking water source in East China[J]. Environmental Science, 2016, 37(7): 2515-2521. |
| [7] | Gao P, Munir M, Xagoraraki I. Correlation of tetracycline and sulfonamide antibiotics with corresponding resistance genes and resistant bacteria in a conventional municipal wastewater treatment plant[J]. Science of the Total Environment, 2012, 421-422: 173-183. DOI:10.1016/j.scitotenv.2012.01.061 |
| [8] | Zhang Q Q, Jia A, Wan Y, et al. Occurrences of three classes of antibiotics in a natural river basin:association with antibiotic-resistant Escherichia coli[J]. Environmental Science & Technology, 2014, 48(24): 14317-14325. |
| [9] | Zhang Y J, Boyd S A, Teppen B J, et al. Role of tetracycline speciation in the bioavailability to Escherichia coli for uptake and expression of antibiotic resistance[J]. Environmental Science & Technology, 2014, 48(9): 4893-4900. |
| [10] | Pruden A, Pei R t, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants:studies in Northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [11] |
罗义, 周启星. 抗生素抗性基因(ARGs)——一种新型环境污染物[J]. 环境科学学报, 2008, 28(8): 1499-1505. Luo Y, Zhou Q X. Antibiotic resistance genes (ARGs) as emerging pollutants[J]. Acta Scientiae Circumstantiae, 2008, 28(8): 1499-1505. |
| [12] | Su J Q, Wei B, Xu C Y, et al. Functional metagenomic characterization of antibiotic resistance genes in agricultural soils from China[J]. Environment International, 2014, 65: 9-15. DOI:10.1016/j.envint.2013.12.010 |
| [13] | Gao P, He S, Huang S L, et al. Impacts of coexisting antibiotics, antibacterial residues, and heavy metals on the occurrence of erythromycin resistance genes in urban wastewater[J]. Applied Microbiology and Biotechnology, 2015, 99(9): 3971-3980. DOI:10.1007/s00253-015-6404-9 |
| [14] | Bengtsson-Palme J, Larsson D G J. Antibiotic resistance genes in the environment:prioritizing risks[J]. Nature Reviews Microbiology, 2015, 13(6): 396. |
| [15] |
李侃竹, 吴立乐, 黄圣琳, 等. 污水处理厂中红霉素抗药性基因的污染特征及选择性因子[J]. 环境科学, 2014, 35(12): 4589-4595. Li K Z, Wu L L, Huang S L, et al. Investigation of pollution characteristics of erythromycin resistance genes in a sewage treatment plant and the relevant selective factors[J]. Environmental Science, 2014, 35(12): 4589-4595. |
| [16] |
张兰河, 王佳佳, 哈雪姣, 等. 北京地区菜田土壤抗生素抗性基因的分布特征[J]. 环境科学, 2016, 37(11): 4395-4401. Zhang L H, Wang J J, Ha X J, et al. Distribution characteristics of antibiotic resistance genes in vegetable soils in Beijing[J]. Environmental Science, 2016, 37(11): 4395-4401. |
| [17] | Calero-Cáceres W, Melgarejo A, Colomer-Lluch M, et al. Sludge as a potential important source of antibiotic resistance genes in both the bacterial and bacteriophage fractions[J]. Environmental Science & Technology, 2014, 48(13): 7602-7611. |
| [18] | Xie W Y, McGrath S P, Su J Q, et al. Long-term impact of field applications of sewage sludge on soil antibiotic resistome[J]. Environmental Science & Technology, 2016, 50(23): 12602-12611. |
| [19] | Appels L, Baeyens J, Degrève J, et al. Principles and potential of the anaerobic digestion of waste-activated sludge[J]. Progress in Energy and Combustion Science, 2008, 34(6): 755-781. DOI:10.1016/j.pecs.2008.06.002 |
| [20] | Huang H N, Chen Y G, Zheng X, et al. Distribution of tetracycline resistance genes in anaerobic treatment of waste sludge:the role of pH in regulating tetracycline resistant bacteria and horizontal gene transfer[J]. Bioresource Technology, 2016, 218: 1284-1289. DOI:10.1016/j.biortech.2016.07.097 |
| [21] | Pei J, Yao H, Wang H, et al. Comparison of ozone and thermal hydrolysis combined with anaerobic digestion for municipal and pharmaceutical waste sludge with tetracycline resistance genes[J]. Water Research, 2016, 99: 122-128. DOI:10.1016/j.watres.2016.04.058 |
| [22] | Zhang J Y, Chen M X, Sui Q W, et al. Fate of antibiotic resistance genes and its drivers during anaerobic co-digestion of food waste and sewage sludge based on microwave pretreatment[J]. Bioresource Technology, 2016, 217: 28-36. DOI:10.1016/j.biortech.2016.02.140 |
| [23] |
魏欣, 薛顺利, 杨帆, 等. 零价铁对污泥高温厌氧消化过程中四环素抗性基因及第一类整合子的消减影响[J]. 环境科学, 2017, 38(2): 697-702. Wei X, Xue S L, Yang F, et al. Effect of zero valent iron on the decline of tetracycline resistance genes and class 1 integrons during thermophilic anaerobic digestion of sludge[J]. Environmental Science, 2017, 38(2): 697-702. |
| [24] | Gao P, Gu C C, Wei X, et al. The role of zero valent iron on the fate of tetracycline resistance genes and class 1 integrons during thermophilic anaerobic co-digestion of waste sludge and kitchen waste[J]. Water Research, 2017, 111: 92-99. DOI:10.1016/j.watres.2016.12.047 |
| [25] | von Wintersdorff C J, Penders J, van Niekerk J M, et al. Dissemination of antimicrobial resistance in microbial ecosystems through horizontal gene transfer[J]. Frontiers in Microbiology, 2016, 7: 173. |
| [26] | Ma Y J, Wilson C A, Novak J T, et al. Effect of various sludge digestion conditions on sulfonamide, macrolide, and tetracycline resistance genes and class I integrons[J]. Environmental Science & Technology, 2011, 45(18): 7855-7861. |
| [27] | Ghosh S, Ramsden S J, LaPara T M, et al. The role of anaerobic digestion in controlling the release of tetracycline resistance genes and class 1 integrons from municipal wastewater treatment plants[J]. Applied Microbiology and Biotechnology, 2009, 84(4): 791-796. DOI:10.1007/s00253-009-2125-2 |
| [28] |
付胜涛, 于水利, 严晓菊, 等. 剩余活性污泥和厨余垃圾的混合中温厌氧消化[J]. 环境科学, 2006, 27(7): 1459-1463. Fu S T, Yu S L, Yan X J, et al. Co-digestion of waste activated sludge and kitchen garbage[J]. Environmental Science, 2006, 27(7): 1459-1463. |
| [29] |
段妮娜, 董滨, 李江华, 等. 污泥和餐厨垃圾联合干法中温厌氧消化性能研究[J]. 环境科学, 2013, 34(1): 321-327. Duan N N, Dong B, Li J H, et al. High-solids anaerobic co-digestion of sludge and kitchen garbage under mesophilic conditions[J]. Environmental Science, 2013, 34(1): 321-327. |
| [30] | Stalder T, Barraud O, Casellas M, et al. Integron involvement in environmental spread of antibiotic resistance[J]. Frontiers in Microbiology, 2012, 3: 119. |
| [31] | Feng Y H, Zhang Y B, Quan X, et al. Enhanced anaerobic digestion of waste activated sludge digestion by the addition of zero valent iron[J]. Water Research, 2014, 52: 242-250. DOI:10.1016/j.watres.2013.10.072 |
| [32] |
郝晓地, 魏静, 曹达啟. 废铁屑强化污泥厌氧消化产甲烷可行性分析[J]. 环境科学学报, 2016, 36(8): 2730-2740. Hao X D, Wei J, Cao D Q. Feasibility analysis of enhancing anaerobic digestion for methane production by waste iron scrap[J]. Acta Scientiae Circumstantiae, 2016, 36(8): 2730-2740. |
| [33] | Guo X C, Liu S, Wang Z, et al. Metagenomic profiles and antibiotic resistance genes in gut microbiota of mice exposed to arsenic and iron[J]. Chemosphere, 2014, 112: 1-8. DOI:10.1016/j.chemosphere.2014.03.068 |
 2018, Vol. 39
2018, Vol. 39


