利用微生物法修复石油污染土壤具有成本低、效果好以及对环境的二次污染小等优点, 被认为是最有前景的修复技术[1, 2].常用的微生物修复技术包括生物刺激(biostimulation)和生物强化(bioaugmentation)等[3~6].目前许多文献报道了利用生物刺激[7~12]或者生物强化技术[13, 14]修复石油污染土壤的研究工作.
尽管利用微生物强化技术修复石油污染土壤取得了一系列进展, 但在实际应用过程中仍存在许多问题.一些筛选出的降解菌在适宜的环境条件下可以有效降解石油烃, 但在进行实际场地修复时, 外源微生物容易受到污染体系温度、水含量、pH、营养物质供给因素的不规律波动以及土著微生物的竞争作用而失去降解能力.另一方面, 当地的气候环境也会影响外源微生物在石油污染地的生长[15, 16].这些因素制约着生物强化技术的应用, 致使许多微生物强化修复技术仍处于实验室研究阶段[17~19].
为解决低温条件下利用微生物强化技术修复油污土壤时投加的降解菌活性较低等问题, 本研究利用腐熟期的有机堆肥作为固体培养基对石油烃降解菌进行扩大培养, 将获得的降解菌-堆肥制剂施入石油污染土壤中进行修复处理, 通过利用堆肥对土壤的保水、保温及透气能力, 解决外源微生物在低温环境下的不规律波动及难以存活问题, 以期为低温条件下提高微生物强化修复效果提供方法和理论依据.
1 材料及方法 1.1 降解菌和堆肥研究中所用到的5株降解菌是前期工作中从西北地区石油污染土壤中分离获得的, 包括1株铜绿假单胞菌、2株施氏假单胞菌、1株假单胞菌和1株不动细菌[20].菌株的详细情况见表 1.
|
|
表 1 实验中用到的5株石油烃降解菌 Table 1 Identity of the 16S rRNA gene sequences of TPH-degrading bacteria |
所用腐熟堆肥取自西北农林科技大学, 其基本性质如表 2所示.
|
|
表 2 堆肥的基本性质/mg·kg-1 Table 2 Selected chemical characteristics of the compost/mg·kg-1 |
1.2 利用堆肥扩大培养降解菌
混合菌悬液的制备:用接种针挑取保存在斜面上的5株降解菌分别置于150 mL牛肉膏蛋白胨液体培养基中, 25℃、150 r·min-1条件下振荡培养48 h, 5 000 r·min-1离心分离并利用PBS缓冲液洗涤菌株3次, 用PBS缓冲液调节每株菌悬液的D值为1.0(600 nm条件下测定), 取等量的5株降解菌悬液混匀, 即得到混合菌悬液.
实验室条件下对堆肥分别进行如下所示的处理.
C-1:不作任何处理的150 g堆肥(作为控制实验); C-2:向150 g堆肥中加入6 mL原油(原油含量为3.5%, 对堆肥中的土著微生物进行驯化); C-3:向150g堆肥中加入1mL D600=1.0的混合降解菌悬液(利用堆肥作为固体基质培养降解菌); C-4:向150 g堆肥中同时加入6 mL原油和1 mLD600=1.0的混合降解菌悬液(利用原油驯化堆肥中的土著微生物, 同时以堆肥作为固体基质培养降解菌).
将处理过的堆肥放在实验室内, 在室温为20℃条件下进行培养, 利用菌落计数法对不同处理堆肥中的微生物数量进行连续7 d测定.每个处理做3个平行.
1.3 利用降解菌-堆肥制剂修复石油污染土壤石油污染土壤取自陕北地区某油井附近, 土壤基本理化性质测定方法:pH值:电位计法; 有机质:重铬酸钾氧化-滴定法; 总有机碳:非色散红外吸收法; 总盐含量:电导法; 阳离子交换容量:EDTA-乙酸铵盐交换法.基本理化性质测定结果为:pH 8.1, 有机质13.0%, 总有机碳11.2%, 总盐含量2 585 mg·kg-1, 阳离子交换容量4.87 cmol·kg-1.土壤中总石油烃、烷烃和多环芳烃的含量分别为88 400、68 800和9 120 mg·kg-1.
土壤经碎散、除杂、混匀后过2 mm筛.称取15份油污土壤, 每份质量为1 kg, 分装于15个花盆中, 以在自然条件下放置不作修复处理的油污土壤作为控制实验(S-1), 对土壤分别进行表 3所示的修复处理.每个处理3个平行.
|
|
表 3 石油污染土壤的修复方案 Table 3 Remediation of petroleum-polluted soil with different treatments |
将15盆土壤置于室外进行模拟自然条件下的修复, 放置时间为2016年2月4日至2016年3月4日.当月最低气温为-5℃, 月平均温度为2℃, 降水量为10 mm.
修复1个月后取样进行测定.将每盆土充分混合均匀, 利用梅花形布点采样法从每盆土中取5 g土样, 利用超声波萃取法提取土壤中的总石油烃并利用重量法进行测定[21].利用中性氧化铝柱(10×150 mm, Agilent Technologies)对提取的总石油烃进行层析分离, 获得烷烃和多环芳烃组分.利用气相色谱-质谱分析仪(7000B型, 美国安捷伦公司)测定烷烃和多环芳烃含量[22].
1.4 土壤微生物群落结构研究利用OMEGA试剂盒(D5625-01 Soil DNA Kit, USA)提取土壤中总DNA, 利用琼脂糖凝胶电泳检验DNA的完整性.
利用341F/805R(341F引物:5′-CCCTACACGA CGCTCTTCCGATCTG-3′; 805R引物:5′-GACTGGA GTTCCTTGGCACCCGAGAATTCCA-3′)对提取的总DNA进行PCR扩增.扩增步骤:94℃预变性3 min, 94℃变性30 s, 45℃退火20 s, 65℃延伸30 s, 重复5个循环; 94℃变性20 s, 55℃退火20 s, 72℃延伸30 s, 重复20个循环; 引入Illumina桥式PCR兼容引物, 95℃预变性30 s, 95℃变性15 s, 55℃退火15 s, 72℃延伸30 s, 重复5个循环.
纯化回收所需的PCR产物, 利用Qubit 2.0 DNA检测试剂盒对回收的DNA精确定量.利用Illumina MiSeq平台进行高通量分析测定, 最终上机测序的量为20 pmol.
1.5 数据统计与分析利用SPSS 19.0软件对数据进行统计分析和计算, 利用Usearch软件校正测序结果并将所有样本按照序列间的距离进行聚类分析, 利用RDP classifier和Mothur软件进行物种分类和Alpha多样分析.
2 结果与讨论 2.1 降解菌在堆肥中的扩大培养降解菌在堆肥中的扩大培养结果如图 1所示.堆肥中土著微生物数量约为2.4×105 CFU·g-1(C-1), 并且在连续7 d的测定中基本保持不变.向堆肥中加入原油对土著微生物进行驯化时(C-2), 与同期的控制实验(C-1) 相比, 驯化第1 d的堆肥中总菌数量降低了1个数量级, 驯化第2 d总菌量增加, 并在随后的几天内基本保持不变, 但总体上菌量相对C-1减少.说明在150 g堆肥中加入6 mL原油后, 初始阶段原油对堆肥中的土著微生物有毒性作用, 经过适应期后, 堆肥中的大部分微生物可以适应石油的刺激作用继续生长.利用堆肥作为固体基质扩大培养石油烃降解菌(C-3), 与同期的C-1相比, 堆肥中的微生物数量在初期略有降低, 在培养的第3 d数量急剧增加, 第4 d后开始减少.说明在培养初期, 堆肥中的土著微生物和加入的降解菌之间存在竞争和拮抗作用, 经过适应期后, 堆肥中丰富的养分为降解菌的生长提供了有利条件, 使得降解菌在堆肥中迅速生长, 在第3 d时数量达到最大, 到第4 d时开始凋亡, 第5 d时凋亡达到最大.向堆肥中同时加入原油和降解菌的处理中(C-4), 总菌量在培养的第4 d时达到最大, 但是微生物数量远小于不加原油的C-3处理, 说明在堆肥中同时加入原油和降解菌, 初期阶段不利于降解菌在堆肥中的生长, 但在后期(5~7 d), 原油的刺激作用可以使得降解菌在堆肥中的凋亡速度变缓.

|
图 1 降解菌在堆肥中的扩大培养结果 Fig. 1 Total microbial counts in differently treated composts |
根据2.1节的研究结果可知, 石油烃降解菌可以在腐熟有机堆肥中扩大培养.将获得的降解菌-堆肥制剂(C-3, 培养3 d)低温条件下施入重度石油污染土壤中进行修复处理(表 3), 所得结果如图 2、图 3所示.

|
图 2 不同处理土壤中的石油烃、烷烃、多环芳烃含量 Fig. 2 Concentrations of TPH, total alkanes, and PAHs in different treatments |

|
图 3 不同处理土壤中的石油烃、烷烃、多环芳烃去除率 Fig. 3 Degradation efficiencies of TPH, total alkanes, and PAHs in different treatments |
根据图 2、图 3可知, 原土壤(0 d)中总石油烃、烷烃和多环芳烃的含量分别为88 400、68 800、9 120 mg·kg-1.将土壤在自然条件下放置30 d(S-1), 土壤中石油烃、烷烃、多环芳烃含量分别为84 400、62 300、8 910 mg·kg-1, 与原土壤相比, 土壤中石油烃、烷烃、多环芳烃去除率分别为4.5%、9.5%、2.3%;向土壤中直接投加降解菌进行强化修复30 d(S-2), 土壤中的石油烃、烷烃、多环芳烃含量分别为86 000、66 300、8 810 mg·kg-1, 去除率分别为2.7%、3.6%、3.4%;加入150 g堆肥进行修复处理(S-3) 的土壤中的石油烃、烷烃、多环芳烃含量为75 300、60 100、8 280 mg·kg-1, 去除率分别为14.8%、12.6%、9.2%;加入降解菌-堆肥制剂进行修复处理(S-5) 的土壤中石油烃、烷烃、多环芳烃含量分别为64 500、55 300、8 210 mg·kg-1, 去除率分别为27.0%、19.6%、10.0%.结果表明自然衰减(S-1) 和直接投菌法(S-2) 对土壤中石油烃的去除效果较差, 而利用降解菌-堆肥复合制剂对土壤中的石油烃有较好的去除效果.
笔者以前的研究工作发现, 在室温条件下(20~35℃)利用直接投菌法对西北地区油污土壤进行修复处理, 对土壤中石油烃可起到较好的去除作用.吴蔓莉等对陕北子长地区石油污染土壤进行生物强化修复8周, 土壤中总石油烃的去除率为33.0%~58.0%[22~24]; 叶茜琼等[25]在实验室条件下利用生物强化合并生物刺激对甘肃庆阳石油污染土壤进行18周的修复研究, 土壤中烷烃和多环芳烃去除率分别为50.8%和45.2%.本研究中, 将石油污染土壤置于室外低温条件下(月最低气温为-5℃, 月平均温度为2℃)修复30 d时, 直接投加降解菌的土壤中(S-2) 总石油烃、烷烃、多环芳烃的去除率分别为2.7%、3.6%、3.4%, 利用降解菌-堆肥制剂对土壤进行处理(S-5)30 d, 土壤中石油烃、烷烃、多环芳烃去除率分别为27.0%、19.6%、10.0%.结果说明利用堆肥的保水保温能力可以提高降解菌在低温条件下对土壤中石油烃的去除效果.
2.3 修复处理对土壤菌群微生态的影响 2.3.1 高通量测序结果的质量控制利用PCR技术扩增细菌的16S rRNA基因, 并进行高通量测序分析.对测序结果进行优化序列统计及测序质量评价, 所得结果如表 4所示. S-1~S-5处理中所测得的原始序列总数分别为31 693、32 622、29 906、28 909和30 104.利用Rear软件和Prinseq软件对原始序列进行结构、质量剪切等处理后, 得到质控后剩余序列数目分别为31 666、32 587、29 871、28 884和30 070.利用usearch软件去除预处理后序列中的嵌合体与靶区域外序列后, 最终得到的有效序列数分别为31 349、30 774、26 802、26 911和27 407.序列优化率在89%以上, 说明本次序列优化步骤合理.
|
|
表 4 优化序列统计及测序质量评价 Table 4 Sequence statistic and quality estimation of five soil samples |
根据97%相似度对处理后剩余序列进行OTU分类, 利用Mothur软件进行rarefaction分析, 并利用R语言工具绘制稀疏性曲线.所得结果如图 4所示.当序列数目大于25 000时, 曲线趋于平坦, 本次测序数据量为30 000左右, 说明本次测序深度足够.

|
图 4 测序结果的稀疏性曲线 Fig. 4 Rarefaction curves of high throughput sequencing |
土壤微生物群落多样性测定结果如表 5所示. S-1~S-5处理中微生物的OTUs数量分别为842、1 092、1 121、1 023和1 177.分别归属于31、33、29, 27、30个门, 门水平上多数为5种处理样品所共有, 而在属水平上多数为不同处理样品独有.
|
|
表 5 不同处理土壤的微生物群落多样性1) Table 5 Biodiversity of petroleum-polluted soil remediated by different treatments |
根据表 5, 向土壤中加入降解菌-堆肥制剂进行修复处理的土壤(S-5) Ace指数和Chao1指数值均为最大, 其次为加入堆肥(S-3) 和加入降解菌处理(S-2) 的油污土壤; 自然放置(S-1) 和加入原油-堆肥处理的土壤(S-4), Ace指数和Chao1指数值较小.比较不同修复处理土壤的香农指数和辛普森多样性指数可知, 与未经处理的土壤(S-1) 相比, 向土壤中加入降解菌-堆肥制剂(S-5) 可提高土壤的微生物多样性.
多样性指数是用于评价土壤微生物丰富度和群落均匀度的综合指标. Ace指数和Chao1指数用来评价土壤微生物丰富度, Ace指数和Chao1指数值越大, 土壤微生物丰富度越大.香农指数值越大, 辛普森指数值越小, 土壤微生物均匀度越高[26, 27].根据测定结果可知, 向油污土壤中施入降解菌-堆肥制剂可同时提高土壤微生物群落的丰富度和均匀度.
2.3.3 土壤微生物群落结构变化对不同处理土壤前20种优势菌门的相对丰度进行了分析, 所得结果如图 5所示, 在未经修复处理的油污土壤中(S-1), 所占丰度最大的菌门是变形菌门(Proteobacteria), 其次为放线菌门(Actinobacteria), 其他一些所占丰度较大的菌门包括拟杆菌门(Bacteroidetes)和酸杆菌门(Acidobacteria)等.

|
图 5 不同处理土壤中微生物在门水平上的相对丰度 Fig. 5 Relative abundance of the 20 most abundant phyla in different treatments |
与未经修复处理的油污土壤相比, 经过修复处理的土壤(S-2~S-5) 中不同微生物所占丰度在门水平上均发生了较大变化.变形菌门(Proteobacteria)和放线菌门(Actinobacteria)所占丰度明显减小, 拟杆菌门(Bacteroidetes)所占丰度在经过处理的土壤中显著提高.
不同处理土壤中微生物在属水平上的相对丰度测定结果如图 6所示.未经处理的石油污染土壤(S-1) 中, 粒杆黏细菌属(Chondromyces)、交替赤细菌(Altererythrobacter)、Saccharibacteria、诺卡氏菌属(Nocardioides)、假黄单胞菌属(Pseudoxanthomonas)、原小单孢菌属(Promicromonospora)、Rheniheimera、Alkanibacter、Alkanindiges、根瘤菌属(Rhizobium)、假单胞菌属(Pseudomonas)等所占丰度相对较大.经过修复处理后, 土壤微生物在属水平上所占丰度发生明显变化.
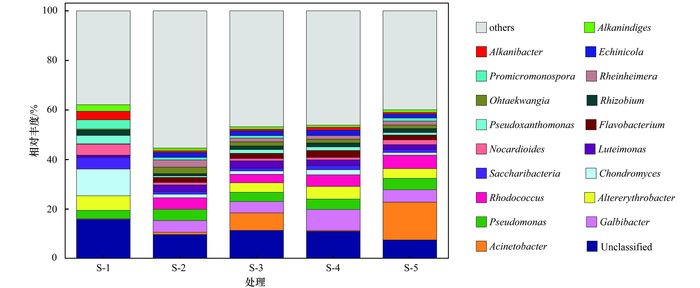
|
图 6 不同处理土壤中微生物在属水平上的相对丰度 Fig. 6 Frequency of the 20 most abundant genera in different treatments |
向土壤中加入降解菌-堆肥制剂修复30 d后(S-5), 土壤中的不动细菌属(Acinetobacter)和假单胞菌属(Pseudomonas)与不经修复处理的土壤(S-1) 相比, 所占丰度分别由0.02%和3.39%提高至15.2%和4.57%.
笔者以前的研究发现, 西北地区石油污染土壤中主要的石油烃降解菌为假单胞菌属(Pseudomonas)和不动细菌属(Acinetobacter)[20, 24].本次研究中使用的石油烃降解菌包括了4株假单胞菌和1株不动细菌(表 1).在向油污土壤直接投加这些降解菌时(S-2), 不动细菌属与假单胞菌属所占丰度分别为0.9%和4.54%, 与未经处理的土壤(S-1) 比, 略有增加; 在加入降解菌-堆肥制剂的土壤中(S-5), 不动细菌属和假单胞菌属所占丰度增加为15.2%和4.57%, 不动细菌属所占丰度增加明显.说明在气温较低的环境条件下向油污土壤中直接投加降解菌进行修复时, 降解菌在土壤中较难存活或者活性较差; 将降解菌在腐熟堆肥中扩大培养制成降解菌-堆肥制剂后施入油污土壤, 不动细菌KF453955的存活率显著提高.
3 结论(1) 利用堆肥作为固体基质扩大培养石油烃降解菌, 堆肥中的微生物总量在培养初期降低, 培养一定时间后迅速增加.降解菌可以利用腐熟堆肥作为培养基质生长.
(2) 低温条件下直接投加降解菌对油污土壤进行生物强化修复30 d, 土壤中石油烃、烷烃、多环芳烃去除率分别为2.7%、3.6%、3.4%, 施加降解菌-堆肥制剂进行修复处理的土壤中3种烃的去除率分别为27.0%、19.6%、10.0%.利用降解菌-堆肥制剂可对土壤中石油烃起到较好去除作用.
(3) 向油污土壤中施入降解菌-堆肥制剂可改变土壤的微生物群落结构, 提高土壤微生物群落的丰富度和均匀度, 土壤中石油烃的去除效率与石油烃降解菌所占丰度有关.
| [1] | 姜昌亮. 石油污染土壤的物理化学处理-生物修复工艺与技术研究[D]. 沈阳: 中国科学院沈阳应用生态研究所, 2001. http://d.wanfangdata.com.cn/Thesis/Y424983 |
| [2] | Leahy J G, Colwell R R. Microbial degradation of hydrocarbons in the environment[J]. Microbiological and Molecular Biology Reviews, 1990, 54(3): 305-315. |
| [3] | Abed R M M, Al-Sabahi J, Al-Maqrashi F, et al. Characterization of hydrocarbon-degrading bacteria isolated from oil-contaminated sediments in the Sultanate of Oman and evaluation of bioaugmentation and biostimulation approaches in microcosm experiments[J]. International Biodeterioration & Biodegradation, 2014, 89: 58-66. |
| [4] | Taccari M, Milanovic V, Comitini F, et al. Effects of biostimulation and bioaugmentation on diesel removal and bacterial community[J]. International Biodeterioration & Biodegradation, 2012, 66(1): 39-46. |
| [5] | Kuyukina M S, Ivshina I B, Kamenskikh T N, et al. Survival of cryogel-immobilized Rhodococcus strains in crude oil-contaminated soil and their impact on biodegradation efficiency[J]. International Biodeterioration & Biodegradation, 2013, 84: 118-125. |
| [6] | Tahhan R A, Ammari T G, Goussous S J, et al. Enhancing the biodegradation of total petroleum hydrocarbons in oily sludge by a modified bioaugmentation strategy[J]. International Biodeterioration & Biodegradation, 2011, 65(1): 130-134. |
| [7] | Lladó S, Solanas A M, de Lapuente J, et al. A diversified approach to evaluate biostimulation and bioaugmentation strategies for heavy-oil-contaminated soil[J]. Science of the Total Environment, 2012, 435-436: 262-269. DOI:10.1016/j.scitotenv.2012.07.032 |
| [8] | Zhang X S, Xu D J, Zhu C Y, et al. Isolation and identification of biosurfactant producing and crude oil degrading Pseudomonas aeruginosa strains[J]. Chemical Engineering Journal, 2012, 209: 138-146. DOI:10.1016/j.cej.2012.07.110 |
| [9] |
韩慧龙, 陈镇, 杨健民, 等. 真菌-细菌协同修复石油污染土壤的场地试验[J]. 环境科学, 2008, 29(2): 454-461. Han H L, Chen Z, Yang J M, et al. Field scale demonstration of fungi-bacteria augmented remediation of petroleum-contaminated soil[J]. Environmental Science, 2008, 29(2): 454-461. |
| [10] |
韩慧龙, 汤晶, 江皓, 等. 真菌-细菌修复石油污染土壤的协同作用机制研究[J]. 环境科学, 2008, 29(1): 189-195. Han H L, Tang J, Jiang H, et al. Synergy between fungi and bacteria in fungi-bacteria augmented remediation of petroleum-contaminated soil[J]. Environmental Science, 2008, 29(1): 189-195. |
| [11] | Zhao D F, Liu C S, Liu L H, et al. Selection of functional consortium for crude oil-contaminated soil remediation[J]. International Biodeterioration & Biodegradation, 2011, 65(8): 1244-1248. |
| [12] | Li G, Huang W, Lerner D N, et al. Enrichment of degrading microbes and bioremediation of petrochemical contaminants in polluted soil[J]. Water Research, 2000, 34(15): 3845-3853. DOI:10.1016/S0043-1354(00)00134-2 |
| [13] |
王悦明, 王继富, 李鑫, 等. 石油污染土壤微生物修复技术研究进展[J]. 环境工程, 2014, 32(8): 157-161, 130. Wang Y M, Wang J F, Li X, et al. Research progress on microbial remediation of petroleum contaminated soil[J]. Environmental Engineering, 2014, 32(8): 157-161, 130. |
| [14] |
吴彬彬, 卢滇楠, 刘铮. 石油污染土壤生物修复过程中氮循环功能基因的动态检测[J]. 环境科学, 2012, 33(6): 2068-2074. Wu B B, Lu D N, Liu Z. Dynamic changes in functional genes for nitrogen cycle during bioremediation of petroleum-contaminated soil[J]. Environmental Science, 2012, 33(6): 2068-2074. |
| [15] |
张胜, 陈立, 崔晓梅, 等. 西北黄土区石油污染土壤原位微生物生态修复试验研究[J]. 微生物学通报, 2008, 35(5): 765-771. Zhang S, Chen L, Cui X M, et al. Experimental study of in-situ microbial ecological remediation for oil contaminated soil in Northwestern Loess region[J]. Microbiology, 2008, 35(5): 765-771. |
| [16] | Steliga T, Jakubowicz P, Kapusta P. Changes in toxicity during in situ bioremediation of weathered drill wastes contaminated with petroleum hydrocarbons[J]. Bioresource Technology, 2012, 125: 1-10. DOI:10.1016/j.biortech.2012.08.092 |
| [17] |
郭婷, 张承东, 张清敏. 生物修复石油污染盐碱土壤小试模拟系统中土壤性质与微生物特性变化[J]. 中国环境科学, 2010, 30(8): 1123-1129. Guo T, Zhang C D, Zhang Q M. Changes of soil properties and microorganism characteristic during bioremediation of petroleum-contaminated saline soil in microscal simulation study[J]. China Environmental Science, 2010, 30(8): 1123-1129. |
| [18] | Wang C Y, Wang F, Wang T, et al. PAHs biodegradation potential of indigenous consortia from agricultural soil and contaminated soil in two-liquid-phase bioreactor (TLPB)[J]. Journal of Hazardous Materials, 2010, 176(1-3): 41-47. DOI:10.1016/j.jhazmat.2009.10.123 |
| [19] | Liu P W G, Chang T C, Chen C H, et al. Effects of soil organic matter and bacterial community shift on bioremediation of diesel-contaminated soil[J]. International Biodeterioration & Biodegradation, 2013, 85: 661-670. |
| [20] |
杨茜, 吴蔓莉, 曹碧霄, 等. 石油降解菌的筛选、降解特性及其与基因的相关性研究[J]. 安全与环境学报, 2014, 14(1): 187-192. Yang Q, Wu M L, Cao B X, et al. Isolation of petroleum degrading strains and determination their degrading and gene characteristics[J]. Journal of Safety and Environment, 2014, 14(1): 187-192. |
| [21] | Wu M L, Dick W A, Li W, et al. Bioaugmentation and biostimulation of hydrocarbon degradation and the microbial community in a petroleum-contaminated soil[J]. International Biodeterioration & Biodegradation, 2016, 107: 158-164. |
| [22] | Wu M L, Li W, Dick W A, et al. Bioremediation of hydrocarbon degradation in a petroleum-contaminated soil and microbial population and activity determination[J]. Chemosphere, 2017, 169: 124-130. DOI:10.1016/j.chemosphere.2016.11.059 |
| [23] |
杨茜, 吴蔓莉, 聂麦茜, 等. 石油污染土壤的生物修复技术及微生物生态效应[J]. 环境科学, 2015, 36(5): 1856-1863. Yang Q, Wu M L, Nie M Q, et al. Effects and biological response on bioremediation of petroleum contaminated soil[J]. Environmental Science, 2015, 36(5): 1856-1863. |
| [24] |
曹文娟, 吴蔓莉, 张明辉, 等. 石油降解菌的筛选优化及其对油污土壤的修复特性[J]. 环境工程学报, 2014, 8(12): 5493-5498. Cao W J, Wu M L, Zhang M H, et al. Isolation and optimization of petroleum degrading bacteria and their bioremediation effects on petroleum-contaminated soil[J]. Chinese Journal of Environmental Engineering, 2014, 8(12): 5493-5498. |
| [25] |
叶茜琼, 吴蔓莉, 陈凯丽, 等. 微生物修复油污土壤过程中氮素的变化及菌群生态效应[J]. 环境科学, 2017, 38(2): 728-734. Ye X Q, Wu M L, Chen K L, et al. Impacts of bioremediation on microbial communities and different forms of nitrogen in petroleum contaminated soil[J]. Environmental Science, 2017, 38(2): 728-734. |
| [26] | Li H, Ye D D, Wang X G, et al. Soil bacterial communities of different natural forest types in Northeast China[J]. Plant and Soil, 2014, 383(1-2): 203-216. DOI:10.1007/s11104-014-2165-y |
| [27] | Sengupta A, Dick W A. Bacterial community diversity in soil under two tillage practices as determined by pyrosequencing[J]. Microbial Ecology, 2015, 70(3): 853-859. DOI:10.1007/s00248-015-0609-4 |
 2017, Vol. 38
2017, Vol. 38

