2. 中国科学院大学, 北京 100049;
3. 河南大学生命科学学院全球变化生态学实验室, 开封 475004
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Laboratory of Global Change Ecology, College of Life Sciences, Henan University, Kaifeng 475004, China
由于人类活动干扰的加剧, 全球气候变化及其生态效应早已成为科学研究的热点.全球气候变化包括气候变暖、降水格局改变等现象, 会显著影响生态系统过程.据IPCC 2013报道, 地球表面平均温度在本世纪末将会升高1.8~4.0℃, 降水格局也会发生显著变化, 包括降水量、降水时间、降水频率等[1].中国北方半干旱温带草原是欧亚草原生物区系的重要组成部分.据预测, 这一地区的年平均温度和降水在未来将继续增加[2].土壤微生物是驱动生物地球化学循环的引擎, 对气候变化响应敏感.为了适应环境的变化, 微生物的生物量、群落结构和功能活性将会改变[3].微生物参与调节的生物地球化学过程可能会随着微生物特征的改变而产生变化, 并对地上植被和生态系统功能产生潜在的正负反馈[4].
半干旱温带草原生态系统中水分是主要的限制因素, 模拟增雨可能会缓解这一地区的水分限制状况, 对生态系统功能有促进作用[5].降水格局的改变对植物的影响之前有过很多研究, 水分可以促进植物的多样性, 改变植物的群落结构[6].水热条件也是影响微生物生长的重要环境因子.对于微生物而言, 增雨可能通过改变土壤含氧量、降低水分抑制性而刺激微生物生长和活性[7], 也可能通过增加土壤pH影响微生物的群落组成[8].一些研究发现, 增雨对多种细菌类群的丰度和群落组成有显著影响.氨氧化古菌(ammonia-oxidizing archaea, AOA)在增雨的条件下丰度保持不变, 而氨氧化细菌(ammonia-oxidizing bacteria, AOB)的丰度可能升高[9].另外, 研究结果显示气候变暖对陆地生态系统中的植物群落结构和组成具有深远影响, 可能会降低植物的多样性[10].气候变暖可能会增加微生物的酶活性, 进而增加微生物的呼吸和分解速率[11].研究发现增温导致真菌与细菌的比例上升[12], 细菌的群落结构也随着增温产生显著变化[13].室内培养实验结果表明AOA更适宜生长在30℃以上的高温环境中; 而对于AOB, 不同温度梯度都会有相应生态位的菌株生长[14], 所以在内蒙古地区, 增温可能会促进AOA的生长, 改变AOB的群落组成.增温可能通过降低土壤含水量与增雨产生拮抗的交互作用影响微生物群落结构[15], 也可能通过促进微生物活性与增雨产生协同的交互作用影响生态系统[16].多种气候变化因子之间的交互作用可能对调节植物群落多样性和预测未来陆地生态系统功能构成巨大的挑战, 因此目前迫切需要明确增温增雨及其交互作用对土壤微生物群落的影响和潜在机制.
测量生物多样性常用的指标包括α多样性指数和β多样性指数. α多样性是一个样品内物种的多样性, 反映物种间内部竞争的结果; 而β多样性是比较不同样品多样性的分布差异, 反映生物对外部环境变化的响应.对于增温、增雨条件下, 微生物和植物多样性之间的相互作用如何变化还不清楚[17].例如, 增雨可能改变微生物的代谢活动, 影响植物的多样性.增温提高微生物酶活, 改变凋落物的降解, 从而影响土壤的养分有效性.区分影响植物群落的生物和非生物因子有助于人们发现植物多样性对气候变化响应的潜在机制.随着免培养的分子生物学技术的发展, 世界范围内已开展了大量的野外控制实验, 从分子水平分析土壤氨氧化微生物的群落组成, 用以评估未来气候变化对土壤氮循环过程的影响[18].然而, 对于微生物对全球变化的响应和适应机制还存在很大的不确定性.鉴于此, 为了研究气候变化对草地生态系统的潜在影响, 笔者提出了以下假设:① 增雨会促进氨氧化微生物的多样性; ② 增温和增雨会对氨氧化微生物的群落结构产生交互作用; ③ 气候变化会通过影响地下微生物的群落而改变地上植物的多样性.本文依托内蒙多伦恢复生态学试验示范研究站这一研究平台, 采用基于DNA的分子生物学手段, 研究模拟全球变化两因素(气候变暖、降水增加)对土壤氨氧化微生物的影响, 并对上述的假设进行验证.
1 材料与方法 1.1 实验样地与设计实验样地位于内蒙古多伦县(东经116°17′, 北纬42°02′, 海拔1324 m), 该区域属于半干旱温带草原[5].气候类型为大陆性季风气候, 年平均降雨量是378 mm, 6~9月的降水量占总体的80%;年平均温度为2.1℃, 温度范围从1月的-17.5℃到7月的18.9℃.根据中国土壤分类, 土壤类型为栗钙土.主要植被类型是克氏针茅(Stipa krylovii)、冷蒿(Artemisia frigida)、星毛委陵菜(Potentilla acaulis)、糙隐子草(Cleistogenes squarrosa)、砂韭(Allium bidentatum)、冰草(Agropyron cristatum)等[10].
模拟气候变暖(增温)及降水增加(增雨)的野外控制实验开始于2005年4月, 采用巢式设计, 增雨为主要因子, 共3个44 m×28 m的区组, 每个区组中有两个10 m×15 m的小区, 分别为增雨和对照处理.每个小区有4个3 m×4 m的亚区, 随机设置成两个增温和两个对照.共4个处理, 每个处理有6个重复, 共24个亚区.每个小区采用6个洒水装置, 在7月和8月每周增雨15 mm, 共增加120 mm的降水.暖化处理采用165 cm×15 cm MSR-2420红外散热器(Kalglo Electronics, Bethlehem, PA, USA)悬挂在地上2.5 m高.增温对照的亚区中设置了一个具有相同形状和大小的“虚拟”加热器来模拟阴影的影响.增温处理的土壤温度大约升高了0.98℃[19].
本研究于2014年5月底采集表层(0~20 cm)土壤样品, 每个亚区用直径7.5 cm的土钻随机取3个样品混合.所有的样品都在3 d内运到实验室.土壤样品通过2 mm筛去除草根和石块, 之后被分为两部分:一部分储存在4℃用于分析理化性质和微生物活性, 另一部分存储在-80℃适用于分子分析.
1.2 土壤理化性质和微生物活性在105℃烘箱中烘干12 h来测量土壤含水量.土壤pH用1 g :2.5 mL的土水比测定.土壤总碳(total carbon, TC)、总氮(total nitrogen, TN)的测定使用杜马斯燃烧法, 用元素分析仪(Vario EL Ⅲ, Elementar, Germany)测定.土壤铵态氮(NH4+-N)和硝态氮(NO3--N)用1 mol ·L-1氯化钾提取(土壤:水=1 g :5 mL), 用连续流动分析仪(SAN++, Skalar, Holand)测定浓度.
硝化潜势(potential nitrification rate, PNR)的测定使用氯酸盐抑制方法:将5.0 g新鲜土壤和20 mL包含1 mmol ·L-1 (NH4)2SO4和50 mg ·L-1 KClO3的磷酸缓冲溶液(PBS:NaCl 8 g ·L-1, KCl 0.2 g ·L-1, Na2HPO4 1.44 g ·L-1, KH2PO4 0.24 g ·L-1, pH 7.4) 添加到50 mL离心管, 在25℃以180 r ·min-1的速度培养24 h, 之后用5 mL氯化钾(2 mol ·L-1)提取亚硝酸盐, 以N-(1-萘胺)乙二胺盐酸盐显色, 并在530 nm波长下用分光光度法测定浓度.土壤呼吸(soil respiration, SR)的测定方法如下:将10 g新鲜土壤添加到120 mL血清瓶中, 在25℃培养24 h, 收集二氧化碳并用气相色谱仪(Agilent 7890A GC System)测定浓度.
1.3 土壤DNA提取和实时定量PCR称取0.3 g冻土, 用PowerSoilTM Total DNA Isolation试剂盒(Mo Bio Laboratories, Inc., San Diego, CA, USA), 参照说明书的方法提取土壤DNA. DNA浓度和纯度用NanoDrop分光光度计(NanoDrop Technologies, USA)测定. DNA样品存储在-20℃, 10倍稀释后用于下游分子实验的模板.古菌16S rRNA、细菌16S rRNA、AOA-amoA、AOB-amoA基因的定量PCR采用iCycler iQ5荧光定量PCR仪(Bio-Rad Laboratories, Inc., USA), 使用的引物和扩增条件在表 1中列出.
|
|
表 1 PCR所用的引物序列和反应条件 Table 1 Primer sequences and reaction conditions used for PCR |
细菌16S rRNA基因采用TaqMan探针法, 25 μL反应体系包含12.5 μL 2×Premix Ex TaqTM (Takara Biotechnology, Japan), 前后引物各0.5 μL(10 μmol ·L-1), 1.5 μmol TaqMan探针(细菌16S rRNA基因)和2 μL的DNA模板(1~10 ng).其他基因采用SYBR GREEN法, 25 μL反应体系包含12.5 μL 2×SYBR Premix Ex TaqTM (Takara Biotechnology, Japan), 前后引物各0.5 μL(10 μmol ·L-1)和2 μL的DNA模板(1~10 ng). 10倍连续稀释的质粒被用作模板制作标准曲线, 所有基因的扩增效率范围在89.2%~100.3%, R2为0.99.利用溶解曲线和琼脂糖凝胶电泳检测扩增的特异性.
1.4 末端限制性片段长度多态性(T-RFLP)T-RFLP的方法被用来分析AOA和AOB的amoA基因群落结构.使用的引物与定量PCR的相同, 但正向引物的5′末端标记了6-carboxyfluorescein(FAM)荧光染料. PCR产物用Wizard SV Gel and PCR Clean-Up System试剂盒纯化(Promega, USA).纯化后的产物用限制性内切酶进行酶切, AOA-amoA基因采用的是内切酶HhaI(Takara Biotechnology, Japan), AOB-amoA基因采用的是内切酶MspI(Takara Biotechnology, Japan).反应体系为20 μL, 包括酶4 U和大约500 ng DNA样品, 反应条件为37℃酶切3 h.酶切产物送至睿博兴科测序公司利用ABI PRISM 3700 DNA分析仪(Applied Biosystems, USA)进行毛细管电泳检测. T-RFLP得到的图谱使用GeneMarker软件(SoftGenetics, USA)进行分析, 选取末端片段长度位于30 bp到目的基因长度之间的片段, 合并长度相差1 bp的片段.用每个片段的峰面积除以总的峰面积得到各T-RFs的相对丰度, 舍去相对丰度低于1%的酶切片段.
1.5 克隆文库和测序分别构建了AOA和AOB的amoA基因的克隆文库.构建过程简述如下:使用与定量PCR相同的引物和条件分别对各土壤样品中的上述基因进行PCR扩增, 随后将24个样品的PCR产物混合, 并进行切胶纯化; 纯化后的产物连接到pMDTM 19-T载体(Takara Biotechnology, Japan), 然后转化入感受态细胞(大肠杆菌JM109) 中进行蓝白斑筛选; 然后从每个文库中随机选取约50个阳性克隆子, 在ABI 3730测序仪(Applied Biosystems, USA)上测序.获得的序列经Bio-Edit 7.0进行编辑、比对后, 利用MOTHUR在97%相似性水平下划分操作分类单位(OTU).从GeneBank上下载标准参比序列及与各代表性OTU高度同源的amoA序列, 使用Mega 6.0软件的neighbor-joining方法构建系统发育树.本研究中获取的AOA-amoA和AOB-amoA基因已提交至NCBI, 其序列号分别为KY118386-KY118431和KY118519-KY118563.
1.6 统计分析基因的拷贝数在进行统计分析之前进行了对数转换以符合数据正态分布.所有统计分析都在R.3.3.2软件(http://cran.r-project.org)中进行, 以P < 0.05作为阈值来判断各土壤处理间的差异显著性.采用单因素方差分析(Duncan检验)进行差异显著性检验.使用双因素方差分析来考察增温、增雨和交互作用对土壤理化性质和微生物活性的影响.用Spearman相关分析考察土壤化学性质之间的相关性.基于T-RFLP的群落组成结果, 计算了α多样性(Shannon指数).增温、增雨对基于Bray-Curtis矩阵的土壤微生物群落的影响也在R软件Vegan包中, 利用排列多元方差分析(PerMANOVA)进行了评估.冗余分析(Redundancy analysis, RDA)用来揭示土壤化学性质(即pH、土壤水分、NH4+-N、NO3--N、TC和TN)和微生物群落结构之间的关系.
基于先验假设, 使用AMOS 20.0(Amos, Development Corporation, Meadville, PA, USA)软件构建结构方程模型(structural equation model, SEM), 来评估增温、增雨对氨氧化微生物和植物群落的α(Shannon指数)和β多样性(基于Bray-Curtis矩阵的主坐标分析的第一轴PCO1) 的直接和间接影响.植物多样性的数据由河南大学生命科学学院万师强老师团队提供.将增温和增雨分别设置为0和1两个外生变量水平, 对α和β多样性分别构建结构方程模型, 模型的拟合优度使用以下指标评价:卡方检验, Akaike讯息指标(AIC), 适配度指数(GFI), 渐进残差均方和平方根(RMSEA).
2 结果与分析 2.1 增温增雨对土壤化学性质和微生物活性的影响土壤化学性质和微生物活性在不同的处理间有显著差异(表 2).在表层土壤中含水量没有显著差异, 而pH在增雨处理中显著升高(P < 0.001).总氮、总碳、氨态氮、硝态氮的含量, 在不同处理间没有显著差异.表层土壤的硝化潜势(PNR)在不同处理间没有显著差异, 而增温显著降低了土壤呼吸(SR,P=0.011). Spearman相关分析发现, 土壤含水量与总氮、总碳、硝态氮显著正相关(分别为ρ=0.78, P < 0.001, n=24; ρ=0.69, P < 0.001, n=24和ρ=0.42, P=0.04, n=24), SR与土壤pH显著负相关(ρ=-0.46, P=0.02, n=24).
|
|
表 2 土壤化学性质和微生物活性 Table 2 Soil chemical properties and microbial activities |
2.2 增温增雨对微生物基因丰度的影响
各相关微生物基因定量PCR的结果见图 1.古菌和细菌的16S rRNA基因、AOA和AOB的amoA基因的丰度在不同处理间均没有显著差异.古菌和细菌的16S rRNA基因丰度范围分别为每克干土0.41×109~1.22×109和4.67×109~3.8×1010拷贝数. AOA和AOB amoA基因丰度范围分别为每克干土3.39×108~13.5×108和1.95×105~1.96×106拷贝数.

|
图中误差线代表 6个重复样品的标准误 图 1 不同处理下微生物的基因丰度 Fig. 1 Abundances of microbial genes under the treatments of increased precipitation and warming |
进一步分析发现增温显著增加了AOA与AOB丰度之比(P=0.046). Spearman相关分析发现, 古菌16S rRNA基因丰度与土壤TN(ρ=0.7, P=0.0, n=24) 和TC(ρ =0.74, P=0.01, n=24) 显著正相关, 细菌16S rRNA基因丰度则与土壤含水量、TN和TC显著正相关(ρ=0.75, P=0.01, n=24; ρ=0.83, P < 0.001, n=24和ρ=0.82, P < 0.001, n=24).
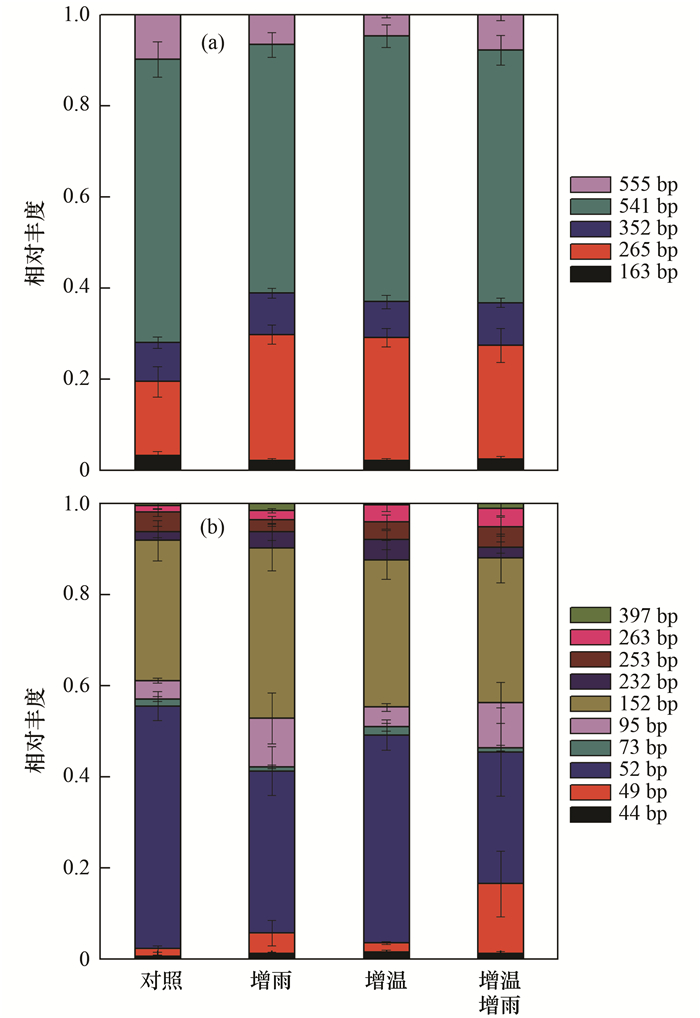
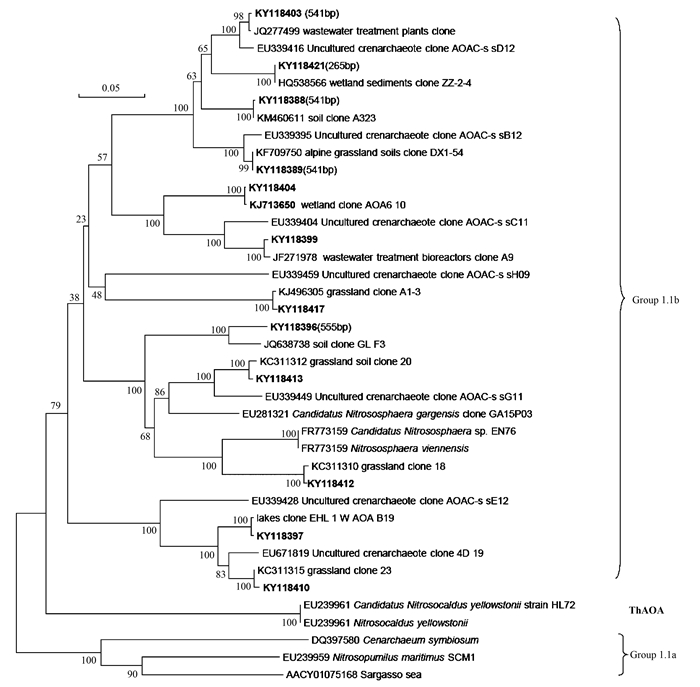
2.3 增温增雨对微生物群落结构的影响土壤AOA-amoA基因用Hha Ⅰ酶切, 共得到5个末端限制片段(T-RFs:即163、265、352、541和555bp),见图 2(a), 其中265 bp和541 bp是优势T-RFs, 平均相对丰度分别为23.96%和57.55%. 47个克隆子的序列基于97%的相似性被分成12个OTU.系统发育分析识别3个T-RFs(265、541、555 bp), 它们都属于Group 1.1b(图 3). PerMANOVA结果表明AOA-amoA群落在不同处理之间没有显著差异, 但265 bp T-RF的相对丰度在增温、增雨的处理中比对照处理高.
|
(a)AOA-amoA基因、(b)AOB-amoA基因; 图中误差线代表 6个重复样品的标准误 图 2 不同处理下AOA-amoA基因、AOB-amoA基因的群落组成 Fig. 2 Relative abundances of T-RFs of AOA-amoA gene and AOB-amoA gene under the treatments of increased precipitation and warming |
|
样品序列信息包括:序列号(加粗)和识别的HhaI酶切片段长度 图 3 基于AOA-amoA基因序列构建的系统发育树 Fig. 3 Phylogenetic tree of AOA-amoA sequences |
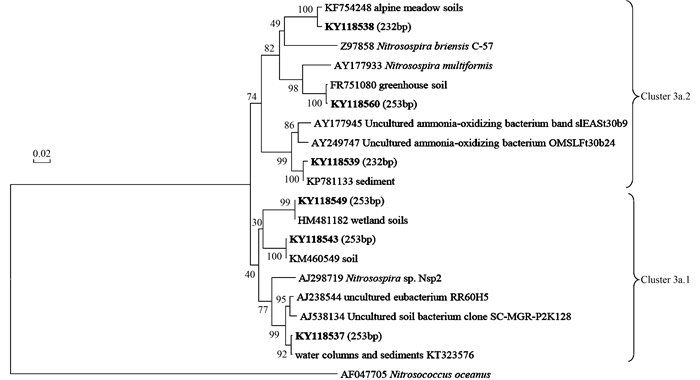
获取的AOB-amoA基因序列经Msp Ⅰ酶切, 共得到10个T-RFs(44、49、52、73、95、152、232、253、263和397bp),见图 2(b), 其中T-RFs 52 bp和152 bp是两个主要的T-RFs, 分别占总T-RFs的40.83%和33.01%. 49个克隆子的序列基于97%的相似性被分成6个OTU.两个T-RFs(232bp和253bp)被系统发育分析识别, 分别属于亚硝化螺菌属(Nitrosospira)的Cluster 3a.1和3a.2(图 4). PerMANOVA结果表明增雨显著改变AOB-amoA基因的群落组成.与对照相比, T-RF 52bp随着增雨而降低, 而49bp和95bp的T-RFs则显示出相反的趋势.
|
样品序列信息包括:序列号(加粗)和识别的MspⅠ酶切片段长度 图 4 基于AOB-amoA基因序列构建的系统发育树 Fig. 4 Phylogenetic tree of AOB-amoA sequences |
本研究用冗余分析(RDA)表征微生物群落和土壤化学性质之间的关系, 发现RDA前两轴共能解释52.55%的AOA群落组成变化, 其中含水量、总氮(TN)和总碳(TC)是影响AOA-amoA群落结构的主要环境因子(P为0.001、0.001和0.001),见图 5(a).对于AOB-amoA, 前两轴的解释量为24.96%, 只有土壤pH显著影响其群落结构[P=0.037,图 5(b)].
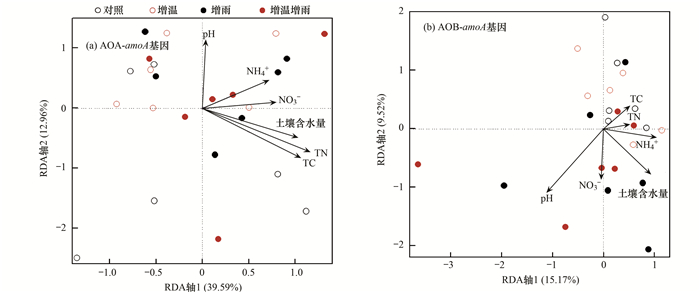
|
图 5 amoA基因、AOB-amoA基因的群落组成和环境变量的冗余分析 Fig. 5 Biplot of redundancy analysis (RDA) of the relationships between soil properties and communities stucture of AOA-amoA gene and AOB-amoA gene |
根据结构方程模型的结果(图 6), 发现增雨可以对土壤pH产生直接的影响, 这与之前方差分析的结果相一致.土壤含水量和硝态氮含量之间呈显著正相关. AOA-amoA基因的α和β多样性明显与土壤含水量和pH显著相关.对于AOB-amoA基因, β多样性被增温增雨直接影响, 同时发现与铵态氮含量显著相关.另外, 检测到地上植物的α多样性与AOA-amoA的α多样性和土壤含水量显著相关, 而β多样性则与AOA-amoA和AOB-amoA基因的β多样性显著相关, 预示着气候变化主要通过影响微生物的多样性间接对植物多样性产生影响.
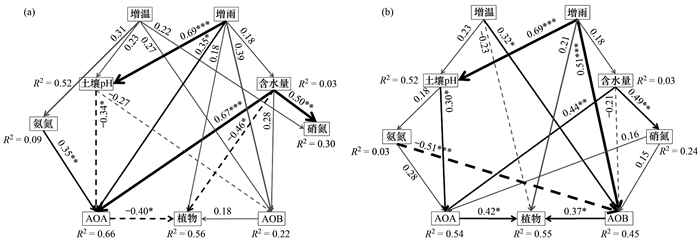
|
(a)α多样性(Shannon指数)和(b)β多样性(主坐标分析第一轴)多样性; 箭头上的数字代表路径系数, 星号表示显著性水平:*表示P < 0.05, **表示P < 0.01和***表示P < 0.001;灰色箭头代表不显著, 箭头宽度代表相关性的大小; R2表示解释量; 模型的显著性检验结果:(a) χ2=11.433; P =0.833; AIC=67.433; GFI=0.909; RMSEA=0.000; (b) χ2=12.652; P=0.759; AIC=68.652; GFI=0.893; RMSEA=0.000 图 6 结构方程模型表征气候变化对氨氧化微生物及植物的α(Shannon指数)和β(主坐标分析第一轴)多样性的直接和间接影响 Fig. 6 Structural equation models (SEM) reveal the direct and indirect effects of climate change on α(Shannon index) and β(first principal coordinate) diversity of ammonia oxidizers and plants |
研究表明, 水分是影响半干旱温带草原生态系统最重要的因素, 增加降水显著提高了土壤的含水量和pH, 这与之前的研究结果一致[2, 5].虽然增雨对各基因丰度的影响都不显著, 但对微生物的群落结构有显著的影响.结构方程模型显示, 增雨与AOA-amoA基因的α多样性有直接的正相关关系, 同时AOA-amoA基因的α多样性与土壤含水量呈显著的正相关, 表明增雨可以通过增加土壤含水量促进AOA的多样性. RDA显示AOA-amoA的群落结构与土壤含水量、TN和TC显著相关, 表明这些土壤性质是影响AOA-amoA群落最重要的因素[24].笔者发现增雨可以直接影响AOB-amoA基因的群落结构, 这与先前的研究结果一致[9].潜在的机制可能有两种, 一是增雨改变了土壤含水量, 因为AOB属于革兰氏阳性菌, 对水分变化更为敏感[25];另一个可能原因是增雨升高了土壤pH, 从而影响氨氧化微生物的群落结构. RDA结果显示AOB-amoA群落结构受到土壤pH的显著影响, 暗示着增雨通过改变pH间接影响微生物群落[8].
增温显著降低了土壤呼吸, 增雨减缓了增温对土壤呼吸的抑制作用.推测可能的解释是增雨增加了土壤含水量, 缓解由于增温导致的土壤干旱, 土壤呼吸与含水量呈正相关[26].土壤呼吸降低的另一种可能原因是增温改变了微生物的群落结构[25].之前也有研究发现土壤温度增加会升高α变形菌门和酸杆菌门的相对丰度之比, 从而刺激土壤呼吸[27].结论存在差异的原因可能是微生物对气候变化的响应随不同生长季节和不同的采样点而有差异[28, 29].另外, 本研究发现AOA和AOB的amoA基因丰度之比随着温度升高而显著增加, 增温导致了AOA-amoA基因丰度更具优势.可能的原因在于AOA比AOB更能适应升温的环境, 温度增长的条件下AOB的丰度会下降[30].和我们的假设相反, 增温增雨对氨氧化微生物的群落结构没有显著的交互作用.
3.2 气候变化对植物多样性的直接和间接的影响在半干旱温带草原, 地上植物多样性主要受水分可利用性限制[10].根据SEM结果, 增雨可以通过改变含水量进而间接降低植物的α多样性, 增加AOA的α多样性, 可能的原因是植物和微生物之间存在对营养物质的竞争关系[31].而增温和增雨可以通过影响AOA和AOB-amoA的群落结构来间接影响植物的β多样性, 表明微生物群落结构的变化可以解释植物对气候变化的响应.之前的研究也显示, 微生物和植物多样性之间存在关联, 微生物对气候变化的响应可以间接地影响植物多样性[32].地上和地下生物可能通过植物凋落物的分解相联系, 而土壤微生物的群落结构可以介导有机质的矿化, 驱动土壤生态系统功能和地上生物量的变化[33].此外, 根据SEM模型的结果, 植物多样性变化的解释量只有56%和53%, 表明还有很多未知的机制没有包含在模型中, 下一步的模型构建中需要纳入更多的环境因素和不同采样时间的调查数据.
4 结论水分的可用性可能是影响半干旱温带草原中氨氧化微生物多样性最重要的因素.增雨可以直接影响AOA-amoA的α多样性, 还可通过改变土壤pH而间接影响AOB-amoA的群落结构.但增温和增雨对氨氧化微生物的群落结构没有显著的交互作用.同时, 长期增温对土壤呼吸的负面影响, 预示着长期的全球气候变暖可能会抑制温室气体CO2的排放. SEM模型显示氨氧化微生物与植物多样性之间存在着一定的相关关系, 表明在半干旱草原生态系统中微生物在调节植物多样性对气候变化的响应中发挥了一定的作用, 为进一步深入探讨气候变化-微生物-植物之间的关系提供了依据.
| [1] | IP CC. Climate change 2013-the physical science basis: working group Ⅰ contribution to the fifth assessment report of the Intergovernmental Panel on Climate Change[M]. Cambridge, UK: Cambridge University Press, 2013. |
| [2] | Bai W M, Wan S Q, Niu S L, et al. Increased temperature and precipitation interact to affect root production, mortality, and turnover in a temperate steppe: implications for ecosystem C cycling[J]. Global Change Biology, 2010, 16(4): 1306-1316. DOI:10.1111/gcb.2010.16.issue-4 |
| [3] |
沈菊培, 贺纪正. 微生物介导的碳氮循环过程对全球气候变化的响应[J]. 生态学报, 2011, 31(11): 2957-2967. Shen J P, He J Z. Responses of microbes-mediated carbon and nitrogen cycles to global climate change[J]. Acta Ecologica Sinica, 2011, 31(11): 2957-2967. |
| [4] |
孙良杰, 齐玉春, 董云社, 等. 全球变化对草地土壤微生物群落多样性的影响研究进展[J]. 地理科学进展, 2012, 31(12): 1715-1723. Sun L J, Qi Y C, Dong Y S, et al. Research progresses on the effects of global change on microbial community diversity of grassland soils[J]. Progress in Geography, 2012, 31(12): 1715-1723. DOI:10.11820/dlkxjz.2012.12.018 |
| [5] | Niu S L, Wu M Y, Han Y, et al. Water-mediated responses of ecosystem carbon fluxes to climatic change in a temperate steppe[J]. New Phytologist, 2008, 177(1): 209-219. |
| [6] | Wu Z T, Dijkstra P, Koch G W, et al. Responses of terrestrial ecosystems to temperature and precipitation change: a meta-analysis of experimental manipulation[J]. Global Change Biology, 2011, 17(2): 927-942. DOI:10.1111/gcb.2010.17.issue-2 |
| [7] | Chen S P, Lin G H, Huang J H, et al. Responses of soil respiration to simulated precipitation pulses in semiarid steppe under different grazing regimes[J]. Journal of Plant Ecology, 2008, 1(4): 237-246. DOI:10.1093/jpe/rtn020 |
| [8] | Zhang X M, Wei H W, Chen Q S, et al. The counteractive effects of nitrogen addition and watering on soil bacterial communities in a steppe ecosystem[J]. Soil Biology and Biochemistry, 2014, 72: 26-34. DOI:10.1016/j.soilbio.2014.01.034 |
| [9] | Chen Y L, Xu Z W, Hu H W, et al. Responses of ammonia-oxidizing bacteria and archaea to nitrogen fertilization and precipitation increment in a typical temperate steppe in Inner Mongolia[J]. Applied Soil Ecology, 2013, 68: 36-45. DOI:10.1016/j.apsoil.2013.03.006 |
| [10] | Yang H J, Wu M Y, Liu W X, et al. Community structure and composition in response to climate change in a temperate steppe[J]. Global Change Biology, 2011, 17(1): 452-465. DOI:10.1111/gcb.2010.17.issue-1 |
| [11] |
张乃莉, 郭继勋, 王晓宇, 等. 土壤微生物对气候变暖和大气N沉降的响应[J]. 植物生态学报, 2007, 31(2): 252-261. Zhang N L, Guo J X, Wang X Y, et al. Soil microbial feedbacks to climate warming and atmospheric N deposition[J]. Journal of Plant Ecology, 2007, 31(2): 252-261. DOI:10.17521/cjpe.2007.0029 |
| [12] | Zhang W, Parker K, Luo Y, et al. Soil microbial responses to experimental warming and clipping in a tallgrass prairie[J]. Global Change Biology, 2005, 11(2): 266-277. DOI:10.1111/gcb.2005.11.issue-2 |
| [13] | Xiong J B, Sun H B, Peng F, et al. Characterizing changes in soil bacterial community structure in response to short-term warming[J]. FEMS Microbiology Ecology, 2014, 89(2): 281-292. DOI:10.1111/fem.2014.89.issue-2 |
| [14] | Wu Y C, Ke X B, Hernández M, et al. Autotrophic growth of bacterial and archaeal ammonia oxidizers in freshwater sediment microcosms incubated at different temperatures[J]. Applied and Environmental Microbiology, 2013, 79(9): 3076-3084. DOI:10.1128/AEM.00061-13 |
| [15] | Zhang N L, Liu W X, Yang H J, et al. Soil microbial responses to warming and increased precipitation and their implications for ecosystem C cycling[J]. Oecologia, 2013, 173(3): 1125-1142. DOI:10.1007/s00442-013-2685-9 |
| [16] | Wang C H, Wan S Q, Xing X R, et al. Temperature and soil moisture interactively affected soil net N mineralization in temperate grassland in Northern China[J]. Soil Biology and Biochemistry, 2006, 38(5): 1101-1110. DOI:10.1016/j.soilbio.2005.09.009 |
| [17] | van der Putten W H, Bardgett R D, Bever J D, et al. Plant-soil feedbacks: the past, the present and future challenges[J]. Journal of Ecology, 2013, 101(2): 265-276. DOI:10.1111/jec.2013.101.issue-2 |
| [18] |
张翠景, 贺纪正, 沈菊培. 全球变化野外控制试验及其在土壤微生物生态学研究中的应用[J]. 应用生态学报, 2016, 27(5): 1663-1673. Zhang C J, He J Z, Shen J P. Global change field manipulative experiments and their applications in soil microbial ecology[J]. Chinese Journal of Applied Ecology, 2016, 27(5): 1663-1673. |
| [19] | Zhou X Q, Chen C R, Wang Y F, et al. Warming and increased precipitation have differential effects on soil extracellular enzyme activities in a temperate grassland[J]. Science of the Total Environment, 2013, 444: 552-558. DOI:10.1016/j.scitotenv.2012.12.023 |
| [20] | Kemnitz D, Kolb S, Conrad R. Phenotypic characterization of rice cluster Ⅲ archaea without prior isolation by applying quantitative polymerase chain reaction to an enrichment culture[J]. Environmental Microbiology, 2005, 7(4): 553-565. DOI:10.1111/emi.2005.7.issue-4 |
| [21] | Suzuki M T, Taylor L T, DeLong E F. Quantitative analysis of small-subunit rRNA genes in mixed microbial populations via 5'-nuclease assays[J]. Applied and Environmental Microbiology, 2000, 66(11): 4605-4614. DOI:10.1128/AEM.66.11.4605-4614.2000 |
| [22] | Tourna M, Freitag T E, Nicol G W, et al. Growth, activity and temperature responses of ammonia-oxidizing archaea and bacteria in soil microcosms[J]. Environmental Microbiology, 2008, 10(5): 1357-1364. DOI:10.1111/j.1462-2920.2007.01563.x |
| [23] | Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker: molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 1997, 63(2): 4704-4712. |
| [24] |
刘若萱, 贺纪正, 张丽梅. 稻田土壤不同水分条件下硝化/反硝化作用及其功能微生物的变化特征[J]. 环境科学, 2014, 35(11): 4275-4283. Liu R X, He J Z, Zhang L M. Response of nitrification/denitrification and their associated microbes to soil moisture change in paddy soil[J]. Environmental Science, 2014, 35(11): 4275-4283. |
| [25] | Hu H W, Macdonald C A, Trivedi P, et al. Water addition regulates the metabolic activity of ammonia oxidizers responding to environmental perturbations in dry subhumid ecosystems[J]. Environmental Microbiology, 2015, 17(2): 444-461. DOI:10.1111/1462-2920.12481 |
| [26] | Liu W X, Zhang Z, Wan S Q. Predominant role of water in regulating soil and microbial respiration and their responses to climate change in a semiarid grassland[J]. Global Change Biology, 2009, 15(1): 184-195. DOI:10.1111/gcb.2009.15.issue-1 |
| [27] | Yergeau E, Bokhorst S, Kang S, et al. Shifts in soil microorganisms in response to warming are consistent across a range of Antarctic environments[J]. The ISME Journal, 2012, 6(3): 692-702. DOI:10.1038/ismej.2011.124 |
| [28] | Luo Y Q, Wan S Q, Hui D F, et al. Acclimatization of soil respiration to warming in a tall grass prairie[J]. Nature, 2001, 413(6856): 622-625. DOI:10.1038/35098065 |
| [29] | Gutknecht J L M, Field C B, Balser T C. Microbial communities and their responses to simulated global change fluctuate greatly over multiple years[J]. Global Change Biology, 2012, 18(7): 2256-2269. DOI:10.1111/j.1365-2486.2012.02686.x |
| [30] | Avrahami S, Bohannan B J M. Response of Nitrosospira sp. strain AF-Like ammonia oxidizers to changes in temperature, soil moisture content, and fertilizer concentration[J]. Applied and Environmental Microbiology, 2007, 73(4): 1166-1173. DOI:10.1128/AEM.01803-06 |
| [31] | Osanai Y, Tissue D T, Bange M P, et al. Plant-soil interactions and nutrient availability determine the impact of elevated CO2 and temperature on cotton productivity[J]. Plant and Soil, 2017, 410(1-2): 87-102. DOI:10.1007/s11104-016-2981-3 |
| [32] | Wang J T, Zheng Y M, Hu H W, et al. Coupling of soil prokaryotic diversity and plant diversity across latitudinal forest ecosystems[J]. Scientific Reports, 2016, 6(1): 19561. DOI:10.1038/srep19561 |
| [33] | Delgado-Baquerizo M, Maestre F T, Reich P B, et al. Microbial diversity drives multifunctionality in terrestrial ecosystems[J]. Nature Communications, 2016, 7: 10541. DOI:10.1038/ncomms10541 |
 2017, Vol. 38
2017, Vol. 38