目前,高盐废水是水处理领域的一大难点,电渗析、焚烧法、膜分离法、蒸汽压缩法等均有一定的处理效果[1~6].但此类方法的投资运行费用较高,易造成二次污染而难以在实际生产过程中推广应用.生化法因处理费用较低、应用范围广且不易造成二次污染而被广泛利用[7].常用的生物法包括传统活性污泥法、SBR法、MBR法、好氧颗粒污泥法以及厌氧生物处理法等[8~12].系统中适量的无机盐不仅促进微生物生长过程中的酶反应[13],还能够维持膜平衡、调节渗透压.但盐度一旦过量,就会对微生物的生长起到抑制作用,甚至会致使微生物死亡,其主要原因在于:废水盐度较高时会使微生物的渗透压升高,使微生物脱水导致细胞原生质分离;盐度较高时会使细胞脱氢酶的活性降低;废水中的Cl-会对微生物有毒害作用,甚至会杀死微生物;使得水的密度增加,造成活性污泥上浮而流失.
国内外很多研究表明,生物法可以高效地处理高盐度废水.但随着系统盐度的增加,微生物需要一段时间的适应过程.系统盐度的变化会引起微生物代谢途径的变化以及微生物群落的显著变化[14, 15].本文通过逐渐提高系统的盐度,探究不同盐度下活性污泥的性质以及微生物群落的变化,并测定不同盐度下活性污泥中微生物的脂肪酸和DNA,对处理实际的含盐废水有较大的意义.
1 材料与方法 1.1 污泥来源与试验材料 1.1.1 污泥来源试验选取青岛李村河生活污水处理厂二沉池污泥,在光学显微镜下对活性污泥进行观察,视野中观察到变形虫、草履虫等微型后生动物、原生动物等,表明现阶段污泥的活性较高.
1.1.2 主要试剂和试验仪器氯化钠、葡萄糖、磷酸二氢钾、硫酸铵、乙酸、氢氧化钾、纳米四氧化三铁、甲基叔丁基醚以及测定氨氮的相关试剂等. COD测定仪、可见分光光度计、台式离心机、脂肪酸鉴定系统等.
1.2 耐盐菌的驯化试验选取李村河污水处理厂二沉池污泥,按污泥:水=1:1的比例配成12 L活性污泥溶液于容器中,每日投加碳源、氮源、磷源分别为:葡萄糖12 g,硫酸铵1.42 g,磷酸二氢钾0.344 g.间歇曝气,每4 h一个周期,曝气3 h,停歇1 h.每天换水一次,投加无碘盐逐步提高系统的盐度,使其盐度从最开始的0,逐渐提升至0.5%、1%、1.5%、2%.每个盐度下分别对活性污泥的活性和降解COD、NH4+-N性能进行测定,探索不同盐度下活性污泥的污泥体积指数SVI的变化规律,同时提取出污泥中微生物的脂肪酸进行分析,得出耐盐驯化过程中活性污泥系统微生物菌群的变化.
1.3 微生物脂肪酸的提取、分析依据微生物脂肪酸的提取步骤,提取所需微生物的脂肪酸,运用MIDI-Sherlock全自动微生物鉴定系统[16]进行菌群的鉴定分析.
1.4 高效降解含盐结晶紫废水活性污泥的驯化取结晶紫粉末于3L烧杯中加蒸馏水,制成2 500 mL质量浓度为5 mg·L-1的模拟染料废水.最初向烧杯中加入50 mL 5 mg·L-1的结晶紫废水,烧杯中加入500 mL活性污泥溶液,加水定容至3 000 mL,曝气驯化培养一周,然后每周逐渐多加200 mL结晶紫废水,共驯化10周,得到可以高效降解含盐结晶紫废水的活性污泥.
1.5 四氧化三铁磁纳米粒子(MNPs)驯化含盐结晶紫废水中的活性污泥将驯化好的500 mL活性污泥放入3 000 mL烧杯中,加入2 500 mL质量浓度为5 mg·L-1的模拟染料废水,再向烧杯中加入0.57 g四氧化三铁磁纳米粒子(MNPs),搅拌,曝气培养8周,即得到试验所需微生物,用于后续的DNA测序分析.
1.6 微生物DNA的提取、分析本试验采用PowerSoil DNA Isolation Kit试剂盒直接提取活性污泥中微生物的DNA,得到高品质的DNA可直接进行PCR扩增,利用高通量测序技术[17, 18],研究不同盐度下微生物群落的变化情况.
PCR仪:ABI GeneAmp® 9700.
测序平台:采用Illumina MiSeq/HiSeq测序平台,不同样品的Tags和OTU数目统计,OUT丰度聚类图,为了研究样品的物种组成及多样性信息,对所有样品的全部Effective Tags序列进行聚类(默认选取Identity为97%),形成OTU.
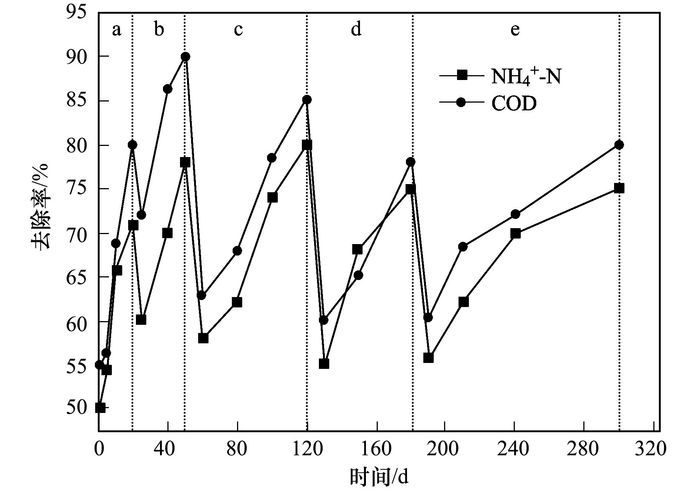
2 结果与讨论 2.1 不同盐度下活性污泥对废水COD、NH4+-N的降解驯化初期系统盐度为0%,由于污泥在容器中处于缺氧环境,活性较差COD去除率仅为55%左右. a阶段(0~20 d)是系统在盐度为0条件下的污泥驯化阶段,20 d时COD和NH4+-N的去除率分别提高至80%和71%;b阶段(20~50 d)是系统在盐度为0.5%条件下的污泥驯化阶段,COD、NH4+-N降解效率先下降后升高,驯化30 d后,COD和NH4+-N的去除率可提高到90%和78%;50d开始将系统的盐度提高到1%,此时进入c阶段(50~120 d),驯化至120 d时污泥COD去除率达85%以上,NH4+-N的去除率在80%左右;120 d后进入d阶段,此时系统的盐度为1.5%,此盐度下驯化维持60 d,COD去除率达到78%左右,NH4+-N去除率75%;180 d继续提高系统盐度至2%,系统进入e阶段(180~300 d),COD、NH4+-N的降解效率下降明显,190 d时,COD去除率低至60.5%,NH4+-N去除率56%,保持盐度2%继续驯化,最终在300 d时测得COD去除率为80%左右,NH4+-N去除率75%,达到预期处理效果,如图 1所示.
|
图 1 COD、NH4+-N降解趋势 Fig. 1 Degradation trends of COD and NH4+-N |
系统盐度每提高一次,污泥对有机物和氨氮的去除效率总是先下降后逐渐恢复的趋势.盐度提高,微生物细胞的渗透压增高,细胞脱氢酶的活性降低,导致活性污泥系统受到冲击,部分微生物由于无法适应高盐环境,甚至出现死亡现象,使得处理效果减弱[19],表现为COD、NH4+-N的去除率下降.随着长时间的不断驯化,系统抗盐度冲击的能力逐渐增强,适应高盐环境的微生物群落开始不断生长繁殖,对COD、NH4+-N的降解效率提高.
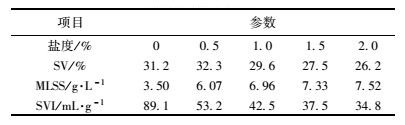
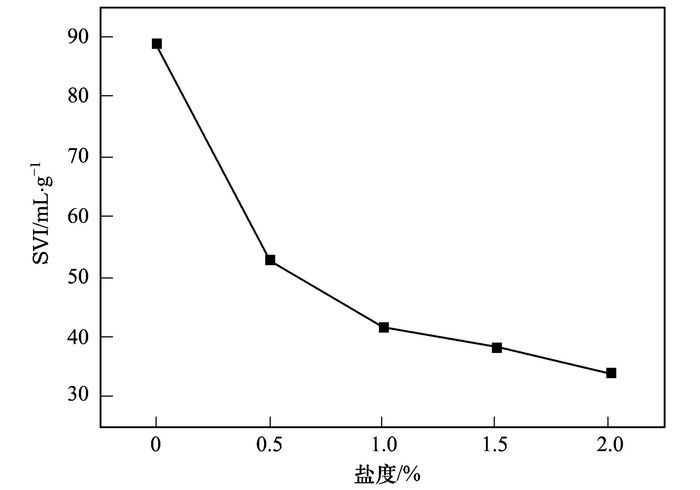
2.2 不同盐度下活性污泥SVI值的变化由表 1和图 2可知,随着盐度的增加,接种时活性污泥的浓度为3.50 g·L-1,经过300 d驯化后,在系统2%的条件下,活性污泥的浓度增加到7.52 g·L-1,而活性污泥的污泥体积指数SVI值不断减小.在无盐的环境中,污泥的SVI值在89 mL·g-1,在盐度0.5%的环境中,污泥的SVI值下降到53 mL·g-1,随着盐度的不断增加,当盐度为2%时,污泥的SVI值不足35 mL·g-1.随着盐度增加,活性污泥SVI值降低,可能是由于:高盐环境下微生物的生长和生物活性均受到抑制,微生物从絮凝体上脱落上浮水中,无法沉降;盐度的冲击改变了污泥的生态环境,游离原生微型后生动物的捕食受到影响而死亡,污泥沉降性减弱[20];高盐冲击使微生物生理活动受阻,胞外分泌物减少,污泥的絮凝性能下降.而有研究认为高盐环境下,微生物为了生存聚集在一起,通过自身调节使胞外多聚物的分泌增加,既可以抵御盐度的毒害又增加了絮凝性能,表现为絮体颗粒变大密实,改善了沉降性能[21].
|
|
表 1 各盐度下活性污泥SV、MLSS、SVI的变化 Table 1 Changes of SV, MLSS and SVI values under different salinity |
|
图 2 各盐度下SVI值 Fig. 2 SVI values under different salinity |
利用MIDI-Sherlock全自动微生物鉴定系统对各个盐度所提取出的脂肪酸进行分析,得到各种脂肪酸分布图谱.将5个盐度下脂肪酸鉴定图谱中各菌群所占含量用Origin软件处理并整理得到图 3.

|
图 3 对应盐度下各菌群所占百分比 Fig. 3 Percentages of bacteria corresponding to different salinity |
从活性污泥微生物的组成来看,主要由AM菌、革兰氏阴性菌、真核生物、放线菌、革兰氏阳性菌和厌氧菌等组成.随着盐度的升高,革兰氏阳性菌数量表现为先增加后减小,其中在盐度为1%时含量最高,为47%,整个盐度梯度革兰氏阳性菌含量均维持在35%以上;而革兰氏阴性菌所占百分比随着盐度的增加,表现出明显下降的趋势,从百分含量45%左右减小到10%左右;真菌和放线菌随着盐度的升高百分含量均有一定程度上增加,其中真菌的增加幅度较为明显,从5%左右升高到30%,放线菌百分含量由5%增加到15%左右;图 3中看出真核生物的百分含量随盐度变化不大,在5%左右波动;AM菌和厌氧菌在活性污泥微生物群落中所占比例很小,约为5%,随着盐度的增加,百分含量下降,盐度1%以上百分含量基本为0%.
革兰氏阳性菌细胞壁较厚,肽聚糖含量丰富,并且含有大量的磷壁酸,这种结构使其可以抵抗较高的盐度冲击,因此革兰氏阳性菌是每个对应盐度下的优势菌种;革兰氏阴性菌细胞壁以及肽聚糖膜要比革兰氏阳性菌要薄得多,因此随着盐度的增加,革兰氏阴性菌越来越少;真菌和放线菌中含有大量的累积相容性物质,如糖类、醇类、脯氨酸、天冬氨酸和谷氨酸等,可大大提高耐盐性,使其随着盐度的增加含量逐渐增大[22].因此,随着不断的驯化,污泥在高盐环境中的活性良好,可高效地处理废水.
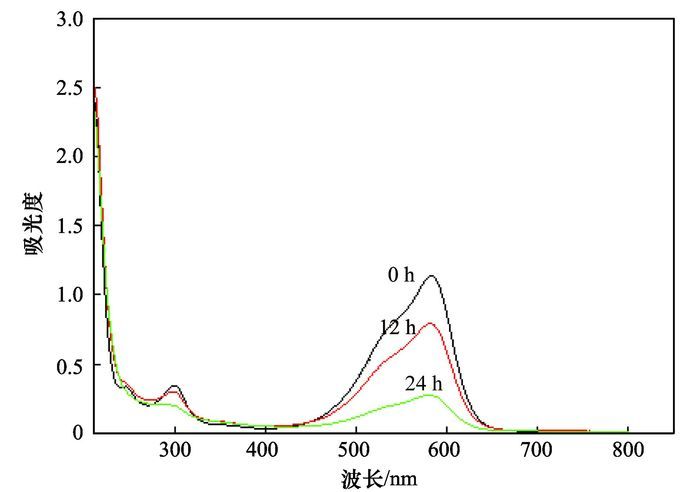
2.4 模拟染料废水降解趋势向驯化好的活性污泥中加入质量浓度为5 mg·L-1的结晶紫染料废水,曝气培养,每隔12 h取样一次,并于紫外-可见光分光光度计上进行波长扫描,扫描出的数据用Origin软件绘图,结果如图 4.从中可以看出,结晶紫在582 nm处有最大吸收峰,在301 nm处有吸收峰. 0 h最大吸收峰处的吸光度值为1.136,12 h最大吸收峰处的吸光度值为0.791,24 h最大吸收峰处的吸光度值为0.274,分别代入标准曲线计算得出结晶紫的浓度分别为5、3.45、1.16mg·L-1.可以直观地看出结晶紫浓度随时间逐渐降低,验证了经高盐结晶紫模拟废水驯化的活性污泥对结晶紫具有显著的降解能力[23].
|
图 4 结晶紫吸收峰变化 Fig. 4 Change of crystal violet absorption peak |
对耐盐驯化阶段的活性污泥(盐度分别为0.5%、1%、1.5%、2%)以及高盐结晶紫染料驯化阶段的污泥和MNPs驯化出的功能微生物进行DNA提取并对提取出的DNA进行16S rDNA测序分析[24].
DNA检测合格后(浓度>ng·μL-1),用一对特异性引物(515F、806R)对16S rDNA的V4区进行PCR扩增,电泳结果如图 5.从中可以看出PCR扩增的条带清晰,大小合适,产物纯度较高,且无杂带,可以进行DNA测序分析.
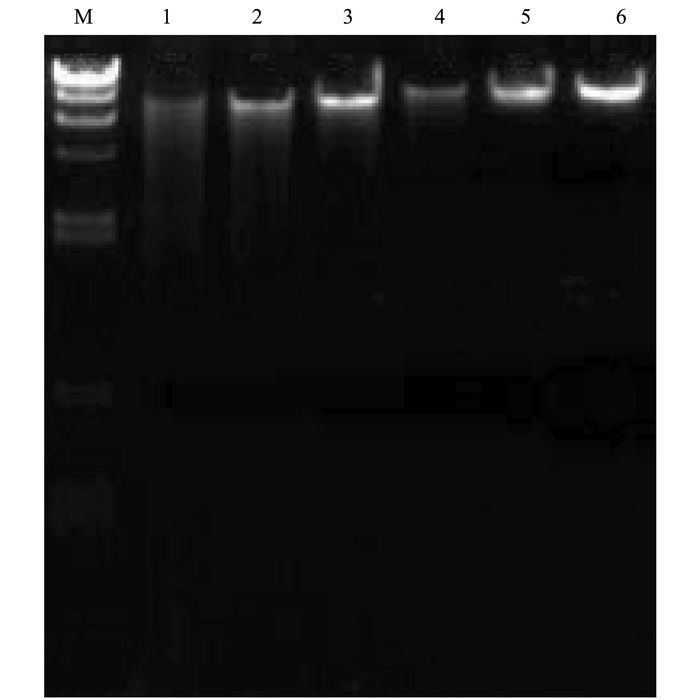
|
图 5 活性污泥细菌16S rDNA基因的PCR产物 Fig. 5 PCR products of 16S rDNA gene from bacteria in activated sludge |
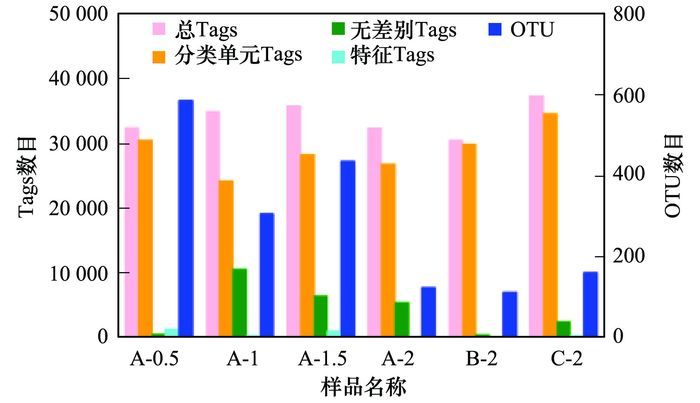
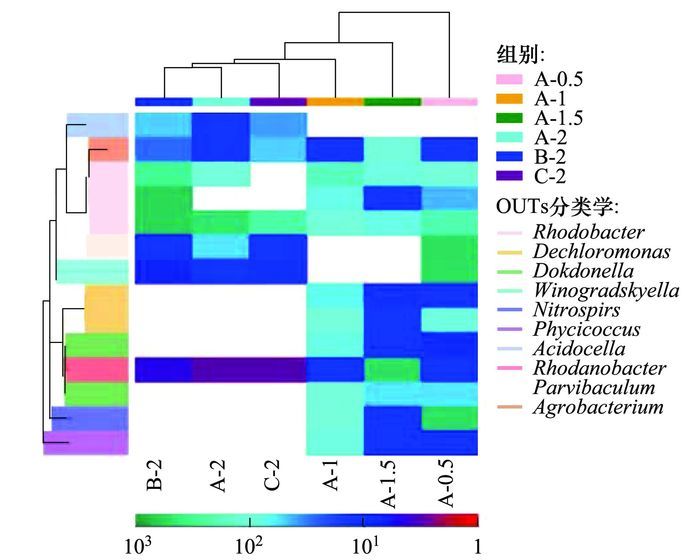
由图 6可以看出样品A-0.5、A-1、A-1.5、A-2中OUT数目分别为600、300、420、160,随着盐度的不断增加,微生物群落的生长受到不同程度的抑制,致使微生物的多样性逐渐降低[25].图 7中横向为样品信息,纵向为OTU ID,其中左侧的聚类树为OTU的系统发生关系及其物种注释,分支的颜色表示OTU所在的属(右边的图例),上方的聚类树为样品聚类树,中间的热图是OTU的相对丰度热图,颜色与相对丰度的关系见图 7中的刻度尺.从中可以看出,随着盐度的不断增加,酸胞菌属(Acidocella)、丽水菌属(Winogradskyella)、产黄杆菌(Rhodanobacter)、红菌(Parvibaculum)含量均有不同程度的升高,并且产黄杆菌(Rhodanobacter)成为优势菌种;而变形菌属(Dechloromonas)、黄色单胞菌(Dokdonella)、硝化螺菌属(Nitrospira)、海藻球菌属(Phycicoccus)含量呈下降趋势,体现了微生物群落随系统的盐度逐步演变的过程.
|
样品编号A-0.5、A-1、A-1.5、A-2、B-2、C-2分别对应盐度为0.5%、1%、1.5%、2%活性污泥样品、结晶紫驯化活性污泥样品和MNPs驯化出的功能微生物样品,下同 图 6 不同样品的Tags和OTU数目统计 Fig. 6 Statistics of Tags and OTU number in different samples |
|
图 7 OUT丰度聚类图 Fig. 7 Clustering of OTU abundance |
将样品B-2(盐度2%的条件下加入结晶紫染料的活性污泥样品)与A-2(盐度2%的活性污泥样品)对比发现:红细菌属(Parvibaculum)、丽水菌属(Winogradskyella)含量有所增加,而产黄杆菌(Rhodanobacter)、农杆菌属(Agrobacterium)含量降低,说明红细菌属(Parvibaculum)、丽水菌属(Winogradskyella)能以结晶紫染料为能源物质,将其分解为其他小分子物质,一方面用于自身的生长代谢,另一方面用于细胞的增殖,使得菌属的含量升高;结晶紫染料不能作为能源物质被产黄杆菌(Rhodanobacter)、农杆菌属(Agrobacterium)所利用,致使没有可以维持其自身生长代谢的物质,从而使得该菌属的数量不断降低.
将样品C-2(加入MNPs的结晶紫-活性污泥样品)与样品B-2对比可以看出:酸胞菌属(Acidocella)、产黄杆菌(Rhodanobacter)含量增加,其原因是由于加入的MNPs有效地提高了污泥抗有毒有害物质冲击的能力,并且MNPs可作为微生物生长栖息的载体,提高了酸胞菌属(Acidocella)、产黄杆菌(Rhodanobacter)的生物活性,便于其生长繁殖,使其含量增加;红细菌属(Parvibaculum)、农杆菌属(Agrobacterium)含量减小,可能由于MNPs的加入改变菌属的生物磁场以及四氧化三铁对该菌属的微毒性,影响其新陈代谢等生命活动,导致数量下降.
3 结论(1) 系统的盐度显著影响活性污泥的活性和沉降性能,随着盐度的增加,系统对废水COD、NH4+-N的去除率以及SVI值均有所降低.经过一定时间驯化后,在系统盐度在2%的条件下,COD和NH4+-N的去除率基本稳定在80%和75%左右,SVI值从开始的89 mL·g-1下降到34 mL·g-1.
(2) 随着系统盐度的增加,革兰氏阴性菌优势菌种的位置逐渐被真菌和放线菌取代,而革兰氏阳性菌依然为优势菌种;盐度对真核生物的生长影响不大,对AM菌和厌氧菌起到抑制作用.
(3) 驯化后的活性污泥可较好的处理含盐结晶紫废水,经24 h生化处理,结晶紫废水的降解率可达76.8%.
(4) 随着盐度的增加微生物的多样性逐渐减少,其中酸胞菌属、丽水菌属、产黄杆菌、红菌含量升高,产黄杆菌成为优势菌种,变形菌属、黄色单胞菌、硝化螺菌属含量下降.
(5) 加入结晶紫染料的活性污泥中红细菌属、丽水菌属含量有所增加,产黄杆菌、农杆菌属含量减小.加入MNPs的结晶紫-活性污泥中酸胞菌属、产黄杆菌含量增加,红细菌属、农杆菌属含量减小.
| [1] | Mabrouk A A, Nafey A S, Fath H E S. Thermoeconomic analysis of some existing desalination processes[J]. Desalination, 2007, 205(1-3): 354–373. DOI: 10.1016/j.desal.2006.02.059 |
| [2] | Lee K P, Arnot T C, Mattia D. A review of reverse osmosis membrane materials for desalination-development to date and future potential[J]. Journal of Membrane Science, 2011, 370(1-2): 1–22. DOI: 10.1016/j.memsci.2010.12.036 |
| [3] | Zejli D, Ouammi A, Sacile R, et al. An optimization model for a mechanical vapor compression desalination plant driven by a wind/PV hybrid system[J]. Applied Energy, 2011, 88(11): 4042–4054. DOI: 10.1016/j.apenergy.2011.04.031 |
| [4] | Afonso M D, Bórquez R. Review of the treatment of seafood processing wastewaters and recovery of proteins therein by membrane separation processes-prospects of the ultrafiltration of wastewaters from the fish meal industry[J]. Desalination, 2002, 142(1): 29–45. DOI: 10.1016/S0011-9164(01)00423-4 |
| [5] | Roy D, Rahni M, Pierre P, et al. Forward osmosis for the concentration and reuse of process saline wastewater[J]. Chemical Engineering Journal, 2016, 287: 277–284. DOI: 10.1016/j.cej.2015.11.012 |
| [6] | Khajavi S, Jansen J C, Kapteijn F. Production of ultra pure water by desalination of seawater using a hydroxy sodalite membrane[J]. Journal of Membrane Science, 2010, 356(1-2): 52–57. DOI: 10.1016/j.memsci.2010.03.026 |
| [7] | 徐寒莉, 梁志伟, 毛巍, 等. 盐分对生物脱氮工艺中硝化反应的影响与机理[J]. 应用生态学报, 2014, 25(7): 2132–2140. Xu H L, Liang Z W, Mao W, et al. Impact of salinity on nitrification in the process of biological nitrogen removal and its mechanism:a review[J]. Chinese Journal of Applied Ecology, 2014, 25(7): 2132–2140. |
| [8] | Zhang Y, Li B, Xu R X, et al. Effects of pressurized aeration on organic degradation efficiency and bacterial community structure of activated sludge treating saline wastewater[J]. Bioresource Technology, 2016, 222: 182–189. DOI: 10.1016/j.biortech.2016.10.005 |
| [9] | Taheri E, Khiadani M H, Amin M M, et al. Treatment of saline wastewater by a sequencing batch reactor with emphasis on aerobic granule formation[J]. Bioresource Technology, 2012, 111: 21–26. DOI: 10.1016/j.biortech.2012.01.164 |
| [10] | Tan S W, Cui C Z, Chen X C, et al. Effect of bioflocculation on fouling-related biofoulants in a membrane bioreactor during saline wastewater treatments[J]. Bioresource Technology, 2017, 224: 285–291. DOI: 10.1016/j.biortech.2016.10.066 |
| [11] | 汪善全, 原媛, 孔云华, 等. 好氧颗粒污泥处理高含盐废水研究[J]. 环境科学, 2008, 29(1): 145–151. Wang S Q, Yuan Y, Kong Y H, et al. Experimental investigation of high saline wastewater treatment using aerobic granules[J]. Environmental Science, 2008, 29(1): 145–151. |
| [12] | Yurtsever A, Calimlioglu B, G rür M, et al. Effect of NaCl concentration on the performance of sequential anaerobic and aerobic membrane bioreactors treating textile wastewater[J]. Chemical Engineering Journal, 2016, 287: 456–465. DOI: 10.1016/j.cej.2015.11.061 |
| [13] | 操庆, 曹海生, 魏晓兰, 等. 盐胁迫对设施土壤微生物量碳氮和酶活性的影响[J]. 水土保持学报, 2015, 29(4): 300–304. Cao Q, Cao H S, Wei X L, et al. Effect of salt stress on carbon and nitrogen of microbial biomass and activity of enzyme in greenhouse soil[J]. Journal of Soil and Water Conservation, 2015, 29(4): 300–304. |
| [14] | Wagner M, Loy A, Nogueira R, et al. Microbial community composition and function in wastewater treatment plants[J]. Antonie van Leeuwenhoek, 2002, 81(1-4): 665–680. |
| [15] | Lee T H, Kurata S, Nakatsu C H, et al. Molecular analysis of bacterial community based on 16S rDNA and functional genes in activated sludge enriched with 2, 4-dichlorophenoxyacetic acid(2, 4-D) under different cultural conditions[J]. Microbial Ecology, 2005, 49(1): 151–162. DOI: 10.1007/s00248-003-1035-6 |
| [16] | Vandamme P, Pot B, Gillis M, et al. Polyphasic taxonomy, a consensus approach to bacterial systematics[J]. Microbiology and Molecular Biology Reviews, 1996, 60(2): 407–438. |
| [17] | Simon C, Daniel R. Metagenomic analyses:past and future trends[J]. Applied and Environmental Microbiology, 2011, 77(4): 1153–1161. DOI: 10.1128/AEM.02345-10 |
| [18] | Voordouw G, Voordouw J K, Jack T R, et al. Identification of distinct communities of sulfate-reducing bacteria in oil fields by reverse sample genome probing[J]. Applied and Environmental Microbiology, 1992, 58(11): 3542–3552. |
| [19] | 周鹏. 盐度冲击对活性污泥系统性能影响的研究[J]. 环境科学与技术, 2011, 34(5): 65–68. Zhou P. Effect of salinity shock on activated sludge system[J]. Environmental Science & Technology, 2011, 34(5): 65–68. |
| [20] | 宋晶, 孙德栋, 王一娜, 等. 直接驯化嗜盐菌处理高盐废水的研究[J]. 环境污染与防治, 2010, 32(2): 51–54, 58. Song J, Sun D D, Wang Y N, et al. Acclimatization of halophilic bacteria for biology treatment of high-salt wastewater[J]. Environmental Pollution and Control, 2010, 32(2): 51–54, 58. |
| [21] | 赵凯峰, 王淑莹, 叶柳, 等. NaCl盐度对耐盐活性污泥沉降性能及脱氮的影响[J]. 环境工程学报, 2010, 4(3): 570–574. Zhao K F, Wang S Y, Ye L, et al. Effects of salinity on salt-tolerant activated sludge settling performance and nitrogen removal[J]. Chinese Journal of Environmental Engineering, 2010, 4(3): 570–574. |
| [22] | Hahne H, Mäder U, Otto A, et al. A comprehensive proteomics and transcriptomics analysis of Bacillus subtilis salt stress adaptation[J]. Journal of Bacteriology, 2010, 192(3): 870–882. DOI: 10.1128/JB.01106-09 |
| [23] | Chen C H, Chang C F, Ho C H, et al. Biodegradation of crystal violet by a Shewanella sp. NTOU1[J]. Chemosphere, 2008, 72(11): 1712–1720. DOI: 10.1016/j.chemosphere.2008.04.069 |
| [24] | Jiang J L, Alderisio K A, Singh A, et al. Development of procedures for direct extraction of Cryptosporidium DNA from water concentrates and for relief of PCR inhibitors[J]. Applied and Environmental Microbiology, 2005, 71(3): 1135–1141. DOI: 10.1128/AEM.71.3.1135-1141.2005 |
| [25] | Guo X C, Miao Y, Wu B, et al. Correlation between microbial community structure and biofouling as determined by analysis of microbial community dynamics[J]. Bioresource Technology, 2015, 197: 99–105. DOI: 10.1016/j.biortech.2015.08.049 |
 2017, Vol. 38
2017, Vol. 38