2. 寒旱地区水资源综合利用教育部工程研究中心, 兰州 730070;
3. 甘肃农业大学资源与环境学院, 兰州 730070
2. Engineering Research Center for Cold and Arid Regions Water Resource Comprehensive Utilization, Ministry of Education, Lanzhou 730070, China;
3. College of Natural Resources and Environment, Gansu Agricultural University, Lanzhou 730070, China
在我国西北以丘陵沟壑为主要地貌的村镇地区,地表水资源匮乏,长期存在着人畜饮水安全危机.为了解决西北干旱缺水地区的群众生活饮用水问题,从20世纪80年代开始实施了以“饮水解困”为目标的集雨水利用工程,实现了农村“饮水解困”目标.然而,随着“饮水解困”目标的实现,由窖水的水质污染问题引发的“饮水安全”问题逐步凸显,已成为制约该地区人民生活水平进一步提高的主要因素之一.经课题组多年的研究实践发现,雨水经集流面汇集入窖后,窖水水质状况发生了较大变化,与通常的蓄水容器蓄积水相比未发现窖水发臭、变质等现象,相反表征窖水中污染物的各项水质指标均出现下降趋势,水质在一定程度上逐渐变好.经研究,窖水水质变化经历了3个阶段:初期沉降阶段 (1~15 d)、中间过渡阶段 (15~100 d) 和后期稳定阶段 (100~250 d),在初期沉降阶段各污染物浓度迅速降低,影响窖水水质变化的主要因素有泥沙物理沉降和生物降解作用,但以泥沙沉降为主; 在中间过渡阶段以生物降解为主,在窖水水质的自然变化过程中,微生物发挥了非常重要的作用[1].
微生物的生态特征可分为结构特征和功能特征,结构特征是决定生物群落生态功能的关键,也是认识与解决环境问题的有效切入点.近年来,借助生态学多样性测度方法研究微生物多样性、群落结构及功能在诸如海洋、湖泊、水库、污水处理系统等领域的水环境污染及修复问题中的应用日益广泛.研究手段已经从依赖分离钝化培养的形态学及生物化学的分析技术,以及如变性梯度凝胶电泳 (PCR-DGGE) 技术、温度梯度凝胶电泳 (TGGE)、实时荧光定量PCR技术 (Q-PCR)、限制性片段长度多态性分析 (RFLP)、末端标记限制性片段长度多态性分析 (T-RFLP) 等分子生物学的传统分析技术向高通量测序技术分析逐渐转化[2].
水体中微生物是水域生态系统的重要组成部分,微生物群落状况与水质状况有着密切的联系,其结构的变化对水质污染负荷积累有很好的响应,它们在生源要素循环、有机物分解和污染物净化等方面起着非常重要的作用[3~6].目前,对于窖水水质变化过程中的生物降解作用,即窖水细菌多样性及群落结构与功能和其主要水质因子之间关系的研究还十分缺乏.本文应用16S rRNA基因-Illumina MiSeq高通量测序技术,分析研究窖水细菌多样性及群落结构与功能及其与主要水质因子之间关系.
1 材料与方法 1.1 水样采集本研究根据我国干旱半干旱典型集雨地区集雨现状,针对不同集流面和不同水窖类型的组合进行了现场取样.不同类型的集流面和水窖类型组合取平行样3个,分别用数字进行编号,其中编号1、2、3为黄土地集流面+红黏土水窖组 (A),编号4、5、6为黄土地集流面+混凝土水窖组 (B),编号7、8、9为混凝土集流面+混凝土水窖组 (C),编号10、11、12为混凝土集流面+红黏土水窖组 (D).
在水窖水面下2 m处取1 000 mL的窖水水样经0.22 μm聚碳酸酯膜 (Millipore, Cork, Ireland) 负压过滤,收集滤膜,用事先灭菌且经过75%的酒精消毒的医用剪刀剪碎后,置于2 mL的无菌离心管中,于-80℃的超低温冰箱中保存至DNA提取.
1.2 细菌基因组DNA的提取及质量检测窖水水样的细菌基因组DNA采用Water DNA Isolation Kit试剂盒 (成都福际生物技术有限公司) 进行提取,之后进行基因组DNA浓度、纯度检测及完整性检测.基因组DNA浓度、纯度检测待DNA完全溶解后,将TE缓冲液设为空白对照,移取2 μL,使用超微量紫外可见分光光度 (美国Quawell Q5000) 计检测其浓度,并根据D260/D280的值检测其纯度.基因组DNA完整性检测根据浓度检测结果,移取3 μL DNA,采用0.8%琼脂糖凝胶电泳检测,在凝胶成像系统上拍照观察分析DNA样品的完整性,电压为120 V,电泳时间约为20 min.
1.3 高通量测序及数据分析 1.3.1 MiSeq高通量测序将提取到的基因组DNA, 对16S rRNA V4区进行扩增, 以12个水窖水样样本中提取的12个DNA原液作为PCR模板,利用细菌16S rDNA通用引物进行扩增.通用引物序列为F:520F:(5′-AYTGGGYD TAAAGNG-3′), R:802R:(5′-TACNVGGGTATC TAATCC-3′). PCR反应条件为:98℃预变性3 min;98℃变性30 s;50℃退火30 s;72℃延伸30 s;27个循环;72℃后保温5 min,于4℃保存.反应结束后配制2%琼脂糖凝胶电泳检测PCR产物.
根据PCR扩增图谱,扩增后的条带比较清晰明显且条带位置一致,背景干净,DNA浓度达到了扩增要求,可直接用于后续分析.委托上海派森诺生物科技有限公司进行Illumina MiSeq高通量测序.
1.3.2 测序数据优化处理保证分析结果的准确性,运用Qiime (version 1.9.0,http://qiime.org/) 进行序列过滤,数据过滤标准为:去除5′端引物错配碱基数 > 1的序列;去除含有N (模糊碱基) 的序列;去除含有连续相同碱基数 > 8的序列;去除长度≤150 bp的序列;去除嵌合体序列.运用Mothur软件 (version1.31.2,http://www.mothur.org/) 中Uchime的方法去除嵌合体序列,得到最终用于后续分析的优质序列.
1.3.3 OTU聚类分析及注释在Qiime中调用Uclust的方法对优质序列按序列相似度0.97进行聚类,选取每个类中最长的序列为代表序列.在Qiime中调用Blast的方法对序列数据库进行比对,获得每个OTU代表序列的分类学信息.注释数据库为:Greengene (Release 13.8,http://greengenes.secondgenome.com/).之后对OTU进行精简处理,去掉丰度值小于总的序列条数的0.001%的OTU,得到后续的分析使用精简后的OTU列表.
1.3.4 多样性及群落结构分析根据OTU列表中的各样品物种丰度情况,应用软件Mothur中的summary. single命令,计算种群丰富度指数Chao指数和ACE指数, 计算群落多样性指数Shannon指数和Simpson指数.组内3个平行样数据批量导入Qiime软件,对OTU表进行组间差异性分析,生成不同分类水平上 (门、纲) 的物种丰度表和多样品物种分布图, 冗余分析 (RDA) 研究水体细菌群落和水体环境因子之间的关系.
2 结果与分析 2.1 窖水水体理化性质不同类型水窖水体物理化学因子由表 1所示.总体上窖水水质呈弱碱性,浊度明显超标,尤其是TN、TP指标超标严重.
|
|
表 1 不同类型水窖水体物理化学指标 Table 1 Physicochemical parameters of cellar water of different types |
2.2 微生物群落多样性分析
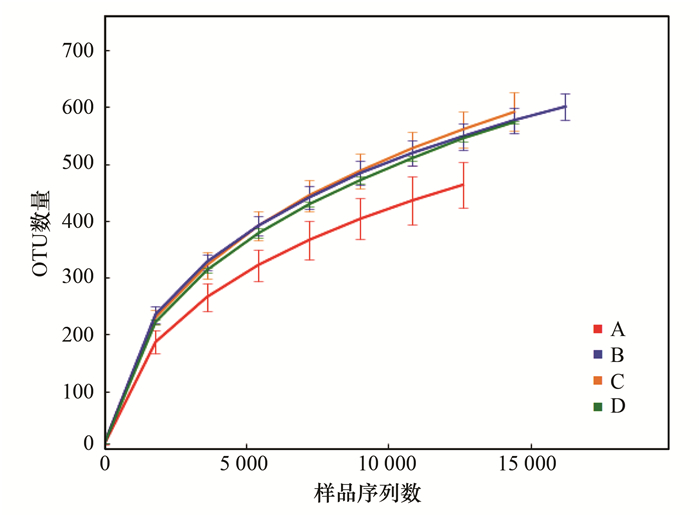
利用高通量测序技术,经序列过滤和去除嵌合体序列,4个窖水样品最终得到用于后续分析的优质序列201 240条,优质率为92.99%.优质序列的序列长度分布在220~230 nt,其中以225 nt的序列居多.采用Qiime,在97%相似度下将其聚类为用于物种分类的OTU,统计各样品在不同OTU中的丰度信息,4个样品共产生1 605条OTU.其中A、B、C、D 4组样品中包含的OTU数目分别为842、898、887、924.其中,D样品的OTU最多,为924个;A样品的OTU最少,为842个 (表 2).其中,4个样本共有的OTU数目为245个,A、B、C、D特有的OTU数目分别为109、91、175、111,其余OTU为样本两两共有的.其中样本C中特有的OTU数目最多,预示着较多的特有微生物种类. B与D共有的OTU数目较多,为564条,见图 1.对OTU利用Qiime生成从门到属依次进行分类, 结果见表 2.本研究共获得窖水细菌群落22门、42纲、71目、115科、146属.
|
|
表 2 窖水水体细菌16S DNA序列、丰富度及多样性 Table 2 Pearson's correlation coefficient of biodiversity and abundance of bacterial 16S DNA in cellar water |
|
图 1 OTU分布维恩图 Fig. 1 Venn diagram showing the distribution of OTUs |
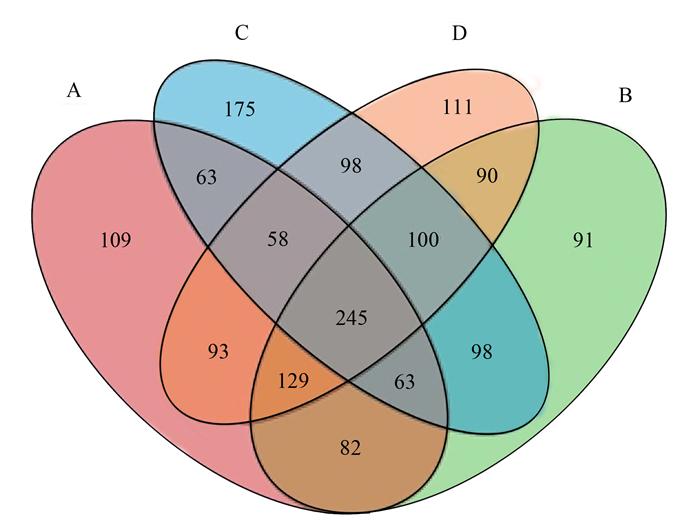
采用对测序序列进行随机抽样的方法, 以抽到的序列数与它们所能代表的数目构建稀释性曲线 (图 2).从图 2中可以看出, 4个样品的稀释曲线在0.97相似性水平下趋于平坦, 但仍未达到饱和,说明4个样品获取了绝大多数样本信息,基本能够反映窖水的微生物群落组成,但还有必要在后续研究中增加测序深度.
|
图 2 OTU稀释曲线 Fig. 2 Rarefaction curves of OTUs |
在97%分类水平上,通过应用Mothur软件计算各样品的丰富度和多样性指数 (见表 2). Chao或ACE是用来衡量群落丰度的指数,指数越大,说明群落丰富度越高.由表 2可见,4组样品的Chao和ACE指数大小顺序为D > B > C > A, 说明了4组水样中细菌群落丰度高低,OTU聚类结果也印证了该结果的准确性.对于群落的多样性,D样品的Simpson指数最小,A、B、C的Simpson指数变幅不大,说明D样品多样性高于其他组的多样性,Shannon指数的大小也印证了这一点. 4组水样细菌的多样性高低为D > C > B > A.
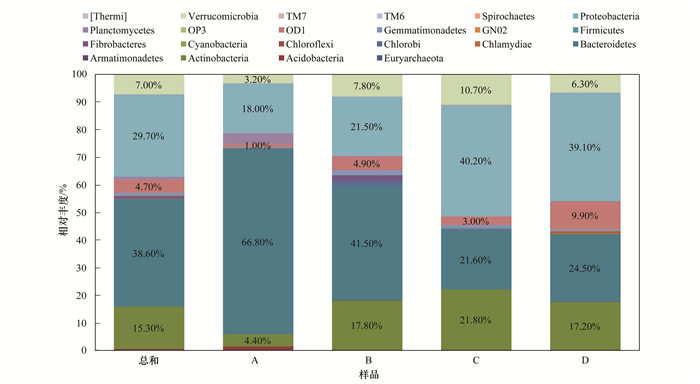
2.3 窖水优势菌群落分布 2.3.1 门、纲水平的优势菌群分布特征窖水水样在门水平上的细菌群落组成如图 3所示. 4组窖水水样细菌群落结构在门分类水平上具有较高的多样性,均达到12个门以上.主要包括拟杆菌门 (Bacteroidetes)、变形菌门 (Proteobacteria)、放线细菌门 (Actinobacteria)、疣微菌门 (Verrucomicrobia)、OD1、浮霉菌门 (Planctomycetes)、酸杆菌门 (Acidobacteria)、芽单胞菌门 (Gemmatimonadetes),并以拟杆菌门、变形菌门、放线细菌门、OD1、疣微菌门的微生物为主要类群,五者的相对丰度比例约占87.1%~94.8%.就平均而言,拟杆菌门在窖水占有最大的丰度比例,为38.6%,其次是变形菌门、放线菌门、疣微菌门,分别达到了29.70%、15.30%、7.00%.
|
图 3 样品中门水平细菌群落结构及分布 Fig. 3 Bacterial community structure and distribution of the samples at phylum level |
各组窖水水样在门分类水平上的菌群落丰度存在一定的差异.从图 3累积柱状图中的微生物相对丰度数据标签可知:拟杆菌门在水窖各组水样中相对丰度高低为A > B > C > D,变形菌门在水窖各组水样中相对丰度高低为C > D > B > A, 放线菌门在水窖各组水样中相对丰度高低为C > B > D > A, 疣微菌门在水窖各组水样中相对丰度高低为C > B > D > A, OD1在水窖各组水样中相对丰度高低为D > B > C > A.
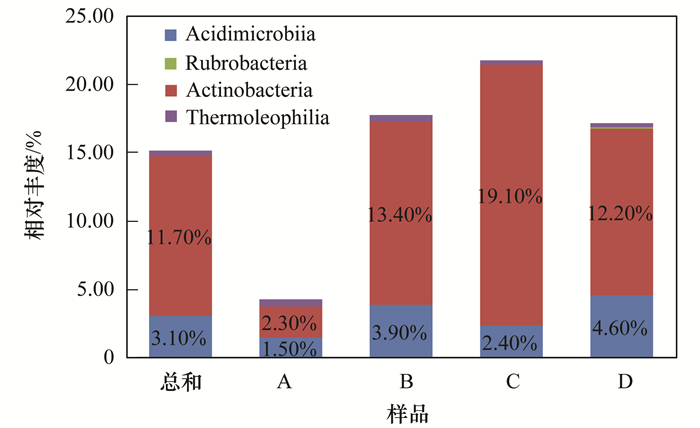
进一步对窖水主要微生物群落拟杆菌门、变形菌门、放线细菌门、OD1、疣微菌门菌群在纲水平上分布特征进行分析,结果见图 4~8.
|
图 4 放线菌门群落结构及分布 Fig. 4 Community structure and distribution of the Actinobacteria |
|
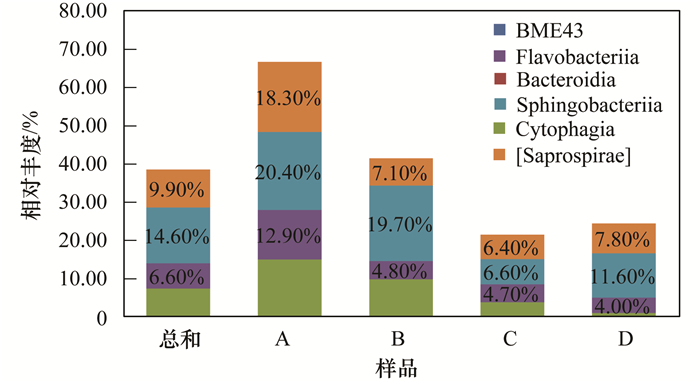
图 5 拟杆菌门群落结构及分布 Fig. 5 Community structure and distribution of the Bacteroidetes |
|
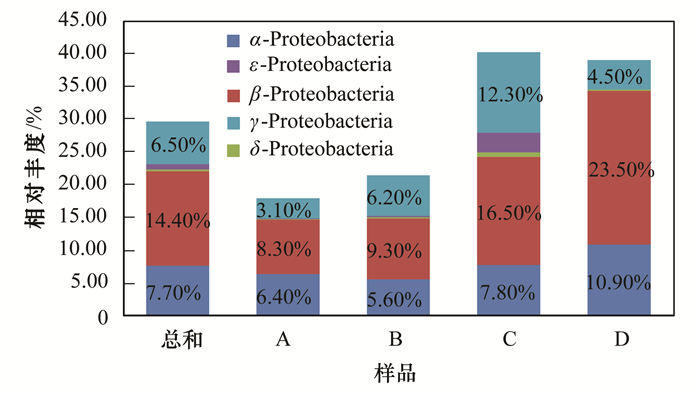
图 6 变形菌门群落结构及分布 Fig. 6 Community structure and distribution of the Proteobacteria |
|
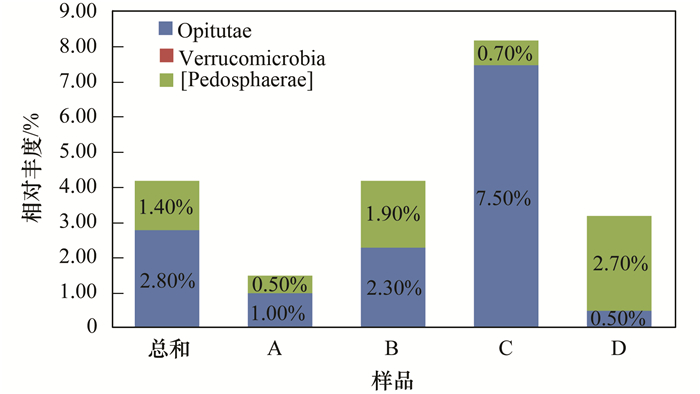
图 7 疣微菌门群落结构及分布 Fig. 7 Community structure and distribution of the Verrucomicrobia |
|
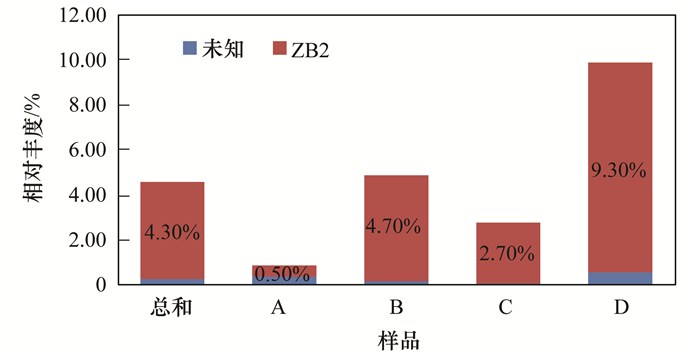
图 8 OD1群落结构及分布 Fig. 8 Community structure and distribution of the OD1 |
4组水样中放线细菌纲 (Actinobacteria) 是放线菌门丰度最大的菌群,其比例约占2.30%~19.10%,除此之外酸微菌纲 (Acidimicrobiia) 占有丰度比例也较高,而红色杆菌纲 (Rubrobacteria)、嗜热油菌纲 (Thermoleophilia) 的丰度较低;组成拟杆菌门的菌群有6个纲,丰度比例较高的有噬纤维菌纲 (Cytophagia)、黄杆菌纲 (Flavobacteriia)、鞘脂杆菌纲 (Sphingobacteriia)、Saprospirae,平均相对比例分别达到了7.30%、6.60%、14.60%、9.90%;变形菌门中α-变形菌纲 (α-Proteobacteria)、β-变形菌纲 (β-Proteobacteria)、γ-变形菌 (γ-Proteobacteria) 纲是相对占优势的变形菌,其相对比例分别占5.60%~10.90%、8.30%~23.50%、3.10%~12.30%,而δ-变形菌纲 (δ-Proteobacteria) 和ε-变形菌纲 (ε-Proteobacteria) 所占比例较低;疣微菌门中主要有丰收神菌纲 (Opitutae)、疣微菌纲 (Verrucomicrobiae)、Pedosphaerae,其所占比例分别为0.50%~7.50%、0.50%~2.70%、1.70%~3.60%;所有样品中ZB2是OD1中丰度最大的菌群,所占比例为0.50%~9.30%.
2.3.2 属水平的优势菌群分布特征以属为分类单元分析4组 (12个) 水样的细菌群落结果如图 9所示. 12个水样中细菌覆盖了146个属, 其中相对丰度大于1%的包括21个属,分别是黄杆菌属 (Flavobacterium, 5.70%)、沉积物杆状菌属 (Sediminibacterium, 5.40%)、噬酸菌属 (Acidovorax,3.90%)、多核杆菌属 (Polynucleobacter,3.80%)、Aquirestis(2.30%)、不动杆菌属 (Acinetobacter, 2.10%)、铁细菌属 (Crenothrix, 1.90%)、红细菌属 (Rhodobacter, 1.50%)、丰收神菌属 (Opitutus, 1.20%) 及未知属12个.共发现未知属55个 (56.80%).
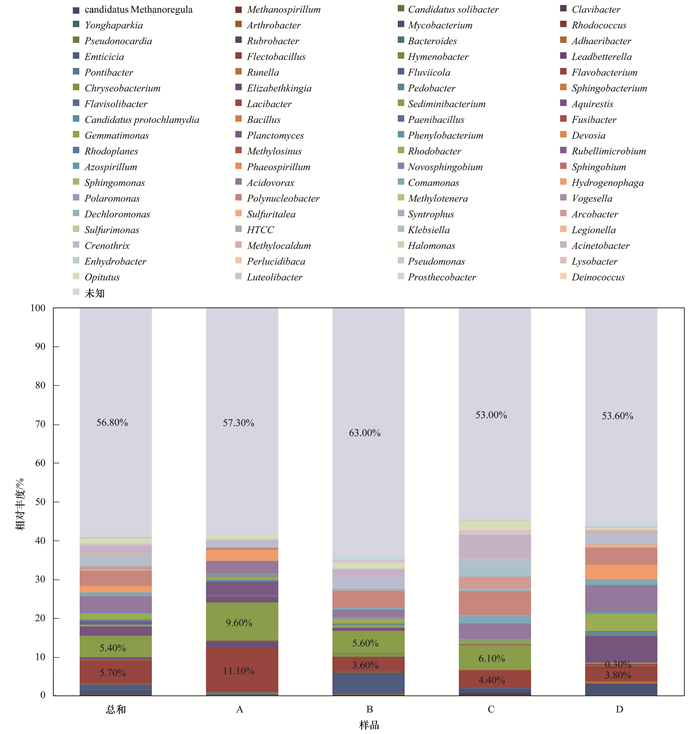
|
图 9 水样中属水平细菌群落结构及分布 Fig. 9 Bacterial community structure and distribution of the samples at genus level |
另外,4组窖水水样中共检测到WHO《饮用水水质准则》及基于Taylor等[7]总结的病原菌数据库中提及的潜在的水源性病原菌属9种,分别是黄杆菌属(Flavobacterium,5.70%)、不动杆菌属 (Acinetobacter,2.10%)、分枝杆菌属 (Mycobacterium,0.90%)、克雷伯氏菌属 (Klebsiella,0.90%)、弓形菌属 (Arcobacter,0.80%)、金黄杆菌属 (Chryseobacterium,0.20%)、弯曲杆菌属 (Flectobacillus,0.10%)、军团菌属 (Legionella,0.10%)、鞘氨醇单孢菌属 (Sphingomonas,0.10%).
如图 10,相对丰度大于1%的属水平上的未知菌群主要隶属于C111(2.70%)、放线细菌目 (Actinomycetales,2.20%)、ACK-M1(7.70%)、噬纤维菌科 (Cytophagaceae, 5.50%)、鞘脂杆菌目 (Sphingobacteriales,4.50%)、鞘脂杆菌科 (Sphingobacteriaceae,10.00%)、Saprospiraceae (1.60%)、柄杆菌科 (Caulobacteraceae, 1.00%)、嗜甲基菌科 (Methylophilaceae, 1.20%)、Cerasicoccaceae (1.20%)、R4-41B (2.00%) 的菌群.
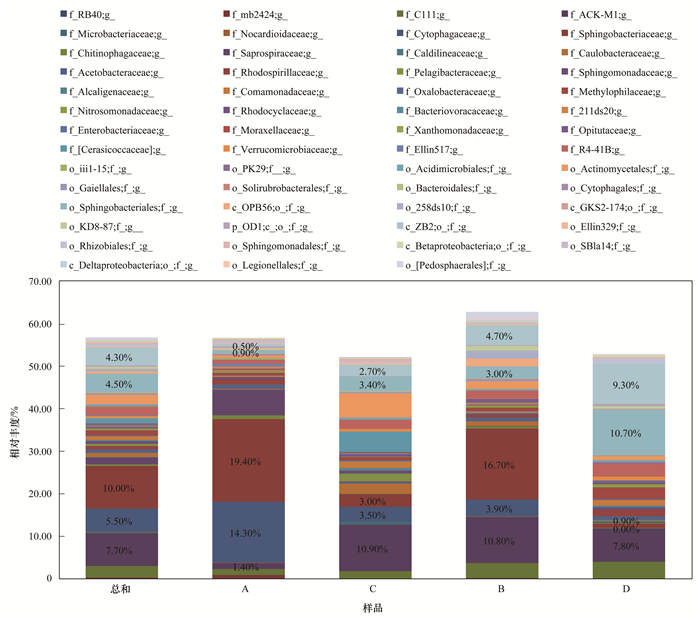
|
图 10 属水平上的未知菌群落结构及分布 Fig. 10 Bacterial community structure and distribution of the unclassified bacteria at genus level |
据以上物种聚类结果显示,窖水中细菌的丰度总体较高, 但不同类型的水窖存贮的窖水中的微生物多样性存在一定差异.即少数的常见菌群相对比例较高,为4组水样共同的绝对优势菌群,相对比例较低的细菌群落多样性各不相同,有显著的差别.
2.4 水窖优势细菌类群生态功能及细菌类群组成影响因子微生物群落的鲁棒性是微生物群落最主要的特性之一.微生物群落的鲁棒性主要表现为结构鲁棒性和功能鲁棒性, 其中功能鲁棒性是指微生物群落发挥着其固有的功能, 不会因为外界环境的变化发生大的改变.
对窖水优势种属中的功能菌属的占比情况进行统计分析,结果显示 (见表 3),窖水中的19种优势菌功能菌中,15种为具有脱氮除磷功能的微生物,占绝大多数.其中红螺菌科 (Rhodospirillaceae)、噬纤维菌科 (Cytophagaceae)、鞘脂杆菌科 (Sphingobacteriaceae)、亚硝化单胞菌科 (Nitrosomonadaceae)、红细菌属 (Rhodobacter)、脱氮单孢菌属 (Dechloromonas)、黄杆菌属 (Flavobacterium) 是常见的反硝化细菌,不动杆菌属 (Acinetobacter)、丛毛单孢菌属 (Comamonas)、克雷伯氏菌属 (Klebsiella)、假单孢菌属 (Pseudomonas)、嗜氢菌属 (Hydrogenophaga) 为异养硝化-好氧反硝化菌.除此之外,在磷的转化和循环过程中起作用的微生物有假单孢菌属 (Pseudomonas)、脱氮单孢菌属 (Dechloromonas)、Saprospiraceae、铁细菌属 (Crenothrix).
|
|
表 3 窖水样品优势菌及功能菌分布特征/% Table 3 Characteristics of dominant and functional bacteria in the samples of cellar water/% |
微生物的群落结构不仅受到生物因素的调控,也与非生物因素 (如环境因子等) 的影响密不可分.根据微生物群落结构和环境因子的RDA分析,可以把主要菌群分为3簇 (Ⅰ、Ⅱ、Ⅲ),如图 11.从RDA分析图中环境因子箭头的连线长度及环境因子与微生物之间箭头的夹角可以看出环境因子对微生物群落的影响.第Ⅱ簇内的菌属红螺菌科 (Rhodospirillaceae)、嗜氢菌属 (Hydrogenophaga)、醋杆菌科 (Acetobacteraceae)、calciphila、红细菌属 (Rhodobacter)、铁细菌属 (Crenothrix)、嗜甲基菌科 (Methylophilaceae)、facilis、分枝杆菌属 (Mycobacterium)、鞘脂杆菌目 (Sphingobacteriales)、R4-41B、ZB2、C111与UV254、高锰酸盐指数、浊度、温度、BOD5呈正相关关系;第Ⅲ簇内的菌属丛毛单孢菌属 (Comamonas)、多核杆菌属 (Polynucleobacter)、ACK-M1、克雷伯氏菌属 (Klebsiella)、柄杆菌科 (Caulobacteraceae)、不动杆菌属 (Acinetobacter)、Cerasicoccaceae、放线菌目 (Actinomycetales)、丰收神菌属 (Opitutus) 与pH、TN、NO2--N、NO3--N、TP、NH4+-N呈显著正相关,另外第Ⅲ簇内的菌属丛毛单孢菌科 (Comamonadaceae)、丛毛单孢菌属 (Comamonas)、多核杆菌属 (Polynucleobacter)、ACK-M1还与BOD5呈正相关关系;第Ⅰ簇内的菌属菌保所杆菌属 (Emticicia)、鞘脂杆菌科 (Sphingobacteriaceae)、噬纤维菌科 (Cytophagaceae)、黄杆菌属 (Flavobacterium)、浮霉状菌属 (Planctomyces)、Saprospiraceae与pH、TN、NO2--N、NO3--N、TP、NH4+-N、BOD5、温度、浊度呈负相关.从微生物群落结构和环境因子的RDA分析图中还可以看出,第Ⅱ、Ⅲ簇内的菌属之间呈正相关关系,而第Ⅰ簇内的菌属与第Ⅱ、Ⅲ簇内的菌属之间呈负相关关系.
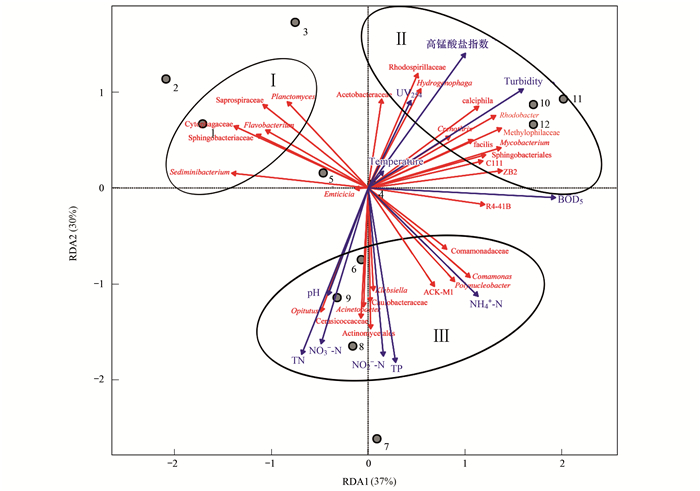
|
图 11 微生物群落结构和环境因子的RDA分析 Fig. 11 RDA analysis of environmental factors and microbial community structure |
本研究利用Illumina MiSeq高通量测序平台,对我国干旱半干旱典型集雨饮水地区的4组 (12个) 窖水样品进行测序. 4个窖水样品最终得到优质序列201 240条, 共产生1 605条OTU, 获得窖水细菌群落22门、42纲、71目、115科、146属.稀释曲线趋于平坦, 说明获取了绝大多数样本信息,基本能够反映研究区的微生物群落组成.不同样品的多样性指数 (Ace指数、Chao指数、Shannon、Simpson多样性指数) 的变化与OTUs具有相同的变化得到了相互印证.
对窖水4组水样细菌群落在门、纲、属分类层次上的优势菌群分布特征分析显示,细菌群落多样性模式呈现出,少数的常见菌群相对比例较高,为4组水样共同的绝对优势菌群,相对比例较低的细菌群落多样性各不相同,有显著的差别.窖水细菌菌群的这种多样性分布特点,印证了描述群落结构的生态学最经典的物种多度分布模式,即物种在地球上的分布并非是均匀的,几乎所有的群落,从海洋生物到亚马逊热带雨林,都呈现出大量的稀有种和少数常见种的模式,并且这种模式重复出现在各种生物类群中[28, 29].另外McGill等[29]认为微生物群落也应该符合大量稀有种和少数常见种的多度分布模式,并且会存在很长的稀有种“尾巴”.
3.2 窖水细菌群落生态功能及细菌类群组成影响因子水体中微生物是水域生态系统的重要组成部分,在水体生态系统中发挥着重要的生态作用,不同微生物群落具有其独特的生态功能, 构成了微生物功能多样性.分析窖水脱氮除磷菌的生态功能发现,有多种细菌属于好氧反硝化菌,比如Rhodobacter(红细菌属)、Dechloromonas(脱氮单孢菌属)、Flavobacterium(黄杆菌属) 等,这类细菌与传统的反硝化细菌相比具有可在有氧条件下生长,大多数菌种在较高的溶解氧浓度有反硝化活性及同时具有异养硝化及代谢难降解有机物的能力,生长周期短,生长速率远大于自养菌,环境适应性强等生态功能[30~34].值得深入研究的是,窖水中发现的异养硝化-好氧反硝化菌 (见表 3).近年来,异养硝化细菌和好氧反硝化细菌的发现打破了传统理论认为的硝化反应只能由自养细菌完成而反硝化反应只能在厌氧条件下进行的观点,异养硝化-好氧反硝化生物脱氮理论已成为国内外研究的热点.譬如,孙庆花等[12]从海底沉积物中分离出1株异养硝化-好氧反硝化菌,并鉴定为克雷伯氏菌属,经研究证明该菌具有高效的异养硝化和好氧反硝化作用.王兆阳等[35]从垃圾渗滤液活性污泥中筛选鉴定出1株异养硝化-好氧反硝化菌为假单孢菌属,经研究显示该菌具高效脱氮特性,可实现同步硝化反硝化.苏婉昀等[14]和乔森等[36]对异养硝化-好氧反硝化菌的脱氮特性进行了总结,与传统脱氨细菌相比,异养硝化-好氧反硝化菌在脱氮和去除有机物方面具有较大优势,碳源、碳氮比和DO是影响异养硝化-好氧反硝化菌脱氮的主要因素.窖水的溶解氧经长期监测,其浓度值比较稳定,在7.10 mg ·L-1左右,说明水窖水体属于好氧环境.因此,研究由异养硝化-好氧反硝化菌参与的受污染水源水体的自然修复有重要的意义.
不同的环境因子对微生物群落丰度具有不同的影响.如在美国的俄亥俄河水中无机氮与可溶性磷对微生物群落结构的影响较大[37];而在滇池水体中氨氮、总氮、硝态氮和叶绿素对浮游细菌群落的分布影响最大[38];贵阳南明河城区河段中,由于河水中氮磷含量的增高,导致乳酸杆菌属、Microvirgula、Kaistobacter、Gillisia、拟杆菌属和芽孢杆菌属细菌丰度明显增高,城区上游的南明河河段,由于河水中的氮磷量较低,导致微生物受到营养的限制,细菌多样性较低[39].从RDA分析中可以看出 (图 11),窖水不同环境因子对微生物群落也有着不同的影响,且这种影响有着明显的分簇, 即簇内菌属生态功能的相似性及簇间菌属生态功能的差异性.据水窖优势细菌类群生态功能分析,第Ⅱ簇内的嗜氢菌属 (Hydrogenophaga)、红细菌属 (Rhodobacter)、分枝杆菌属 (Mycobacterium) 除了有脱氮功能外,还对多种有机物具有良好的降解作用[40],而红螺菌科 (Rhodospirillaceae)、醋杆菌科 (Acetobacteraceae)、嗜甲基菌科 (Methylophilaceae) 属于变形菌门,国内外研究表明,大多数变形菌门的细菌都在脱氮、生物除磷及诸多有机物降解过程中起重要作用[41],也具有降解有机物的功能,这与第Ⅱ簇内的菌属主要与反映窖水有机物含量的指标之间呈正相关关系可得到验证.第Ⅲ簇内的菌属丛毛单孢菌属 (Comamonas)、克雷伯氏菌属 (Klebsiella)、不动杆菌属 (Acinetobacter) 是典型的具有脱氮功能的细菌,而放线菌目 (Actinomycetales) 下的ACK-M1与Mycobacterium(分枝杆菌属) 也具有脱氮功能,这与第Ⅲ簇内的菌属主要与反映窖水氮、磷水平的指标正相关得到验证.这说明,微生物群落的生态功能具有多样性和独特性的特征.
3.3 窖水人饮细菌类病原微生物风险评价如表 4所示为水窖中检出的9种潜在的水源性病原菌属分布特征.可以看出,这9种病原菌属分别隶属于变形菌门 (Proteobacteria)、拟杆菌门 (Bacteroidetes)、放线细菌门 (Actinobacteria),这三门同样是水窖微生物优势菌门.
|
|
表 4 窖水样品中潜在病原菌分布特征/% Table 4 Characteristics of potentially pathogenic microorganisms in the samples of cellar water/% |
进一步深入分析发现,分枝杆菌属 (Mycobacterium)、黄杆菌属 (Flavobacterium)、克雷伯氏菌属 (Klebsiella)、不动杆菌属 (Acinetobacter)、弓形菌属 (Arcobacter) 在窖水4组水样中均有检出,同时也是占有比例较高的属,就平均而言分别占0.90%、5.70%、0.90%、2.10%.而弯曲杆菌属 (Flectobacillus)、金黄杆菌属 (Chryseobacterium)、鞘氨醇单孢菌属 (Sphingomonas)、军团菌属 (Legionella) 仅在个别样品中检出,其丰度较低.此外,值得注意的是,约有56.8%的序列,不能划分到确定的属,其中可能包含对人畜有致病性的潜在病原菌.目前,地表水中化学污染物和致病微生物的污染日趋严重,其中致病微生物污染引起的水质卫生安全问题尤为突出,微生物污染是影响水质的主要原因之一[42].水环境受各种致病微生物污染可引起多种介水传染病,尤其是肠道传染病发生[43].根据WHO《饮用水水质准则》,不动杆菌属 (Acinetobacter) 尚没有关于人群因饮用含不动杆菌属细菌的饮用水而引起胃肠道感染的证据,但有可能通过饮用水引起易感个体的非胃肠道感染的传播;而弯曲杆菌属 (Flectobacillus) 是引起全球急性胃肠炎最重要的病因之一,污染的饮用水是弯曲菌病暴发的重要感染源,自患者及其饮用的饮用水中分离到同样的菌株,证实了该病的水源性传播;克雷伯氏菌属 (Klebsiella) 属于肠杆菌科,其中的产酸克雷伯菌也已被确认为致病菌.非肠源性的微生物,如军团菌属 (Legionella) 被认为是潜在的人类病菌,噬肺军团菌是军团菌病的主要水源性病原体,该菌有可能侵入饮用水系统,所以应采取控制措施减少该菌的存活及繁殖,而分枝杆菌属 (Mycobacterium) 主要的感染途径可能是吸入、接触以及饮用被污染的水,多种菌的感染都与该菌在饮用水中存在有关.虽然,可以通过检测指示菌对水体微生物污染状况及安全进行评估,但是越来越多的研究表明,指示微生物与病原微生物相关性不强[44, 45].由此可见,鉴于人饮安全考虑,进一步开展窖水细菌类病原微生物污染的人饮健康风险评估有很大的必要性.
4 结论(1) 采用16S rRNA基因Illumina MiSeq高通量测序技术对4组窖水样品进行测序.经统计分析得到用于物种分类的OTU 1 605条,获得窖水细菌群落22门、42纲、71目、115科、146属.稀释性曲线在0.97相似性水平下趋于平坦, 可基本反映窖水的微生物群落组成. Chao、ACE指数及OTU聚类结果显示4组样品的细菌群落丰度大小为D > B > C > A,Simpson、Simpson指数说明,4组水样细菌的多样性高低为D > C > B > A.
(2) 物种聚类结果显示,窖水中细菌的丰度总体较高,但不同类型的水窖存贮的窖水中的微生物多样性存在一定差异,即少数的常见菌群相对比例较高,如拟杆菌门 (Bacteroidetes)、变形菌门 (Proteobacteria)、放线细菌门 (Actinobacteria)、疣微菌门 (Verrucomicrobia)、OD1的微生物相对丰度比例约占87.1%~94.8%,为4组水样共同的绝对优势菌群,相对比例较低的细菌群落多样性各不相同,有显著的差别,这与OTU分布维恩图可得到印证.另外,窖水细菌聚类到黄杆菌属 (Flavobacterium) 等9种潜在的水源性病原菌属.
(3) 水窖中的优势菌属大多数为具有脱氮除磷功能的反硝化细菌和异养硝化-好氧反硝化菌.窖水不同环境因子对微生物群落的影响有着明显的分簇, 即第Ⅱ簇内的菌属主要与反映窖水有机物含量的指标UV254、高锰酸盐指数、BOD5之间呈显著正相关关系,而第Ⅲ簇内的菌属主要与反映窖水氮、磷水平的指标TN、NO2--N、NO3--N、TP、NH4+-N显著正相关,体现了生态功能的簇内相似性及簇间差异性.
| [1] | 武福平, 夏传, 王艳琴, 等. 西北典型村镇集雨窖水水质变化及特性[J]. 环境工程学报, 2014, 8(9): 3541–3545. Wu F P, Xia C, Wang Y Q, et al. Quality and its variation of water in rainwater collection cellar of rural areas of northwest China[J]. Chinese Journal of Environmental Engineering, 2014, 8(9): 3541–3545. |
| [2] | 王绍祥, 杨洲祥, 孙真, 等. 高通量测序技术在水环境微生物群落多样性中的应用[J]. 化学通报, 2014, 77(3): 196–203. Wang S X, Yang Z X, Sun Z, et al. Application of high throughput sequencing in the diversity of water microbial communities[J]. Chemistry, 2014, 77(3): 196–203. |
| [3] | 柴晓娟, 骆大伟, 吴春笃, 等. 水体中微生物分布及与环境因素的相关性研究[J]. 人民长江, 2008, 39(3): 45–47. Chai X J, Luo D W, Wu C D, et al. Research on relation of micro-organism distribution in water and environmental factor[J]. Yangtze River, 2008, 39(3): 45–47. |
| [4] | 任丽娟, 何聃, 邢鹏, 等. 湖泊水体细菌多样性及其生态功能研究进展[J]. 生物多样性, 2013, 21(4): 421–432. Ren L J, He D, Xing P, et al. Bacterial diversity and ecological function in lake water bodies[J]. Biodiversity Science, 2013, 21(4): 421–432. |
| [5] | Newton R J, Jones S E, Eiler A, et al. A guide to the natural history of freshwater lake bacteria[J]. Microbiology and Molecular Biology Reviews, 2011, 75(1): 14–49. DOI: 10.1128/MMBR.00028-10 |
| [6] | Zwart G, Crump B C, Kamst-van Agterveld M P, et al. Typical freshwater bacteria:an analysis of available 16S rRNA gene sequences from plankton of lakes and rivers[J]. Aquatic Microbial Ecology, 2002, 28(2): 141–155. |
| [7] | Taylor L H, Latham S M, Woolhouse M E J. Risk factors for human disease emergence[J]. Philosophical Transactions of the Royal Society B:Biological Sciences, 2001, 356(1411): 983–989. DOI: 10.1098/rstb.2001.0888 |
| [8] | 肖晶晶, 郭萍, 霍炜洁, 等. 反硝化微生物在污水脱氮中的研究及应用进展[J]. 环境科学与技术, 2009, 32(12): 97–102. Xiao J J, Guo P, Huo W J, et al. Application of denitrifying microbes to wastewater denitrification[J]. Environmental Science & Technology, 2009, 32(12): 97–102. |
| [9] | Krustok I, Truu J, Odlare M, et al. Effect of lake water on algal biomass and microbial community structure in municipal wastewater-based lab-scale photobioreactors[J]. Applied Microbiology and Biotechnology, 2015, 99(15): 6537–6549. |
| [10] | Fukami K, Yuzawa A, Nishijima T, et al. Isolation and properties of a bacterium inhibiting the growth of Gymnodinium nagasakiense[J]. Nippon Suisan Gakkaishi, 1992, 58(6): 1073–1077. DOI: 10.2331/suisan.58.1073 |
| [11] | 段亮, 夏四清, 宋永会, 等. 高密度微阵列基因芯片技术在微生物分子生态学研究中的运用[J]. 环境科学, 2009, 30(12): 3691–3697. Duan L, Xia S Q, Song Y H, et al. Using high-density universal 16S rRNA microarray in microbial molecular ecology research[J]. Environmental Science, 2009, 30(12): 3691–3697. |
| [12] | 孙庆花, 于德爽, 张培玉, 等. 1株海洋异养硝化-好氧反硝化菌的分离鉴定及其脱氮特性[J]. 环境科学, 2016, 37(2): 647–654. Sun Q H, Yu D S, Zhang P Y, et al. Identification and nitrogen removal characteristics of a heterotrophic nitrification-aerobic denitrification strain isolated from marine environment[J]. Environmental Science, 2016, 37(2): 647–654. |
| [13] | 李小义, 王丽萍, 杜雅萍, 等. 好氧反硝化微生物多样性及其反硝化功能初步研究[J]. 氨基酸和生物资源, 2016, 38(2): 37–45. Li X Y, Wang L P, Du Y P, et al. Diversity of aerobic denitrifying microbes from various environment and analysis of their denitrification ability[J]. Amino Acids & Biotic Resources, 2016, 38(2): 37–45. |
| [14] | 苏婉昀, 高俊发, 赵红梅. 异养硝化-好氧反硝化菌的研究进展[J]. 工业水处理, 2013, 33(12): 1–5. Su W Y, Gao J F, Zhao H M. Research progress in heterotrophic nitrification-aerobic denitrification bacteria[J]. Industrial Water Treatment, 2013, 33(12): 1–5. |
| [15] | 柯娜, 肖昌松, 应启锋, 等. 一个可降解直链烷基苯磺酸盐的新种[J]. 微生物学报, 2003, 43(1): 1–7. Ke N, Xiao C S, Ying Q F, et al. A new species of the genus phenylobacterium for the degradation of LAS (Linear Alkylbenzene Sulfonate)[J]. Acta Microbiologica Sinica, 2003, 43(1): 1–7. |
| [16] | 王硕, 徐巧, 张光生, 等. 完全混合式曝气系统运行特性及微生物群落结构解析[J]. 环境科学, 2017, 38(2): 665–671. Wang S, Xu Q, Zhang G S, et al. Operational performance and microbial community structure in a completely mixed aeration system[J]. Environmental Science, 2017, 38(2): 665–671. |
| [17] | 郑林雪, 李军, 胡家玮, 等. 同步硝化反硝化系统中反硝化细菌多样性研究[J]. 中国环境科学, 2015, 35(1): 116–121. Zheng L X, Li J, Hu J W, et al. Analysis of denitrifying bacteria community composition in simultaneous nitrification and denitrification systems[J]. China Environmental Science, 2015, 35(1): 116–121. |
| [18] | Vasiliadou I A, Pavlou S, Vayenas D V. Dynamics of a chemostat with three competitive hydrogen oxidizing denitrifying microbial populations and their efficiency for denitrification[J]. Ecological Modelling, 2009, 220(8): 1169–1180. DOI: 10.1016/j.ecolmodel.2009.02.009 |
| [19] | Nalcaci O O, Böke N, Ovez B. Potential of the bacterial strain Acidovorax avenae subsp. avenae LMG 17238 and macro algae Gracilaria verrucosa for denitrification[J]. Desalination, 2011, 274(1-3): 44–53. |
| [20] | Bae H S, Rash B A, Rainey F A, et al. Description of Azospira restricta sp. nov., a nitrogen-fixing bacterium isolated from groundwater[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 57(7): 1521–1526. |
| [21] | Calvó L, Vila X, Abella C A, et al. Use of the ammonia-oxidizing bacterial-specific phylogenetic probe Nso1225 as a primer for fingerprint analysis of ammonia-oxidizer communities[J]. Applied Microbiology and Biotechnology, 2004, 63(6): 715–721. DOI: 10.1007/s00253-003-1433-1 |
| [22] | Wang H B, Hu C, Hu X X, et al. Effects of disinfectant and biofilm on the corrosion of cast iron pipes in a reclaimed water distribution system[J]. Water Research, 2012, 46(4): 1070–1078. DOI: 10.1016/j.watres.2011.12.001 |
| [23] | 李哿.活性污泥-生物膜复合系统脱氮除磷试验研究[D].济南:山东建筑大学, 2011. Li G. An experimental study on nitrogen and phosphorous removal of the hybrid activated sludge and biofilm system[D]. Ji'nan:Shandong Jianzhu University, 2011. |
| [24] | 赵文莉, 郝瑞霞, 王润众, 等. 复合碳源填料反硝化脱氮及微生物群落特性[J]. 中国环境科学, 2015, 35(10): 3003–3009. Zhao W L, Hao R X, Wang R Z, et al. Denitrification of composite carbon filler and character of microbial community[J]. China Environmental Science, 2015, 35(10): 3003–3009. |
| [25] | Brakstad O G, Bonaunet K. Biodegradation of petroleum hydrocarbons in seawater at low temperatures (0-5℃) and bacterial communities associated with degradation[J]. Biodegradation, 2006, 17(1): 71–82. DOI: 10.1007/s10532-005-3342-8 |
| [26] | Van Hamme J D, Singh A, Ward O P. Recent advances in petroleum microbiology[J]. Microbiology and Molecular Biology Reviews, 2003, 67(4): 503–549. DOI: 10.1128/MMBR.67.4.503-549.2003 |
| [27] | 张雪梅, 佘跃惠, 黄金凤, 等. 大庆油田聚合物驱后油藏微生物多样性研究[J]. 应用与环境生物学报, 2008, 14(5): 668–672. Zhang X M, She Y H, Huang J F, et al. Microbial diversity of the Daqing oilfield after polymer flooding[J]. Chinese Journal of Applied and Environmental Biology, 2008, 14(5): 668–672. |
| [28] | Magurran A E. Measuring biological diversity[M]. Malden, MA: Blackwell, 2004. |
| [29] | McGill B J, Etienne R S, Gray J S, et al. Species abundance distributions:moving beyond single prediction theories to integration within an ecological framework[J]. Ecology Letters, 2007, 10(10): 995–1015. DOI: 10.1111/ele.2007.10.issue-10 |
| [30] | Zhou Q, Takenaka S, Murakami S, et al. Screening and characterization of bacteria that can utilize ammonium and nitrate ions simultaneously under controlled cultural conditions[J]. Journal of Bioscience and Bioengineering, 2007, 103(2): 185–191. DOI: 10.1263/jbb.103.185 |
| [31] | Kim M, Jeong S Y, Yoon S J, et al. Aerobic denitrification of Pseudomonas putida AD-21 at different C/N ratios[J]. Journal of Bioscience and Bioengineering, 2008, 106(5): 498–502. DOI: 10.1263/jbb.106.498 |
| [32] | Zhao B, He Y L, Hughes J, et al. Heterotrophic nitrogen removal by a newly isolated Acinetobacter calcoaceticus HNR[J]. Bioresource Technology, 2010, 101(14): 5194–5200. |
| [33] | Kim J K, Park K J, Cho K S, et al. Aerobic nitrification-denitrification by heterotrophic Bacillus strains[J]. Bioresource Technology, 2005, 96(17): 1897–1906. |
| [34] | Joo H S, Hirai M, Shoda M. Characteristics of ammonium removal by heterotrophic nitrification-aerobic denitrification by Alcaligenes faecalis No. 4[J]. Journal of Bioscience and Bioengineering, 2005, 100(2): 184–191. |
| [35] | 王兆阳, 陈国耀, 姜珂, 等. 1株耐冷兼性嗜碱好氧反硝化菌的分离鉴定及反硝化特性[J]. 环境科学, 2014, 35(6): 2341–2348. Wang Z Y, Chen G Y, Jiang K, et al. Identification and denitrification characteristics of a psychrotolerant facultative basophilic aerobic denitrifier[J]. Environmental Science, 2014, 35(6): 2341–2348. |
| [36] | 乔森, 刘雪洁, 周集体. 异养硝化-好氧反硝化在生物脱氮方面的研究进展[J]. 安全与环境学报, 2014, 14(2): 128–135. Qiao S, Liu X J, Zhou J T. Research progress of heterotrophic nitrification-aerobic denitrification in biological denitrification[J]. Journal of Safety and Environment, 2014, 14(2): 128–135. |
| [37] | Rubin M A, Leff L G. Nutrients and other abiotic factors affecting bacterial communities in an Ohio River (USA)[J]. Microbial Ecology, 2007, 54(2): 374–383. DOI: 10.1007/s00248-007-9209-2 |
| [38] | Kent A D, Smith D J, Benson B J, et al. Web-based phylogenetic assignment tool for analysis of terminal restriction fragment length polymorphism profiles of microbial communities[J]. Applied and Environmental Microbiology, 2003, 69(11): 6768–6776. |
| [39] | 唐婧, 徐小蓉, 商传禹, 等. 南明河城区河段细菌多样性与环境因子的关系[J]. 微生物学报, 2015, 55(8): 1050–1059. Tang J, Xu X R, Shang C Y, et al. Association of bacterial diversity in city area of Nanming river with environmental factors[J]. Acta Microbiologica Sinica, 2015, 55(8): 1050–1059. |
| [40] | 王丽华, 吕铮, 郝春博, 等. 某石油污染场地地下水中降解菌群落结构研究[J]. 环境科学与技术, 2013, 36(7): 1–8, 146. Wang L H, Lv Z, Hao C B, et al. Degrading bacteria community structure in groundwater of a petroleum-contaminated site[J]. Environmental Science & Technology, 2013, 36(7): 1–8, 146. |
| [41] | Nguyen H T T, Le V Q, Hansen A A, et al. High diversity and abundance of putative polyphosphate-accumulating Tetrasphaera-related bacteria in activated sludge systems[J]. FEMS Microbiology Ecology, 2011, 76(2): 256–267. DOI: 10.1111/fem.2011.76.issue-2 |
| [42] | Cole D, Long S C, Sobsey M D. Evaluation of F+RNA and DNA coliphages as source-specific indicators of fecal contamination in surface waters[J]. Applied and Environmental Microbiology, 2003, 69(11): 6507–6514. |
| [43] | 张振兴, 王江权, 郑祥. 水体病原微生物定量风险评价:历史、现状与发展趋势[J]. 环境科学学报, 2016, 36(1): 2–15. Zhang Z X, Wang J X, Zheng X. Quantitative microbial risk assessment of waterborne pathogens:history and progress[J]. Acta Scientiae Circumstantiae, 2016, 36(1): 2–15. |
| [44] | 孙启明, 郑仁淑, 李援, 等. 东北地区某流域河流污染指示菌调查[J]. 环境与健康杂志, 2011, 28(5): 409–411. Sun Q M, Zheng R S, Li Y, et al. Investigation of pollution Indicator bacteria in rivers of northeast China[J]. Journal of Environment and Health, 2011, 28(5): 409–411. |
| [45] | 谌志强, 王新为, 金敏, 等. 珠江水域微生物的分布特征[J]. 环境与健康杂志, 2012, 29(10): 913–916. Shen Z Q, Wang X W, Jin M, et al. Investigation and analysis on microbial indicators in Pearl River[J]. Journal of Environment and Health, 2012, 29(10): 913–916. |
 2017, Vol. 38
2017, Vol. 38

