2. 华南理工大学工业聚集区污染控制与生态修复教育部重点实验室, 广州 510006;
3. 华南理工大学广东省大气环境与污染控制重点实验室, 广州 510006
2. The Key Laboratory of Pollution Control and Ecosystem Restoration in Industry Clusters, Ministry of Education, South China University of Technology, Guangzhou 510006, China;
3. Guangdong Provincial Key Laboratory of Atmospheric Environment and Pollution Control, South China University of Technology, Guangzhou 510006, China
20世纪90年代,荷兰的Mulder等[1]在处理工业废水的反硝化流化床中发现了一种新型细菌——厌氧氨氧化细菌 (anaerobic ammonia oxidation bacteria,AnAOB).该类细菌可以在缺氧条件下以亚硝酸盐为电子受体将氨氮转化为氮气. 1999年,Strous等[2]采用密度梯度离心法成功分离了AnAOB菌株,并通过16S rRNA基因测序,确认了AnAOB是浮霉状菌的成员.
与传统的硝化反硝化工艺相比,厌氧氨氧化 (anaerobic ammonia oxidation,ANAMMOX) 工艺具有无需外加碳源和供氧动力消耗,反应过程中CO2排放量少和剩余污泥产量少等优点[3].目前该工艺已经用于高氨氮工业废水的处理中,如污泥消化液、垃圾渗滤液和养猪场废水等[4~6].
厌氧氨氧化工艺虽然有诸多优点,但AnAOB生长缓慢,倍增时间为7~22 d,导致ANAMMOX反应器启动时间长[7],是限制厌氧氨氧化工艺大规模应用的最主要因素[8].因此,选择合适的反应器,减少AnAOB的流失,是成功应用厌氧氨氧化工艺的关键[9].目前,用于厌氧氨氧化技术的反应器有上流式污泥床反应器 (USB)[10]、批序式间歇反应器 (SBR)[11]和厌氧折流板反应器 (ABR)[12]等.本实验选择上流式厌氧过滤床反应器 (upflow blanket filter, UBF),UBF反应器是由 (上流式污泥床) USB和厌氧过滤器 (AF) 构成的复合式反应器,具有良好的生物截留能力和易形成颗粒污泥等特点.
AnAOB至今仍未获得人工的纯培养,而高通量测序技术的发展为鉴定环境中的微生物群落的多样性提供了有力的工具[13~15].本研究采用Illumina Hisep PE250测序平台对UBF厌氧氨氧化反应器内的微生物进行高通量测序,分析了ANAMMOX反应器内微生物的分布情况,以期为ANAMMOX反应器内微生物群落相对丰度及群落结构变化提供理论依据.
1 材料与方法 1.1 实验装置反应器为UBF厌氧氨氧化反应器,由有机玻璃制成,呈圆柱形,总容积为3.94 L,有效容积为2.1 L,直径为8 cm,总高度为66 cm,内置20 cm高的填料层,外裹黑布,避免光对厌氧氨氧化菌的抑制.废水通过蠕动泵从反应器的底部连续泵入,与污泥混合后,反应生成的气体从反应器顶部的排气孔排出,出水由反应器上部的溢流堰流出.进水pH控制在7.1~7.8,在常温条件下启动.实验装置如图 1所示.
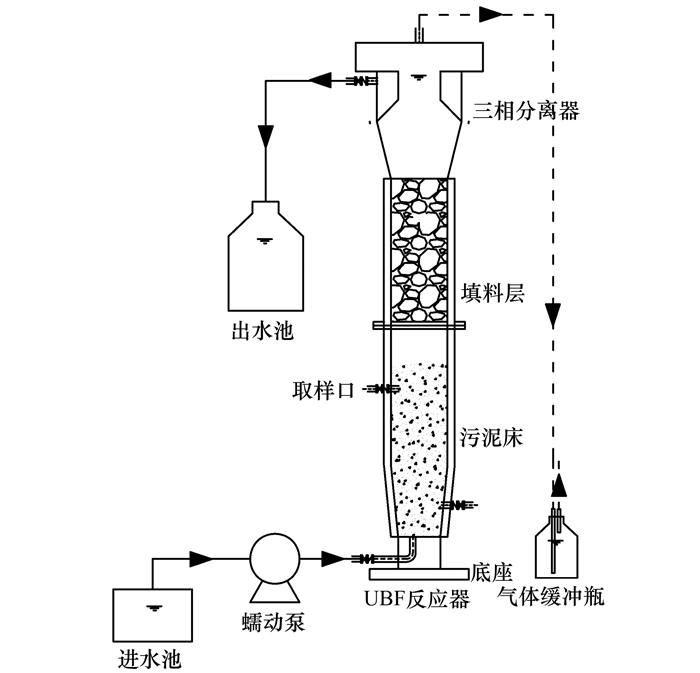
|
图 1 实验装置示意 Fig. 1 Schematic diagram of experimental equipment |
反应器接种污泥来自广州市沥滘污水处理厂缺氧池,接种量为800 mL.
实验采用模拟废水,主要成分为NH4Cl和NaNO2,其浓度按实验需要添加.其它成分为:NaHCO3 1 000 mg ·L-1,MgSO4 ·7H2O 473 mg ·L-1,CaCl2 ·2H2O 180 mg ·L-1,KH2PO4 27 mg ·L-1,微量元素Ⅰ1 mg ·L-1和微量元素Ⅱ 1 mg ·L-1.其中微量元素Ⅰ为:EDTA 5 000 mg ·L-1和FeSO4 ·7H2O 5 000 mg ·L-1.微量元素Ⅱ为:ZnSO4 ·7H2O 430 mg ·L-1,GuSO5 ·5H2O 240 mg ·L-1,MnCl2 ·4H2O 990 mg ·L-1,NiCl2 ·6H2O 190 mg ·L-1,CoCl2·6H2O 24 mg ·L-1.进水pH值用盐酸调节为7.1~7.8.
1.3 水样检测方法氨氮:纳氏试剂分光光度法;亚硝氮:N-(1-萘基)-乙二胺光度法;硝氮:紫外分光光度法[16];总氮为氨氮、亚硝氮和硝氮之和;pH值采用pH计测定.
1.4 微生物群落多样性检测方法 1.4.1 DNA提取反应器运行至第54 d时取下层污泥20 mL (标记为CYR110,文中表示富集阶段),此时污泥高度为19.6 cm.第83 d时分别取上层污泥 (标记为CYR111) 和下层污泥 (标记为CYR112,文中表示稳定运行阶段) 各20 mL,此时污泥高度为24.4 cm.下层污泥的取样口距离底部4.4 cm,上层污泥的取样口距离底部20.7 cm.采用Powersoil土壤DNA提取试剂盒提取基因组DNA.提取的DNA使用琼脂糖凝胶电泳检测DNA纯度,使用Nanodrop检测DNA浓度.
1.4.2 PCR扩增采用16S rRNA V4区通用引物,引物序列为515F 5′-GTGCCAGCMGCCGCGGTAA-3′和806R 5′-GGACTACHVGGGTWTCTAAT-3′,扩增片段大小为300bp左右.采用PCR仪对细菌16S rRNA基因进行PCR扩增,扩增反应体系 (60 μL) 为10x Ex Taq Buffer 6 μL,dNTP 6 μL,BSA 0.6 μL,Ex Taq 0.3 μL,Primer F 1.2 μL,Primer R 1.2 μL,DNA 1 μL,ddH2O 43.7 μL.按照PCR反应条件 (94℃ 5 min,94℃ 30 s,52℃ 30 s,72℃ 45 s,72℃ 10 min) 进行,31 Cycles PCR完成后利用琼脂糖凝胶电泳检测扩增产物.
1.4.3 Illumina高通量测序采用Illumina Hisep PE250测序平台对采集的样品进行高通量测序.
1.4.4 数据处理分析方法采用Origin 7.5和AutoCAD 2007软件绘制图,并采用SPSS 19.0(IBM Inc,USA) 软件对数据进行分析.
2 结果与讨论 2.1 反应器的脱氮性能反应器运行至第54d时,保持HRT为8h不变,以提高进水NH4+-N和NO2--N浓度的方式增大反应器的容积负荷,此时NH4+-N、NO2--N和TN的去除率分别为76.18%、53.47%和55.66%,总氮容积负荷和去除负荷分别为0.56 kg ·(m3 ·d)-1和0.29 kg ·(m3 ·d)-1.当反应器运行至第83 d时,NH4+-N、NO2--N和TN的去除率分别为99.4%、93.1%和82.8%,总氮容积负荷和去除负荷分别为1.25 kg ·(m3 ·d)-1和1.04 kg ·(m3 ·d)-1,说明反应器启动成功.由图 2可以看出,NH4+-N、NO2--N浓度和NLR呈梯度升高.当反应器的总氮容积负荷由0.52 kg ·(m3 ·d)-1增大至2.75 kg ·(m3 ·d)-1时,TN的去除率由55.66%增大至82.96%.蒋军等[17]研究表明,当反应器的容积负荷由0.21 kg ·(m3 ·d)-1增大至0.84 kg ·(m3 ·d)-1时,TN的去除率由67.98%减小至50.4%,与本研究结果有较大差异,可能与实验装置和反应条件有关.实验的前40多天,出水中的NH4+-N和NO2--N浓度较稳定,分别在0~30 mg ·L-1和4~53 mg ·L-1之间波动.当进水NH4+-N和NO2--N浓度分别增大至240 mg ·L-1和300 mg ·L-1时,出水中的NH4+-N和NO2--N浓度有所增大,但增大幅度较小,通过降低进水的pH值,出水中的NH4+-N和NO2--N浓度经过1 d分别降低至0~10 mg ·L-1和15~30 mg ·L-1之间.随着进水NH4+-N和NO2--N浓度的继续增大,当进水NH4+-N和NO2--N浓度分别达到330 mg ·L-1和430mg ·L-1时,反应器出水水质突然恶化,出水的NH4+-N和NO2--N浓度分别增大至89 mg ·L-1和163 mg ·L-1.此时NH4+-N和NO2--N的去除率分别为73.0%和62.1%,此时厌氧氨氧化菌的活性受到抑制. Dapena-Mora等[18]研究发现当NO2--N浓度达到350mg ·L-1时,厌氧氨氧化菌的活性会降低50%.在不同研究中,NO2--N对厌氧氨氧化菌的抑制浓度是不同,这可能与菌种及操作条件有关.为了较快地恢复反应器的脱氮性能,将进水NH4+-N和NO2--N浓度分别降低至310 mg ·L-1和400 mg ·L-1.经过7 d的恢复期,出水中的NH4+-N和NO2--N浓度分别降低至16 mg ·L-1和56 mg ·L-1,NH4+-N和NO2--N的去除率分别恢复至95.0%和86.6%.研究表明,NO2--N毒性显著强于NH4+-N,在进水NO2--N浓度较高或进水NO2--N/NH4+-N相对较高的条件下,反应器性能易因基质抑制而失稳[19, 20].
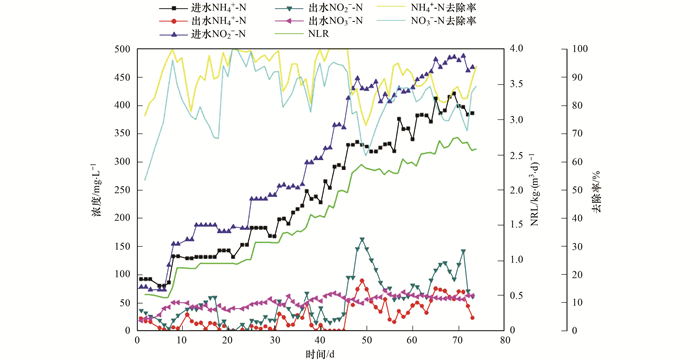
|
图 2 ANAMMOX反应器的脱氮性能 Fig. 2 Nitrogen removal performance of ANAMMOX reactor |
由Observed species和Chao1指数可知 (见表 1),富集阶段反应器内的微生物群落相对丰度要高于稳定运行阶段,而在稳定运行阶段,反应器内的上层微生物群落相对丰度要高于下层,说明反应器内的微生物经过一段时间的驯化后,部分微生物适应不了无机厌氧环境而逐渐死亡,造成微生物的丰度降低.由PD whole tree、Shannon和Simpson指数可知 (见表 1),富集阶段反应器内的微生物群落多样性要高于稳定运行阶段,而稳定运行阶段反应器内的上层微生物群落多样性要高于下层,说明反应器内的AnAOB经过一段时间的驯化后形成了优势种群,导致微生物群落多样性的减少.由于废水是从反应器的底部泵入,因此反应器下层的AnAOB最先被驯化成优势种群,因而下层的微生物群落多样性要低于上层.
|
|
表 1 生物多样性分析 Table 1 Biodiversity analysis |
2.2.2 门水平物种相对丰度分析
由图 3可知,所测得的相对丰度大于等于1%的门隶属于14个门.其中,富集阶段反应器中的微生物主要以变形菌门Proteobacteria、拟杆菌门Bacteroidetes、绿弯菌门Chloroflexi、泉古菌门Crenarchaeota和广古菌门Euryarchaeota为主,稳定运行阶段反应器中的微生物主要以变形菌门Proteobacteria、拟杆菌门Bacteroidetes、绿弯菌门Chloroflexi和浮霉菌门Planctomycetes为主,反应器中的浮霉菌门和变形菌门有了明显的增加,分别由1.1%、27.9%增加到了26.4%、39.9%.而拟杆菌门、绿弯菌门、泉古菌门和广古菌门都明显减少.反应器上下层物种相对丰度有明显的差异.底部的浮霉菌门明显比上层多,而上层的拟杆菌门要比底部多.
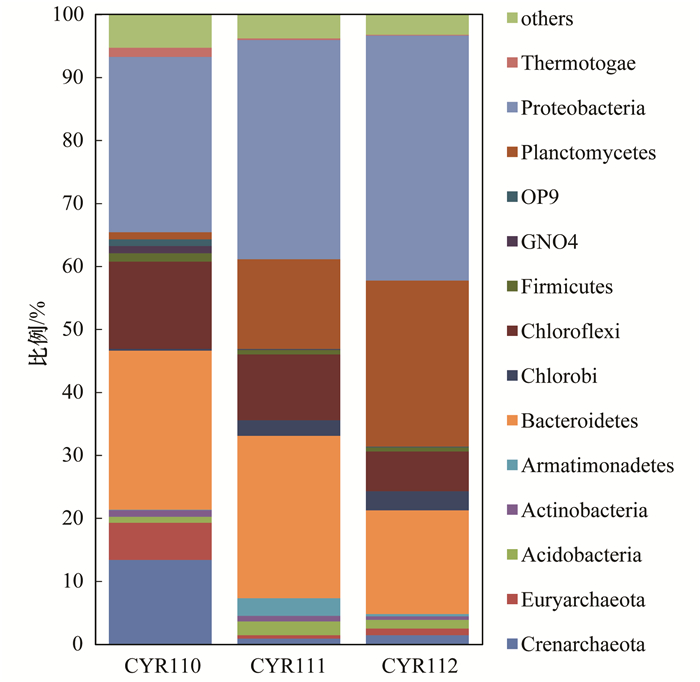
|
图 3 门水平物种相对丰度分布 Fig. 3 Relative abundance distribution at phylum level |
由图 4可知,所测得的相对丰度大于等于1%的科隶属于21个科,其中,富集阶段反应器中的微生物主要以厌氧蝇菌科Anaerolinaceae、红环菌科Rhodocyclaceae、丛毛单孢菌科Comamonadaceae为主.稳定运行阶段反应器中的上层污泥主要以厌氧蝇菌科Anaerolinaceae、红环菌科Rhodocyclaceae、丛毛单孢菌科Comamonadaceae和Brocadiaceae为主,下层污泥主要以厌氧蝇菌科Anaerolinaceae、红环菌科Rhodocyclaceae、丛毛单孢菌科Comamonadaceae、黄单胞菌科Xanthomonadaceae和Brocadiaceae为主.反应器中的厌氧蝇菌科Anaerolinaceae、甲烷丝状菌科Methanosaetaceae、WSA2、腐螺旋菌科Saprospiraceae、热孢菌科Thermotogaceae和互营菌科Syntrophaceae有明显的减少,其中WSA2、腐螺旋菌科Saprospiraceae、热孢菌科Thermotogaceae和互营菌科Syntrophaceae在稳定运行阶段几乎看不到.而红环菌科Rhodocyclaceae、丛毛单孢菌科Comamonadaceae、黄单胞菌科Xanthomonadaceae和Brocadiaceae有了明显的增加,其中Brocadiaceae增加最为显著,由0.074 6%增加到了24.572 7%.反应器上层Brocadiaceae占12.486 9%,下层占24.572 7%,下层Brocadiaceae的丰度明显比上层高. Brocadiaceae属于浮霉菌门下的一个科,已出现的厌氧氨氧化菌主要来自于这个科[21].由此可知,厌氧氨氧化反应器经过一段时间的运行后,反应器中的厌氧氨氧化微生物明显增加了.而反应器下层的厌氧氨氧化微生物明显比上层高.
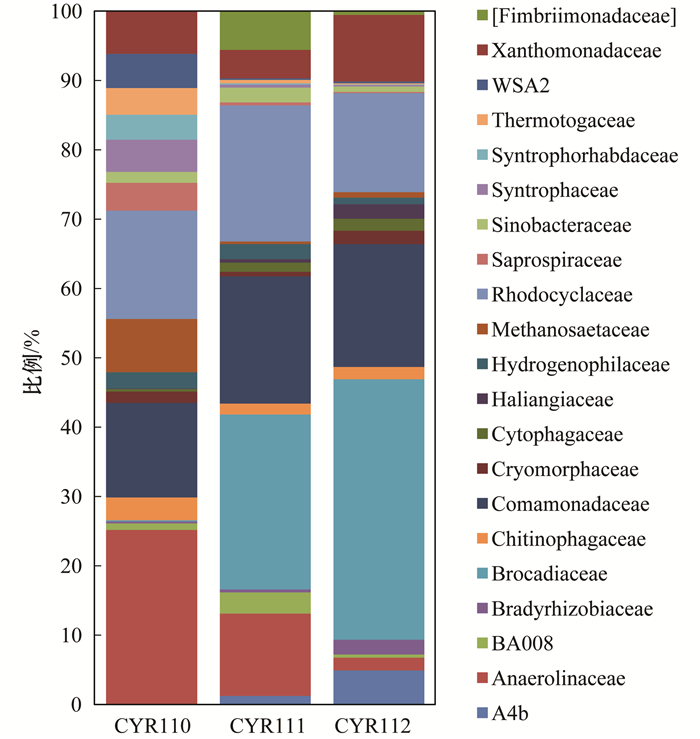
|
图 4 科水平物种相对丰度分布 Fig. 4 Relative abundance distribution at family level |
由图 5可知,稳定运行阶段的下层污泥中的Brocadiaceae、噬纤维菌科Cytophagaceae、丛毛单胞菌科Comamonadaceae、SB1、黄单胞菌科Xanthomonadaceae、慢生根瘤菌科Bradyrhizobiaceae、A4b、Haliangiaceae、Cryomorphaceae、生丝微菌科Hyphomicrobiaceae群落相对丰度要比上层高,其中,上层污泥中的Brocadiaceae、噬纤维菌科Cytophagaceae、丛毛单胞菌科Comamonadaceae、SB1群落相对丰度要比富集阶段高.相反,富集阶段反应器中的热袍菌科Thermotogaceae、互营杆菌科Syntrophobacteraceae、Syntrophorhabdaceae、互营菌科Syntrophaceae、腐螺旋菌科Saprospiraceae、WSA2、甲烷丝状菌科Methanosaetaceae、甲烷杆菌科Methanobacteriaceae、厌氧蝇菌科Anaerolinaceae、暖蝇菌科Caldilineaceae、肉杆菌科Carnobacteriaceae群落相对丰度要比稳定运行阶段高.总体来看,富集阶段反应器内的微生物群落结构与稳定运行阶段相比相差很大,而稳定运行阶段反应器上下层的微生物群落结构也有较大的差异. Chen等[12]研究表明ABR厌氧氨氧化反应器各隔室的微生物群落结构存在较大的差异, 与本研究结果相符.
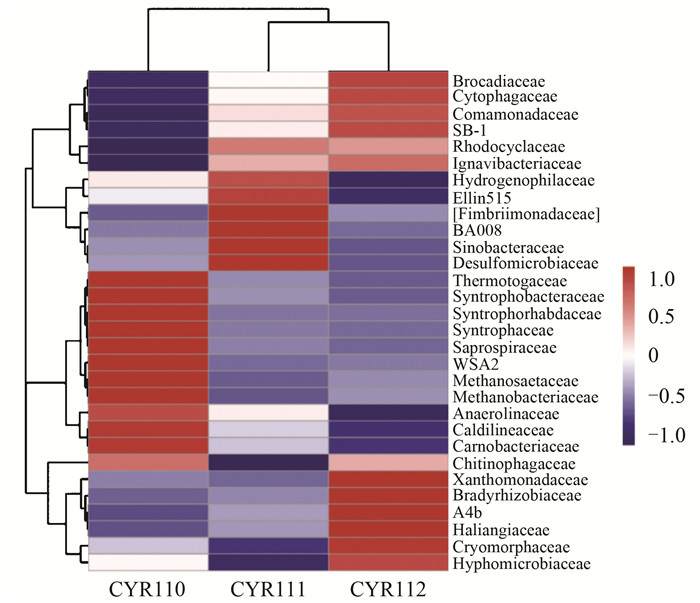
|
图 5 物种相对丰度聚类 (科水平) Fig. 5 Clustering of relative abundance |
通过高通量的扩增子测序,共鉴定出门的细菌40种.其中,涉及到脱氮细菌的有变形菌门、浮霉菌门和硝化螺旋菌门3种,变形菌门丰度最高,与多数厌氧氨氧化反应器微生物群落分布情况相一致[9, 22, 23].
在变形菌门中,富集阶段的样品中共鉴定出属的细菌67种,占据比例较高的是陶厄氏菌属Thauera(1.532%)、Dok59(1.318%)、Thibacillus硫杆菌属 (0.888%) 和互营杆菌属Syntrophobacter(0.650%).稳定运行阶段的上层污泥样品中共鉴定出属的细菌56种,占据比例较高的是Dok59属 (4.644%)、Thauera属 (2.560%)、Thibacillus属 (1.074%) 和脱硫微菌属Desulfomicrobium(0.923%),稳定运行阶段的下层污泥样品中共鉴定出属的细菌54种,占据比例较高的是Dok59属 (6.014%)、Thauera属 (1.447%)、Thibacillus属 (0.638%) 和Thermomonas属 (0.503%).变形菌门中属的细菌多样减小. Thauera属、Dok59属和Thibacillus属丰度最高,其中Thauera属和Thibacillus属是β-变形菌纲中的反硝化细菌.此外,富集阶段的样品中只检测出Nitrosovibrio这一种氨氧化细菌 (AOB),仅占0.011%,属于β-变形菌亚纲. AOB属于能够在有氧条件下将氨氧化为亚硝酸盐的化能无机自养型细菌.本实验装置没有专门的除氧设备,因此会伴随有少量的氧气进入反应器中为AOB提供有氧环境.由图 6可以看出,ΔNO2--N/ΔNH4+-N值在最初的10 d由0.60增大至1.31,然后在1.05~1.41之间波动. ΔNO3--N/ΔNH4+-N值在最初的10 d由0.22增大至0.44,然后逐渐减小至0.15.说明富集阶段部分NH4+-N被AOB转化为亚硝酸盐.而稳定运行阶段的ΔNO2--N/ΔNH4+-N值较接近理论值1.32[24],说明此时NH4+-N几乎全部由AnAOB所利用.

|
图 6 3种氮素的比值变化 Fig. 6 Ratio variation of three forms of nitrogen |
硝化螺旋菌门是测序得到的三大脱氮细菌门中所占比例最小的,在0.035%~0.188%之间,富集阶段的含量最高 (0.188%).推测富集阶段的AOB将部分氨转化为亚硝酸盐,导致剩余亚硝酸盐的积累,而硝化螺旋菌门是亚硝化反应的主要微生物,因此含量高于稳定运行阶段.在硝化螺旋菌门中,共鉴定出2种属的细菌,分别为GOUTA19和Nitrospira,其中,Nitrospira为亚硝盐氧化细菌 (NOB),所占比例在0.023%~0.174%之间,富集阶段的含量最高 (0.174%).推测富集阶段的AOB将部分氨转化为亚硝酸盐,导致剩余亚硝酸盐的积累,而NOB可将亚硝酸盐氧化为硝酸盐,因此含量高于稳定运行阶段.
在浮霉菌门中共鉴定出属的细菌5种,分别为Gemmatia、A17、Pirellula、Planctomyces、Candidatus brocadia,其中,Candidatus brocadia所占的比例最高,在0.011%~1%之间.稳定运行阶段Candidatus brocadia所占的比例 (1%) 高于富集阶段 (0.011%),与稳定运行阶段的去除负荷1.04kg ·(m3 ·d)-1高于富集阶段0.29 kg ·(m3 ·d)-1相一致. Candidatus brocadia属是第一个被富集鉴定的AnAOB[25],属于Brocadiaceae科.而属于Brocadiaceae科的96%序列通过BLAST进行序列比对发现与多条序列同源性相似,因此无法确定是哪种属.推测未分类菌属中可能含有未鉴定出的AnAOB新种.
3 结论(1) 采用提高进水NH4+-N和NO2--N浓度的方式将ANAMMOX反应器的容积负荷由0.52 kg ·(m3 ·d)-1增大至2.75 kg ·(m3 ·d)-1,NH4+-N、NO2--N和TN的去除率分别从76.18%、53.47%、55.66%增大至94.04%、86.97%、82.96%,AnAOB成功富集.
(2) 由Alpha多样性指数分析可知,反应器富集阶段的微生物群落相对丰度和群落多样性要高于稳定运行阶段,而在稳定运行阶段,反应器内的上层微生物群落相对丰度和群落多样性要高于下层.
(3) 由门水平物种相对丰度分析可知,反应器中的浮霉菌门和变形菌门有明显的增加,其中,浮霉菌门的丰度增大最为显著.反应器底部的浮霉菌门明显比上层多,而上层的拟杆菌门要比底部多.由科水平物种相对丰度分析可知,反应器富集阶段以厌氧蝇菌科的相对丰度最高,为9.47%.稳定运行阶段Brocadiaceae的相对丰度最高,达到了24.57%,成为优势菌群.
(4) 由物种相对丰度热图分析可知,富集阶段的微生物群落结构与稳定运行阶段相比相差很大,而稳定运行阶段反应器上下层微生物群落结构也有较大的差异.
(5) 由测序结果可知,稳定运行阶段UBF厌氧氨氧化反应器中的脱氮细菌较为丰富,其中变形菌门、浮霉菌门和硝化螺旋菌门分别占39.9%、26.4%和0.188%.变形菌门中占据比例较高的是Dok59属、Thauera属、Thibacillus属和Thermomonas属.浮霉菌门主要包含Candidatus brocadia.
| [1] | Mulder A, van de Graaf A A, Robertson L A, et al. Anaerobic ammonium oxidation discovered in a denitrifying fluidized bed reactor[J]. FEMS Microbiology Ecology, 1995, 16(3): 177–184. DOI: 10.1111/fem.1995.16.issue-3 |
| [2] | Strous M, Fuerst J A, Kramer E H M, et al. Missing lithotroph identified as new planctomycete[J]. Nature, 1999, 400(6743): 446–449. DOI: 10.1038/22749 |
| [3] | Lotti T, van der Star W R L, Kleerebezem R, et al. The effect of nitrite inhibition on the Anammox process[J]. Water Research, 2012, 46(8): 2559–2569. DOI: 10.1016/j.watres.2012.02.011 |
| [4] | Liang Z, Liu J X. Landfill leachate treatment with a novel process:anaerobic ammonium oxidation (Anammox) combined with soil infiltration system[J]. Journal of Hazardous Materials, 2008, 151(1): 202–212. DOI: 10.1016/j.jhazmat.2007.05.068 |
| [5] | van Dongen U, Jetten M S M, Van Loosdrecht M C M. The sharon-anammox process for treatment of ammonium rich wastewater[J]. Water Science and Technology, 2001, 44(1): 153–160. |
| [6] | 王欢, 李旭东, 曾抗美. 猪场废水厌氧氨氧化脱氮的短程硝化反硝化预处理研究[J]. 环境科学, 2009, 30(1): 114–119. Wang H, Li X D, Zeng K M. Nitritation-denitrification process as a pretreatment of ANAMMOX to remove nitrogen from piggery wastewater[J]. Environmental Science, 2009, 30(1): 114–119. |
| [7] | Kartal B, de Almeida N M, Maalcke W J, et al. How to make a living from anaerobic ammonium oxidation[J]. FEMS Microbiology Reviews, 2013, 37(3): 428–461. DOI: 10.1111/1574-6976.12014 |
| [8] | 丁爽, 郑平, 唐崇俭, 等. 三种接种物启动Anammox-EGSB反应器的性能[J]. 生物工程学报, 2011, 27(4): 629–636. Ding S, Zheng P, Tang C J, et al. Performances of Anammox-EGSB bioreactors started up with three different seeding sludges[J]. Chinese Journal of Biotechnology, 2011, 27(4): 629–636. |
| [9] | 陈重军, 张海芹, 汪瑶琪, 等. 基于高通量测序的ABR厌氧氨氧化反应器各隔室细菌群落特征分析[J]. 环境科学, 2016, 37(7): 2652–2658. Chen C J, Zhang H Q, Wang Y Q, et al. Characteristics of microbial community in each compartment of ABR ANAMMOX reactor based on high-throughput sequencing[J]. Environmental Science, 2016, 37(7): 2652–2658. |
| [10] | 秦玉洁, 周少奇, 朱明石. 厌氧氨氧化反应器微生态的研究[J]. 环境科学, 2008, 29(6): 1638–1643. Qin Y J, Zhou S Q, Zhu M S, et al. Microecology of the anaerobic ammonium oxidation reactor[J]. Environmental Science, 2008, 29(6): 1638–1643. |
| [11] | De Lille M V, Berkhout V, Fröba L, et al. Ammonium estimation in an ANAMMOX SBR treating anaerobically digested domestic wastewater[J]. Chemical Engineering Science, 2015, 130: 109–119. DOI: 10.1016/j.ces.2015.03.018 |
| [12] | Chen C J, Sun F Q, Zhang H Q, et al. Evaluation of COD effect on anammox process and microbial communities in the anaerobic baffled reactor (ABR)[J]. Bioresource Technology, 2016, 216: 571–578. DOI: 10.1016/j.biortech.2016.05.115 |
| [13] | 唐霁旭, 王志伟, 马金星, 等. 454高通量焦磷酸测序法鉴定膜生物反应器膜污染优势菌种[J]. 微生物学通报, 2014, 41(2): 391–398. Tang J X, Wang Z W, Ma J X, et al. Identifying predominant strains causing membrane biofouling by using 454 high-throughput pyrosequencing[J]. Microbiology China, 2014, 41(2): 391–398. |
| [14] | Isanta E, Bezerra T, Fernández I, et al. Microbial community shifts on an anammox reactor after a temperature shock using 454-pyrosequencing analysis[J]. Bioresource Technology, 2015, 181: 207–213. DOI: 10.1016/j.biortech.2015.01.064 |
| [15] | Cao S B, Du R, Li B K, et al. High-throughput profiling of microbial community structures in an ANAMMOX-UASB reactor treating high-strength wastewater[J]. Applied Microbiology and Biotechnology, 2016, 100(14): 6457–6467. DOI: 10.1007/s00253-016-7427-6 |
| [16] | 国家环境保护总局. 水和废水监测分析方法[M]. (第四版). 北京: 中国环境科学出版社, 2002. |
| [17] | 蒋军, 吴立波, 付丽霞, 等. 容积负荷对厌氧氨氧化反应器运行影响的研究[J]. 环境工程学报, 2010, 4(2): 283–287. Jiang J, Wu L B, Fu L X, et al. Study on influence of volume loading on running of ANAMMOX reactor[J]. Chinese Journal of Environmental Engineering, 2010, 4(2): 283–287. |
| [18] | Dapena-Mora A, Fernández I, Campos J L, et al. Evaluation of activity and inhibition effects on Anammox process by batch tests based on the nitrogen gas production[J]. Enzyme and Microbial Technology, 2007, 40(4): 859–865. DOI: 10.1016/j.enzmictec.2006.06.018 |
| [19] | Jin R C, Yang G F, Yu J J, et al. The inhibition of the Anammox process:a review[J]. Chemical Engineering Journal, 2012, 197: 67–79. DOI: 10.1016/j.cej.2012.05.014 |
| [20] | Kimura Y, Isaka K, Kazama F, et al. Effects of nitrite inhibition on anaerobic ammonium oxidation[J]. Applied Microbiology and Biotechnology, 2010, 86(1): 359–365. DOI: 10.1007/s00253-009-2359-z |
| [21] | Krieg N R, Ludwig W, Whitman W, et al. Bergey's manual of systematic bacteriology[M]. (2nd ed). New York: Springer-Verlag, 2010. |
| [22] | Bae H, Chung Y C, Jung J Y. Microbial community structure and occurrence of diverse autotrophic ammonium oxidizing microorganisms in the anammox process[J]. Water Science and Technology, 2010, 61(11): 2723–2732. DOI: 10.2166/wst.2010.075 |
| [23] | 李滨, 赵志瑞, 马斌, 等. 克隆文库方法分析厌氧氨氧化反应器中细菌群落结构[J]. 环境科学与技术, 2012, 35(12): 159–164. Li B, Zhao Z R, Ma B, et al. Analysis on bacterial diversity of an anaerobic ammonium-oxidizing reactor by use of 16S rDNA clone library[J]. Environmental Science & Technology, 2012, 35(12): 159–164. |
| [24] | Strous M, Heijnen J J, Kuenen J G, et al. The sequencing batch reactor as a powerful tool for the study of slowly growing anaerobic ammonium-oxidizing microorganisms[J]. Applied Microbiology and Biotechnology, 1998, 50(5): 589–596. DOI: 10.1007/s002530051340 |
| [25] | Kuenen J G, Jetten M S M. Extraordinary anaerobic ammonium oxidizing bacteria[J]. ASM News, 2001, 67(9): 456–463. |
 2017, Vol. 38
2017, Vol. 38


