污水厂生物除磷稳定性及磷回收研究一直是污水处理领域的研究热点[1].聚磷菌是污水生物除磷中发挥主要作用的一类菌,聚磷菌聚磷与多个基因相关,而直接相关的是编码多聚磷酸盐激酶 (PPK) 的ppk基因,其可表达出多聚磷酸盐激酶,催化ATP上的磷酸基团连接形成多聚磷酸盐颗粒[2].污水除磷系统中已检测出ppk的不同分支[3, 4].构建ppk基因转导的工程菌,细胞内磷含量提高[5],工程菌除磷能力也大大提高[6].在大肠杆菌的研究中也发现,ppk能够影响聚磷颗粒的生成[7].
已从上百种细菌体内发现ppk基因,但它们的种类和人们所认为的PAO种类差别很大[8].即使在PAO候选细菌中,也发现ppk基因种间多样性非常复杂[9].在真核生物中也发现了ppk基因的存在,假丝酵母PPK氨基端多出的370多个氨基酸残基与大肠杆菌的PPK蛋白无同源性[10],推断在蛋白结构上真核生物PPK和原核生物的PPK是有差异的.利用GenBank进行BLAST,笔者也分析了不同生物的ppk基因,发现不同生物种间ppk并不具有同源性.关于ppk基因及其功能还无法得到确切结论.
大肠杆菌是基因操作中常用的细菌之一,其全基因组序列已被测序,是研究基因及其功能的模式菌株.为进一步研究ppk的功能特别是在生物除磷中的功能,本实验通过无痕敲除大肠杆菌ppk基因,比较工程菌株和野生型菌株除磷性能的差异,研究ppk基因在污水除磷中的作用,以期为进一步从分子生物学角度研究除磷的生物学机制提供理论依据.
1 材料与方法 1.1 材料 1.1.1 质粒和菌株pKD4,pKD46,pCP20,野生型菌株大肠杆菌ATCC25922,质粒增殖用宿主菌株DH5α.
1.1.2 主要工具酶及试剂基因敲除用各类酶及DL200Marker、DL500Marker均购自宝生物公司;L-阿拉伯糖、氨苄青霉素、卡那霉素购自上海生工;其它常规试剂均为国产分析纯.
1.1.3 培养基大肠杆菌ppk基因敲除用培养基:营养琼脂培养基,LB液体培养基、SOB培养基、SOC培养基和TSS培养基[11].
生长性能测定与聚磷性能分析用培养基:LB培养基;合成培养基2,成分:无水乙酸钠,0.5g ·L-1; 胰蛋白胨,5 g ·L-1;NH4Cl,0.2 g ·L-1;MgSO4,0.4 g ·L-1;FeSO4 ·7H2O,0.002 g ·L-1;CaCl2,0.08 g ·L-1;NaCl,5 g ·L-1;KH2PO4,视培养液需磷量而定.
1.1.4 引物实验所用引物序列见表 1.
|
|
表 1 实验所用引物序列 Table 1 Primers used in this study |
1.2 方法 1.2.1 大肠杆菌ppk基因缺失工程菌的构建
(1) 含卡那霉素抗性基因的外源DNA片段的构建
设计用于同源重组的引物F-Kan、R-Kan,其5′端50 bp同源臂 (划线部分) 为待敲除目标基因ppk两侧的延伸同源序列.以pKD4质粒为模板,扩增含卡那霉素抗性基因的外源DNA片段,胶回收后用于电转化.
(2) 短臂同源臂介导大肠杆菌ppk基因的无痕敲除
制备大肠杆菌ATCC25922电转化感受态细胞[11].用bio-red电转仪,将pKD46质粒电转化导入大肠杆菌ATCC25922,加入1 mL SOC液体培养基,30℃振荡培养2.5 h,离心后涂布于含氨苄青霉素 (60mg ·L-1) 的固体营养琼脂平板培养15 h.
制备感受态大肠杆菌E. coli/pKD46[11].将外源线性靶DNA片段电转化整合入其中.电转化条件为:电脉冲25 μF,电阻200 Ω、电压分别为1.8、2.0、2.3 kV;转化完毕加入1 mL SOC培养基,培养2.5 h后,涂布于含卡那霉素 (25mg ·L-1) 的营养琼脂平板上培养15 h.挑取单菌落接种于LB培养基中,30℃培养5 h后,升温至42℃继续培养10 h.同时将同一菌落接种于含有氨苄青霉素 (60 mg ·L-1) 的营养琼脂平板上,30℃过夜培养,挑取能够在含卡那霉素平板上生长而不能在氨苄青霉素平板上生长的菌落,即为重组成功菌落,标记为E. coli/ppk- Kan+.分别采用正向引物purn-F/ppx-R和反向引物p-F/p-R对阳性重组子进行扩增并测序,确定外源DNA片段是否整合入大肠杆菌基因组中.
将pCP20质粒电转化入感受态E. coli/ppk- Kan+,以消除卡那霉素抗性基因,转导方法同pKD46的转导.采用分别含有卡那霉素 (25 mg ·L-1)、氨苄青霉素 (60 mg ·L-1) 和无抗生素的LB营养琼脂平板筛选,挑取能够在LB平板上生长而在含有卡那霉素、氨苄青霉素的平板不生长的菌落,标记为E. coli/ppk- Kan-.采用鉴定引物purn-F/ppx-R和p-F/p-R分别进行PCR扩增并测序,验证大肠杆菌ppk基因缺失工程菌株E. coli/ppk- Kan-是否构建成功.
1.2.2 工程菌株和野生型菌株生长性能比较分别挑取工程菌株和野生型菌株的单菌落接种于LB培养基中,37℃过夜培养16 h后,以1%接种量接入并控制两菌初始接种量相同或相近,转接至100 mL LB培养基中,37℃、170 r ·min-1培养32 h,定时测定菌体光密度值,比较两菌生长性能的差异.同时,染色观察是否有聚磷颗粒形成[13].
1.2.3 工程菌株和野生型菌株除磷性能比较(1) 工程菌株和野生型菌株在缺磷诱导/富磷培养基上的除磷性能比较
采用培养基2,核算胰蛋白胨提供磷源量,计算加入KH2PO4量,控制缺磷培养基最终磷浓度为5 mg ·L-1,富磷培养基最终磷浓度为15 mg ·L-1.
培养操作方法同生长性能操作方法.缺磷诱导时,将菌体置于缺磷培养基中培养6 h,然后转入富磷培养基中培养,定时测定菌体光密度值和上清液中磷含量,并分别测定缺磷、富磷时菌体含磷量[13].
(2) 工程菌株和野生型菌株厌氧/好氧交替条件下的除磷性能比较
采用培养基2,调整磷浓度为5 mg ·L-1,厌氧培养6 h,6 000 r ·min-1离心清洗后,接入相同培养基,好氧培养6 h,重复5次,每次培养分别在厌氧末和好氧末测定上清液含磷量和菌体含磷量[11],并在厌氧末和好氧末分别进行PHB染色和异染粒染色[13].
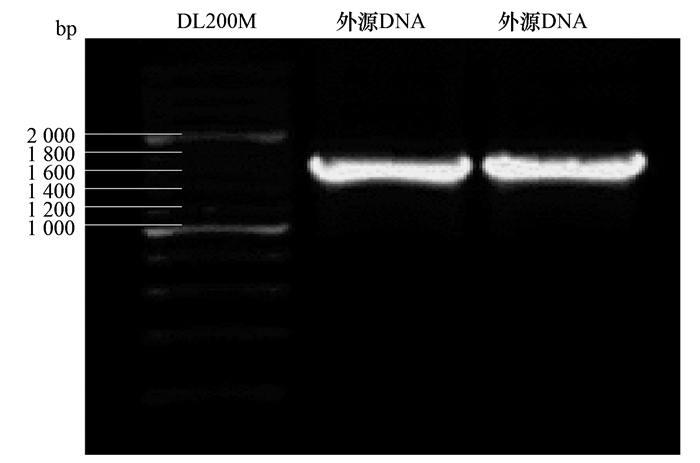
2 结果与分析 2.1 含卡那霉素抗性基因的外源目的DNA片段的构建采用引物F-Kan/R-Kan扩增,电泳得到约1 600 bp长度条带,结果见图 1,长度与预期结果相符.
|
图 1 外源目的DNA的PCR扩增 Fig. 1 PCR amplification of the target gene |
经测序验证外源片段长度为1 613 bp,BLAST结果表明该片段前50bp碱基和后50bp碱基为同源臂序列,中间部分为卡那霉素抗性基因片段,表明含卡那霉素抗性基因的外源目的DNA片段构建成功.
2.2 短臂同源臂介导大肠杆菌ppk基因的无痕敲除外源DNA电转化结果表明,仅当L-阿拉伯糖浓度为15 mmol ·L-1、电压为1.8 kV时,得到了重组菌,升温培养后,重组菌在含氨苄青霉素的平板上不生长而在含卡那霉素的平板上生长良好,初步证明外源基因已整合进大肠杆菌DNA.
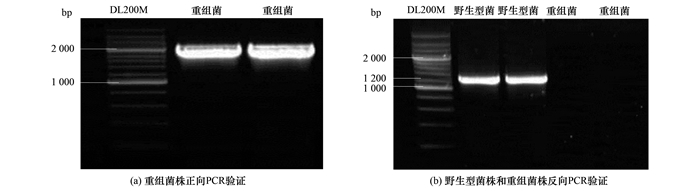
对大肠杆菌基因组序列信息分析表明,需敲除的目的基因ppk上游为purn基因,下游为ppx基因,以此设计鉴定引物,正向鉴定引物purn-F/ppx-R分别设计在purn基因和ppx基因区域,反向鉴定引物p-F/p-R设计在ppk基因内部.分别采用正反向鉴定引物,对重组菌株和野生型菌株进行菌落PCR验证,结果如图 2.
|
图 2 重组菌株正反向PCR验证 Fig. 2 Verification of recombinant strain by PCR |
由图 2可知,采用正向鉴定引物purn-F/ppx-R,重组菌株E. coli/ppk- Kan+的扩增条带约2000bp[图 2(a)],与预期结果相符,测序验证片段长度为2 012 bp.采用反向鉴定引物p-F/p-R,野生型菌株扩增条带大小约为1 200 bp[图 2(b)],与预期结果相符,重组菌未扩增出任何条带,经测序验证及BLAST比对后,表明大肠杆菌重组菌E. coli/ppk- Kan+重组成功,外源DNA已整合入大肠杆菌基因组中.
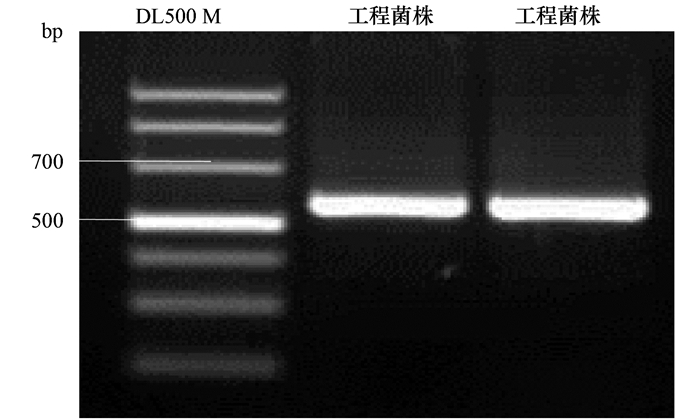
pCP20质粒电转化进入E. coli/ppk- Kan+重组菌株后,挑取只在营养琼脂平板生长而在其它抗性平板不生长的菌落,即为敲除成功的工程菌株.分别采用正反向鉴定引物purn-F/ppx-R和F/R引物进行菌落PCR验证,结果如图 3.
|
图 3 工程菌株正向PCR扩增验证 Fig. 3 Verification of mutant strain by PCR |
由图 3可知,采用purn-F/ppx-R扩增条带约为600bp,与预期结果相符,经测序验证片段长度为635bp.而采用反向鉴定引物p-F/p-R未扩增出片段,经BLAST比对,表明ppk基因缺失工程菌E. coli/ppk- Kan-构建成功.
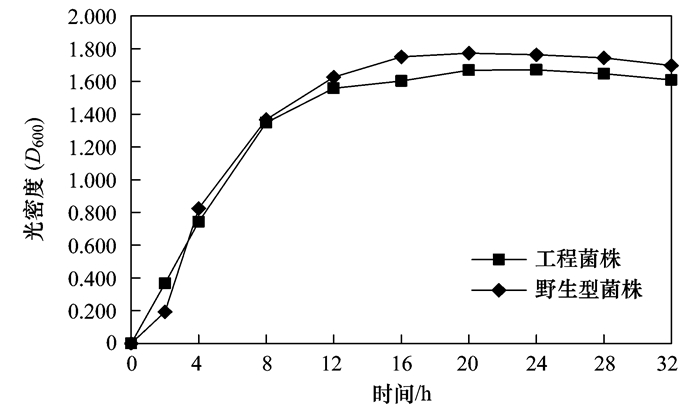
2.3 工程菌株和野生型菌株生长性能比较两株菌在LB培养基中生长性能比较见图 4.
|
图 4 工程菌株和野生型菌株生长特性比较 Fig. 4 Comparison of growth curves between mutant and wild-type strains |
由图 4可知,两株菌生长整体上无明显差异.培养初始,两株菌都处于高速生长状态,8 h后生长减缓,达到静止期,16 h时,菌体光密度值都达到1.5左右并不再增加.但是,前期对数生长期,野生型菌株生长较慢,2 h时菌体光密度值为0.198,而工程菌株为0.402;8 h时,两菌菌体光密度值基本一致,达到1.3左右. 8 h后进入稳定期,工程菌株较野生型菌株生长劣势.异染粒染色结果表明,两菌株在整个生长周期内都没有形成聚磷颗粒.因此,在LB培养基上培养,敲除ppk整体上并不影响大肠杆菌的生长,对聚磷颗粒的生成也无影响.但是,对数期工程菌生长快于野生型菌株,而静止期野生型菌株快于工程菌株.
2.4 工程菌株和野生型菌株在缺磷诱导/富磷培养基上除磷性能比较目前,分离富集聚磷微生物,有研究者采用限磷、富磷培养的蓝白斑筛选法[14];也有研究发现细菌要形成聚磷颗粒,需要经过压抑环境的诱导[15].实验比较了两菌株在经过6 h缺磷诱导后,在富磷培养下的除磷性能,结果见图 5.
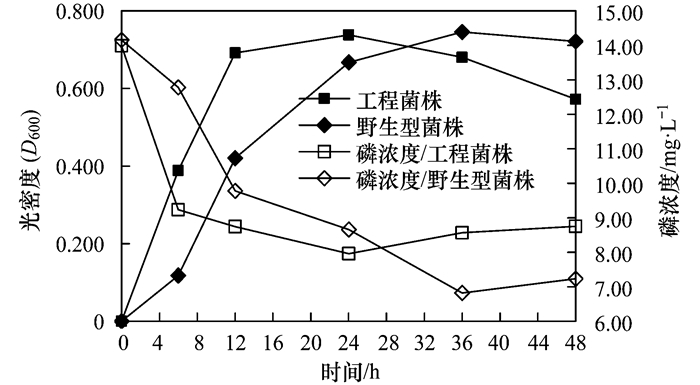
|
图 5 缺磷诱导/富磷培养基中工程菌株和野生型菌株除磷性能比较 Fig. 5 Comparison of phosphate accumulating characteristics between mutant and wild-type strains cultured in the phosphate luxuriant medium after induction in the phosphate lacking medium |
由图 5可知,经缺磷诱导后的工程菌株和野生型菌株接入富磷培养基中,两菌生长趋势与LB培养基上趋势基本一致.也表现出对数期工程菌株生长快于野生型菌株,24 h达到静止期后,工程菌株菌体密度快速下降,生长慢于野生型菌株.上清液磷浓度测定结果显示,对数期时,随着菌体密度上升,两培养基中磷浓度都下降,并且工程菌株相对于野生型菌株除磷能力稍强.稳定期后菌体密度下降,磷浓度下降量减少,除磷率也明显下降,表现出磷浓度降低的快慢与菌体密度成正比的趋势.并没有表现出由于ppk的缺失影响工程菌株除磷率的现象.
实验又比较了两菌株分别在LB培养基、缺磷培养和富磷培养时菌体的含磷量,结果见表 2.
|
|
表 2 工程菌株和野生型菌株生长对数期的菌体含磷量比较/% Table 2 Comparison of intercellular phosphate amount of mutant and wild-type strains in log phase/% |
由表 2可知无论是在缺磷还是经过缺磷诱导后富磷培养基中培养,工程菌和野生型菌株菌体含磷量都维持在一般微生物含磷量水平.结合图 5结果表明,缺磷诱导/富磷培养时,上清液磷浓度变化并不是菌体高效聚磷的结果,而是细菌生长过程同化作用利用磷、死亡时分解磷的结果. ppk缺失并不影响菌体在缺磷诱导/富磷培养中除磷能力.
2.5 工程菌株和野生型菌株厌氧/好氧交替条件下的除磷性能比较Ubukata等[16]认为一般情况下聚磷菌要表现出高效聚磷特性需要经过4次左右的诱导培养.本实验进行5次厌氧/好氧 (A/O) 交替诱导,并对每一个周期末菌体含磷量进行测定.比较两菌株经过诱导菌体含磷量的变化,结果见表 3.
|
|
表 3 厌氧/好氧交替诱导条件下工程菌株和野生型菌株菌体含磷量比较/% Table 3 Comparison of intercellular phosphate amounts of mutant and wild-type strains cultured in alternating anaerobic/aerobic condition/% |
由表 3知,经过5次诱导,野生型菌株和工程菌株菌体含磷量保持在一般细菌菌体含磷量1%~2%的水平,两菌都没有因诱导次数的增加而表现出菌体含磷量增加的趋势. PHB和异染粒染色结果表明,在厌氧培养过程中都未形成PHB,好氧培养过程也未发现聚磷颗粒的积累.诱导条件下,野生型菌株和工程菌株都未表现出明显的聚磷能力,ppk基因的缺失并没有引起细菌除磷性能的下降.
3 讨论在Red重组过程中,外源DNA片段电转化导入受体细胞并发生基因交换不易实现,一般重组效果都不理想,L-阿拉伯糖浓度密切影响重组效率.白光兴等[17]发现L-阿拉伯糖最佳的诱导浓度为30 mmol ·L-1,高于120 mmol ·L-1时细菌生长受到抑制,张雪等[18]认为100 mmol ·L-1的L-阿拉伯糖诱导能得到理想的重组子数量,也有人认为浓度降低到10 mmol ·L-1甚至2 mmol ·L-1效果更好[19].电转化电压也是影响重组效率重要因素[20].本实验对这两个因素进行了正交试验,控制L-阿拉伯糖诱导浓度分别为5、15、30、50 mmol ·L-1,控制电压分别为1.8、2.0和2.3 kV.结果表明仅当L-阿拉伯糖浓度为15 mmol ·L-1、电压为1.8 kV时,得到了重组菌,而其它组合条件均没有得到重组子.
Shi等[21]发现在LB培养基上培养的ppk缺失的赖氨酸芽孢杆菌,ppk的缺失只影响聚磷的积累而不影响细菌的生长,但是缺失菌株在氨基酸缺乏的压力条件下有多聚磷酸盐的积累. Hossain等[22]发现丁香假单胞菌ppk缺失菌,其编码静止期RpoS蛋白的rpoS基因在对数期减少. RpoS蛋白参与静止期基因的表达,使其能维持静止期免受热、高渗、饥饿等不良条件的影响.而当将在不良环境下生长的rpoS缺失菌放在营养丰富培养基上生长时,其能存活下来[23].而Shi等[24]发现大肠杆菌ppk突变导致其适应压力条件的功能受损,从而使其在静止期存活下降.彭亮[12]也证实,ppk缺失,聚磷颗粒合成减少,RpoS合成下降.实验中由于是在LB培养基上,ppk缺失整体上不会影响到生长.而工程菌在对数期表现出来的生长快于野生型菌株的特点,可能是因为对数期营养丰富,ppk缺失并不影响菌体生长,反而可能导致能量合成及利用代谢过程的改变,工程菌株为了生存表现出了能量过度利用而快速生长的特点[25].而到静止期营养受限的压力条件下,由于ppk缺失,影响了rpoS表达,出现生长下降.
杜宏伟等[26]将大肠杆菌ppk基因转入假单胞菌,获得工程菌株其体内聚磷颗粒量明显大于原始菌株,同时,工程菌静止期生长量大于原始菌株,认为是静止期时,外源营养物质下降,工程菌产生的聚磷促进了细胞的生长.王勤等[5]发现转入ppk基因的大肠杆菌其静止期生长也高于原始菌,也认为静止期ppk基因可促进菌体生长. Motlagh等[27]研究EBPR时发现,一些化学压力能够导致ppk表达下降,从而影响除磷效果.这些结果表明ppk对细菌静止期的生长及聚磷颗粒的生成具有促进作用.而李波[28]敲除恶臭假单胞菌ppk后,菌体没有因ppk基因的缺失而表现出聚磷能力的下降.认为该菌可能还存在另外一个与聚磷相关的基因ppk2或者其它的磷代谢路径.目前也发现了ppk2基因的存在并证实了其聚磷功能[29, 30].除了ppk和ppk2外,还有多种酶共同调控聚磷颗粒的合成和分解代谢[31],Shi等[24]研究发现蜡状芽孢杆菌ppk基因缺失菌株聚磷颗粒的含量不到野生菌的5%,而ppx(Exopolyphosphatase) 缺失菌株的聚磷颗粒含量约是野生型的1 000倍,且PPK酶活性提高了10倍.王智[32]敲除了中华根瘤菌的ppx基因,发现其摄磷能力大幅度提高. Jones等[33]也证实,海洋底泥中的聚磷颗粒的降解和ppk2及ppx的表达有密切关系. Graham等[34]研究也证实很多基因都与ppk表达相关,文献[35]从宏基因组学角度分析认为除磷代谢中的糖原是通过EMP途径而不是通过ED途径代谢的;除磷反应器中存在众多的代谢途径,有着更多的基因调控除磷过程[9],Oyserman等[36]也发现聚磷的调控途径众多,认为除磷是这些基因的共同作用导致的最终结果.因此,生物聚磷是生物体内众多相关基因的复杂调控及其代谢的最终表现,本实验中未发现ppk敲除基因菌体的除磷变化,是因为ppk基因敲除后与其代谢相关的其他基因的表达发生变化,代偿因ppk基因缺失而影响微生物的摄磷行为,其具体机制还有必要进一步研究[37].
4 结论利用Red敲除系统,采用短臂同源重组方法,通过无痕敲除,并用正反向引物进行PCR验证,成功构建了大肠杆菌ppk缺失工程菌株.敲除后的工程菌株和野生型菌株生长特性整体没有差异,但是对数期工程菌株生长快于野生型菌株,静止期由于缺乏ppk基因,影响到菌体生长,工程菌株生长慢于野生型菌株.缺磷诱导/富磷条件下,工程菌株没有因为ppk基因的缺失而出现除磷功能下降.厌氧/好氧诱导条件下,工程菌株也并没有表现出菌体含磷量增加的趋势,ppk基因的缺失没有引起工程菌除磷能力的大幅下降. ppk基因的缺失在污水处理中并没有影响到除磷功能.
| [1] | 张顺, 田晴, 汤曼琳, 等. 磷回收对厌氧/好氧交替式生物滤池蓄磷/除磷的影响[J]. 环境科学, 2014, 35(3): 979–986. Zhang S, Tian Q, Tang M L, et al. Effect of phosphorus recovery on phosphorous bioaccumulation/harvesting in an alternating anaerobic/aerobic biofilter system[J]. Environmental Science, 2014, 35(3): 979–986. |
| [2] | 魏峥, 聂琰晖, 刘乐庭, 等. 多聚磷酸盐在原核和真核生物中的研究进展[J]. 生理科学进展, 2009, 40(3): 197–202. Wei Z, Nie Y H, Liu L T, et al. Progress in functional polyphosphate in prokaryotic and eukaryotic living organisms[J]. Progress in Physiological Sciences, 2009, 40(3): 197–202. |
| [3] | He S M, Gall D L, McMahon K D, et al. "Candidatus Accumulibacter" population structure in enhanced biological phosphorus removal sludges as revealed by polyphosphate kinase genes[J]. Applied and Environmental Microbiology, 2007, 73(18): 5865–5874. |
| [4] | Zeng W, Wang A Q, Zhang J, et al. Enhanced biological phosphate removal from wastewater and clade-level population dynamics of "Candidatus Accumulibacter phosphatis" under free nitrous acid inhibition:linked with detoxication[J]. Chemical Engineering Journal, 2016, 296: 234–242. |
| [5] | 王勤, 赵庆顺, 肖琳, 等. 转聚磷激酶基因的大肠杆菌去除水体中的磷[J]. 中国环境科学, 2006, 26(6): 742–745. Wang Q, Zhao Q S, Xiao L, et al. Soluble phosphorus removed by the Escherichia coli transformed with polyphosphate kinase gene[J]. China Environmental Science, 2006, 26(6): 742–745. |
| [6] | 任晶, 肖琳, 郑小红, 等. 转聚磷激酶大肠杆菌诱导表达条件及磷浓度对其除磷能力的影响[J]. 环境保护科学, 2009, 34(6): 1–3, 22. Ren J, Xiao L, Zheng X H, et al. Effect of induction conditions and phosphate concentration on the ability of polyphosphate kinase gene transferred E. coli to remove phosphate[J]. Environmental Protection Science, 2009, 34(6): 1–3, 22. |
| [7] | Kuroda A, Nomura K, Ohtomo R, et al. Role of inorganic polyphosphate in promoting ribosomal protein degradation by the Lon protease in E. coli[J]. Science, 2001, 293(5530): 705–708. |
| [8] | Hooley P, Whitehead M P, Brown M R W, et al. Eukaryote polyphosphate kinases:is the 'Kornberg' complex ubiquitous?[J]. Trends in Biochemical Sciences, 2008, 33(12): 577–582. |
| [9] | Skennerton C T, Barr J J, Slater F R, et al. Expanding our view of genomic diversity in Candidatus Accumulibacter clades[J]. Environmental Microbiology, 2015, 17(5): 1574–1585. |
| [10] | Rao N N, Gómez-García M R, Kornberg A. Inorganic polyphosphate:essential for growth and survival[J]. Annual Review of Biochemistry, 2009, 78(1): 605–647. |
| [11] | 萨姆布鲁克·J著, 黄培堂译.分子克隆实验指南[M]. (第三版).北京:科学出版社, 2002. |
| [12] | 彭亮.聚磷酸盐激酶1在脑膜炎大肠杆菌K1株致病中的相关功能研究[D].广州:南方医科大学, 2011. 10-11, 49. Peng L. Study on the role of polyphosphate kinase 1 in the Pathopoiesis of meningitis Escherichia coli K1[D]. Guangzhou:Southern Medical University, 2011. 10-11, 49. |
| [13] | 国家环境保护总局. 水和废水监测分析方法[M]. (第四版). 北京: 中国环境科学出版社, 2002. |
| [14] | 仲小敏.除磷微生物筛选及除磷条件优化研究[D].无锡:江南大学, 2012. Zhong X M. Screening and optimization of phosphorus removal microorganism[D]. Wuxi:Jiangnan University, 2012. |
| [15] | van Dien S J, Keasling J D. Control of polyphosphate metabolism in genetically engineered Escherichia coli[J]. Enzyme and Microbial Technology, 1999, 24(1-2): 21–25. |
| [16] | Ubukata Y, Takii S. Induction ability of excess phosphate accumulation for phosphate removing bacteria[J]. Water Research, 1994, 28(1): 247–249. |
| [17] | 白光兴, 孙志伟, 黄莺, 等. 利用Red重组系统对大肠杆菌ClpP基因的敲除[J]. 中国生物化学与分子生物学报, 2005, 21(1): 35–38. Bai G X, Sun Z W, Huang Y, et al. Deletion of clpP in chromosome of E. coli by red recombination[J]. Chinese Journal of Biochemistry and Molecular Biology, 2005, 21(1): 35–38. |
| [18] | 张雪, 温廷益. Red重组系统用于大肠杆菌基因修饰研究进展[J]. 中国生物工程杂志, 2008, 28(12): 89–93. Zhang X, Wen T Y. Advances of red recombination system in Escherichia coli gene modification[J]. China Biotechnology, 2008, 28(12): 89–93. |
| [19] | Lee K H, Park J H, Kin T Y, et al. Systems metabolic engineering of Escherichia coli for L-threonine production[J]. Molecular Systems Biology, 2007, 3(1): 149. |
| [20] | 朱森康, 黄磊, 李燕飞, 等. 制备高效大肠杆菌电转化感受态细胞和电转化条件的研究[J]. 生物技术通报, 2011(10): 206–209. Zhu S K, Huang L, Li Y F, et al. Preparation of efficient electrotransformation competent cells of Escherichia coli and condition of electrotransformation[J]. Biotechnology Bulletin, 2011(10): 206–209. |
| [21] | Shi T Y, Ge Y, Zhao N, et al. Polyphosphate kinase of Lysinibacillus sphaericus and its effects on accumulation of polyphosphate and bacterial growth[J]. Microbiological Research, 2015, 172: 41–47. |
| [22] | Hossain M M, Tani C, Suzuki T, et al. Polyphosphate kinase is essential for swarming motility, tolerance to environmental stresses, and virulence in Pseudomonas syringae pv. tabaci 6605[J]. Physiological and Molecular Plant Pathology, 2008, 72(4-6): 122–127. |
| [23] | Wang Y, Kim K S. Effect of rpoS mutations on stress-resistance and invasion of brain microvascular endothelial cells in Escherichia coli K1[J]. FEMS Microbiology Letters, 2000, 182(2): 241–247. |
| [24] | Shi X B, Rao N N, Kornberg A. Inorganic polyphosphate in Bacillus cereus:motility, biofilm formation, and sporulation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(49): 17061–17065. |
| [25] | Gottesman S, Maurizi M R. Surviving starvation[J]. Science, 2001, 293(5530): 614–615. |
| [26] | 杜宏伟, 武俊, 肖琳, 等. 聚磷激酶基因在假单胞菌中的整合和表达[J]. 环境科学, 2009, 30(10): 3011–3015. Du H W, Wu J, Xiao L, et al. Integration and expression of polyphosphate kinase gene in pseudomonas putida[J]. Environmental Science, 2009, 30(10): 3011–3015. |
| [27] | Motlagh A M, Bhattacharjee A S, Goel R. Microbiological study of bacteriophage induction in the presence of chemical stress factors in enhanced biological phosphorus removal (EBPR)[J]. Water Research, 2015, 81: 1–14. |
| [28] | 李波.聚磷菌多聚磷酸盐激酶的分子生物学研究及高效除磷工程菌的构建[D].南京:南京农业大学, 2008. 46. Li B. Molecular biology research of polyphosphate kinase in polyphosphate accumulating organisms and construction of genetical engineered polyphosphate accumulation strains[D]. Nanjing:Nanjing Agricultural University, 2008. 46. |
| [29] | Ishige K, Zhang H Y, Kornberg A. Polyphosphate kinase (PPK2), a potent, polyphosphate-driven generator of GTP[J]. Proceedings of the National Academy of Sciences, 2002, 99(26): 16684–16688. |
| [30] | Sureka K, Sanyal S, Basu J, et al. Polyphosphate kinase 2:a modulator of nucleoside diphosphate kinase activity in mycobacteria[J]. Molecular Microbiology, 2009, 74(5): 1187–1197. |
| [31] | Brown M R W, Kornberg A. The long and short of it-polyphosphate, PPK and bacterial survival[J]. Trends in Biochemical Sciences, 2008, 33(6): 284–290. |
| [32] | 王智.磷代谢关键基因的克隆及高效聚磷工程菌构建的研究[D].北京:中国农业科学院, 2008. 36-48. Wang Z. Cloning of key genes of phosphate metabolism and research of construction of engineering strain with high capability of accumulating polyphosphate[D]. Beijing:Chinese Academy of Agricultural Sciences, 2008. 36-48. |
| [33] | Jones D S, Flood B E, Bailey J V. Metatranscriptomic insights into polyphosphate metabolism in marine sediments[J]. The ISME Journal, 2016, 10(4): 1015–1019. |
| [34] | Graham J E, Wantland N B, Campbell M, et al. Chapter Fourteen-characterizing bacterial gene expression in nitrogen cycle metabolism with RT-qPCR[J]. Methods in Enzymology, 2011, 496: 345–372. |
| [35] | Martín H G, Ivanova N, Kunin V, et al. Metagenomic analysis of two enhanced biological phosphorus removal (EBPR) sludge communities[J]. Nature Biotechnology, 2006, 24(10): 1263–1269. |
| [36] | Oyserman B O, Noguera D R, del Rio T G, et al. Metatranscriptomic insights on gene expression and regulatory controls in Candidatus Accumulibacter phosphatis[J]. The ISME Journal, 2016, 10(4): 810–822. |
| [37] | 孙雪, 朱为静, 王亮, 等. 强化生物除磷系统主要微生物及其代谢机理研究进展[J]. 应用生态学报, 2014, 25(3): 892–902. Sun X, Zhu W J, Wang L, et al. Review on the main microorganisms and their metabolic mechanisms in enhanced biological phosphorus removal (EBPR) systems[J]. Chinese Journal of Applied Ecology, 2014, 25(3): 892–902. |
 2017, Vol. 38
2017, Vol. 38

