2. 杭州市环境监测中心站, 杭州 310007
2. Hangzhou Environmental Monitoring Center, Hangzhou 310007, China
抗生素自19世纪末被发现以来,广泛应用于人类感染性疾病的控制和畜禽养殖、水产养殖.但抗生素的不合理使用和长期滥用使得环境中微生物耐药性产生的选择压力增强,导致环境中抗生素抗性细菌 (antibiotic resistance bacteria,ARB) 和抗性基因 (antibiotic resistance genes,ARGs) 含量不断增加,且抗性基因能通过遗传和水平基因转移在亲代和其他菌种间传播扩散,对人类健康和生态平衡造成危害.抗生素和抗生素抗性基因已经被认为是新型环境污染物[1, 2].污水处理厂是人类活动产生污水的交汇点,同样也是抗性细菌和抗性基因的储存库[3].目前的研究表明,现有的各类污水处理厂工艺对抗性基因有一定的去除效果,但是出水中抗性基因仍处于一个较高的丰度[3~5],存在一定的环境风险.故需要有针对性地研究抗性基因污染问题,结合现有的污水处理工艺,提出合适的控制技术[6~8].
消毒是污水处理过程中杀灭微生物的水处理过程,是保障污水处理厂尾水水质安全必不可少的环节.消毒处理在杀灭微生物的同时,也可能削减抗性基因[9, 10].但是,目前对不同消毒削减抗性基因的影响因素和具体机制仍尚不明确.此外,消毒对抗性基因的削减效果还受到其抗性细菌影响,不同抗性细菌对其携带的抗性基因可能有不同的保护作用[10]. Huang等[11]对1株未携带抗性基因的大肠杆菌和携带tetA的大肠杆菌进行紫外和氯消毒研究发现,携带抗性基因的菌株表现出更高的消毒耐受性.虽然目前对于氯消毒、紫外消毒、臭氧消毒等常规消毒方法杀灭细菌的机制有了较为成熟的理论[12, 13],但抗性基因在消毒过程中的行为特征有待讨论.本研究采用“消毒+DNaseI酶”实验来研究抗性基因在各消毒过程中的行为特征.有研究表明,DNA可游离于细胞体之外,成为游离态DNA,并在环境中维持较长时间[14].游离态DNA上的抗性基因则可能通过基因水平转移途径重新进入细菌,继续产生环境危害[15]. DNaseI酶是1种DNA降解酶,应用其对水环境中胞外游离态DNA进行降解,有助于了解消毒方法削减水中抗性基因的机制[16].
磺胺类药是人工合成的抗生素,自1935年临床应用以来,仅80余年就已产生明显的环境耐药性问题[17, 18].本研究从磺胺类抗性基因入手,分析氯消毒、紫外消毒、臭氧消毒等常规消毒方法对抗性基因的削减作用.从某污水处理厂二级出水中分离出磺胺类抗性细菌,并挑取典型的革兰氏阳性菌和阴性菌各1株,确定其中的磺胺类抗性基因种类及菌株种类.以上述两株细菌为实验对象,污水厂二级出水灭菌后作为实验水样,控制上述细菌浓度投加到实验水样中,比较研究消毒对抗性细菌和抗性基因的削减效果.同时,利用“消毒+DNaseI酶”实验研究磺胺类抗性基因在消毒过程中的行为特征,以期为理解消毒削减抗性基因的机制,并为污水中抗性基因污染的控制方法提供新思路.
1 材料与方法 1.1 实验菌株筛选采集浙江临安某污水处理厂氧化沟出水 (二级出水),采用稀释、涂布分离方法在磺胺类选择培养基平板上筛选磺胺类抗性细菌.磺胺类选择培养基配置方法为:蛋白胨10 g ·L-1,酵母提取物5 g ·L-1,氯化钠10 g ·L-1,调节pH至7后121℃、20 min高压灭菌,在完全凝固前趁温加入磺胺甲唑母液至浓度为50.4 μg ·mL-1[17, 19].母液浓度40 mg ·mL-1,采用1 mol ·L-1 NaOH溶液在避光条件下溶解配置. 30℃、24 h培养后,挑取单菌落分离纯化,采用细菌基因组提取试剂盒TIANamp Bacteria DNA Kit (TIANGEN, China) 提取细菌DNA,由上海睿迪生物科技有限公司测定16S rRNA序列,测序结果通过使用NCBI网站 (http://www.ncbi.nhn.nih.ov/blast/) 的BLAST进行序列同源性检索比对,确定单菌株菌种.选择典型革兰氏阳性菌和革兰氏阴性菌各1株,采用普通PCR法对两株细菌分别进行磺胺类抗性基因 (sul Ⅰ 、sul Ⅱ 、sul Ⅲ 、sulA) 及16S rRNA、intI1基因进行定性检测,分析其基因类型.
PCR体系为:10×PCR buffer (Mg2+)2.5 μL,2.5 mmol ·L-1,dNTP 2.0 μL,10 mmol ·L-1上游引物1.0 μL,10 mmol ·L-1下游引物1.0 μL,5U Taq酶0.3 μL,ddH2O 17.2 μL,DNA样品1.0 μL.各基因引物见表 1.在PCR循环仪 (TIANLONG,China) 中进行扩增,PCR扩增条件为:95℃预变性5 min后,95℃变性15 s,退火30 s,72℃延伸30 s,共35个循环,最后72℃下延伸7 min.普通PCR完成后,取5 μL PCR反应产物,与6×Loading buffer Dye混合均匀后,使用经EB染色的1.5%的琼脂糖凝胶,在120 V电压下电泳30 min检测扩增条带.使用全自动凝胶成像仪 (PEIQING, China) 对琼脂糖凝胶进行拍照,分析所得PCR产物长度并与表 1中提供的扩增片段相比较.
|
|
表 1 各基因引物序列及退火温度 Table 1 Primer sequence and annealing temperature of each gene |
本研究采用菌株为:①琼氏不动杆菌,Acinetobacter junii,革兰氏阴性,16S rRNA序列NCBI登录号:KY120958;②枯草芽孢杆菌,Bacillus subtilis,革兰氏阳性,16S rRNA序列NCBI登录号:KY121111.两者均含有sul Ⅰ、sul Ⅱ ,不含有sul Ⅲ、sulA,故下文对磺胺类抗性基因的研究主要集中于sul Ⅰ、sul Ⅱ .
1.2 实验水样准备将实验菌株培养30℃、24 h后的菌液10 000 r ·min-1离心10 min,弃上清液收集菌体,用无菌PBS重悬,利用0.5号麦氏比浊管控制细菌浓度约为1.5×108 CFU ·mL-1.污水处理厂二级出水采用0.22 μm膜进行过滤灭菌后,将细菌PBS溶液以1 :14的比例投加至水样中,使最终实验水样细菌浓度约为107 CFU ·mL-1(模拟现实水样细菌浓度).
1.3 氯消毒氯消毒剂采用次氯酸钠溶液,将其稀释至有效氯10 g ·L-1,并置于棕色试剂瓶中在4℃下避光保存,实验前测定实际有效氯含量后立即使用.取1 L水样,滴加次氯酸钠溶液,使样品中的有效氯浓度依次达到设定值 (1、2、3、4、5 mg ·L-1).实验水样体积为1 L,氯消毒接触时间为30 min.在氯消毒完成后,投加5 mg ·L-1的硫代硫酸钠溶液终止消毒反应.每次消毒实验重复3次,下同.
1.4 紫外消毒紫外线消毒处理在实验室自主设计的紫外线平行光束仪装置内进行,平行光束仪内含有5根不同功率的低压紫外UV254灯管,灯光水平中心线距离实验水样表面50 cm.紫外线平行光束仪装置示意如图 1所示,从灯管发出的紫外线经平行光柱,均匀地辐照到反应容器 (20 cm×20 cm×5 cm).实验水样体积为1 L,控制紫外灯强度不变,通过调节照射时间以获得不同的紫外剂量 (5、10、15、20、25 mJ ·cm-2).为避免光复活效应,经紫外消毒处理后,立即进行下一步的样品处理和检测.
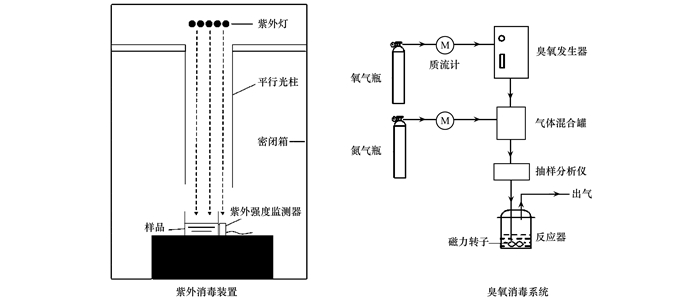
|
图 1 紫外消毒与臭氧消毒装置示意 Fig. 1 Set-up diagram used in ultraviolet and ozone disinfection |
臭氧消毒处理采用VMUS-DG臭氧发生器 (AZCO,Canada).气路上装有流量计和臭氧分析仪UV100(ECO,USA),用以测量进出气体中的臭氧浓度,如图 1所示.实验水样体积为1 L,通过调节气体流速和臭氧浓度,使臭氧消毒接触时间为10 min时,臭氧投加量达到目标设定值 (2、4、6、8、10 mg ·L-1).
1.6 DNaseI处理向消毒处理后的水样中加入500 U单位DNaseI (约可降解250 μg DNA,大于水样中总DNA含量),15℃反应振荡2 h.
1.7 菌落计数法本研究使用菌落计数法来确定水样中的磺胺类抗性细菌数目.用无菌水对水样进行梯度稀释,各取100 μL水样稀释液于磺胺类选择培养基平板上,用玻璃刮刀涂布均匀,平行3份,30℃、24 h培养后,进行菌落计数 (CFU ·mL-1).
1.8 荧光定量PCR本研究使用荧光定量PCR法检测抗性基因的绝对丰度.水样采用FastDNA Spin Kit forsoil试剂盒 (MP,USA) 提取DNA,使用微量蛋白质核酸分析仪 (Nanodrop,USA) 测定DNA浓度,置于-85℃保存.
荧光定量PCR标准曲线的建立:普通PCR扩增得到的目的基因序列使用Biospin凝胶回收试剂盒 (BIOER,China) 进行纯化回收PCR产物,后测量DNA含量、纯度并调节至合适浓度后,连接pMD19-T载体,转化入E. coli感受态细胞DH5α,将感受态细胞涂于含有氨苄青霉素、X-gal和IPTG的LB固体培养基上培养12~16 h;通过蓝白斑筛选挑取白色重组菌斑,挑选阳性克隆子用LB培养液扩大培养,待菌液混浊后,取1 mL菌液测序插入基因片断.测序结果通过使用BLAST进行序列同源性检索比对;其余3~4 mL菌液用来提取质粒,使用QIAGEN质粒专用提取试剂盒 (QIAPREP,Germany) 进行质粒抽提,确保质粒DNA的A260/A280比值在1.8左右.符合要求的质粒,作为标准品计算其浓度[质粒浓度的计算公式为:浓度=(质量÷相对分子质量)×6.02×1023].将已知拷贝数的质粒标准品依次进行10倍稀释,并且保持质粒浓度在108~102之间.标线扩增效率见表 1.
定量PCR反应在Bio-rad IQ5荧光定量PCR仪器 (Bio-Rad,USA) 中进行.定量PCR反应的反应液体系:7.5 μL SYBR Premix Ex Taq溶液,0.3 μL ROX Reference溶液,0.3 μL浓度为10 mmol ·L-1浓度的正向引物,0.3 μL浓度为10 mmol ·L-1浓度的反向引物,4.6 μL ddH2O.反应液体系在100 μL 96孔反应板的反应孔中进行.定量PCR反应程序为预变性95℃下热变性30 s;然后进入40个循环的扩增阶段,包括95℃变性5 s,退火30 s,72℃延伸30 s,延伸的同时扫描荧光信号.溶解曲线程序为55~95℃之间,每0.5℃读数,其间停留30 s.每个样品做3次重复.
1.9 数据处理数据采用Excel 2010和Origin 9.0软件进行分析.
2 结果与讨论 2.1 3种消毒方法对磺胺类抗性细菌的削减效果不同消毒方法对实验水样中的磺胺类抗性菌株均有一定的去除效果,且在低剂量浓度下,抗性细菌的削减效果随着消毒剂量的增加而提高,在实验设置的最高剂量 (5 mg ·L-1氯投加浓度,25 mJ ·cm-2紫外剂量,10 mg ·L-1臭氧投加浓度) 下,实验水样中的抗性细菌大多已被灭活,如图 2所示.当消毒剂量达到一定水平后,抗性细菌均不能被菌落计数法检测.在两种细菌的比较中,实验发现枯草芽孢杆菌 (革兰氏阳性菌) 的耐受性强于琼氏不动杆菌 (革兰氏阴性菌),这可能与革兰氏阳性菌的细胞壁保护作用有关,其能阻碍自由氯和臭氧分子的穿透,一定程度上保护细菌.在紫外消毒中,McKinney等[10]也发现了类似现象,他们深入研究后认为,这可能与两种细菌基因序列中总基因组大小不同有关.紫外消毒导致DNA中的嘧啶经过光化学的破坏,相邻胸腺嘧啶产生二聚作用.细菌的DNA中多数胸腺嘧啶二聚物的形成阻止了DNA复制和最终导致细胞死亡,达到消毒的目的.革兰氏阳性菌的总基因组更小,意味着它们有更少的潜在嘧啶二聚体目标,所以枯草芽孢杆菌对紫外的敏感性更低.

|
G-ARB:琼氏不动杆菌水样;G+ARB:枯草芽孢杆菌水样;ARB存活率为存活量与初始量的比值,两者单位为CFU ·mL-1 图 2 不同消毒方法中磺胺类抗性细菌数量的变化 Fig. 2 Change of ARB's concentration in different disinfections |
对消毒剂消毒效果的比较,可以采用CT值 (concentration-time value) 来衡量[23]. CT值是消毒剂的浓度和作用时间的乘积,用于比较消毒剂杀菌作用的指标,CT值越低消毒效果越好.将氯消毒和臭氧消毒相对比,当琼氏不动杆菌的削减率达到3个数量级左右时,氯消毒的CT值为90 mg ·(min ·L)-1,而臭氧消毒的CT值为20 mg ·(min ·L)-1,可以看出,臭氧的CT值远远小于氯.因此臭氧削减目标细菌所需时间更短,效率更高.此外,污水厂常使用紫外消毒作为深度处理工艺,使用的辐照剂量在30 mJ ·cm-2以下[24, 25].本研究中,25 mJ ·cm-2消毒剂量下,琼氏不动杆菌和枯草芽孢杆菌基本灭活,研究表明通常污水厂所设置的辐射剂量能灭活磺胺类抗性细菌.
2.2 3种消毒方法对磺胺类抗性基因的削减效果目前各消毒方法的评价都是以微生物的削减程度来衡量的,但是根据实验结果,发现削减微生物的量并不直接等同于削减抗性基因的量. 图 3为实验水样在不同消毒剂量下磺胺类抗性基因的变化情况,可知在消毒过程中,磺胺类抗性基因大体上是随着消毒剂量的增加而下降的.氯消毒中削减效果较为明显,在本实验设置的最低浓度1 mg ·L-1时,琼氏不动杆菌水样的sul Ⅱ 绝对丰度削减率为78.9%,枯草芽孢杆菌水样的sul Ⅱ 绝对丰度削减率为70.5%. sul Ⅱ 的削减率要高于sul Ⅰ ,但是随着消毒剂量的上升,sul Ⅰ 基因丰度也有明显的下降,氯消毒浓度达到3 mg ·L-1,枯草芽孢杆菌水样的sul Ⅰ 绝对丰度削减率由2 mg ·L-1的29.7%上升到77.9%.在氯消毒浓度达到4 mg ·L-1时,两种不同目标细菌sul Ⅰ 、sul Ⅱ 绝对丰度削减率都能达到95.0%以上.琼氏不动杆菌水样与枯草芽孢杆菌水样磺胺类抗性基因的基因丰度随氯消毒浓度的变化规律基本一致,但琼氏不动杆菌水样的削减率高于枯草芽孢杆菌水样.结合磺胺类抗性基因丰度和16S rRNA基因丰度之间的相关性分析发现,氯消毒过程中,琼氏不动杆菌水样中sul Ⅱ 基因与16S rRNA基因呈现正相关关系 (P < 0.05),R2为0.90(如图 4);枯草芽孢杆菌水样中sul Ⅰ 、sul Ⅱ 基因均与16S rRNA基因呈现正相关关系 (P < 0.05),R2分别为0.77和0.59. 16S rRNA基因的丰度在一定程度上反映了样品中的微生物量,这表明氯消毒削减磺胺类抗性基因主要和微生物量的降低有关.
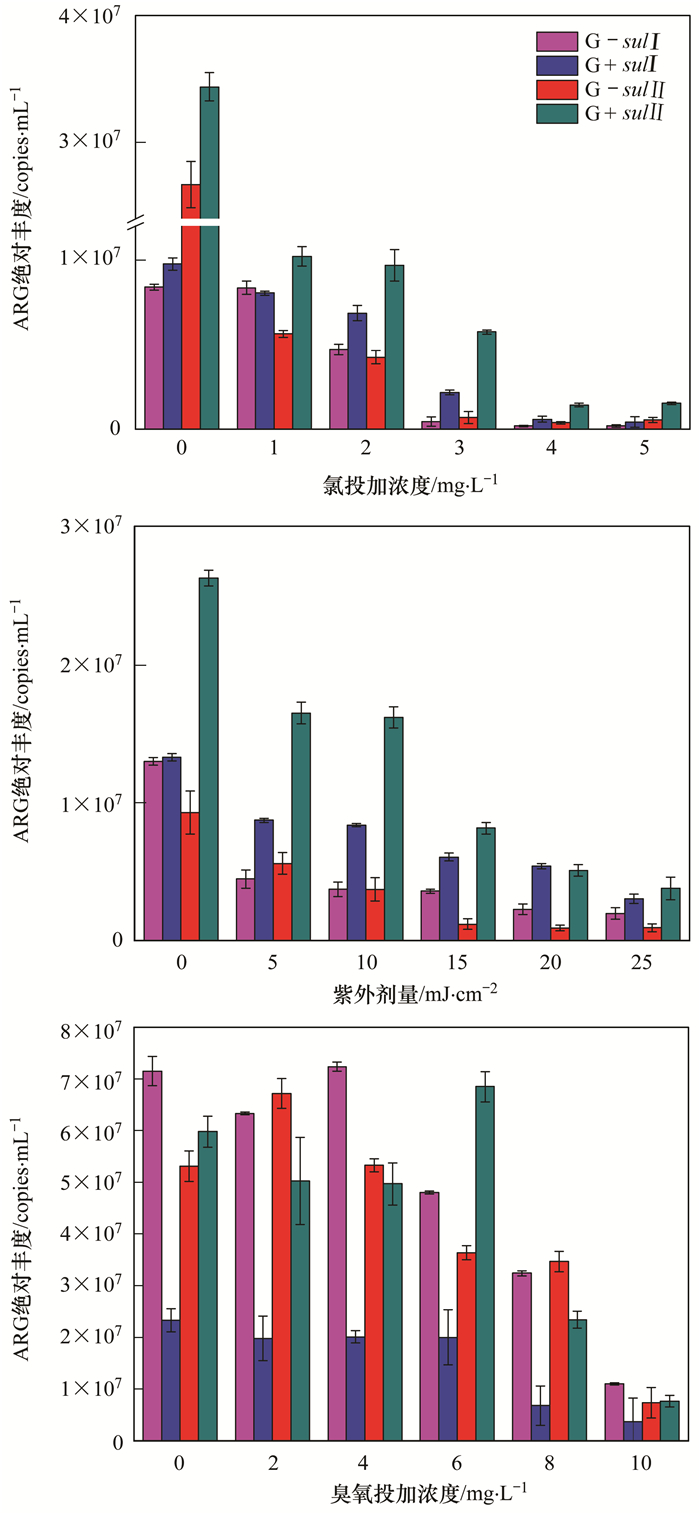
|
G-sulⅠ、G-sulⅡ:琼氏不动杆菌水样中sulⅠ、sulⅡ基因; G+sulⅠ、G+sulⅡ:枯草芽孢杆菌水样中sulⅠ、sulⅡ基因 图 3 不同消毒方法中磺胺类抗性基因绝对丰度的变化 Fig. 3 Change of ARGs'abundance in different disinfections |
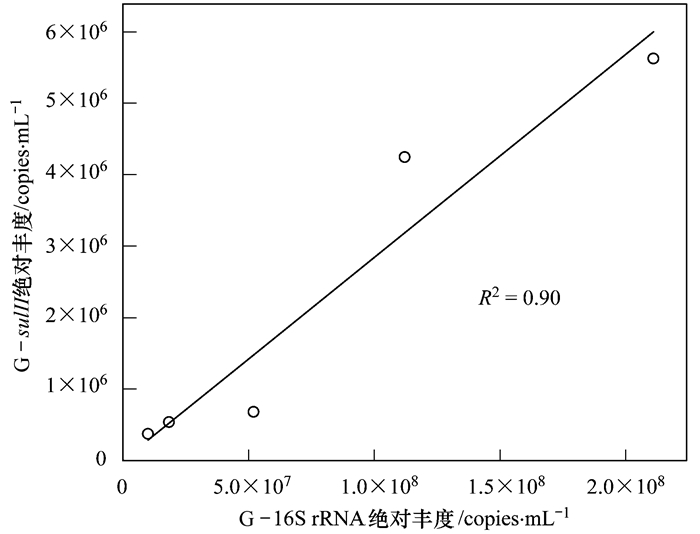
|
图 4 氯消毒中琼氏不动杆菌16S rRNA与sulⅡ拟合曲线 Fig. 4 Fitting curve for abundance ofAcinetobacter junii's 16S rRNA to abundance of sulⅡ in chlorination |
紫外消毒和臭氧消毒对磺胺类抗性基因的削减效果一般,在设置的最高剂量 (25 mJ ·cm-2紫外剂量,10 mg ·L-1臭氧投加浓度) 下,sul Ⅰ 绝对丰度削减率为84.9%、84.6%(琼氏不动杆菌) 和77.4%、84.3%(枯草芽孢杆菌),sul Ⅱ 绝对丰度削减率为89.9%、86.2%(琼氏不动杆菌) 和85.7%、87.2%(枯草芽孢杆菌),抗性基因绝对丰度仅下降不到一个数量级,仍处于较高程度.同时,有研究认为,低剂量氯消毒 ( < 40 mg ·min ·L-1) 促进细菌接合转移[26],在现实环境中,消毒对抗性基因的抑制作用可能更弱.臭氧消毒过程中磺胺类抗性基因的削减规律呈现波动,臭氧消毒浓度为0~6 mg ·L-1时,削减情况呈现波动,对磺胺类抗性基因基本无削减效果 (平均削减率为9.1%),浓度达到8 mg ·L-1时平均削减率升至55.3%,呈现一定下降趋势,可以认为低浓度臭氧消毒对磺胺类抗性基因的削减效果并不明显.比较臭氧消毒和氯消毒对磺胺类抗性基因的削减效果,当枯草芽孢杆菌sul Ⅰ 绝对丰度削减率达到80.0%左右时,氯消毒的CT值为90 mg ·(min ·L)-1,而臭氧消毒的CT值为100 mg ·(min ·L)-1,其他情况下也能分析到相似结论,这与削减抗性细菌的结果截然相反.氯消毒过程中,抗性基因的削减与微生物量减少相关,这表明臭氧消毒中,抗性基因的减少可能不是由于微生物量的减少,抗性细菌减少时抗性基因仍得到一定程度的保存.同时,磺胺类抗性基因丰度和16S rRNA基因丰度的相关性分析表明,两种水样sul Ⅰ 、sul Ⅱ 基因均与16S rRNA基因相关性不显著 (P>0.05).臭氧消毒和紫外消毒中,抗性细菌被杀灭后,抗性基因的行为特征需要进行深入研究.
此外,对两种细菌进行对比发现,两者在氯消毒和紫外消毒过程中的变化规律基本一致,但是琼氏不动杆菌 (革兰氏阴性菌) 中磺胺类抗性基因的削减率高于枯草芽孢杆菌 (革兰氏阳性菌).在3 mg ·L-1氯消毒浓度时,琼氏不动杆菌sul Ⅰ 、sul Ⅱ 基因削减率分别为94.8%和97.4%,而枯草芽孢杆菌sul Ⅰ 、sul Ⅱ 基因削减率为77.8%和83.3%,表明磺胺类抗性基因在不同种类细菌中对氯消毒的耐受性有一定差别,革兰氏阳性菌耐受性强于革兰氏阴性菌,这与抗性细菌的削减情况相同,符合氯消毒削减磺胺类抗性基因主要和微生物量的降低有关的结论.在10 mJ ·cm-2紫外消毒剂量时,琼氏不动杆菌sul Ⅰ 、sul Ⅱ 基因削减率分别为71.4%和60.0%,而枯草芽孢杆菌sul Ⅰ 、sul Ⅱ 基因削减率为37.2%和38.6%,表明紫外消毒中抗性基因的削减仍受到细菌种类的影响.值得注意的是,有研究对污水紫外消毒进行深入研究发现,UV消毒在减小细菌多样性的同时,会提高抗性基因在污水中的相对丰度[27].抗生素抗性细菌和非抗性细菌在紫外消毒过程中的不同表现同样会减少抗性基因的削减率.在臭氧消毒中,两种细菌的削减率变化无明显规律,臭氧消毒可能与细菌种类无明显关系.
2.3 应用DNaseI研究消毒中磺胺类抗性基因削减的机制3种消毒方法均能削减两种目标细菌丰度和抗性基因丰度,但细菌和基因丰度的削减程度相差很多,例如20 mJ ·cm-2紫外消毒下,消毒前后细菌浓度相差5个数量级,而抗性基因的丰度相差仅为0~1个数量级,磺胺类抗性基因丰度仍高达105~106 copies ·mL-1.表明基因在其细菌被杀死后可能仍独立于细胞体外存留,抗生素抗性基因亦可能包含其中,并在特定条件下,又会进入其他细胞中重新表现出耐药性,从而继续传播[28].本研究选择3 mg ·L-1(氯消毒)、10 mJ ·cm-2(紫外消毒)、6 mg ·L-1(臭氧消毒) 剂量消毒后实验水样,进行DNaseI处理,图 5为DNaseI处理前后实验水样中磺胺类抗性基因丰度的变化情况.经过DNaseI处理后,目标细菌磺胺类抗性基因丰度均有明显的下降.其中,臭氧消毒前后磺胺类抗性基因丰度变化最大,例如琼氏不动杆菌氯消毒、紫外消毒水样中sul Ⅰ 基因在DNaseI处理前后比例分别为28.5%和67.5%,而臭氧消毒水样在DNaseI处理后仅剩4.3%,即有95.7%的去除率,明显高于氯消毒和紫外消毒.氯消毒水样中经过DNaseI处理后的去除率基本高于紫外消毒.实验表明,臭氧消毒后,磺胺类抗性基因主要存在于水环境中的游离态DNA.
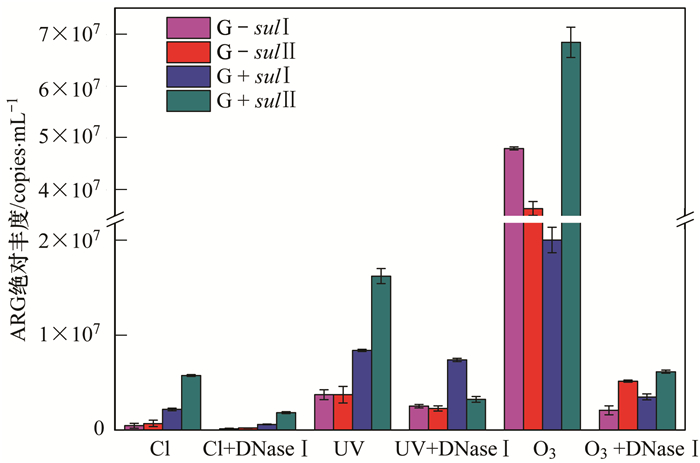
|
图 5 DNaseⅠ处理前后磺胺类抗性基因丰度的变化 Fig. 5 Change of ARGs'abundance before and after disinfection+DNaseⅠtreatment |
对DNaseI处理前后16S rRNA和intⅠ1基因变化分析发现 (见图 6),DNaseI对16S rRNA基因丰度有一定的削减效果 (14.6%~74.0%),总体削减率在50.0%左右.其中在紫外消毒和氯消毒后DNaseI对16S rRNA基因丰度的削减率在42.0%~74.0%,削减率相对于臭氧消毒高.在不同消毒方法处理后DNaseI对intⅠ1基因丰度的削减情况不相同,臭氧消毒后DNaseI对intⅠ1基因丰度削减率最高,氯消毒和紫外消毒后DNaseI对intⅠ1基因丰度削减率低. intⅠ1基因作为1种水平基因转移元件[22],可以认为是抗性基因在游离态DNA中的主要载体之一.结合上文研究,氯消毒过程中磺胺类抗性基因与16S rRNA的丰度显著相关,而游离态DNA中磺胺类抗性基因的削减情况基本与16S rRNA的削减情况一致,这均表明,氯消毒对磺胺类抗性基因的削减主要与减少细菌量有关.在紫外消毒方面,游离态DNA中磺胺类抗性基因的含量不高,而紫外消毒的机制主要是细胞的DNA中的胸腺嘧啶经过光化学的破坏,相邻的嘧啶产生二聚作用.细菌的DNA中多数胸腺嘧啶二聚物的形成阻止了DNA复制和最终导致细胞死亡,达到消毒的目的[10].之前的研究结果同样表明,紫外消毒过程中不同种类细菌所携带抗性基因呈现不同的表现,表明紫外消毒对磺胺类抗性基因的削减主要是直接破坏磺胺类抗性基因,达到削减的效果.
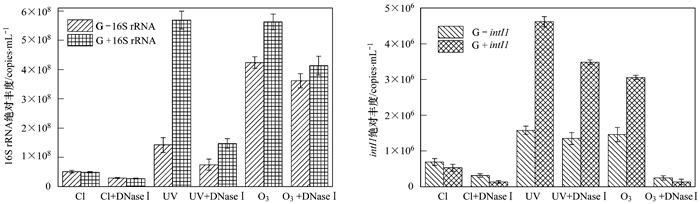
|
G-16S rRNA、G-16S rRNA:琼氏不动杆菌水样中16S rRNA基因;G+intⅠ1、G+intⅠ1:枯草芽孢杆菌水样中intⅠ1基因 图 6 DNaseI处理前后实验水样中16S rRNA和intⅠ1基因 Fig. 6 Change of 16S rRNA's andintⅠ1's abundance before and after disinfection+DNase Ⅰtreatment |
值得关注的是,臭氧消毒中,DNaseI对16S rRNA基因丰度削减率不高 (琼氏不动杆菌水样和枯草芽孢杆菌水样分别为14.6%和25.9%),而对intⅠ1基因丰度的削减相对较高 (琼氏不动杆菌水样和枯草芽孢杆菌水样分别为83.0%和95.5%),这表明游离态DNA中16S rRNA基因含量不高,intⅠ1基因含量高.这与臭氧消毒能有效杀灭目标细菌,而对磺胺类抗性基因削减率不高的实验现象基本符合.目前对臭氧消毒机制观点主要有以下两点:①臭氧能氧化分解细菌内部葡萄糖氧化酶,破坏细胞的核酸,分解DNA、RNA、蛋白质、脂肪和多糖等大分子聚合物,使细菌的物质代谢生长和繁殖过程遭到破坏. ②臭氧能渗透细胞膜组织,侵入细胞膜内作用于外膜脂蛋白和内部的脂多糖,使细胞发生畸变,导致细胞的溶解死亡[23].这表明臭氧消毒的机制可能主要为侵入细胞膜内作用于外膜脂蛋白和内部的脂多糖,使细胞发生畸变,导致细胞的溶解死亡,大量含有抗性基因的游离DNA就散逸到水环境中,仍具有潜在的环境风险.
3 结论(1) 氯消毒、紫外消毒、臭氧消毒对城市污水处理厂尾水中磺胺类抗性细菌有较好的灭菌效果,受到消毒剂量和细菌种类的影响,但对磺胺类抗性基因的控制效果不够理想.
(2) 氯消毒削减磺胺类抗性与细菌量的减少有关,紫外消毒过程中磺胺类抗性基因可能被直接破坏而被削减,而臭氧消毒过程中磺胺类抗性基因随游离态DNA进入到环境中,存在潜在的环境问题,需要引起关注.
| [1] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants:studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445–7450. |
| [2] | 罗义, 周启星. 抗生素抗性基因 (ARGs)——一种新型环境污染物[J]. 环境科学学报, 2008, 28(8): 1499–1505. Luo Y, Zhou Q X. Antibiotic resistance genes (ARGs) as emerging pollutants[J]. Acta Scientiae Circumstantiae, 2008, 28(8): 1499–1505. |
| [3] | Mao D Q, Yu S, Rysz M, et al. Prevalence and proliferation of antibiotic resistance genes in two municipal wastewater treatment plants[J]. Water Research, 2015, 85: 458–466. |
| [4] | Chen H, Zhang M M. Occurrence and removal of antibiotic resistance genes in municipal wastewater and rural domestic sewage treatment systems in eastern China[J]. Environment International, 2013, 55: 9–14. |
| [5] | Oh J, Salcedo D E, Medriano C A, et al. Comparison of different disinfection processes in the effective removal of antibiotic-resistant bacteria and genes[J]. Journal of Environmental Sciences, 2014, 26(6): 1238–1242. DOI: 10.1016/S1001-0742(13)60594-X |
| [6] | 佟娟, 魏源送. 污水处理厂削减耐药菌与抗性基因的研究进展[J]. 环境科学学报, 2012, 32(11): 2650–2659. Tong J, Wei Y S. State-of-the-art removal of antibiotic resistance bacteria (ARB) and antibiotic resistance gene (ARG) in wastewater treatment plants (WWTPs)[J]. Acta Scientiae Circumstantiae, 2012, 32(11): 2650–2659. |
| [7] | Zhang Y Y, Zhuang Y, Geng J J, et al. Inactivation of antibiotic resistance genes in municipal wastewater effluent by chlorination and sequential UV/chlorination disinfection[J]. Science of the Total Environment, 2015, 512-513: 125–132. DOI: 10.1016/j.scitotenv.2015.01.028 |
| [8] | Pruden A, Larsson D G J, Amézquita A, et al. Management options for reducing the release of antibiotics and antibiotic resistance genes to the environment[J]. Environmental Health Perspectives, 2013, 121(8): 878–885. DOI: 10.1289/ehp.1206446 |
| [9] | Dodd M C. Potential impacts of disinfection processes on elimination and deactivation of antibiotic resistance genes during water and wastewater treatment[J]. Journal of Environmental Monitoring, 2012, 14(7): 1754–1771. |
| [10] | McKinney C W, Pruden A. Ultraviolet disinfection of antibiotic resistant bacteria and their antibiotic resistance genes in water and wastewater[J]. Environmental Science & Technology, 2012, 46(24): 13393–13400. |
| [11] | Huang J J, Hu H Y, Wu Y H, et al. Effect of chlorination and ultraviolet disinfection on tetA-mediated tetracycline resistance of Escherichia coli[J]. Chemosphere, 2013, 90(8): 2247–2253. |
| [12] | Anastasi E M, Wohlsen T D, Stratton H M, et al. Survival of Escherichia coli in two sewage treatment plants using UV irradiation and chlorination for disinfection[J]. Water Research, 2013, 47(17): 6670–6679. |
| [13] | Lee O M, Kim H Y, Park W, et al. A comparative study of disinfection efficiency and regrowth control of microorganism in secondary wastewater effluent using UV, ozone, and ionizing irradiation process[J]. Journal of Hazardous Materials, 2015, 295: 201–208. DOI: 10.1016/j.jhazmat.2015.04.016 |
| [14] | Niemeyer J, Gessler F. Determination of free DNA in soils[J]. Journal of Plant Nutrition and Soil Science, 2002, 165(2): 121–124. |
| [15] | Ferro G, Guarino F, Castiglione S, et al. Antibiotic resistance spread potential in urban wastewater effluents disinfected by UV/H2O2 process[J]. Science of the Total Environment, 2016, 560-561: 29–35. DOI: 10.1016/j.scitotenv.2016.04.047 |
| [16] | Romanowski G, Lorenz M G, Wackernagel W, et al. Adsorption of plasmid DNA to mineral surfaces and protection against DNase I[J]. Applied and Environmental Microbiology, 1991, 57(4): 1057–1061. |
| [17] | Pei R T, Kim S C, Carlson K H, et al. Effect of River Landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J]. Water Research, 2006, 40(12): 2427–2435. |
| [18] | 金明兰, 刘凯, 徐莹莹, 等. 污水处理厂中磺胺类抗生素、抗性菌、抗性基因的特性[J]. 环境工程, 2015, 33(11): 1–4. Jin M L, Liu K, Xu Y Y, et al. Characteristics of sulfonamide antibiotics, resistant bacteria, resistance genes in sewage treatment plant[J]. Environmental Engineering, 2015, 33(11): 1–4. |
| [19] | Gao P, Munir M, Xagoraraki I. Correlation of tetracycline and sulfonamide antibiotics with corresponding resistance genes and resistant bacteria in a conventional municipal wastewater treatment plant[J]. Science of the Total Environment, 2012, 421-422: 173–183. DOI: 10.1016/j.scitotenv.2012.01.061 |
| [20] | Luo Y, Mao D Q, Rysz M, et al. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environmental Science & Technology, 2010, 44(19): 7220–7225. |
| [21] | Gaze W H, Zhang L H, Abdouslam N A, et al. Impacts of anthropogenic activity on the ecology of class 1 integrons and integron-associated genes in the environment[J]. The ISME Journal, 2011, 5(8): 1253–1261. DOI: 10.1038/ismej.2011.15 |
| [22] | Goldstein C, Lee M D, Sanchez S, et al. Incidence of class 1 and 2 integrases in clinical and commensal bacteria from livestock, companion animals, and exotics[J]. Antimicrobial Agents and Chemotherapy, 2001, 45(3): 723–726. DOI: 10.1128/AAC.45.3.723-726.2001 |
| [23] | Pak G J, Salcedo D E, Lee H, et al. Comparison of antibiotic resistance removal efficiencies using ozone disinfection under different pH and suspended solids and humic substance concentrations[J]. Environmental Science & Technology, 2016, 50(14): 7590–7600. |
| [24] | Hallmich C, Gehr R. Effect of pre-and post-UV disinfection conditions on photoreactivation of fecal coliforms in wastewater effluents[J]. Water Research, 2010, 44(9): 2885–2893. DOI: 10.1016/j.watres.2010.02.003 |
| [25] | 景明, 王磊. 污水回用中主要病原菌解析及其紫外消毒效应[J]. 环境科学, 2016, 37(2): 622–629. Jing M, Wang L. Analysis of pathogenic bacteria in reclaimed water and impact of UV disinfection on the removal of pathogenic bacteria[J]. Environmental Science, 2016, 37(2): 622–629. |
| [26] | Guo M T, Yuan Q B, Yang J. Distinguishing effects of ultraviolet exposure and chlorination on the horizontal transfer of antibiotic resistance genes in municipal wastewater[J]. Environmental Science & Technology, 2015, 49(9): 5771–5778. |
| [27] | Hu Q, Zhang X X, Jia S Y, et al. Metagenomic insights into ultraviolet disinfection effects on antibiotic resistome in biologically treated wastewater[J]. Water Research, 2016, 101: 309–317. DOI: 10.1016/j.watres.2016.05.092 |
| [28] | 郭美婷, 袁青彬, 杨健. 环境中抗药细菌及其抗药基因的研究进展[J]. 环境科学与技术, 2012, 35(11): 87–92. Guo M T, Yuan Q B, Yang J. Antibiotic resistant bacteria and its antibiotic resistant genes in environment:a review[J]. Environmental Science & Technology, 2012, 35(11): 87–92. |
 2017, Vol. 38
2017, Vol. 38


