2. 中国环境科学研究院环境基准与风险评估国家重点实验室, 北京 100012;
3. 中国环境科学研究院流域水生态保护技术研究室, 北京 100012
2. State Key Laboratory of Environmental Criteria and Risk Assessment, Chinese Research Academy of Environmental Sciences, Beijing 100012, China;
3. Laboratory of Riverine Ecological Conservation & Technology, Chinese Research Academy of Environmental Sciences, Beijing 100012, China
辽河流域 (东经116°30′~125°47′,北纬38°43′~45°00′之间) 位于我国东北地区南部,流经吉林、辽宁、河北省和内蒙古自治区的部分市域,全流域面积21.9×104 km2.该流域由两个水系所组成:一为东、西辽河,于福德店汇流后为辽河干流,经双台子河由盘山入海;另一为浑河、太子河,于三岔河汇合后经大辽河由营口入海.真核浮游藻类在辽河流域分布极为广泛,大多为光合自养的单细胞藻类,其群落的特定组成是反映诸如营养盐输入、全球气候变化以及人类活动影响等环境变化的敏感指标[1].国内曾有人研究了辽河流域真核浮游藻类的群落分布特征及其对水质的评价[2~7],这些研究均需要借助显微镜观察藻细胞的形态学特征以确定种类.对真核浮游藻类的种类鉴定需要非常丰富的分类学经验,基于形态特征精确鉴定到种甚至需要借助扫描电子显微镜等高分辨率的仪器.在研究中由于鉴定错误而误导生态评价结果的状况也时有发生[8].
基于高通量测序技术的DNA宏条形码研究使得评估环境样品的生物多样性及复杂的群落结构成为可能[9].目前用于真核浮游藻类的DNA条形码基因包括核基因ITS、18S rRNA基因和28S rRNA基因,叶绿体rbcL基因,线粒体cob基因等[10~13].其中一些分子标记已被用于DNA宏条形码研究,例如18S rRNA基因的V4区或V9区,28S rRNA基因的D2/D3区,rbcL基因等[14~16].Amaral-Zettler等[17]通过设计两对引物扩增出18S r RNA基因的V9可变区,利用高通量测序技术成功得到了海洋原生生物的组成结构,验证了高通量技术在生物群落组成中的可行性.Zimmermann等[18]采用形态学和18S rRNA V4区的宏条形码技术,对奥德拉河和尼萨河的源头到河口不同采样点的硅藻物种多样性和丰度进行了比较,发现宏条形码技术更能反映环境样品中的生物多样性,并提出了环境样品中物种鉴别的技术流程.
本研究采集了辽河干流12个采样点的表层水样,对水环境样品的18S rRNA基因V4可变区进行高通量测序,注释得到的OTUs与光学显微镜观察的结果进行比较分析.本研究的主要目标是:①比较高通量测序技术与显微镜观察在研究真核浮游藻类多样性和群落结构特征方面的异同;②评估18S rRNA基因的V4可变区在真核浮游藻类不同分类阶元进行系统发育构建的有效性;③揭示影响该区域真核浮游藻类群落结构特征的重要环境因子.本研究结果可以为在辽河流域开展水环境中浮游植物监测以及水生态系统健康评价提供参考和借鉴.
1 材料与方法 1.1 样品采集研究组于2015年9月期间在辽河干流采集水样,共采集12个点位样品,如图 1所示,表层水采自水体0.5m深.水温 (T)、总溶解固体 (TDS) 和电导率 (EC) 数据由YSI水质分析仪 (Horiba) 测量.在各样点采集的水样 (2 L),置于低温保温箱中 (4℃),于48 h内带回实验室,根据标准方法[19],测定以下水化指标:氨氮 (NH4+-N)、活性磷 (PO43--P)、硝态氮 (NO3-)、总磷 (TP)、总氮 (TN) 和总碳 (TC).浮游真核藻类样品的采集是在每个采样点的3个不同生境的等体积水混合,使用纱绢对所采集水样进行预过滤除去浮游动物和较大颗粒物质,用0.45 μm孔径的聚碳酸酯膜 (Millipore公司) 过滤采集的水样,过滤时保持压力小于0.03 MPa,过滤时间限制在2 h内,过滤体积为2 L;过滤好的膜装在冻存管中并液氮保存,送实验室进一步处理;光学显微镜观察的真核浮游藻类的种类和数量来自2013年9月的采集资料.
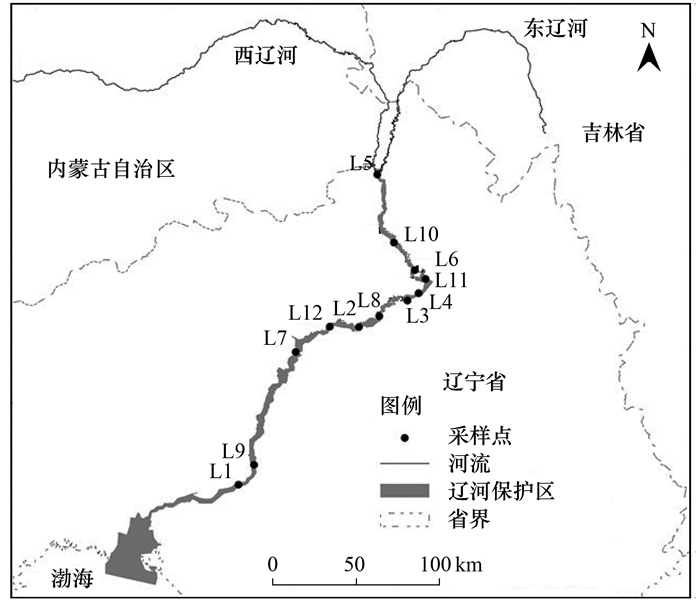
|
图 1 辽河干流水样采集点分布示意 Fig. 1 Location map of sampling sites in Liaohe River |
根据Yuan等[20]的方法提取环境样品总DNA,结合使用酶、DNA裂解液和FastPrep-24细胞破碎仪的方法裂解和破碎样品.选取euk-F:5′-GGCAAG TCTGGTGCCAG-3′,euk-R:5′-GACTACGACGGTATC TRATCRTCTTCG-3′作为真核浮游藻类第一轮PCR扩增的引物[21].PCR的反应条件为:95℃ 2 min,95℃ 30 s,55℃ 30 s,72℃ 30 s,共23个循环,最后72℃终延伸7 min.在设计第二轮PCR引物时,在引物euk-F的5′端加上10 bp的区分不同样本的标签,并在正反向引物的5′端引入焦磷酸测序的正反向引物.使用DNA胶回收试剂盒 (Takara公司) 对PCR产物进行纯化,纯化后的PCR产物使用dsDNA定量检测试剂盒 (Promega公司) 进行定量,通过QC-PCR及Agilent 2100生物分析仪分析PCR产物的质量.按照等摩尔比将不同样品的PCR产物混合并进行乳液PCR,构建高通量测序文库.使用Roche 454高通量基因测序系统GS FLX+(厦门大学海洋基因组学平台) 对文库进行测序.
1.3 数据处理生物信息学分析应用QIIME (quantitative insights into microbial ecology) 软件对获得的原始序列过滤分析,存在简并碱基、低质量得分、嵌合体以及短于150 bp的噪声序列均被剔除.然后用QIIME软件对高质量序列在相似性≥97%(默认为种水平的相似度) 的划分标准上进行最佳分类单元 (operational taxonomic unit, OTU) 的划分.根据NCBI nr数据库中的18S rRNA基因序列,通过BLAST,在80%的相似性阈值对得到的OTU的代表性序列进行分类学上的注释.
使用Mothur软件分析真核浮游藻类群落的α多样性指数 (Chao1指数、Ace指数、Shannon-Wiener指数和Simpson指数)[22~24].本研究得到的OTUs的代表性序列,结合GenBank (http://www.ncbi.nlm.nih.gov/nuccore) 中下载的代表物种序列,使用ClustalX 2.0对序列进行比对后,运用MEGA 7.0软件中的临接法 (neighbor-joining,NJ),结合iTOL (interactive tree of life) 程序[25]构建系统发育树.使用Bootstrap法 (重复1 000次) 对系统发育树进行评估.
统计学分析对不同采样点的真核浮游藻类相对多度数据进行除趋势对应分析 (detrended correspondence analysis,DCA),发现矩阵第一轴的梯度长度 < 2,即不同点位真核浮游藻类群落结构特征与水环境因子具有线性相关关系.选择冗余分析 (redundancy analysis,RDA) 计算其相关性,通过Canoco5.0软件和蒙特卡罗筛选检验分析藻类群落结构与水环境因子的相关性.
2 结果与分析 2.1 测序数据分析及多样性指数不同样品的18S rRNA基因V4可变区的第一轮PCR扩增产物如图 2所示,所扩增的基因片段条带单一,长度约为500 bp,并且不同采样点的电泳条带的亮度较为一致.
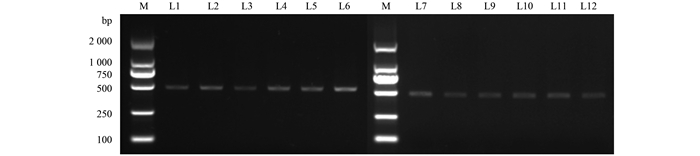
|
M:Marker 图 2 不同样品中18S rRNA基因V4可变区第一轮PCR产物电泳图 Fig. 2 Agarose gel electrophoresis of PCR products for 18S rRNA gene V4 variable region in different samples |
测序共获得18S rRNA基因V4可变区的785 554条原始序列,对其过滤分析后,共得到167 901个高质量序列,不同样品的序列数为4 590~49 841.将序列进行随机抽样,所抽到的序列数代表的OTU数目构建稀释性曲线,如图 3所示,各个样本的稀释性曲线趋于平缓,表明测序数据趋于饱和,测序深度已足够反映样品中的所有OTU信息[26].所有样本中有11.9%的序列不能注释,剩余的147 959条序列注释为424种OTU,去掉代表原生动物、真菌和真核生物的不能注释的OTUs,得到注释为134种真核浮游藻类的OTU.
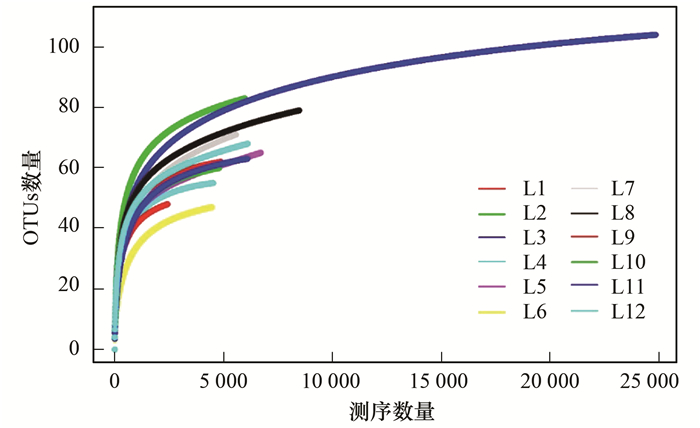
|
图 3 不同样本中OTUs稀释曲线 Fig. 3 Rarefaction curves of OTUs in different samples |
对各采样点的丰富度和多样性指数进行分析,如表 1所示.Shannon-Wiener指数和Simpson指数表示个体分布的均匀度,各物种之间个体分配越均匀,值就越大,如果每一个体都属于不同的物种,则指数就最高,本研究中指数越高表示文库中OTUs分布越不均匀,Shannon-Wiener多样性指数的变化范围是1.87~2.94,Simpson指数的范围是0.740 9~0.891 8,表明各样品间OTUs数目的分布差异不大;Chao1指数和ACE指数是估计群落中含OTU数目的指数,表示物种的丰富度,值越大代表物种总数越多.ACE指数的变化范围是46~105,Chao1指数的变化范围是46.6~106.25,Chao1指数和ACE指数最小值的点位均为L6,该点位出现的种类数为47种,也为各采样点最小,最大值出现的点位分别为L5和L3,种类数分别为65种和104种,点位L3和L5的藻类种类数多于L6点.
|
|
表 1 不同采样点真核浮游藻类的多样性指数 Table 1 Diversity indices of eukaryotic planktonic algae in different sampling sites |
2.2 真核浮游藻类的多样性和群落结构分析
对所得到的OTUs序列进行物种注释后发现,12个采样点的真核浮游藻类多样性丰富,涵盖了7门134种,不等鞭毛门 (Heterokontophyta)34种,占总种类数的比例为25.4%,其中金藻纲 (Chrysophyceae)23种;硅藻门 (Bacillariophyta)24种,比例为17.9%;绿藻门 (Chlorophyta)24种,比例为17.9%;隐藻门 (Cryptophyta)18种,比例为13.4%;甲藻门 (Pyrroptata)17种,比例为12.7%;轮藻门 (Charophyta)13种,比例为9.7%;定鞭藻门 (Haptophyta)4种,比例为2.9%.高通量测序结果中,12个采样点的占优势类群的核心OTU略有不同,其中采样点L4的核心OTU是Chlorophyta Trebouriophyceae other,采样点L3和L8的核心OTU是Chlorophyta Chlorophyceae other,其他点位的核心OTU均为硅藻纲的Chaetoceros sp..
显微镜观察结果中的裸藻门 (Euglenophyta) 在高通量测序结果中未观察到.显微镜观察共鉴定出真核浮游藻类4门63种,其中硅藻门32种,占总种类数的比例为50.8%;绿藻门23种,比例为36.5%;裸藻门7种,比例为11.1%;甲藻门1种,比例均为1.6%.镜检结果中,采样点L1、L2、L4、L5、L6、L9和L12出现数量最多的物种分别是齿牙栅藻Scenedesmus denticulatus、梅尼小环藻Cyclotella meneghiniana、小片菱形藻Nitzschia frustulum、小环藻Cyclotella sp.、针形菱形藻Nitzschia acicularis、韦氏藻Westellopsis sp.和集星藻Actinastrum hantzschii.采样点L3、L7和L10数量最多的种类是颗粒直链藻Melosira granulata,采样点L8和L11数量最多的种类是四尾栅藻Scenedesmus quadricauda.显微镜观察结果中丰度最高的5个物种是颗粒直链藻Melosira granulata、梅尼小环藻Cyclotella memeghiniana、四尾栅藻Scenedesmus quadricauda、针形菱形藻Nitzschia acicularis和集星藻Actinastrum hantzschii(如图 4).

|
A.颗粒直链藻Melosira granulata; B.梅尼小环藻Cyclotella memeghiniana; C.四尾栅藻Scenedesmus quadricauda; D.集星藻Actinastrum hantzschii; E.针形菱形藻Nitzschia acicularis 图 4 显微镜观察藻类照片 Fig. 4 Microscopic images of algae |
浮游硅藻、浮游绿藻、浮游甲藻和不等鞭毛类均是淡水生态系统中的重要初级生产力,12个采样点的高通量测序结果中可能包含的浮游硅藻种类有:Synura petersenii、Mallomonassp.、Chaetoceros sp.、Skeletonemasp.、Thalassiosira sp.、Aulacoseira ambigua及其近缘种等.浮游绿藻种类有:Tetraselmis sp.、Carteriasp.、Chlamydomonas sp.、Chloromonassp.、Desmodesmussp.、Golenkiniasp.、Neochlorosarcinasp.、Pseudopediastrum boryanum、Radiococcussp.、Wislouchiella planctonica、Nephroselmis sp.、Pedinomonassp.、Amphikrikossp.及其近缘种等.浮游甲藻的种类有:Gymnodiniumsp.、Gyrodinium sp.、Alexandriumsp.、Ceratiumsp.、Lingulodinium polyedrum.、Gymnodinium sp.、Diplopsalis lenticula、Peridiniumsp.及其近缘种等.高通量测序结果注释得到而显微镜未观察到的轮藻门、隐藻门、定鞭藻门和金藻纲的信息如表 2所示.
|
|
表 2 轮藻门、隐藻门、定鞭藻门、金藻纲的物种注释 Table 2 Species annotation of Charophyta, Cryptophyta, Haptophyta and Chrysophyceae |
高通量测序结果和显微镜观察结果比较发现,这两种方法的结果在群落组成上存在相同点,如图 5所示,均以硅藻门和绿藻门为优势类群.高通量测序结果发现,硅藻门和绿藻门的丰度在比例上分别占28.70%和46.20%,甲藻门、轮藻门、隐藻门、定鞭藻门、不等鞭毛门和甲藻门的丰度在比例上分别占0.48%、0.05%、17.51%、3.93%和3.12%.而显微镜观察结果中的硅藻门和绿藻门的丰度在比例上分别占51.54%和39.90%,裸藻门和甲藻门的丰度在比例上分别占7.59%和0.97%.
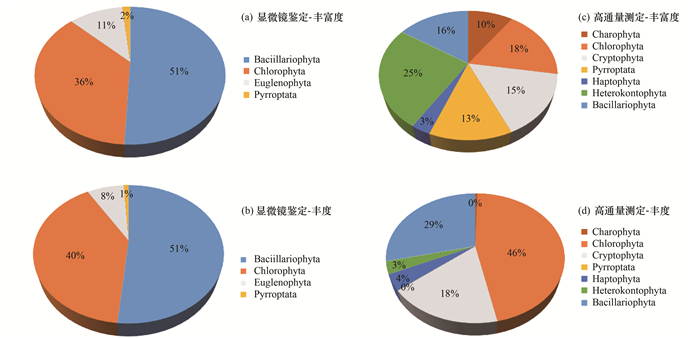
|
图 5 基于高通量测序和显微镜观察比较真核浮游藻类多样性及群落组成 Fig. 5 Comparison of eukaryotic planktonic algae diversity and community composition based on high-throughput sequencing and microscopic observation |
采用邻接法,将高通量测序结果注释得到的OTUs代表性序列以及从NCBI下载的真核浮游藻类18S rRNA基因序列构建系统发育树,结果如图 6显示,18S rRNA基因的V4区可以在门这一较高分类阶元对隐藻门和甲藻门进行聚类,但是对于其他5个门而言,18S rRNA基因的V4区序列并未按照物种的实际分类情况进行聚类.
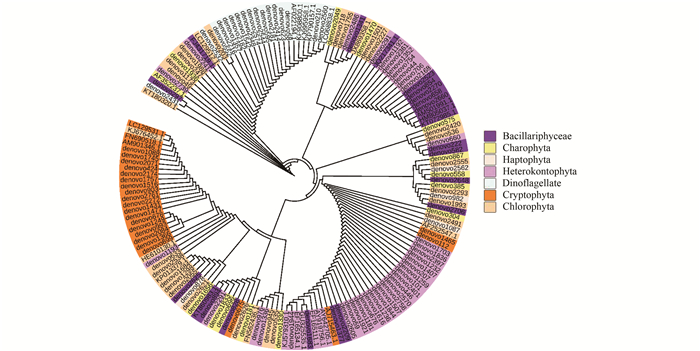
|
图 6 基于邻接法构建的真核浮游藻类的系统发育分析 Fig. 6 Phylogenetic tree for the eukaryotic planktonic algae based on Neighbor-Joining method of 18S rRNA gene |
利用高通量测序结果注释得到的隐藻门的OTUs以及从NCBI下载的隐藻门18S rRNA基因序列,采用邻接法进行系统发育分析,结果如图 7所示,可以在较低的分类阶元分别对隐藻门的Goniomonas属、Cryptomonas属、Chroomonas属、Rhodomonas属及Katablepharid属进行聚类和区分.由表示不同OTU丰度情况的热图 (图 7) 所示,deovo2071(注释为未培养的katablepharid) 和denovo1745(注释为Cryptophyceae Other1) 的18S rRNA基因的拷贝数远高于隐藻门中其他种类该基因的拷贝数.
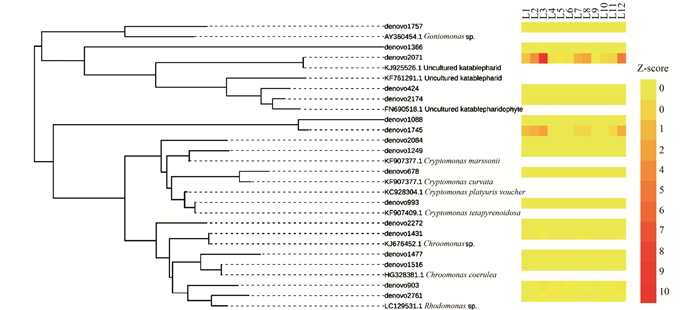
|
图 7 隐藻门的系统发育分析及各OTU的丰度情况 Fig. 7 Phylogenetic tree for Cryptophyta and the abundance of various OTUs |
采用邻接法,将高通量测序结果注释得到的甲藻门的OTUs代表性序列及从NCBI下载的甲藻门18S rRNA基因序列进行系统发育树构建,结果如图 8所示,可以将部分OTUs在科或属水平上进行区分和聚类,例如,Suessiaceae科、Perdinium属和Alexandrium属各自明显聚为一支.由表示不同OTU丰度情况的热图 (图 8) 所示,deovo58(注释为Suessiaceae other)、denovo2181(注释为Peridiniales Other)、denovo2544(注释为Gymnodinium Other) 和denovo2414(注释为Symbiodinium Other) 的18S rRNA基因的拷贝数,远高于甲藻门中其他种类该基因的拷贝数.
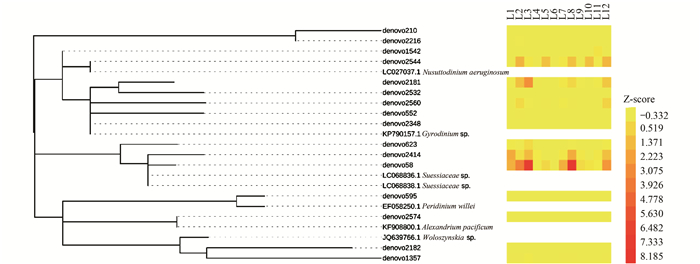
|
图 8 甲藻门的系统发育分析及各OTU的丰度情况 Fig. 8 Phylogenetic tree for Pyrroptata and the abundance of various OTUs |
在门这一分类阶元并不能将硅藻门的OTUs聚为一支,硅藻门的OTUs在图 6所示的系统发育树上较为分散.鉴于18S rRNA基因V4区曾用于系统发育构建,本研究将高通量测序结果注释得到的硅藻门的OTUs以及从NCBI下载的硅藻门18S rRNA基因序列,采用邻接法构建系统发育树,其在纲这一分类阶元即不能有效区分和聚类.总之,18S rRNA基因的V4区可以将本研究中的隐藻门在属水平上有效聚类和区分,甲藻门可以在部分科或属水平聚类和区分,尚存在误差.
2.4 群落结构特征的影响指标用CANOCO 5.0软件分析不同真核藻类群落结构变化与氨氮 (NH4+-N)、活性磷 (PO43--P)、硝态氮 (NO3-)、电导率 (EC)、总磷 (TP)、总氮 (TN)、总碳 (TC)、水温 (T) 和总溶解固体 (TDS) 共9个环境因子的相关性,如图 9所示.DCA的结果显示样品矩阵第一轴的梯度长度 < 2, 因此本研究中的不同真核浮游藻类多样性与环境因子具有线性相关关系,选择冗余分析 (RDA) 计算其相关性,通过软件和蒙特卡罗筛选检验发现真核浮游藻类的群落结构变化和活性磷 (PO43--P)、氨氮 (NH4+-N) 及硝态氮 (NO3-) 的浓度显著相关 (P < 0.05).前两轴的特征值分别为0.247 8和0.175 4,共解释了42.32%真核浮游藻类的多样性差异,活性磷是最主要的影响因子.因而,活性磷、氨氮和硝态氮是影响辽河干流真核浮游藻类群落变化的主要因素.
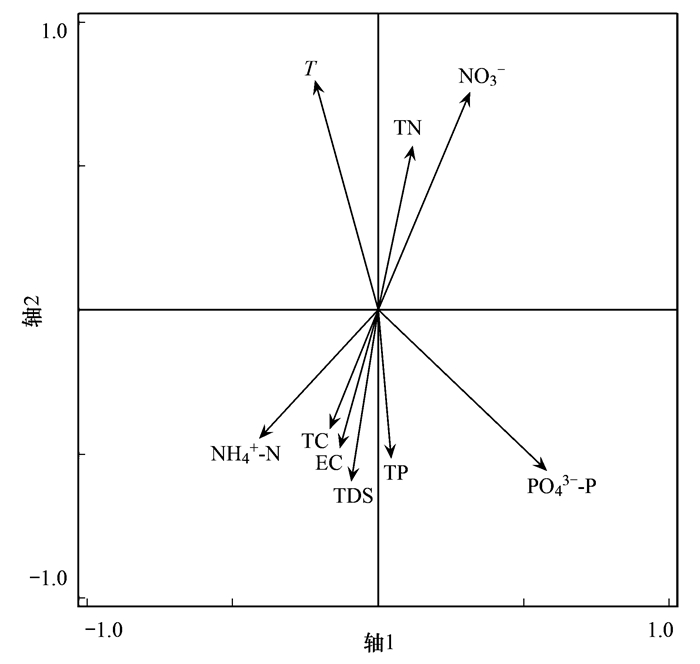
|
图 9 浮游藻类群落与水环境因子的RDA分析 Fig. 9 Redundancy analysis of species-environmental factors relationship |
环境样品的高通量测序技术具备检测稀有的、个体小且易碎物种的潜力[27],其对生物多样性监测和量化已经被证明是可行的[24, 28].目前辽河流域浮游真核藻类的研究主要建立在光学显微镜观察鉴定的基础上[2~7],本研究采用高通量测序技术对辽河干流的真核浮游藻类进行多样性和群落结构组成的研究,并与光学显微镜观察的结果进行比较,发现高通量测序技术对辽河真核浮游藻类多样性有极高的还原能力,得出18S rRNA基因的V4可变区对隐藻门和甲藻门的部分科/属进行系统发育构建的有效性,揭示出影响该区域真核浮游藻类群落变化的重要环境因子为氨氮、活性磷和硝态氮.
3.1 高通量测序技术对辽河真核浮游藻类多样性的揭示18S rRNA基因全长1 787~1 813 bp,由能反映物种间亲缘关系的保守区和能体现物种间差异的可变区交替排列组成,其中可变区序列总长678~699bp.当前用做真核浮游藻类宏条形码的18S rRNA基因主要集中在使用V4区和V9区这两个可变区,它们所得出的结果也存在差异.相比于18S rRNA基因V9区 (~150bp),18S rRNA基因V4区序列较长,可以使分类鉴定更加可靠,系统发育分析也较为严格[17].本研究采用高通量测序技术,消除了对微型真核藻类分离的依赖,共得到167 901个高质量序列,利用GenBank中的18S rRNA基因序列还原出环境样本中134个OTUs.无论对OTU的物种注释还是系统发育分析都表明,高通量测序技术弥补了显微镜观察研究中丢失的大量物种信息,本研究提高了对真核浮游藻类监测的能力.由于真核浮游藻类的种类极为广泛,并且数据库中缺乏某些种类的条形码基因序列,很难对环境序列准确定位[29].当前利用高通量测序对它们的研究主要注释到科或者属,因而本研究结果中只有14.7%的真核浮游藻类OTU可以被注释到种,62.2%可以被注释到属.
3.2 辽河真核浮游藻类不同类群的丰度差异本研究通过高通量测序和光学显微镜观察发现,无论从种类多样性比例,还是丰度比例,这两种方法的结果类似,得出辽河干流浮游真核藻类的优势群落是硅藻门和绿藻门.李庆南等[2]于2011年5月和8月对辽河、太子河水系的浮游植物进行了野外调查,共鉴定出浮游植物195种,其中硅藻门种类最多,有97种; 绿藻门次之,共鉴定出55种;李芳芳等[3]在2010年对大辽河水系浮游植物群落结构和水质状况进行了调查,共鉴定出浮游植物5门124种或变种,其中以绿藻和硅藻为主,优势物种与本研究结果一致.本研究共鉴定出真核浮游藻类7门134种,其中轮藻门、隐藻门、定鞭藻门和金藻纲,是辽河干流相同采样点从未采到过,可见本方法可以监测到显微镜镜检方法检测不到的生物物种,弥补光学显微镜观察结果的不足.光学显微镜观察结果中的裸藻门 (Euglenophyta) 在高通量测序结果中未找到,这可能与18S rRNA基因V4可变区引物的特异性有关,需要结合其他的条形码基因弥补丢失的信息.至于隐藻门中deovo2071和denovo1745,甲藻门中deovo58、denovo2181、denovo2544和denovo2414的18S rRNA基因较高的拷贝数,可能在一定程度上反映了这些种类较高的丰度,还需要进一步研究证实.各个采样点占优势类群的OTU在高通量测序结果和显微镜观察结果中是不同的,这可能是由于所选择分子标记的普适性不足,或者许多本地种的18S rRNA基因序列在NCBI数据库中是不存在的,这也反映出构建本土淡水水生态系统中浮游植物的DNA条形码数据库的必要性.
3.3 18S rRNA基因V4区对隐藻门和甲藻门进行系统发育构建的有效性本研究通过高通量测序技术可以基本还原亲缘关系较远的各类真核浮游藻类的多样性.本研究中使用的18S rRNA的引物可有效地扩增和区分复杂群落的不同的种类.特别针对隐藻门和甲藻门,在系统发育关系上不仅能将它们各自在较高分类阶元上聚在一起,还具有一定的属区分能力.目前多数研究将18S rRNA基因的V4可变区用作硅藻的宏条形码基因,例如,Zimmermann等[18]研究发现,对18S rRNA基因V4区的高通量测序能有效区分硅藻的不同种,包括区分Sellaphora属中的混合种;Luddington等[30]用18S rRNA基因的V4可变区对硅藻纲3个目进行研究,分析了76种硅藻的272个序列,验证了18S rRNA基因V4区作为硅藻条形码基因的可靠性.本研究中通过重建系统发生关系验证了18S rRNA基因对隐藻门和甲藻门内的部分类群的区分具有更好的分辨力,但是对其他门类的分辨力较低,因此接下来采用多基因宏条形码技术可能是一个更合理的方案.
3.4 水环境因子对真核浮游藻类群落结构的影响藻类是河流的初级生产者,进行光合作用时需要氮、磷作为基础的营养元素.相关研究发现,河流中氮和磷的含量对着生藻类群落有着显著的影响:孟睿等[31]在2011年调查了长江中下游草型湖泊浮游植物群落,并对藻类群落结构特征和水环境因子进行分析,结果表明,总磷是影响长江中下游草型湖泊藻类群落结构的主要影响因子;苟婷等[32]为了解贝江浮游藻类的分布特征与水质状况,于2013年的枯水期和丰水期对浮游藻类群落结构的时空动态进行调查和分析,RDA结果显示,显著影响浮游藻类群落结构的环境因子是氮磷营养盐.这一现象在本研究中得到了验证,本研究表明 (NH4+-N)、活性磷 (PO43--P)、(NO3-) 是影响辽河浮游藻类群落结构的重要影响因子,3个环境因子总共解释了总体变量的42.32%;与殷旭旺等[6, 7]2011年和2013在辽河流域调查得到的结果一致.
本研究结果表明,运用基于18S rRNA基因的高通量技术揭示出的辽河环境样本中真核浮游藻类的多样性及群落组成,其结果可弥补光学显微镜观察结果的不足,在生物监测和水质评价方面有巨大的应用潜力.当然,高通量测序技术在分析环境样品中生物群落组成时仍然存在一些不足,例如,真核浮游藻类细胞内的SSU rRNA基因的拷贝数在不同物种中的变化很大[33],需要建立基因序列拷贝数与生物量之间的关系;PCR扩增的引物偏好性也会导致不同种类的丰度不同.将来,建立针对辽河流域真核浮游藻类的条形码基因和标准操作流程,根据自己实验室已有研究的数据和NCBI等数据库的数据,采用高通量测序技术对条形码基因进行测序,从而在实现快速评估该区域的生物多样性、群落结构和水质评价中具有重要的意义.
4 结论(1) 在对辽河干流环境样本的浮游真核藻类研究中,高通量测序技术和光学显微镜观察的丰富度和丰度的结果类似,优势群落均是硅藻门和绿藻门,并且高通量测序技术弥补了光学显微镜观察中丢失的种类信息.
(2) 通过构建系统发育树可以在门分类阶元对隐藻门和甲藻门聚类.18S rRNA基因V4可变区对隐藻门和甲藻门内的部分类群的区分具有较好的分辨力.
(3) 影响辽河干流真核浮游藻类群落结构的主要环境因子是氨氮、活性磷和硝态氮.
致谢: 感谢厦门大学近海海洋环境科学国家重点实验室林森杰教授与李凌高级工程师的实验指导与帮助.| [1] | Eiler A, Drakare S, Bertilsson S, et al. Unveiling distribution patterns of freshwater phytoplankton by a next generation sequencing based approach[J]. PLoS One, 2013, 8(1): e53516. DOI: 10.1371/journal.pone.0053516 |
| [2] | 李庆南, 赵文, 殷旭旺, 等. 辽河太子河水系的浮游植物种类多样性研究[J]. 大连海洋大学学报, 2011, 26(4): 322–327. Li Q N, Zhao W, Yin X W, et al. Phytoplankton diversity in Taizi River tributary in Liao River[J]. Journal of Dalian Fisheries University, 2011, 26(4): 322–327. |
| [3] | 李芳芳, 董芳, 段梦, 等. 大辽河水系夏季浮游植物群落结构特征及水质评价[J]. 生态学杂志, 2011, 30(11): 2489–2496. Li F F, Dong F, Duan M, et al. Phytoplankton community structure and water quality of Daliaohe river system in summer[J]. Chinese Journal of Ecology, 2011, 30(11): 2489–2496. |
| [4] | 耿文华, 陈继淼, 冯剑丰, 等. 辽河浮游植物群落及生物多样性基准验证[J]. 中国环境科学, 2014, 34(1): 239–245. Geng W H, Chen J M, Feng J F, et al. Phytoplankton community and validation of phytoplankton biodiversity criteria in Liaohe River[J]. China Environmental Science, 2014, 34(1): 239–245. |
| [5] | 王宏伟, 陈莹, 张晓明, 等. 辽河太子河流域藻类植物群落结构及其季节变化[J]. 湖泊科学, 2013, 25(6): 936–942. Wang H W, Chen Y, Zhang X M, et al. The algal community structure and seasonal variation in Taizi River catchment, Liaohe River basin[J]. Lake Sciences, 2013, 25(6): 936–942. DOI: 10.18307/2013.0618 |
| [6] | 殷旭旺, 张远, 渠晓东, 等. 浑河水系着生藻类的群落结构与生物完整性[J]. 应用生态学报, 2011, 22(10): 2732–2740. Yin X W, Zhang Y, Qu X D, et al. Community structure and biological integrity of periphyton in Hunhe River water system of Liaoning Province, Northeast China[J]. Chinese Journal of Applied Ecology, 2011, 22(10): 2732–2740. |
| [7] | 殷旭旺, 张远, 渠晓东, 等. 太子河着生藻类群落结构空间分布特征[J]. 环境科学研究, 2013, 26(5): 502–508. Yin X W, Zhang Y, Qu X D, et al. Spatial community structure of periphyton assemblages in the Taizihe River Basin[J]. Research of Environmental Sciences, 2013, 26(5): 502–508. |
| [8] | Morales E A, Siver P A, Trainor F R. Identification of diatoms (Bacillariophyceae) during ecological assessments:comparison between light microscopy and scanning electron microscopy techniques[J]. Proceedings of the Academy of Natural Sciences of Philadelphia, 2009, 151: 95–103. |
| [9] | Zhan A, Hulák M, Sylvester F, et al. High sensitivity of 454 pyrosequencing for detection of rare species in aquatic communities[J]. Methods in Ecology and Evolution, 2013, 4(6): 558–565. DOI: 10.1111/2041-210X.12037 |
| [10] | Ellegaard M, Godhe A, Härnström K, et al. The species concept in a marine diatom:LSU rDNA-based phylogenetic differentiation in Skeletonema marinoi/dohrnii (Bacillariophyceae) is not reflected in morphology[J]. Phycologia, 2008, 47(2): 156–167. DOI: 10.2216/07-09.1 |
| [11] | Wayne L R, Vandersea M W, Kibler S R, et al. Recognizing dinoflagellate species using ITS rDNA sequences[J]. Journal of Phycology, 2007, 43(2): 344–355. DOI: 10.1111/jpy.2007.43.issue-2 |
| [12] | Lin S J, Zhang H, Hou Y B, et al. High-level diversity of dinoflagellates in the natural environment, revealed by assessment of mitochondrial cox1 and cob genes for dinoflagellate DNA barcoding[J]. Applied and Environmental Microbiology, 2009, 75(5): 1279–1290. DOI: 10.1128/AEM.01578-08 |
| [13] | Stern R F, Andersen R A, Jameson I, et al. Evaluating the ribosomal internal transcribed spacer (ITS) as a candidate dinoflagellate barcode marker[J]. PLoS One, 2011, 7(8): e42780. |
| [14] | de Vargas C, Audic S, Henry N, et al. Eukaryotic plankton diversity in the sunlit ocean[J]. Science, 2015, 348(6237): 1261605. DOI: 10.1126/science.1261605 |
| [15] | Hamsher S E, Evans K M, Mann D G, et al. Barcoding diatoms:exploring alternatives to COI-5P[J]. Protist, 2011, 162(3): 405–422. DOI: 10.1016/j.protis.2010.09.005 |
| [16] | Samanta B, Bhadury P. A comprehensive framework for functional diversity patterns of marine chromophytic phytoplankton using rbcL phylogeny[J]. Scientific Reports, 2016, 6: 20783. DOI: 10.1038/srep20783 |
| [17] | Amaral-Zettler L A, McCliment E A, Ducklow H W, et al. A method for studying protistan diversity using massively parallel sequencing of V9 hypervariable regions of small-subunit ribosomal RNA genes[J]. PLoS One, 2009, 4(7): e6372. DOI: 10.1371/journal.pone.0006372 |
| [18] | Zimmermann J, Glöckner G, Jahn R, et al. Metabarcoding vs. morphological identification to assess diatom diversity in environmental studies[J]. Molecular Ecology Resource, 2015, 15(3): 526–542. DOI: 10.1111/1755-0998.12336 |
| [19] | 国家环境保护总局. 水和废水监测分析方法[M]. ((第四版)). 北京: 中国环境科学出版社, 2002. |
| [20] | Yuan J, Li M Z, Lin S J. An improved DNA extraction method for efficient and quantitative recovery of phytoplankton diversity in natural assemblages[J]. PLoS One, 2014, 10(7): e0133060. |
| [21] | Bråte J, Logares R, Berney C, et al. Freshwater Perkinsea and marine-freshwater colonizations revealed by pyrosequencing and phylogeny of environmental rDNA[J]. The ISME Journal, 2010, 4(9): 1144–1153. DOI: 10.1038/ismej.2010.39 |
| [22] | Wang Q, Garrity G M, Tiedje J M, et al. Naïve Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16): 5261–5267. DOI: 10.1128/AEM.00062-07 |
| [23] | DeSantis T Z, Hugenholtz P, Larsen N, et al. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J]. Applied and Environmental Microbiology, 2006, 72(7): 5069–5072. DOI: 10.1128/AEM.03006-05 |
| [24] | Sogin M L, Morrison H G, Huber J A, et al. Microbial diversity in the deep sea and the underexplored "rare biosphere"[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(32): 12115–12120. DOI: 10.1073/pnas.0605127103 |
| [25] | Letunic I, Bork P. Interactive tree of life (iTOL):an online tool for phylogenetic tree display and annotation[J]. Bioinformatics, 2007, 23(1): 127–128. DOI: 10.1093/bioinformatics/btl529 |
| [26] | 田地, 马欣, 李玉娥, 等. 利用高通量测序对封存CO2泄漏情景下土壤细菌的研究[J]. 环境科学, 2013, 34(10): 4096–4104. Tian D, Ma X, Li Y E, et al. Research on soil bacteria under the impact of sealed CO2 leakage by high-throughput sequencing technology[J]. Environmental Science, 2013, 34(10): 4096–4104. |
| [27] | Hajibabaei M, Shokralla S, Zhou X, et al. Environmental barcoding:a next-generation sequencing approach for biomonitoring applications using river benthos[J]. PLoS One, 2011, 6(4): e17497. DOI: 10.1371/journal.pone.0017497 |
| [28] | Meusnier I, Singer G A, Landry J F, et al. A universal DNA mini-barcode for biodiversity analysis[J]. BMC Genomics, 2008, 9: 214. DOI: 10.1186/1471-2164-9-214 |
| [29] | 谢薇薇, 龚伊, 王志伟, 等. 利用流式细胞仪分选技术研究湖泊真核超微藻的遗传多样性[J]. 环境科学, 2013, 34(4): 1485–1491. Xie W W, Gong Y, Wang Z W, et al. Use of flow cytometric sorting to assess the diversity of eukaryotic picophytoplankton of lakes[J]. Environmental Science, 2013, 34(4): 1485–1491. |
| [30] | Luddington I A, Kaczmarska I, Lovejoy C. Distance and character-based evaluation of the V4 region of the 18S rRNA gene for the identification of diatoms (Bacillariophyceae)[J]. PLoS One, 2012, 7(9): e45664. DOI: 10.1371/journal.pone.0045664 |
| [31] | 孟睿, 何连生, 过龙根, 等. 长江中下游草型湖泊浮游植物群落及其与环境因子的典范对应分析[J]. 环境科学, 2013, 34(7): 2588–2596. Meng R, He L S, Guo L G, et al. Canonical correspondence analysis between phytoplankton community and environmental factors in Macrophtic lakes of the middle and lower reaches of Yangtze River[J]. Environmental Science, 2013, 34(7): 2588–2596. |
| [32] | 苟婷, 马千里, 许振成, 等. 贝江浮游藻类群落特征及富营养化风险分析[J]. 环境科学, 2015, 36(3): 946–954. Gou T, Ma Q L, Xu Z C, et al. Phytoplankton community structure and eutrophication risk assessment of Beijiang River[J]. Environmental Science, 2015, 36(3): 946–954. |
| [33] | Godhe A, Asplund M E, Härnström K, et al. Quantification of diatom and dinoflagellate biomasses in coastal marine seawater samples by real-time PCR[J]. Applied and Environmental Microbiology, 2008, 74(23): 7174–7182. DOI: 10.1128/AEM.01298-08 |
 2017, Vol. 38
2017, Vol. 38


