2. 中国科学院生态环境研究中心, 环境生物技术重点实验室, 北京 100085
2. Key Laboratory of Environmental Biotechnology, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China
多种工业废水及市政污水都同时含有碳、氮和硫系化合物.含氮化合物具有毒性和臭味,而且能够引起水体的富营养化[1].基于目前的生物处理技术,铵盐通常在好氧条件下被氧化为硝酸盐,然后硝酸盐在厌氧条件下通过反硝化作用被还原成氮气[2].自然界多种异养微生物都能够利用各种有机物作为碳源和能源进行反硝化作用.它们具有丰富的生物多样性,通常属于变形菌门中不同的属,例如:Rhodoferax、Dechloromonas和Sulfuritalea[3].
硫化物具有强烈的毒性和臭味,它的超标排放是目前面临的严峻的环境问题之一.它能够和细胞色素中的金属离子进行反应,进而抑制细胞的呼吸作用[4].此外,它还具有腐蚀性并产生很高的化学需氧量(chemical oxygen demand,COD).硫化物通常在产甲烷的过程中伴随产生,它也产生于多种工业加工过程,例如:石油化工、造纸、制革等[5].许多物理、化学和电化学方法已经用来处理气体和水中的硫化物,例如:沉淀法、气提、离子交换、电催化氧化、有机溶剂和化学氧化[6].其中,利用无色硫细菌的生物处理技术具有低成本、低能耗和产物无害等优势.因此,它是一种国际上日益关注的热门技术.Thiobacillus denitrificans被发现能够利用无机硫化合物作为能量来源,无机碳化合物作为碳源进行生长[7, 8].左剑恶等[9]在升流式生物膜反应器中,利用无色硫细菌处理废水中的硫化物,去除率为90%,单质硫转化率为100%.这类能够利用无机硫化合物的自养反硝化细菌具有较高的研究价值,因为它在反硝化过程中不需要再额外添加有机碳源并节约经济成本.相关技术目前已经得到广泛的应用,例如:市政污水、地表水和垃圾渗滤液的处理[10, 11].
近年来,多单元联合生物技术快速发展并应用于处理含有碳、氮和硫系化合物的废水.此技术的过程原理为:厌氧发酵阶段硫酸盐还原所产生的硫化物及少量剩余COD在反硝化单元被来自硝化单元的硝酸盐氧化去除.在香港特别行政区,应用此技术已经成功建立了示范工程,主要进行沿海地区高硫酸盐生活污水的脱氮处理[12, 13].脱氮单元中的活性污泥通常含有自养和异养反硝化细菌,其中有些物种能够利用含硫化合物还原硝化单元所产生的硝酸盐[14].除能以含硫化合物作为能源的自养反硝化细菌以外,一些异养细菌也被发现具有这样的功能.它们能够利用硫化物和硝酸盐进行呼吸作用,并产生单质硫和氮气作为反应产物[15].这种生物基单质硫具有亲水的特性,能够作为生产肥料和杀虫剂的原料,具有较高经济价值[16].
本研究分离1株能够利用硝酸盐作为电子受体,乙酸盐和硫化物作为电子供体进行生长代谢的细菌.在硫化物氧化过程中,单质硫为主要的反应产物.已有的研究结果显示Thauera属的物种是污水处理系统中最活跃的反硝化细菌[17],但是其硫氧化的生理特性极少有研究报道.本研究揭示菌株HDD1在生态学以及污水处理技术应用中的重要意义.
1 材料与方法 1.1 菌株分离与培养条件活性污泥来源于实验室运行的污水处理生物反应器.分离培养基包括以下物质(g·L-1):Na2S·9H2O,1.5;CH3COONa,0.387 5;KNO3,0.757 5;NH4Cl,1.0;KH2PO4,1.8;Na2HPO4·12H2O,3.0;MgSO4·7H2O,0.1;Agar,1.5.培养基灭菌后加入过滤灭菌的微量元素液[18].采用亨盖特厌氧培养技术结合稀释灭绝法进行菌株的分离和纯化[19].活性污泥经过梯度稀释以后,接种到含有固体培养基的厌氧管中.在30℃条件下静置培养7 d后,单克隆被接种到液体培养基中进行培养.液体培养基经过煮沸和充入氩气达到厌氧条件,加入L-半胱氨酸(0.5 g·L-1)和1 mL浓度为0.2%的刃天青作为除氧剂和指示剂.通过在固-液分离培养基中反复转接得到细菌培养物.通过显微镜观察和16S rRNA基因测序技术检测其是否为纯培养.
1.2 菌株鉴定采用Bacterial DNA Mini Kit (TianGen,中国)提取细菌基因组DNA.提取的基因组DNA经分光光度计(Nanodrop 2000,Thermo,美国)定量后作为聚合酶链式反应的模板.采用细菌16S rRNA基因通用引物F27(5′-AGAGTTTGATCCTGGCTCAG-3′)和R1492(5′-GGTTACCTTGTTACGACTT-3′)进行扩增反应[20].反应体系(50 μL)包括Q5 Hot Start High-Fidelity 2X Master Mix (25 μL)(NEB,美国),浓度为20 μmol·L-1的上下游引物(2 μL),20 ng DNA模板(1 μL)和蒸馏水(22 μL).采用GeneAmp PCR system (9700,ABI,美国)进行扩增反应,具体步骤设置如下:94℃起始变性3 min,94℃变性30 s,55℃退火30 s,72℃延伸1 min,30个循环.扩增产物经测序后,用BLAST-N程序和原核生物数据库进行比对分析[21].利用MEGA 5.0软件包[22],采用邻位相连算法构建系统进化树[23].进化树的拓扑结构经过1 000次引导重复取样检验[24].
1.3 生理学和化学分析细菌经离心收集后,用磷酸盐缓冲液清洗3次,用2.5%戊二醛固定30min.用30%、50%、70%、90%、100%的乙醇脱水后,以饱和叔丁醇做介质进行真空干燥.利用扫描电子显微镜(Quanta 200 FEG,FEI,美国)观察细胞形态.利用生化试剂条(API 20NE,BioMérieux,法国)检测菌株的生理特征.细胞经过标准革兰氏染色后,用氢氧化钾消散法复检[25].菌体蛋白浓度测定采用Bradford Protein Assay Kit (Takara,日本)的标准方法.CH3COO-、NO3-、NO2-、SO42-和S2O32-的浓度由离子色谱(ICS-90A,DIONEX,美国)测定.S2-的测定采用标准亚甲基蓝分光光度法[26].利用能量散射谱仪(EDAX,美国)测定单质硫及其含量.菌株的16S rRNA基因序列的GenBank登录号为KX242545.
2 结果与分析 2.1 菌株的分离与鉴定经过5轮固-液分离培养基转接,得到菌株HDD1.在显微镜视野下,细菌的形态呈统一的杆状;16S rRNA基因测序结果中不存在噪声信号,这表明菌株HDD1为纯培养.提交长度为1 397bp的16S rRNA基因序列到数据库进行比对分析,结果显示HDD1与Thauera aminoaromatica S2的相似度最高,达到98.7%.进化树结果显示HDD1在Thauera属中形成一个单源的进化枝,并与Thauera humireducens和Thauera terpenica形成一个进化群(图 1).用最大似然算法对系统进化树验证,得到相同结果.这些结果表明菌株HDD1可能是Thauera属的一个新种,但是还需多相分类结果进行验证.
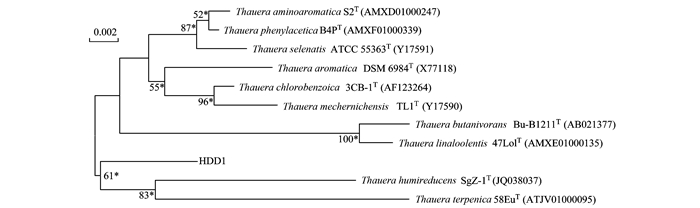
|
图 1 邻位相连法构建16S rRNA基因系统进化树 Fig. 1 Neighbour-joining phylogenetic tree based on 16S rRNA gene |
菌株HDD1革兰氏染色呈阴性,细胞呈杆状(图 2).反硝化作用、吲哚反应和有机酸同化作用呈阳性;糖类水解酸化反应呈阴性(表 1).通常Thauera属的各个种是一类兼性厌氧,专性进行呼吸作用的细菌.它们能够利用氧气、氮氧化物作为电子受体,在好氧呼吸和反硝化作用之间转化代谢状态.有些种在厌氧条件下可以还原硒酸盐[27].很多种能够利用有机酸、氨基酸、芳香和脂肪类化合物进行生长.它们通常在污水处理厂、江河、池塘沉积物等被污染的地区被发现.菌株HDD1的生理特征结果和伯杰氏系统微生物学手册中Thauera属的描述相同[28].

|
图 2 菌株HDD1扫描电子显微镜图像 Fig. 2 Transmission electron micrograph of Thauerasp. HDD1 |
|
|
表 1 菌株HDD1的生理学特性 Table 1 Physiological properties of Thauerasp. HDD1 |
2.3 菌株的代谢特性
菌株HDD1在厌氧条件下利用硝酸盐作为电子受体氧化乙酸盐和硫化物.在15 h之内,CH3COO-基本被完全代谢,其浓度由300 mg·L-1下降到20 mg·L-1,菌体蛋白浓度由1.5 μg·mL-1上升到11 μg·mL-1[图 3(a)],生物量有所增加;NO3-浓度由487 mg·L-1下降到38 mg·L-1,NO2-在第15~20 h之间有短暂的积累,最高浓度为67.7 mg·L-1 [图 3(b)];浓度为200 mg·L-1的S2-被完全代谢,S2O32-(18.4~36.2 mg·L-1)和SO42-(85.5~127.8 mg·L-1)的浓度略有上升[图 3(c)].S2O32-和SO42-的背景值可能是由于在灭菌过程中发生轻微的氧化导致的.在0~5 h,CH3COO-浓度快速地由300 mg·L-1下降到128 mg·L-1,菌体蛋白浓度由1.5 μg·mL-1上升到5.7 μg·mL-1.在代谢反应初期,活化细菌直接进入对数生长期,生物量快速增加,CH3COO-浓度相应地快速下降.在此阶段,NO3-(487~283 mg·L-1)作为电子受体并没有相应地大幅减少.这是由于在对数生长期,大部分CH3COO-通过同化作用直接合成细胞物质,只有少部分通过与NO3-耦合进行呼吸作用.进入稳定期和衰亡期后,细菌同化作用速率减小,需要产生大量能量维持细胞的生命活动.CH3COO-和S2-作为电子供体耦合NO3-进行呼吸作用,大部分NO3-(283~38 mg·L-1)被代谢消耗.为了限制S2-过度氧化成S2O32-和SO42-,增加单质硫的产量,电子受体NO3-浓度(487 mg·L-1)的初始设置较低.它也是工艺实际运行过程中一个重要的参数,根据进水COD含量和活性污泥状态而进行调整.随着硫化物被逐渐氧化,所形成的单质硫逐渐聚集成为直径不同的颗粒[图 4(a)].这些颗粒主要是由C、O、S和P等元素组成.其中S元素占总含量的20%;由于样品中存在菌体等有机物,C元素占总量的68.6%;由于培养基中存在磷酸盐等物质,O元素和P元素分别占总含量的9.2%和2.1%[图 4(b)].由于S2O32-和SO42-含量很低,根据化学反应元素平衡原理,硫化物氧化的主要产物为单质硫.硫酸盐被还原成硫化物,硫化物再被氧化,是硫元素生物地球化学循环中重要的过程[29].这个过程涉及到多种复杂的反应、硫细菌和酶.无色硫细菌分为4个系统进化世系,3个属于细菌域,1个属于古细菌域.多数的无色硫细菌属于Proteobacteria门的Gammaproteobacteria纲[30].根据碳源和能量代谢方式,无色硫细菌可以分为不同的生理类型,包括:专性化能无机营养型、兼性化能无机营养型、化能无机异养型和化能有机异养型.化能无机异养型是指能够利用还原性含硫化合物作为能量来源,但是不能固定二氧化碳的细菌[31].根据生理学特性,菌株HDD1不能利用光能,且只能利用乙酸盐等有机物作为碳源,所以它属于化能异养型微生物.在氧化硫化物过程中其是否获得能量还需进一步验证.原位检测技术结果显示Thauera属在污水处理系统中是一类活跃的反硝化细菌[32],但是很少有研究报道它们的硫氧化功能.微生物群落分析表明Thauera属在反应器的生态系统中具有很高的丰度,但是由于缺乏菌株的纯培养,深入的生理学特征研究很难开展[33].在生理学研究中,菌株HDD1可以作为研究Thauera属硫氧化功能的模式种为进一步的研究提供基础;在生态学研究中,它的相关功能基因可以作为分子标记,为搜索复杂群落中的功能微生物提供技术支撑.目前,造纸厂、煤气厂和制药厂等工业废水都含有大量的硫化物.生物法短程氧化硫化物并回收单质硫作为资源具备很多优势.首先,它是化学和化肥工业的原料.其次,和物理化学法相比,生物法更加节约能源和成本.分离并研究功能菌株的生理学特性是这项生物技术应用的重要保障.在这项技术中,菌株HDD1和其他常见的硫氧化细菌,例如:Thiobacillus denitrificans和Paracoccus denitrificans起到关键作用并有待更深入地研究.
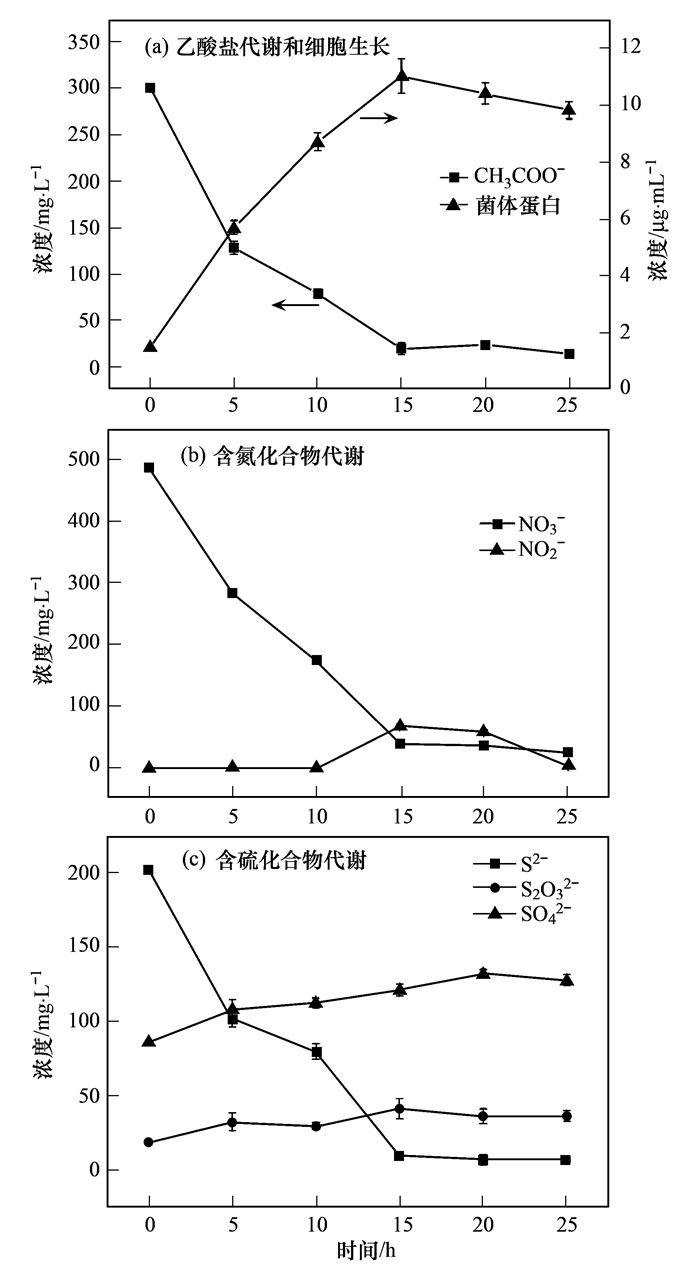
|
图 3 菌株HDD1的碳氮硫化合物代谢 Fig. 3 Metabolism of C, N and S contaminants by Thauerasp. HDD1 |
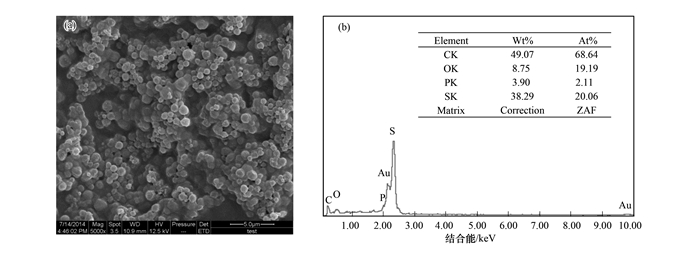
|
(a)扫描电子显微镜图像;(b)能量色散谱分析 图 4 菌株HDD1代谢产物分析 Fig. 4 Analysis of metabolic products of sulfide oxidation by Thauerasp. HDD1 |
在本研究中,从反应器活性污泥中分离纯化出1株细菌HDD1.基于16S rRNA基因的系统进化分析显示,它与Thauera属的物种具有亲缘关系并形成一个单源的进化枝.生理特征实验结果验证表明菌株HDD1是Thauera属的一个种.菌株HDD1能够利用硝酸盐作为电子受体同步氧化硫化物和乙酸盐.在15 h之内,浓度为300 mg·L-1的CH3COO-、200 mg·L-1的S2-和487 mg·L-1的NO3-被完全代谢去除.根据其特殊的生理特征,菌株HDD1可以同时应用于处理含有碳、氮、硫系化合物的工业废水及硫元素的资源化回收.
| [1] | Baker L A. Design considerations and applications for wetland treatment of high-nitrate waters[J]. Water Science and Technology, 1998, 38(1) : 389–395. DOI: 10.1016/S0273-1223(98)00425-9 |
| [2] | Reyes-Avila J, Razo-Flores E, Gomez J. Simultaneous biological removal of nitrogen, carbon and sulfur by denitrification[J]. Water Research, 2004, 38(14-15) : 3313–3321. DOI: 10.1016/j.watres.2004.04.035 |
| [3] | McIlroy S J, Starnawska A, Starnawski P, et al. Identification of active denitrifiers in full-scale nutrient removal wastewater treatment systems[J]. Environmental Microbiology, 2016, 18(1) : 50–64. DOI: 10.1111/emi.2016.18.issue-1 |
| [4] | Visser J M, Robertson L A, Van Verseveld H W, et al. Sulfur production by obligately chemolithoautotrophic thiobacillus species[J]. Applied and Environmental Microbiology, 1997, 63(6) : 2300–2305. |
| [5] | Manconi I, Carucci A, Lens P, et al. Simultaneous biological removal of sulphide and nitrate by autotrophic denitrification in an activated sludge system[J]. Water Science and Technology, 2006, 53(12) : 91–99. DOI: 10.2166/wst.2006.410 |
| [6] | Selvaraj H, Chandrasekaran K, Gopalkrishnan R. Recovery of solid sulfur from hydrogen sulfide gas by an electrochemical membrane cell[J]. RSC Advances, 2016, 6(5) : 3735–3741. DOI: 10.1039/C5RA19116E |
| [7] | Baalsrud K, Baalsrud K S. Studies on Thiobacillus denitrificans[J]. Archiv für Mikrobiologie, 1954, 20(1) : 34–62. DOI: 10.1007/BF00412265 |
| [8] | Claus G, Kutzner H J. Physiology and kinetics of autotrophic denitrification by Thiobacillus denitrificans[J]. Applied Microbiology and Biotechnology, 1985, 22(4) : 283–288. |
| [9] | 左剑恶, 袁琳, 胡纪萃, 等. 利用无色硫细菌氧化废水中硫化物的研究[J]. 环境科学, 1995, 16(6) : 7–10. Zuo J E, Yuan L, Hu J C, et al. Biotechnological removal of sulfides in the effluent from sulfate reducing reactor[J]. Environmental Science, 1995, 16(6) : 7–10. |
| [10] | Kuai L, Verstraete W. Autotrophic denitrification with elemental sulphur in small-scale wastewater treatment facilities[J]. Environmental Technology, 1999, 20(2) : 201–209. DOI: 10.1080/09593332008616809 |
| [11] | Zhang T C, Lampe D G. Sulfur:limestone autotrophic denitrification processes for treatment of nitrate-contaminated water:batch experiments[J]. Water Research, 1999, 33(3) : 599–608. DOI: 10.1016/S0043-1354(98)00281-4 |
| [12] | Wu D, Ekama G A, Chui H K, et al. Large-scale demonstration of the sulfate reduction autotrophic denitrification nitrification integrated (SANI®) process in saline sewage treatment[J]. Water research, 2016, 100 : 496–507. DOI: 10.1016/j.watres.2016.05.052 |
| [13] | Hao T W, Luo J H, Wei L, et al. Physicochemical and biological characterization of long-term operated sulfate reducing granular sludge in the SANI® process[J]. Water Research, 2015, 71 : 74–84. DOI: 10.1016/j.watres.2014.12.051 |
| [14] | Chen C, Ren N Q, Wang A J, et al. Simultaneous biological removal of sulfur, nitrogen and carbon using EGSB reactor[J]. Applied Microbiology and Biotechnology, 2008, 78(6) : 1057–1063. DOI: 10.1007/s00253-008-1396-3 |
| [15] | Tan W B, Jiang Z, Chen C, et al. Thiopseudomonas denitrificans gen. nov., sp. nov., isolated from anaerobic activated sludge[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(1) : 225–229. |
| [16] | van den Bosch P L F, van Beusekom O C, Buisman C J, et al. Sulfide oxidation at halo-alkaline conditions in a fed-batch bioreactor[J]. Biotechnology and Bioengineering, 2007, 97(5) : 1053–1063. DOI: 10.1002/(ISSN)1097-0290 |
| [17] | Thomsen T R, Kong Y H, Nielsen P H. Ecophysiology of abundant denitrifying bacteria in activated sludge[J]. FEMS Microbiology Ecology, 2007, 60(3) : 370–382. DOI: 10.1111/fem.2007.60.issue-3 |
| [18] | Pfennig N, Lippert K D. über das vitamin B12-bedürfnis phototropher Schwefelbakterien[J]. Archiv für Mikrobiologie, 1966, 55(3) : 245–256. DOI: 10.1007/BF00410246 |
| [19] | Hungate R. The anaerobic mesophilic cellulolytic bacteria[J]. Bacteriological Reviews, 1950, 14(1) : 1–49. |
| [20] | Heuer H, Krsek M, Baker P, et al. Analysis of actinomycete communities by specific amplification of genes encoding 16S rRNA and gel-electrophoretic separation in denaturing gradients[J]. Applied and Environmental Microbiology, 1997, 63(8) : 3233–3241. |
| [21] | Kim O S, Cho Y J, Lee K, et al. Introducing EzTaxon-e:a prokaryotic 16S rRNA gene sequence database with phylotypes that represent uncultured species[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(3) : 716–721. |
| [22] | Tamura K, Peterson D, Peterson N, et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10) : 2731–2739. DOI: 10.1093/molbev/msr121 |
| [23] | Saitou N, Nei M. The neighbor-joining method:a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4(4) : 406–425. |
| [24] | Felsenstein J. Confidence limits on phylogenies:an approach using the bootstrap[J]. Evolution, 1985, 39(4) : 783–791. DOI: 10.2307/2408678 |
| [25] | Cerny G. Studies on the aminopeptidase test for the distinction of gram-negative from gram-positive bacteria[J]. European Journal of Applied Microbiology and Biotechnology, 1978, 5(2) : 113–122. DOI: 10.1007/BF00498805 |
| [26] | Trüper H G, Schlegel H G. Sulphur metabolism in Thiorhodaceae Ⅰ. Quantitative measurements on growing cells of Chromatium okenii[J]. Antonie van Leeuwenhoek, 1964, 30(1) : 225–238. DOI: 10.1007/BF02046728 |
| [27] | Krafft T, Bowen A, Theis F, et al. Cloning and sequencing of the genes encoding the periplasmic-cytochrome B-containing selenate reductase of Thauera selenatis[J]. DNA Sequence, 2000, 10(6) : 365–377. DOI: 10.3109/10425170009015604 |
| [28] | Garrity G, Staley J T, Boone D R, et al. Bergey's manual of systematic bacteriology:volume two:the Proteobacteria[M]. New York: Springer Science & Business Media, 2005. |
| [29] | Jørgensen B B. A thiosulfate shunt in the sulfur cycle of marine sediments[J]. Science, 1990, 249(4965) : 152–154. DOI: 10.1126/science.249.4965.152 |
| [30] | Muyzer G, Kuenen J G, Robertson L A. Colorless sulfur bacteria[A]. In:Rosenberg E, DeLong E F, Lory S, et al (Eds.). The Prokaryotes[M]. Berlin Heidelberg:Springer, 2013. 555-588. |
| [31] | Robertson L A, Kuenen J G. The colorless sulfur bacteria[A]. In:Balows A, Trüper H G, Dworkin M, et al (Eds.). The Prokaryotes:A Handbook on the Biology of Bacteria:Ecophysiology, Isolation, Identification, Applications, vol. Ⅰ. 2nd ed[M]. New York:Springer-Verlag, 1992. 385-413. |
| [32] | Ginige M P, Keller J, Blackall L L. Investigation of an acetate-fed denitrifying microbial community by stable isotope probing, full-cycle rRNA analysis, and fluorescent in situ hybridization-microautoradiography[J]. Applied and Environmental Microbiology, 2005, 71(12) : 8683–8691. DOI: 10.1128/AEM.71.12.8683-8691.2005 |
| [33] | Cytryn E, van Rijn J, Schramm A, et al. Identification of bacteria potentially responsible for oxic and anoxic sulfide oxidation in biofilters of a recirculating mariculture system[J]. Applied and Environmental Microbiology, 2005, 71(10) : 6134–6141. DOI: 10.1128/AEM.71.10.6134-6141.2005 |
 2017, Vol. 38
2017, Vol. 38


