2. 中国科学院南京土壤研究所, 土壤与农业可持续发展国家重点实验室, 南京 210008;
3. 中国科学院大学, 北京 100049
2. State Key Laboratory of Soil and Sustainable Agriculture, Institute of Soil Science, Chinese Academy of Sciences, Nanjing 210008, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
随着经济的增长,人们对石油资源的勘探和开采不断增加,对全球环境造成严重的污染[1]. 在一些石油污染严重的场地,每克土壤中检测到的石油含量达到数百微克[2]. 长期的石油污染改变了环境微生物群落的组成,降低了微生物群落多样性,对生态造成了严重的威胁[3]. 污染历史悠久的重质有机污染土壤中,易生物降解的低碳直链烷烃含量很低,多为难降解的高碳烷烃(14~40碳数的正构和异构烷烃)、多环芳烃(萘、菲、芘、、苯并芘及其烷基取代物)、胶质、沥青等[2]. 这类复杂污染物的降解依赖于多种微生物的协同作用. 因此,需要深入研究污染土壤中功能微生物相互作用关系,为开展石油污染土壤的生物修复奠定理论基础.
高通量测序技术[4]和基因芯片技术[5] 的飞速发展为人们理解微生物群落的多样性和复杂性提供了检测平台. 但由于微生物的相互作用复杂多变且难于观察,同时缺乏足够的数据和相应的数据挖掘方法,目前的研究主要集中在分析石油污染土壤中微生物群落多样性水平[6]、各门类微生物分布特征和与环境因素的相互关系之上,而对有机污染土壤中微生物物种间相互作用关系的研究却很少. 生态网络分析技术是一种基于随机矩阵理论分析生态系统中内在作用关系的系统分析方法,用于描绘和表征微生物群落的分子生态网络,为理解复杂的微生物群落的潜在相互作用提供了一种可靠的方法[7]. Liang等[8]运用这种方法构建了我国百色油田酸性土壤中微生物的分子生态网络,研究表明石油污染对酸性土壤微生物群落的相互作用产生显著影响. 然而,我国有大面积的石油污染土壤为盐碱化土壤(如胜利油田、塔里木油田等),需要进一步探索石油污染对典型盐碱化土壤中微生物相互作用关系的影响.
本研究以胜利油田盐碱化土壤为对象,构建了石油污染土壤和未污染土壤中的微生物功能基因组的分子生态网络,探讨了典型盐碱化土壤中石油污染对微生物群落功能网络的影响,确定了网络结构中的“关键基因”以及在石油降解过程中物种之间的相互作用关系,以期为微生物修复盐碱化石油污染土壤提供理论依据.
1 材料与方法 1.1 样品采集与基因芯片杂交胜利油田(37°28′N,118°29′E)地处山东北部渤海之滨的黄河三角洲地带,主要分布在东营、滨州、德州、济南、潍坊、淄博、聊城、烟台等8个城市的28个县(区)境内,工作范围约4.4×104 km2,是中国的第二大油田. 其年平均降水量为550 mm,属于温带大陆性半湿润季风气候. 于2008年6月对胜利油田土壤(SL)进行样品采集,总共采集20个样品. 其中10个石油污染土壤样品(SL-C),采自与原油抽油井邻近并且长期受污染的土壤; 另外10个样品采自离污染场地5 km未受污染的土壤(SL-U). 对于每个采样点,在面积为0.015 m2、高10 cm的土壤上钻5个直径为2.5 cm的孔,收集这些土壤并混合均匀. 采集的土壤样品一部分用于分析土壤的理化性质(见表 1),污染土壤和未污染土壤的理化性质基本接近,另一部分用于土壤微生物的功能基因组成分析. 采集的土壤样品密封在无菌真空采样袋里,并冷冻运回到实验室.
|
|
表 1 石油污染土壤和未污染土壤的理化性质 1) Table 1 Geochemical data of uncontaminated (U) and contaminated (C) soils |
利用酚-氯仿-丁醇萃取法[9, 10]提取土壤微生物基因组DNA. 从每个DNA样品中取2 μg进行标记,42℃条件下杂交12 h[11, 12]. 获得的芯片数据进行数据标准化、信噪比等处理[13].
1.2 网络构建与特征参数为了阐明石油污染对整个微生物生态网络的影响,分别对污染土壤和未污染土壤构建了功能分子生态网络. 构建网络和获取特征参数均在美国俄克拉荷马大学环境基因组学研究所网站(http://ieg2.ou.edu/MENA)上完成. 本研究中使用的功能基因芯片(GeoChip),含有24 243个50-mer的寡核苷酸基因探针,覆盖了10 000多个功能基因,用于编码多于150种碳、氮、硫、磷循环,金属还原和有机物降解相关的关键酶. 选用该技术既可以从功能基因的角度研究石油污染对微生物生态功能的潜在影响,并将微生物的功能结构与其它环境因子联系起来; 同时,本研究中使用的功能基因芯片含有大量编码降解烷烃、苯、联苯、二甲苯、萘、芘及多环芳烃代谢中间产物儿茶酚、原儿茶酸等的功能基因,可以用以评价油田土壤微生物对污染物的降解能力. 网络构建过程如下:将GeoChip功能基因芯片数据进行lg转换,构建皮尔逊相关性矩阵[14],进一步转换成相似性矩阵; 根据随机矩阵理论,通过施加合适的阈值从相似矩阵导出邻接矩阵,用邻接矩阵来编码每对节点之间的连接强度. 可以通过分析相关矩阵特征值的最近邻间距分布预测生态群落. 两个极端分布是高斯正交系综和泊松分布,高斯正交系综反映了一个复杂系统分布的随机性,泊松分布则反映复杂系统的特定性和非随机性[15]. 阈值是从高斯正交系综到泊松分布的转变[7].
用Cytoscape 2.6.0软件对网络进行可视化处理[17],从而得到网络结构图和相关的信息. 描述网络的特征参数包括[7],连通度即计算每个基因(节点)与其他连接着的基因之间的连接数之和,表示一个基因与其他基因的连通强度,是最常用的网络指标之一; 路径长度即两个基因之间的最短距离; 聚类系数即表示一个节点与其他节点之间连通程度的好坏; 以及每个网络的模块性[16],即描述分子生态网络中模块化特性,一个生态网络被分成多个模块,每个模块是生物系统的一个功能单元. 节点i的作用由模块内连通度(Zi)与模块间连通度(Pi)来表征[17].
在石油污染环境中,参与某种化合物降解的微生物功能基因可以指示相应化合物的生物降解潜力和它们的生物利用度[18]. 功能基因还可以用于监测污染物降解菌的动态变化和评估污染物的可生物降解性. 为了进一步了解与石油污染物降解相关的功能微生物群落的相互作用,选择GeoChip的alk基因(alkB、alkH、alkJ和alkK)和多环芳烃降解功能基因(bphA、bphB、bphC、bphD、dfbA、nahA、nahB、nahF、phdA、phdCI、phdJ、phdK和qorL)构建了烷烃和多环芳烃降解功能分子生态网络,其构建过程与整体分子生态网络过程相同,并用Cytoscape 2.6.0软件进行可视化处理[19].
1.3 微生物相互作用网络与环境因子的关系利用皮尔逊相关性分析微生物相互作用网络和环境因子之间的相关性. 用污染土壤和未污染土壤的理化性质和石油含量作为环境因子. 由于不同理化性质的测量单位不同,先把所有理化数据进行标准化再进行计算.
2 结果与分析 2.1 石油污染对盐碱化土壤微生物分子生态网络的影响分别用1 434个和871个基因构建了石油未污染土壤和污染土壤的分子生态网络,研究石油污染对盐碱化土壤微生物功能群落相互作用关系的影响(表 2). 用网络节点数(网络中的基因数)、连接数(不同基因之间的相互作用)、连通度和模块数等来表征污染土壤和未污染土壤中微生物分子生态网络结构的特征. 结果表明,平均路径长度、平均聚类系数和模块性等这些网络指标在污染土壤和未受污染土壤之间显著不同(P<0.001). 在长期石油污染的影响下,盐碱化土壤微生物群落的整体网络结构与未污染土壤明显不同.
|
|
表 2 石油污染土壤和未污染土壤的微生物群落的分子生态网络和随机分子生态网络的拓扑性质 1) Table 2 Topological properties of the empirical molecular ecological networks (MENs) of microbial communities in uncontaminated (U) and contaminated (C) soils of the oil-contaminated site and their associated random MENs |
2.2 石油污染改变了网络节点的拓扑角色
通过计算污染土壤和未污染土壤的模块内连通度(Zi)和模块间连通度(Pi)来分析石油污染对不同节点的拓扑学角色的影响(图 1). 网络节点一般分为4类[20]:①外围节点(Zi≤2.5,Pi≤0.62),连接数很少,且基本连接模块内的节点; ②连接器(Zi≤2.5,Pi>0.62),与一些模块高度相连; ③模块枢纽(Zi>2.5,Pi≤0.62),在模块内部与许多节点高度连接; ④网络枢纽(Zi>2.5,Pi>0.62),既是模块枢纽又是连接器. 模块枢纽基因、连接器详细信息见表 3和表 4. 污染土壤(98.6%)和未污染土壤(99.2%)中的大部分基因是外围节点,在模块内部有很多连接数. 未污染土壤的分子生态网络中有5个模块枢纽,污染土壤分子生态网络结构中有3个模块枢纽; 未污染土壤中没有连接器基因,污染土壤中有3个连接器基因; 在污染和未污染土壤中均没有网络枢纽基因. 在污染土壤和未污染土壤样品中,几乎没有模块枢纽基因与连接器基因重叠.
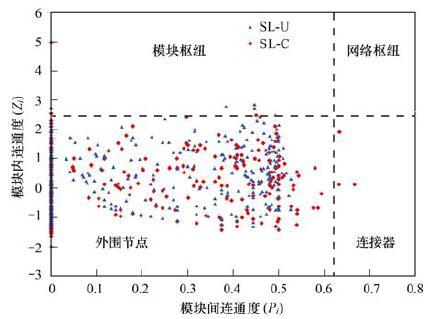
|
图 1 基于污染土壤和未污染土壤的拓扑学角色的基因分布 Fig. 1 plot showing the distribution of genes in contaminated and uncontaminated soils based on their topological roles from SL |
|
|
表 3 模块枢纽基因的信息 Table 3 Information of module hub genes |
|
|
表 4 连接器基因的信息 Table 4 Information of connector genes |
2.3 烷烃、多环芳烃降解功能基因的分子生态网络
烷烃和多环芳烃是石油污染场地中原油污染的主要成分[2, 21]. 为了进一步了解与降解烷烃和多环芳烃相关的微生物功能基因的相互作用,选择GeoChip的alk和多环芳烃降解功能基因构建了污染土壤分子生态网络,其可视化网络结构见图 2和图 3. 在可视化网络中,不同颜色的节点代表不同的基因类别,节点上的数字代表基因序列号. 连接数、基因序列号、基因名、来源等详细信息见表 4和表 5.
在污染土壤中检测到功能基因alkB、alkH、alkJ和alkK,这些基因主要编码降解烷烃的关键酶. 烷烃降解基因中,有68.5%为负相互作用(图 2和表 4),这可能反映了烷烃降解微生物功能群落的竞争行为. 连通性最高的功能基因为alkK (68546649, Corynebacterium sp.),有28个连接数.
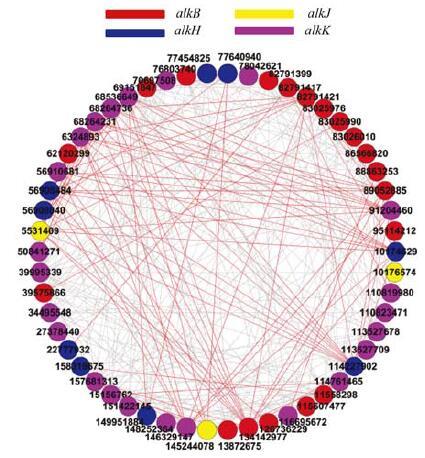
|
图 2 石油污染土壤中烷烃基因的网络相互作用 Fig. 2 Network interactions of the alk genes in oil contaminated soils |
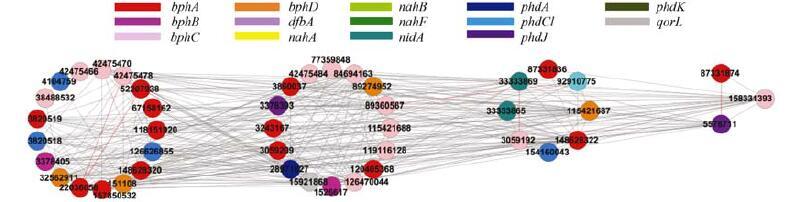
|
图 3 石油污染土壤中的多环芳烃基因的网络相互作用 Fig. 3 Network interactions of the PAH genes in oil contaminated soils |
在石油污染场地检测到高浓度的多环芳烃,如萘、菲、芘、苯并芘以及它们的烷基衍生物,因此,进一步探索参与降解多环芳烃的微生物功能基因的相互作用关系. 多环芳烃降解功能基因的所有相互作用中,98.1%为负相互作用关系. 功能基因bphA (148628322, 未培养细菌) 和 bphD (115421687, Bordetella sp.)的连通性最高,分别有30个连接数(图 3和表 5).
|
|
表 4 石油污染土壤中的烷烃基因的节点信息 Table 4 Nodes information of the alk genes in oil contaminated soils |
|
|
表 5 石油污染土壤中多环芳烃PAH基因的节点信息 Table 5 Nodes information of the PAH genes in oil contaminated soils |
2.4 网络结构与环境特性的关系
为了分析微生物网络相互作用、石油污染和土壤理化性质之间的关系,对基因度和环境因子进行相关性分析(表 6). 结果表明,污染土壤中的基因度和石油浓度呈显著负相关(P<0.01),这表明石油污染可能对盐碱化土壤微生物群落的网络相互作用产生显著的影响. 在污染土壤中,与基因度显著相关的理化性质比未污染土壤中的少.
|
|
表 6 基因度与环境因子之间的相关性 1) Table 6 Pearson correlations between gene degrees and environmental factors |
3 讨论 3.1 整体微生物网络结构的改变
研究微生物群落的网络相互作用及其对环境因子的响应是从根本上研究石油污染对微生物生态系统的影响,而不是单一的分析物种的数量和丰度. 与未污染土壤相比,污染土壤样品中的网络属性发生了变化. 连通度在石油污染的影响下呈显著降低的趋势. 石油污染胁迫下,污染土壤的节点数比未污染土壤的节点数少,分别为436和615,污染土壤每个节点的平均连接数也比未污染土壤少,说明石油污染使微生物之间的相互作用减少,使网络结构的复杂程度降低. 这表明石油污染不仅改变了微生物的群落结构,也改变了整体的网络结构[22]. 石油污染可能会导致土壤微生物功能群落稳定性降低,改变微生物物种在生态系统中的角色,从而引起群落生态功能的潜在变化.
酸性石油污染土壤的分子生态网络的[8]节点数为256,连接数为957,平均连通度为7.48,模块性为0.46,比盐碱性石油污染土壤分子生态网络的节点数、连接数多,而连通度、模块性少,这表明盐碱化石油污染土壤和酸性石油污染土壤的网络结构不同,盐碱化石油污染土壤和酸性污染土壤中微生物之间的相互作用方式不同.
通过运用分子生态网络方法,揭示了石油进入土壤中通过影响土壤环境因子来改变土壤微生物功能基因的网络结构,使对土壤微生物群落的认识从单纯的丰度和多样性提升到了结构的可视化水平.
3.2 潜在关键物种的确定由于微生物的多样性较高且大多数微生物不可培养,因此很难确定一个群落的关键物种[22]. 功能分子生态网络提供了参考信息. 本研究定义了两种类型的关键基因. 其中一类是基于网络拓扑结构和它们的模块组成的关系,在整体网络中起着关键作用的基因(如模块枢纽基因和连接器基因). 在胜利油田污染土壤中的模块枢纽基因比未污染土壤中的模块枢纽基因少,污染土壤中出现3个模块枢纽基因:rubisco(152146172,Beggiatoa sp. SS)、ArsC(6460527,Deinococcus radiodurans R1)、pcc(149370135,unidentified eubacterium),而未污染土壤中有5个模块枢纽基因:proO(118685870,Marinomonas sp. MWYL1)、CODH(68201857,Frankia sp. EAN1pec)、endochitinase(88778030,Reinekea sp. MED297)、dmsA(109641682,Desulfitobacterium hafniense DCB-2)、ArsC(73748096,Dehalococcoides sp. CBDB1),在污染土壤和未污染土壤中没有重叠基因. 石油污染引起土壤中碳氮比严重失衡,造成土壤微生物种间竞争关系增加,改变了整体生态网络,在长期石油污染胁迫下,石油污染土壤中的关键模块枢纽基因发生变化. 这些模块枢纽基因有些是参与碳循环的基因,如未污染土壤中来自于Frankia sp. EAN1pec、Reinekea sp. MED297的CODH、endochitinase基因和污染土壤中来自于Beggiatoa sp. SS、unidentified eubacterium的rubisco、pcc基因. 在污染和未污染土壤中,这些枢纽基因分别来自不同的微生物. 因此,由污染引起的关键模块枢纽基因的变化可能是微生物对环境胁迫的响应. 相反,在污染土壤中有3个连接器基因:nifH(3157562,unidentified nitrogen-fixing bacteria)、gyrB(82798482,Acholeplasma cavigenitalium)、hydrogenase(144943287,Geobacter bemidjiensis Bem),而在未污染土壤中没有连接器基因. 因此,石油污染改变了土壤生态网络中的关键基因.
Liang等[8]研究发现,酸性石油污染土壤中出现2个模块枢纽基因和23个连接器基因,只有基因nifH、gyrB在盐碱化和酸性石油污染土壤中作为连接器基因同时出现. 由此可见,盐碱化石油污染土壤中的关键物种与酸性石油污染土壤中的不同. 这些关键基因对于石油污染场地的微生物群落的网络结构和功能起着重要的作用.
第二类关键基因是参与降解烷烃、多环芳烃且在网络中高度连接的节点. 在功能分子生态网络中,高度连接的节点对于网络的稳定性有至关重要的作用. 如果高度连接的节点丢失,那么该网络将会发生巨大的变化. 因此,这些高度连接的节点可能类似于微生物的“关键物种”. 在盐碱化石油污染土壤中,排在前六位的烷烃基因是alkK(68536649,28个节点)、alkK(157681313,25个节点)、alkK(114761465,24个节点)、alkB(69151647,23个节点)、alkB(13872675,21个节点)、alkK(15156762,21个节点). 烷烃降解基因网络中的许多基因来源于红球菌、副球菌、假单胞菌,这些微生物均能降解石油污染环境中的烷烃[23~25]. 排在前六位的多环芳烃基因是bphA(148628322,30个节点)、bphD(115421687,30个节点)、bphC(126470044,26个节点)、bphD(32562911,26个节点)、bphC(84694163,24个节点)、bphC(158331393,24个节点). 多环芳烃基因主要来源于鞘氨醇、结核分枝杆菌,这些微生物能够降解石油污染环境中的多环芳烃[24, 27]. 本研究表明,这些关键功能基因对于保持生物网络的稳定性发挥着重要的作用.
在酸性石油污染土壤中,排在前六位的烷烃基因是alkB(83025976)、alkK(89890831)、alkK(111018496)、alkK(39647387)、alkB(134142943)alkH(154155441),酸性石油污染土壤中节点数较多的烷烃降解基因与盐碱化石油污染土壤中的烷烃降解基因不同. 排在前六位的多环芳烃基因是nidA(33333869)、oxoH(2072733)、bphD(115421687)、bphA(3820519)、phdCI(126626855)、bphC(84694163),其中bphD(115421687)和bphC(84694163)在盐碱性石油污染土壤和酸性石油污染土壤中均为降解多环芳烃的关键基因[7]. 由此可见,酸性石油污染土壤中的关键烷烃降解基因、关键多环芳烃降解基因与盐碱化石油污染土壤中的不同. 因此,在对石油污染土壤进行微生物修复时,盐碱化土壤和酸性土壤应采取不同的措施,从而提高修复土壤的效率.
3.3 污染场地微生物分子生态网络的研究展望分子生态网络为预测污染场地复杂的微生物群落相互作用关系提供了一条有效的途径. 不同污染场地的环境特性、土壤类型、微生物组成差异较大,因此,有必要比较不同污染类型土壤微生物的分子生态网络. 此外,大多数基于统计学的生态网络推理方法并不能完全反映真实状态下微生物的的生态功能和相互作用[22]. 需要构建微宇宙实验,模拟可控条件下物种多样性-生态系统功能关系,进一步验证污染对微生物群落的相互作用和功能过程的影响.
4 结论本研究以盐碱化石油污染土壤为对象,利用高通量功能基因芯片分析方法和随机矩阵网络构建理论,分析了石油污染对不同物种间相互关系的影响. 结果表明:①石油污染降低了盐碱化土壤网络结构的复杂程度,土壤不同物种间的相互作用减少; ②石油污染使在盐碱化土壤整体网络中起着关键作用的基因发生变化,即土壤中的关键物种发生变化; ③确定了盐碱化石油污染中降解烷烃和多环芳烃的关键基因; ④盐碱化石油污染土壤与酸性石油污染土壤的整体网络结构、在整体网络中起关键作用的基因以及降解烷烃、多环芳烃的关键基因不同.
| [1] | 刘健, 宋雪英, 孙瑞莲, 等. 胜利油田采油区土壤石油污染状况及其微生物群落结构[J]. 应用生态学报 , 2014, 25 (3) : 850–856. |
| [2] | Liang Y T, Zhang X, Wang J, et al. Spatial variations of hydrocarbon contamination and soil properties in oil exploring fields across China[J]. Journal of Hazardous Materials , 2012, 241-242 : 371–378. DOI:10.1016/j.jhazmat.2012.09.055 |
| [3] | Liang Y T, Van Nostrand J D, Deng Y, et al. Functional gene diversity of soil microbial communities from five oil-contaminated fields in China[J]. The ISME Journal , 2011, 5 (3) : 403–413. DOI:10.1038/ismej.2010.142 |
| [4] | Simon C, Daniel R. Metagenomic analyses:past and future trends[J]. Applied and Environmental Microbiology , 2011, 77 (4) : 1153–1161. DOI:10.1128/AEM.02345-10 |
| [5] | He Z L, Van Nostrand J D, Zhou J Z. Applications of functional gene microarrays for profiling microbial communities[J]. Current Opinion in Biotechnology , 2012, 23 (3) : 460–466. DOI:10.1016/j.copbio.2011.12.021 |
| [6] | 杨萌青, 李立明, 李川, 等. 石油污染土壤微生物群落结构与分布特性研究[J]. 环境科学 , 2013, 34 (2) : 789–794. |
| [7] | Zhou J Z, Deng Y, Luo F, et al. Functional molecular ecological networks[J]. mBio , 2010, 1 (4) : e00169–10. |
| [8] | Liang Y T, Zhao H H, Deng Y, et al. Long-term oil contamination alters the molecular ecological networks of soil microbial functional genes[J]. Frontiers in Microbiology , 2016, 7 : 60. |
| [9] | Zhou J Z, Bruns M A, Tiedje J M. DNA recovery from soils of diverse composition[J]. Applied and Environmental Microbiology , 1996, 62 (2) : 316–322. |
| [10] | Montoya J M, Pimm S L, Solé R V. Ecological networks and their fragility[J]. Nature , 2006, 442 (7100) : 259–264. DOI:10.1038/nature04927 |
| [11] | Liang Y T, He Z L, Wu L Y, et al. Development of a common oligonucleotide reference standard for microarray data normalization and comparison across different microbial communities[J]. Applied and Environmental Microbiology , 2010, 76 (4) : 1088–1094. DOI:10.1128/AEM.02749-09 |
| [12] | Wu L Y, Liu X D, Schadt C W, et al. Microarray-based analysis of subnanogram quantities of microbial community DNAs by using whole-community genome amplification[J]. Applied and Environmental Microbiology , 2006, 72 (7) : 4931–4941. DOI:10.1128/AEM.02738-05 |
| [13] | He Z L, Deng Y, Van Nostrand J D, et al. GeoChip 3^0 as a high-throughput tool for analyzing microbial community composition,structure and functional activity[J]. The ISME Journal , 2010, 4 (9) : 1167–1179. DOI:10.1038/ismej.2010.46 |
| [14] | Horvath S, Dong J. Geometric interpretation of gene coexpression network analysis[J]. PLoS Computational Biology , 2008, 4 (8) : e1000117. DOI:10.1371/journal.pcbi.1000117 |
| [15] | Luo F, Yang Y F, Zhong J X, et al. Constructing gene co-expression networks and predicting functions of unknown genes by random matrix theory[J]. BMC Bioinformatics , 2007, 8 (1) : 299. DOI:10.1186/1471-2105-8-299 |
| [16] | Clauset A, Newman M E J, Moore C. Finding community structure in very large networks[J]. Physical Review E , 2004, 70 (6) : 066111. DOI:10.1103/PhysRevE.70.066111 |
| [17] | Guimerà R, Amaral L A N. Functional cartography of complex metabolic networks[J]. Nature , 2005, 433 (7028) : 895–900. DOI:10.1038/nature03288 |
| [18] | 唐景春, 吕宏虹, 刘庆龙, 等. 石油烃污染及修复过程中的微生物分子生态学研究进展[J]. 微生物学通报 , 2015, 42 (5) : 944–955. |
| [19] | Cline M S, Smoot M, Cerami E, et al. Integration of biological networks and gene expression data using Cytoscape[J]. Nature Protocols , 2007, 2 (10) : 2366–2382. DOI:10.1038/nprot.2007.324 |
| [20] | 汪峰, ZhouJ Z, 孙波. 我国东部土壤氮转化微生物的功能分子生态网络结构及其对作物的响应[J]. 科学通报 , 2014, 59 (4-5) : 387–396. |
| [21] | 杨茜, 吴蔓莉, 聂麦茜, 等. 石油污染土壤的生物修复技术及微生物生态效应[J]. 环境科学 , 2015, 36 (5) : 1856–1863. |
| [22] | Faust K, Raes J. Microbial interactions:from networks to models[J]. Nature Reviews Microbiology , 2012, 10 (8) : 538–550. DOI:10.1038/nrmicro2832 |
| [23] | 张玉秀, 蒙小俊, 柴团耀. 苯酚降解菌红球菌(Rhodococcus sp^) P1的鉴定及其在焦化废水中的应用[J]. 微生物学报 , 2013, 53 (10) : 1117–1124. |
| [24] | 赵翠, 孙庆华, 柏耀辉, 等. 副球菌BW001的生理特性及其对吡啶的降解[J]. 环境污染与防治 , 2008, 30 (11) : 17–22. |
| [25] | 徐金兰, 黄廷林, 唐智新, 等. 高效石油降解菌的筛选及石油污染土壤生物修复特性的研究[J]. 环境科学学报 , 2007, 27 (4) : 622–628. |
| [26] | 周丽沙, 李慧, 张颖, 等. 石油污染土壤鞘氨醇单胞菌遗传多样性[J]. 土壤学报 , 2011, 48 (4) : 804–812. |
| [27] | Chaillan F, Le Flècheb A, Bury E, et al. Identification and biodegradation potential of tropical aerobic hydrocarbon-degrading microorganisms[J]. Research in Microbiology , 2004, 155 (7) : 587–595. DOI:10.1016/j.resmic.2004.04.006 |
 2016, Vol. 37
2016, Vol. 37


