2. 瑞典乌普萨拉大学进化生物学中心环境毒理学系, 瑞典 SE-752 36;
3. 南开大学环境科学与工程学院, 天津 300092
2. Environmental Toxicology of Evolutionary Biology Center, Uppsala University, Uppsala SE-752 36, Sweden;
3. College of Environmental Science and Engineering, Nankai University, Tianjin 300092, China
CYP1A是细胞色素P450 (cytochrome P450,CYP) 家族中的重要成员,主要和芳基烃受体(AhR) 调控的信号途径有关; 在生物体对多环芳烃(PAHs)、 多氯联苯(PCBs)和二 英(Dioxins)等有机污染物的代谢和脱毒作用中起重要作用[1, 2, 3]. CYP1A 被外源化合物诱导的机制主要是通过诱导剂与细胞内的芳香烃受体(AhR) 结合,进而通过一系列反应结合、 转位而与CYP1A靶基因的启动区的反应元件结合,从而启动CYP1A的转录. 当有机污染物配体与胞浆中的AhR结合,使AhR 定位到细胞核内,与芳烃受体核转录因子(ARNT) 蛋白结合后,这种配体-AhR-ARNT复合体能够与CYP1A 上游的外化物反应元件(XREs)结合,从而最终激活 CYP1A 基因的表达[1, 4, 5]. 由于生物暴露于PAHs、 PCBs等污染物的水平与体内CYP1A1表达水平具有显著相关性,其表达水平甚至以几何级数增加; 因此,在对水生系统污染监测中,检测鱼类CYP1A基因的诱导表达已经被作为一种灵敏的早期预警手段[6, 7].
在鱼类CYP1家族当中,除了 CYP1A 之外,还有其它3种 CYP1 亚家族成员,包括 CYP1B、 1C及1D [8, 9, 10]. 在斑马鱼中,PCB126与TCDD均可以诱导 CYP1A、 1B及1C 基因的表达,但不能诱导 CYP1D [10, 11]. 在虹鳟鱼中, CYP1 基因包括 CYP1As基因(1A1与1A3)、 CYP1B1及CYP1Cs(1C1、 1C2与1C3 )[12, 13]. 此前已有许多报道叙述了不同 CYP1 基因可诱导性,Denison等[14]曾报道过,很多AhR激动剂可以诱导 CYP1A mRNA与蛋白的表达; 另外,在鱼胚胎发育及成鱼期, CYP1B1与CYP1C s基因也可以受AhR激动剂的诱导[11, 15, 16, 17]. 另据报道,在斑马鱼、 鳉鱼(Fundulus heteroclitus)及非洲爪蟾(Xenopus tropicalis)体内新发现的 CYP1D1 基因在转录水平没有出现被典型AhR激动剂(如TCDD、 PCB-126等)诱导的现象,表明 CYP1D1 不是受AhR途径介导的[10, 17, 18]. 考虑到对污染物反应效果强弱的不同,鱼类 CYP1A、 1B、 1C 基因表达反应模式可作为评价其所处水环境污染程度的重要生物标记物.
目前我国鱼类 CYP1在水环境污染及其效应监测中的应用研究还处于起步阶段,研究利用鱼鳃 CYP1基因表达模式监测海洋污染更是鲜见报道. 因此,针对我国水环境污染状况,建立与国际先进技术相接轨的生物标志物监测技术体系迫在眉睫. 本研究旨在利用实时荧光定量核酸扩增检测系统(qPCR)对模式生物虹鳟鱼(Oncorhynchus mykiss) 进行分析,通过在天津市内不同流域现场点位监测实验,考察多种 CYP1 活性与水环境污染程度的相关性,及根据不同基因表达模式及其生物效应的可行性,探讨该方法在实际监测中应用的可能性.
1 材料与方法 1.1 材料虹鳟鱼(Oncorhynchus mykiss)购于北京顺通虹鳟鱼养殖中心,体重126 g±23 g,体长21 cm±1.3 cm. 每日用充气机连续充气,投喂人工饵料,每天更换新鲜,暂养期间水温 10~11℃,pH 7.9,暂养 5 d 后选取对鱼随机分组进行野外监测实验.
1.2 野外监测实验为使外部条件(天气、 河水温度等)对实验影响最小化,实验时间选取在3月中旬,虹鳟鱼孵化场、 暂存实验室、 及待放置河水温度温差小(具体参数见表 1).
| 表 1 受试生物在不同监测点位的水环境指标 Table 1 Aquatic parameters at different caging sites |
将3组置于笼子(55 cm×55 cm×55 cm)中实验动物(N=6)分别放置于天津境内3个不同位置,分别是海河干流葛沽G(位于民俗文化中心河口处,N38.995 010°,E 117.507 866°),海河入海口处H(位于航一路天津市水运工程材料试验检测中心旁,N 38.999 060°,E 117.701 841°),马厂减河桥下M(马厂减河桥与天津大道交汇处,N38.979 850°,E117.548 699°). 实验周期为1 d,暴露实验后,剪断鳃弓,放出血液,立即打开腹腔,取出完整鳃弓与肝脏组织,用滤纸擦净表面血污后放入RNA稳定溶液RNAlater(购于Ambion公司)中. 回到实验室后,将样品置于4℃冰箱保存. 实验期间,全部实验动物未发现明显不适或死亡现象. 暂存实验室虹鳟鱼设定为实验对照组.
1.3 总RNA 提取使用总RNA脂肪和纤维组织试剂盒与DNase酶(购自美国伯乐公司)提取总RNA,提取产物经紫外分光光度计(微量核酸蛋白测定仪,ND1000)检测,260/280与260/230吸光值比率在2-2.1范围内. 经 1%的琼脂糖凝胶电泳后(TAE缓冲液EDTA,购自Sigma公司),显示总RNA样品没被降解,完整性好. 严格按cDNA合成试剂盒(iScript cDNA 合成试剂盒,购自Biorad伯乐公司)操作规范进行操作合成cDNA.
1.4 qPCR分析虹鳟鱼靶基因定量PCR引物(qPCR产物长度在75-150 bp之间)由Sigma公司合成(mRNA 序列见表 2). 使用iQ SYBR Green Supermix试剂盒(购于Bio-Rad公司)及Rotor-Gene 6000荧光实时定量PCR仪(Qiagen公司)进行定量PCR检测分析. cDNA定量分析操作过程严格按规范进行,具体过程如下: cDNA样品同时进行二孔平行分析,程序首先为95℃ 10 min,然后进入95℃ 15 s,62℃ 15 s的35个循环反应,最后溶解曲线分析阶段(温度从55~95℃,从低至高,温度每5 s提高1℃).
| 表 2 实时定量PCR分析基因表达所用的引物(5′-3′) Table 2 Sequences (5′-3′) of the gene-specific real-time PCR primers used in the experiments |
实验使用EΔΔCT法分析计算相对mRNA的表达表达量[19, 20]. PCR扩增效率通过LinRegPCR程序进行分析[21, 22],所得效率值在1.85 ~1.95之间. 实验选取 ER1α、 l13 与β-actin作为内源参照基因来确定鱼鳃与肝脏内目标基因的相对表达量; 由于 ER1α 在不同组织间稳定性高且基因表达量不受实验影响, ER1α 被最终确定为最佳参照基因.
1.5 数据分析为使变量保持正态分布,实验数据首先经过对数变换,然后利用参数检验方法进行统计学分析. 实验数据以平均值±标准偏差(mean±SD)表示并采用GraphPad Prism5软件的Students t test与One-way ANOVA进行方差分析与显著性检验,若 P<0.05(以*表示),表示有显著性差异,若 P<0.001(以***表示),表示有极显著性差异.
2 结果与分析在野外实验中,不同位置河水对鱼鳃 CYP1 基因表达有明显诱导作用(图 1、 2). 诱导性最强的是 CYP1A3 ,最弱的是 CYP1C2 . 与实验对照组相比, CYP1A1、 1A3、 1B1 表达明显增加,差异均有显著性(***P<0.001). 比较鱼鳃 CYP1 mRNA表达模式在不同实验笼放置位置,葛沽与入海口有相似的表达模式,除了入海口 CYP1C2 没有产生诱导反应; 虽然表达模式相近似,但是反应级别有所不同,在葛沽检测位置 CYP1 mRNA表达低于入海口位置. 在葛沽的反应模式与入海口、 马厂减河存在部分相似. 与其他位置相比较,马厂减河 CYP1C 没有受暴露实验而产生的诱导作用. 为了明确数据是否受到运输过程影响,实验比较了虹鳟鱼运输前后(在虹鳟鱼孵化场及暂存实验室) CYP1 基因表达量变化; 结果显示,所测基因表达量没有受到运输过程影响(图 2).
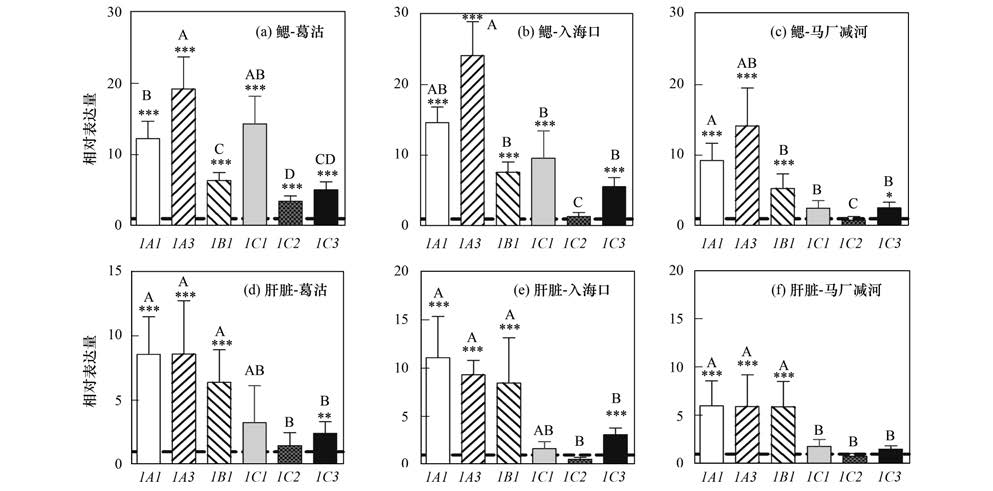 | 图 1 在不同监测点位虹鳟鱼鳃及肝脏相对 CYP1A mRNA 表达水平 Fig. 1 Relative CYP1 s mRNA expression patterns in gills and liver of rainbow trout 将3组虹鳟鱼(N=6)悬挂在不同点位进行野外监测实验选取 ER1α 作为内源参照基因来确定鱼鳃与肝脏内目标基因的相对表达量,暂存实验室虹鳟鱼(实验对照组)作为参照因子,虚线表示实验对照组的表达水平(1倍的相对表达量)利用one way ANOVA进行差异显著性分析: 使用Tukeys post hoc算法比较同一点位不同基因的差异(以不同字母表示,P<0.05); 使用Bonferronis post hoc算法比较不同基因与实验对照组相应基因的差异(***P<0.001,** P<0.01) |
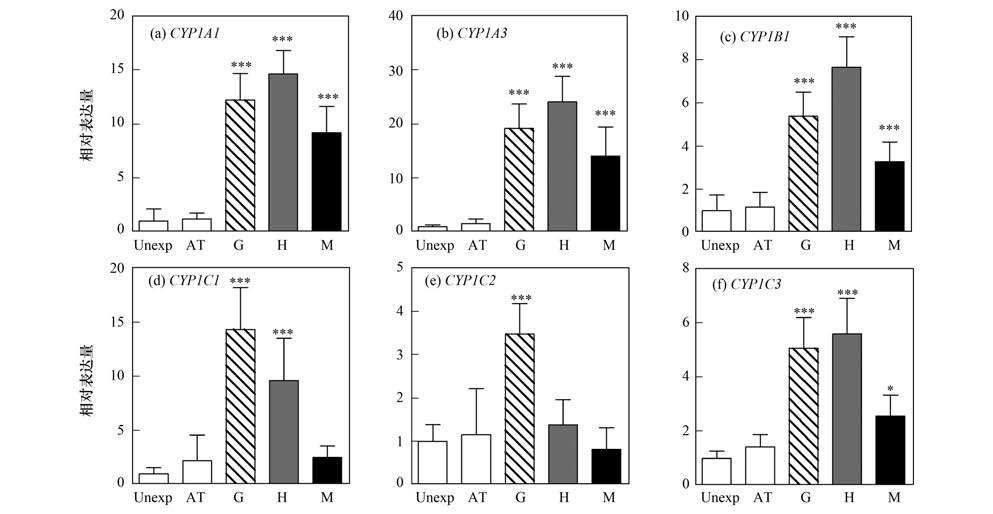 | 图 2 虹鳟鱼鳃 CYP1A、 CYP1B、 CYP1C 亚家族mRNA 表达模式 Fig. 2 The mRNA expression patterns for CYP1A,CYP1B,CYP1C subfamily genes in gills of rainbow trout 虹鳟鱼(N=6)悬挂在不同点位进行野外监测实验选取 ER1α 作为内源参照基因来确定鱼鳃目标基因的相对表达量,暂存实验室虹鳟鱼(实验对照组)作为参照因子; 利用one way ANOVA进行差异显著性分析,使用Bonferronis post hoc算法比较不同基因与实验对照组相应基因的差异 |
在不同监测位置,肝脏 CYP1 表达差异性不大(图 1). 与实验对照组相比, CYP1A1、 1A3、 1B1 表达明显增加,但3种 CYP1C 基因没有受河水影响而产生诱导效应. 与鱼鳃中 CYP1 的相对表达量比较,肝脏 CYP1 表达量低于鱼鳃.
3 讨论在以鱼类鳃 CYP1A 为生物标志物进行的野外监测实验中,有两种方法被广泛应用,一种是直接在实验海区采集受试鱼类,这种方法有监测时间快的优点,但存在捕获的鱼类种类、 来源不确定性和个体、 年龄大小难于统一的缺点. 另外一种方法是将监测鱼类悬挂在特定污染海区进行野外监测实验,这种方法需要购买和暂养监测鱼类,但能够更好地反映特定监测海域的污染生物效应状况[23]. 本研究采用了后者的实验方法.
鱼鳃作为直接暴露于水环境的组织,在石油类污染发生的第一时间与污染物直接接触,而鳃丝中存在的CYP1酶对环境有机污染物反应灵敏,可在短时间、 低浓度的污染物暴露条件下就受到显著诱导[7, 24, 25]. 因此,利用鱼类鳃丝CYP1的变化量检测方法可应用于监测水质污染及其生化效应状况. 例如,在国际上,Abrahamson等[7, 26]利用虹鳟和鳕鱼进行水环境PAHs污染监测和石油污染海区的实地监测. 其研究结果显示: 虹鳟鳃丝CYP1A在污染水域暴露1 d后即可显著诱导,在PAHs浓度较高的排污口附近水域挂养的受试虹鳟鳃丝CYP1A活性高于排污口上游水域15 km处对照组的酶活性; 挂养在离石油钻井平台较近海域的鳕鱼鳃丝EROD 活性(7-乙氧基异吩 唑-O-脱乙基酶,其活性表示可评价对 CYP1A1 的诱导作用)则显著高于离钻井平台较远的实验组的活性. 在我国,CYP1A 蛋白表达量及其活性的在水环境污染及其效应监测中的应用研究也受到广泛关注,霍传林等[27]报道了利用鱼肝 EROD 活性监测海洋环境PCBs污染的作用; 张玉生等[23]报道了石油类及其 PAHs 污染对褐菖鲉(Sebastiscus marmoratus)肝CYP1A表达的影响; 余铭恩[28]探讨了利用鱼肝微粒体 EROD 活性监测海洋环境石油类污染的可行性等. 由此可见,CYP1A的诱导表达可作为指示海洋环境中石油类及其 PAHs 等有机污染物污染的有效而敏感的生物标志物. 本研究的实验结果也表明,在不同监测位置河水中存在引起虹鳟鱼代谢解毒系统响应的污染物,造成一定的毒性效应,应引起重视.
实验中还发现 CYP1A 在肝脏中的表达量比鱼鳃中要低,表明水中很可能含有容易被降解的物质,例如PAHs. 此前的实验证明一些PAH既是 CYP1A/1B 的诱导物也是其作用的底物. Jönsson等[25, 29]曾经证明虹鳟鱼吸收的苯并[a]芘在鳃组织中会产生首过效应,即只有一部分被鳃吸收苯并[a]芘可以到达肝脏组织. 另一方面,Brammell等[30]曾调查了生产PCB工厂废水对虹鳟鱼的影响. 结果发现,在肯塔基州泥河流域距离PCB工厂排污口2 km区域挂养的受试虹鳟鱼肝脏CYP1A1表达量比对照组显著升高,而鳃丝CYP1A1的表达量很低. 可以看出,在泥河挂养的受试虹鳟鱼主要暴露于不易于降解的PCB,而在乌普萨拉区域的受试虹鳟鱼主要暴露于PAH,由此可以推断,鳃丝与肝脏中不同的反应模式是由诱导物的代谢难易差异造成的.
虹鳟鱼 CYP1C 基因是近年来发现细胞色素P450 家族中的新成员,但相关领域的研究开展甚少,目前还不能明确其他3种被克隆的cDNA所编码的蛋白是否像CYP1A一样有着与EROD的高相关性. 从野外监测实验的结果表明,鳃丝 CYP1C1 与 CYP1A1 的表达量有着很高的相近性. CYP1C1 与 CYP1C3 表达模式的相似性高于 CYP1C1/C3 与 CYP1C2 之间的相似性. Jönsson等[25]利用虹鳟鱼进行PCB126污染物水体暴露实验研究表明, CYP1A 与CYP1C1 的表达模式非常相似; 但进行野外监测实验时,发现不同 CYP1C 亚家族的表达模式有明显不同. 本实验的结果表明,在不同监测位点中,葛沽与入海口的6种 CYP1 基因表达模式很相近,但表达量不同,有可能是由于水中污染物负荷不同造成,表明海河入海口的受污染程度更高. 此外, CYP1C1/C3 在葛沽与入海口受到明显诱导作用,但在马厂减河却不然,表明马厂减河中存在的污染物种类不同于其它监测位点.
4 结论综上所述,本实验利用虹鳟鱼鳃丝与肝脏多种 CYP1 基因的表达模式用于现场水域污染监测中,为水质混合污染物生物监测工作提出了一种新的方法. 通过对虹鳟鱼的6个 CYP1 基因在不同监测区域mRNA 的监测,发现所有基因均可被诱导,其中 CYP1A1、 1A3 和1C1 基因的反应最敏感,而基因最高诱导率出现在海河入海口处. 在马厂减河水域中 CYP1 mRNA 的反应规律显著不同于在其他两地得到的结果,说明在这两处存在不同的 CYP1 诱导剂/抑制剂. 下一步将建立并解释化学混合物成分与生物反应模式之间关系,对单体以及复合污染物产生的生物效应进行归纳,为不同水域污染早期评价方法提供基础理论依据.
| [1] | Denison M S, Pandini A, Nagy S R,et al.Ligand binding and activation of the Ah receptor [J].Chemico-Biological Interactions, 2002, 141 (1-2): 3-24. |
| [2] | Williams T D, Wu H F, Santos E M,et al.Hepatic Transcriptomic and Metabolomic Responses in the Stickleback (Gasterosteus aculeatus) Exposed to Environmentally Relevant Concentrations of Dibenzanthracene [J].Environmental Science & Technology, 2009, 43 (16): 6341-6348. |
| [3] | 许友卿, 张青红, 张茜, 等.对多氯联苯敏感的水生动物生物标志物 [J].水产科学, 2015, 34 (1): 66-70. |
| [4] | Mandal P K.Dioxin: a review of its environmental effects and its aryl hydrocarbon receptor biology [J].Journal of Comparative Physiology B, 2005, 175 (4): 221-230. |
| [5] | Whitlock J P Jr.Induction of cytochrome p4501A1 [J].Annual Review of Pharmacology and Toxicology, 1999, 39: 103-125. |
| [6] | 林茂, 杨先乐, 纪荣兴.草鱼体外培养细胞中β-萘黄酮对CYP1A基因表达的诱导 [J].水产学报, 2010, 34 (6): 885-890. |
| [7] | Abrahamson A, Andersson C, Jönsson M E,et al.Gill EROD in monitoring of CYP1A inducers in fish-A study in rainbow trout (Oncorhynchus mykiss) caged in Stockholm and Uppsala waters [J].Aquatic Toxicology, 2007, 85 (1): 1-8. |
| [8] | Leaver M J, George S G.A cytochrome P4501B gene from a fish, Pleuronectes platessa [J].Gene, 2000, 256 (1-2): 83-91. |
| [9] | Godard C A J, Goldstone J V, Said M R,et al.The new vertebrate CYP1C family: Cloning of new subfamily members and phylogenetic analysis [J].Biochemical and Biophysical Research Communications, 2005, 331 (4): 1016-1024. |
| [10] | Goldstone J V, Jönsson M E, Behrendt L,et al.Cytochrome P450 1D1: A novel CYP1A-related gene that is not transcriptionally activated by PCB126 or TCDD [J].Archives of Biochemistry and Biophysics, 2009, 482 (1-2): 7-16. |
| [11] | Jönsson M E, Jenny M J, Woodin B R,et al.Role of AHR2 in the expression of novel cytochrome p450 1 family genes, cell cycle genes, and morphological defects in developing zebra fish exposed to 3,3',4,4',5-pentachlorobiphenyl or 2,3,7,8-tetrachlorodibenzo-p-dioxin [J].Toxicological Sciences, 2007, 100 (1): 180-193. |
| [12] | Råbergh C M I, Vrolijk N H, Lipsky M M,et al.Differential expression of two CYP1A genes in rainbow trout (Oncorhynchys mykiss) [J].Toxicology and Applied Pharmacology, 2000, 165 (3): 195-205. |
| [13] | Berndtson A K, Chen T T.2 Unique CYP1 genes are expressed in response to 3-methylcholanthrene treatment in rainbow trout [J].Archives of Biochemistry and Biophysics, 1994, 310 (1): 187-195. |
| [14] | Denison M S, Nagy S R.Activation of the aryl hydrocarbon receptor by structurally diverse exogenous and endogenous chemicals [J].Annual Review of Pharmacology and Toxicology, 2003, 43 (1): 309-334. |
| [15] | Jönsson M E, Orrego R, Woodin B R,et al.Basal and 3,3',4,4',5-pentachlorobiphenyl-induced expression of cytochrome P450 1A, 1B and 1C genes in zebrafish [J].Toxicology and Applied Pharmacology, 2007, 221 (1): 29-41. |
| [16] | Wang L, Scheffler B E, Willett K L.CYP1C1 messenger RNA expression is inducible by benzo[a]pyrene in Fundulus heteroclitus embryos and adults [J].Toxicological Sciences, 2006, 93 (2): 331-340. |
| [17] | Zanette J, Jenny M J, Goldstone J V,et al.New cytochrome P450 1B1, 1C2 and 1D1 genes in the killifish Fundulus heteroclitus: Basal expression and response of five killifish CYP1s to the AHR agonist PCB126 [J].Aquatic Toxicology, 2009, 93 (4): 234-243. |
| [18] | Uno Y, Uehara S, Murayama N,et al.CYP1D1, pseudogenized in human, is expressed and encodes a functional drug-metabolizing enzyme in cynomolgus monkey [J].Biochemical Pharmacology, 2010, 81 (3): 442-450. |
| [19] | Livak K J, Schmittgen T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT Method [J].Methods, 2001, 25 (4): 402-408. |
| [20] | Schmittgen T D, Livak K J.Analyzing real-time PCR data by the comparative CT method [J].Nature Protocols, 2008, 3 (6): 1101-1108. |
| [21] | Ramakers C, Ruijter J M, Deprez R H L,et al.Assumption-free analysis of quantitative real-time polymerase chain reaction (PCR) data [J].Neuroscience Letters, 2003, 339 (1): 62-66. |
| [22] | Ruijter J M, Ramakers C, Hoogaars W M H,et al.Amplification efficiency: linking baseline and bias in the analysis of quantitative PCR data [J].Nucleic Acids Research, 2009, 37 (6): e45. |
| [23] | 张玉生, 郑榕辉, 陈清福.褐菖鲉肝CYP 1A作为生物标志物监测厦门海域石油污染状况 [J].生态学报, 2011, 31 (19): 5851-5859. |
| [24] | Mdegela R, Myburgh J, Correia D,et al.Evaluation of the gill filament-based EROD assay in African sharptooth catfish (Clarias gariepinus) as a monitoring tool for waterborne PAH-type contaminants [J].Ecotoxicology, 2006, 15 (1): 51-59. |
| [25] | Jönsson E M, Abrahamson A, Brunström B,et al.Cytochrome P4501A induction in rainbow trout gills and liver following exposure to waterborne indigo, benzo[a]pyrene and 3, 3, 4, 4, 5-pentachlorobiphenyl [J].Aquatic Toxicology, 2006, 79 (3): 226-232. |
| [26] | Abrahamson A, Brandt I, Brunström B,et al.Monitoring contaminants from oil production at sea by measuring gill EROD activity in Atlantic cod (Gadus morhua) [J].Environmental Pollution, 2008, 153 (1): 169-175. |
| [27] | 霍传林, 王菊英, 韩庚辰, 等.鱼体内EROD活性对多氯联苯类的指示作用 [J].海洋环境科学, 2002, 21 (1): 5-8. |
| [28] | 余铭恩.褐菖鲉(Sebastiscus marmoratus)肝微粒体细胞色素P-450单加氧酶的研究——动力学反应条件、诱导和监测[D].厦门: 国家海洋局第三海洋研究所, 2003.1-63. |
| [29] | Jönsson M E, Gao K, Olsson J A,et al.Induction patterns of new CYP1 genes in environmentally exposed rainbow trout [J].Aquatic Toxicology, 2010, 98 (4): 311-321. |
| [30] | Brammell B F, McClain J S, Oris J T,et al.CYP1A expression in caged rainbow trout discriminates among sites with various degrees of polychlorinated biphenyl contamination [J].Archives of Environmental Contamination and Toxicology, 2010, 58 (3): 772-782. |
 2015, Vol. 36
2015, Vol. 36


