2. 中国科学院生态环境研究中心, 北京 100085;
3. 江西省科学院能源研究所, 南昌 330096
2. Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China;
3. Institute of Energy, Jiangxi Academy of Sciences, Nanchang 330096, China
由于抗生素广泛用于人类医疗、 畜禽养殖、 水产养殖等,大量残留的抗生素进入环境,致使微生物在持续抗生素选择压力下产生了耐药性,形成了耐药菌[1]. 抗性基因是耐药菌具有抗性的主要原因之一,它可以通过多种形式的可移动遗传元件如质粒、 整合子、 转座子、 插入序列等,突破细菌之间的种属关系广泛传播,加重抗性污染[2]. 近几年,国内外细菌的耐药率成逐年上升的趋势[3, 4, 5],并且国内外已有报道指出,耐药菌和抗性基因可能成为一种新型的环境污染物,其风险将比抗生素药物本身的污染风险更高[6],这将对环境和人类健康构成严重的威胁[7, 8].
大环内酯与林可霉素、 链阳菌素类抗生素具有细菌染色体上重叠的作用位点,因此导致一类抗性基因对所有这些抗生素具有抗药性,称为大环内酯类-林可霉素类-链阳菌素(macrolide-lincosamide-streptogramin,MLS)抗性基因[9]. 已有研究表明,MLS抗性基因不仅能在G+和G-菌之间传播[10, 11],而且广泛存在于污水中,例如,MLS抗性基因广泛存在于污水处理厂和海河流域的河水[12]、 养猪废水[13]和临床医院废水[14],其中在临床医院废水中分离出的红霉素耐药链球菌(Streptococcus)中有78.7%携带MLS抗性基因[14]. MLS抗性基因宿主广泛,且抗生素制药废水通常含有非常高的微生物量和丰富的营养,是耐药菌、 MLS抗性基因产生和扩散传播的高发区. 抗生素废水有机物浓度高,难于处理,目前人们主要关注的是常规污染物(如COD、 氨氮)的去除效果,缺乏对抗生素废水抗性污染控制的深入研究.
本文以江苏无锡某螺旋霉素生产废水为研究对象,通过现场采样分析,重点考察耐药菌(antibiotic resistance bacteria,ARB)与抗性基因(antibiotic resistance genes,ARG)在螺旋霉素抗生素废水处理过程中的分布和转归特征,以期为抗生素制药废水的抗性污染防治提供科技支撑. 1 材料与方法 1.1 样品采集
本研究的样品采集于江苏省无锡市某制药废水处理站,主要采用活性污泥法处理螺旋霉素生产废水,污水处理流程为:调节池-厌氧池-缺氧池-好氧池-二沉池. 本研究采样分两次进行,分别于2014年4月(春季)和2014年9月(秋季)采集不同污水处理单元(调节池、 厌氧池、 缺氧池、 二沉池)的出水样品,分别标记为A1~A4和S1~S4. 采集的样品置于便携式冰箱运回实验室,并于4℃冰箱中保存待用. 1.2 样品的处理 1.2.1 水质分析
根据国家标准方法[15]对污水的常规指标(SS、 COD、 氨氮、 硝酸盐、 亚硝酸盐、 磷酸盐等)进行检测. 1.2.2 样品总DNA提取
由于污水样品为泥水混合物,为了使泥水能够分离,同时又不会把水样中的微生物沉淀下去,并考虑到泥水混合物中大颗粒物与微生物的沉降速度不同,故通过低速离心使其分离,根据各采样点的水质不同分别取20~100 mL经过0.22 μm滤膜过滤,将滤膜剪碎,采用试剂盒Fast DNA Spin Kit for soil (USA)对滤膜上截留的生物量提取基因组总DNA. 提取的DNA溶液在Nanodrop分光光度计(Nanodrop,US)上测定浓度和质量,并进行琼脂糖凝胶电泳(TBE缓冲液,质量体积比1%). 1.2.3 样品中可培养细菌和肠球菌丰度的分析
总异养菌浓度的分析主要采用R2A培养基进行培养计数[16],用1 mL 灭菌移液吸管吸取污水样品1 mL,缓慢注入含有9 mL灭菌PBS缓冲液的试管内,振荡试管混合均匀,制成10-1 稀释菌液. 同法依次连续稀释至10-2、 10-3、 10-4、 10-5稀释菌液. 每个采样点水样选择3个适宜稀释度,每个稀释梯度做3个平行样,吸取0.1 mL稀释菌液,加在已经倒入灭菌培养基并凝固备用的平板上. 涂布法涂布均匀,37℃恒温生化培养箱中培养2 d后计数,以上无菌操作均在超净台中进行.
肠球菌的浓度分析主要采用滤膜法测定[17],污水样品同上经过稀释,不同采样点的样品采用PBS缓冲液稀释成适宜的稀释液,用孔径0.45 μm醋酸纤维滤膜过滤,将滤膜以截留面朝上贴附在灭菌滤膜肠球菌培养基并凝固备用的平板上,平板倒置于生化培养箱中,37℃恒温培养2~5d,根据菌落生长情况确定培养时间(t). 计数每个平板上的菌落数,并根据稀释倍数计算样品中肠球菌的丰度. 挑取乳白色菌落划线于胆盐七叶苷琼脂培养基上,挑取有棕色光泽的菌落保存进行下一步实验. 1.3 菌株药敏测试
药敏测试采用CLSI 推荐的Kirby-Bauer 纸片法[18],将待检菌株菌液的浊度用无菌生理盐水调节至0.5麦氏浊度后,用棉签蘸取菌液涂布于Muller-Hinton 琼脂(Oxoid)平板上,贴上4种抗生素药敏纸片(Oxoid):阿奇霉素(AZI)、 红霉素(ERY)、 螺旋霉素(SP)、 克拉霉素(CLR). 将平板倒置于生化培养箱中,37℃恒温培养24 h,测定抑菌圈直径,采用CLSI 标准判断菌株的抗性,该研究以ATCC25922菌株为质控菌株. 1.4 抗性基因和转移元件的检测 1.4.1 抗性基因定性和肠球菌携带抗性基因的检测
采用PCR方法检测大环内酯类抗性基因ermB、 ermF、 ermX、 mefA、 ereA、 mphB和转移元件ISCR1、 intI1、 Tn916/1545,PCR所用引物见表 1. 抗性基因定性检测反应体系为:10 μL Green qPCR Master Mix,0.5 μL引物(F),0.5 μL引物(R),DNA模板1 μL,加ddH2O至20 μL. 反应程序为:94℃ 5 min; 94℃ 30 s,55℃ 30 s,72℃ 1 min,34次循环; 72℃ 5 min,并在4℃保存. 每次运行使用无菌水做阴性对照,PCR产物以1%的琼脂凝胶电泳分析.
| 表 1 抗性基因和转移元件所用引物序列 Table 1 Primers for resistance genes and mobile elements |
采用菌液PCR的方法检测肠球菌携带抗性基因ermB、 mefA和转移元件intI1的情况,基因引物同上. 菌液PCR反应体系为:10 μL Green qPCR Master Mix,0.5 μL引物(F),0.5 μL引物(R),菌液0.5 μL,加ddH2O至20 μL. 反应程序为:94℃ 15 min; 94℃ 30 s,55℃ 30 s,72℃ 1 min,34次循环; 72℃ 5 min,并于4℃保存. 每次运行使用无菌水做阴性对照,PCR产物以1%的琼脂凝胶电泳分析. 1.4.2 抗性基因的定量检测
采用SYBR-Green实时定量PCR方法对各基因进行定量分析,检测仪器为StepOne型荧光定量PCR仪(ABI,美国). PCR产物经过克隆测序确认后,使用生工质粒提取试剂盒SK1131从阳性克隆子中提取质粒,用作标准曲线. 使用NanoDrop微量分光光度计(Thermo Scientific,美国)测定质粒浓度. 制作标准曲线时按照10倍梯度稀释构建好的各质粒,于90 μL稀释液中加入10 μL 质粒,做4~6个点,通过预实验选取合适标准品用于制备标准曲线. 标准质粒、 环境样品、 阴性对照均做3个平行,取平均值进行计算.
质粒拷贝数换算公式(copies ·μL-1)=[质粒浓度(ng ·μL-1)×10-9×6.02×1023]/[克隆产物碱基数×660(g ·mol-1)]
载体(pMD 18-T)碱基数=2 692 bp,载体(pGEMX-T Easy)碱基数=3 023 bp
荧光定量PCR反应体系为:12.5 μL 2×SYBR,1.0 μL DNA (10 ng ·μL-1),0.5 μL 引物 F (10 mol ·L-1),0.5 μL引物 R (10 mol ·L-1),10.5 μL 水,总体积 25.0 μL. 荧光定量PCR反应程序为:① 50℃,2 min,② 95℃,5 min,③ 95℃,20 s,④ 退火,30 s,⑤ 72℃,31 s,⑥ Plate read,重复③~⑤,39 次重复,⑦Melt-curve 分析:60℃ to 95℃. 每隔0.2℃采集一次荧光以生成溶解曲线,根据溶解曲线的变化检测扩增结果的特异性. 退火温度和反应时间根据引物不同进行调整. 2 结果与讨论 2.1 水质净化效果
从表 2可知,进水的COD和氨氮浓度较高,经过厌氧池、 缺氧池和好氧池之后,逐级下降,二沉池出水中COD和氨氮的浓度分别为142.5 mg ·L-1和26.1 mg ·L-1,COD和氨氮的去除率分别为90%~94%和87%~90%,基本达到了发酵类制药工业水污染物排放标准(GB 21903-2008)的要求.
| 表 2 抗生素制药废水处理过程的常规化学指标 Table 2 Chemical characteristics of wastewater treatment process of antibiotic production/mg ·L-1 |
图 1显示了总异养菌和肠球菌在4个污水处理单元的去除效果. 调节池出水中总异养菌浓度(CFU ·100 mL-1)介于8.2~8.4 logs,肠球菌浓度(CFU ·100 mL-1)介于5.3~5.6 logs,这表明春季和秋季进水中都有着丰富的异养菌和肠球菌.
 | 图 1 抗生素制药废水处理过程中总异养菌和肠球菌的丰度分布 Fig. 1 Amount of enterococci and cultivable bacteria during wastewater treatment process of antibiotic production |
以往的研究表明,污水处理过程中可以去除0~5个数量级的总异养菌和肠球菌[17]. 本研究表明,经过厌氧池、 缺氧池、 好氧池处理阶段后,总异养菌和肠球菌都大幅度地被去除,二沉池出水中总异养菌和肠球菌的浓度分别降低了1.6~2.1 logs和3.7 logs,有效降低了细菌抗性的传播风险. 2.2 肠球菌的耐药率
从春季和秋季采集的4个污水生物处理单元的出水中分离出了94株肠球菌,它们对4种大环内酯类抗生素的耐药率变化情况如表 3所示,所有的肠球菌对阿奇霉素、 克拉霉素、 红霉素和螺旋霉素都具有抗性. 然而经过污水处理之后,肠球菌的耐药率不仅没有得到削减,而且在春季和秋季没有显著差异,这说明肠球菌的耐药率与季节并没有直接的相关性. Ma等[23]认为污水生物处理能有效削减微生物量包括病原菌,但却不能有效削减一些携带抗性基因和整合子的细菌. 本研究结果表明,螺旋霉素生产废水生物处理能有效地削减总异养菌和肠球菌的数量,但却不能削减耐药菌的比例.
| 表 3 肠球菌的耐药率 Table 3 Antibiotic resistance rates of Enterococci in this study |
从4个污水生物处理单元出水中分离出来的94株肠球菌均对大环内酯类抗生素螺旋霉素、 阿奇霉素、 克拉霉素、 红霉素具有抗性. 通过检测这94株肠球菌抗性菌所携带的MLS抗性基因ermB、 mefA和转移元件intI1,结果显示(见表 4),有75株肠球菌对ermB抗性基因呈阳性,但全部94株肠球菌对mefA和intI1都呈阴性. 已有研究报道ermB抗性基因的检出率高于MLS抗性基因的检出率[24]. 虽然Luna等[25]分离出的肠球菌携带抗性基因mef,但是Portillo等[26]分离的肠球菌却没有检测到抗性基因mef,这与本研究的结果一致,他认为不同环境中抗性基因mef的分布特征不同. 春季分离出的30株肠球菌中有73%携带抗性基因,而秋季分离出的64株肠球菌中有79%携带ermB抗性基因,这说明气温对肠球菌携带抗性基因没有显著的影响.
| 表 4 肠球菌携带抗性基因的检测结果 Table 4 Detection of antibiotic resistance genes in Enterococci |
在4个污水处理单元中ermB的检出率分别为84%、 73%、 76%、 75%. ermB抗性基因在厌氧池有一定的去除效果,但在缺氧池开始出现了反弹. 总体来说,经过污水处理,肠球菌中抗性基因ermB的检出率下降了9%. ermB抗性基因常常与一些质粒、 转座子等转移元件相关联[24],在缺氧池中出现反弹的原因很可能与转座子、 质粒的相关联有关,然而环境中的生物千变万化,也可能与微生物量的变化有着紧密的联系,具体原因有待进一步研究. 2.3.2 抗性基因的分布和转归特征
利用荧光定量PCR的方法检测了大环内酯类抗性基因ermB、 ermF、 ermX、 mefA、 ereA、 mphB和转移元件ISCR1、 intI1、 Tn916/1545在4个污水处理单元中不同季节的分布特征和变化情况(图 2). 从图 2(a)中可知,春季进水中抗性基因和转移元件的浓度范围为4.53×105~3.39×107 copies ·mL-1,二沉池出水中抗性基因的浓度范围为0~7.53×106 copies ·mL-1,污水处理对抗性基因ermB、 ermF、 mefA、 ereA、 mphB和转移元件Tn916/1545都有一定的去除效果,削减范围为0.4~6 logs,抗性基因和转移元件的削减效果依次为mefA>Tn916/1545> mphB> ereA> ermF>ermB,其中mefA在二沉池出水中未检出,去除了6 logs,去除效果最好; 而ermX、 intI1、 ISCR1不但没有被去除,反而出现了反弹. 如图 2(b)中所示,秋季进水中抗性基因和转移元件的含量范围为 4.17×104~2.95×107 copies ·mL-1,二沉池出水中抗性基因和转移元件的含量范围为1.86×103~8.19×106 copies ·mL-1,抗性基因ermB、 ermF、 mefA、 ereA、 mphB和转移元件Tn916/1545有一定的去除效果,出水中下降了0.55~2.1 logs,其中mefA去除效果较好,下降了2.1 logs,而ermX、 intI1、 ISCR1出现了反弹.
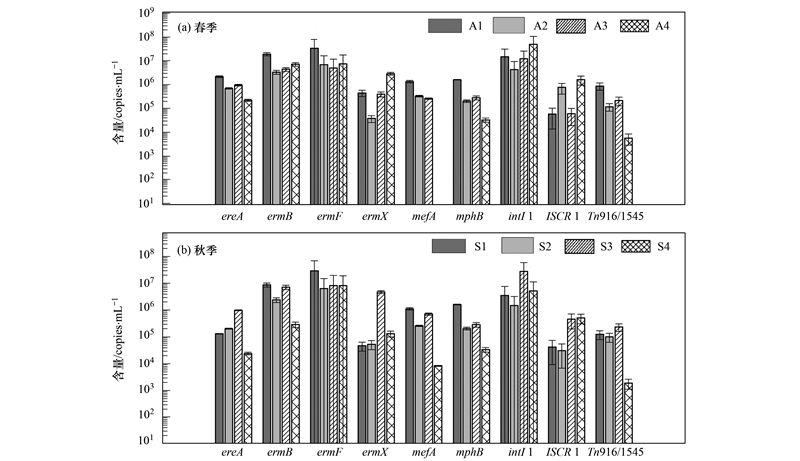 | 图 2 抗生素制药废水处理过程中抗性基因和转移元件的变化 Fig. 2 Changes of resistance genes and transfer elements during wastewater treatment of antibiotic production |
从上述结果可知,春季和秋季中ermB和ermF抗性基因的浓度均为最高. 已有研究证实了erm-TR抗性基因的检出率最高,是最广泛存在的大环内酯类抗性基因[13]. ermB和 ermF的丰度高,一个原因是宿主广泛,已报道的研究中表明宿主覆盖了革兰氏阳性菌(G+)和革兰氏阴性菌(G-)、 好氧菌和厌氧菌,另一个原因是这些基因常常与水平转移因子有关,比如可连接的转座子和质粒. 抗性基因mefA 为外排泵基因,在春季和秋季样品中的去除效果均是最好的. 但是广泛的宿主和潜在的水平转移能力与该基因在系统中的低浓度似乎有些矛盾. 可能解释的原因是由于 G-菌存在外膜,螺旋霉素不能进入 G-菌细胞,G-细菌具有固有抗性,因此不需要外排泵作用[27]. 根据水质情况分析,春季中进水的COD明显比秋季进水高,已有研究表明有机物浓度的增加利于耐药菌和抗性基因的传播扩散. 本研究中春季样品中抗性基因丰富的原因很可能与高浓度的有机污染物存在有关[28].
将各抗性基因归一化到16S rRNA,考察抗性基因在污水处理过程中的相对丰度变化,可以了解抗性基因在总DNA中的相对表达量,结果见图 3. 如图 3(a)所示春季样品在污水处理过程中抗性基因ermF、 mefA、 ereA、 mphB和转移元件Tn916/1545的丰度分别降低了25.24%、 100%、 65.14%、 93.31%和97.90%,而抗性基因ermB、 ermX和转移元件intI1、 ISCR1的丰度均不同程度上升了. 如图 3(b)所示,秋季样品在污水处理过程中抗性基因ermB、 mefA、 ereA和转移元件Tn916/1545的丰度降低了82.73%、 93.31%、 3.97%和92.25%,而抗性基因ermF、 mphB、 ermX和转移元件intI1、 ISCR1的丰度均不同程度上升了.
 | 图 3 各污水样品中 MLS 抗性基因和转移因子的相对丰度 Fig. 3 Relative abundance of MLS resistance genes and mobile elements in wastewater samples |
污水处理过程中只可以去除部分抗性基因和转移元件,但同时也使部分抗性基因发生了传播. mefA、 ereA、 Tn916/1545的丰度在春季和秋季样品中有显著的差异,这些抗性基因和转移元件在春季样品中的去除效果明显好于秋季. 目前,抗性基因在何种环境和条件下会水平转移,在何种情况下会削减尚无一致定论,还有待今后更深入地研究. 3 结论
(1)螺旋霉素制药废水生物处理(厌氧-缺氧-好氧)能有效削减总异养菌和病原菌肠球菌的数量,总异养菌和肠球菌的浓度分别降低了1.6~2.1 logs和3.7 logs.
(2)分离出来的94株肠球菌均对大环内酯类抗生素螺旋霉素、 阿奇霉素、 克拉霉素、 红霉素具有抗性. 虽然污水处理对肠球菌的耐药率没有削减作用,但经过污水处理后肠球菌中ermB的检出率下降了9%.
(3)抗生素制药废水生物处理对抗性基因ermB、 ermF、 mefA、 ereA和转移元件Tn916/1545的含量有一定的削减作用,但抗性基因ermX和intI1、 ISCR1的含量反而发生了一定程度的反弹.
(4)抗生素制药废水生物处理过程中mefA、 ereA、 Tn916/1545的丰度降低了,且春季mefA、 ereA、 Tn916/1545的去除效果明显好于秋季,而ermX和转移元件intI1、 ISCR1的丰度分别增加了.
| [1] | 佟娟, 魏源送. 污水处理厂削减耐药菌与抗性基因的研究进展[J]. 环境科学学报, 2012, 32 (11): 2650-2659. |
| [2] | Zhang T, Zhang X X, Ye L. Plasmid metagenome reveals high levels of antibiotic resistance genes and mobile genetic elements in activated sludge[J]. PLoS ONE, 2011, 6 (10): e26041. |
| [3] | Thiele-Bruhn S, Beck I C. Effects of sulfonamide and tetracycline antibiotics on soil microbial activity and microbial biomass[J]. Chemosphere, 2005, 59 (4): 457-465. |
| [4] | Borg M A, Zarb P, Scicluna E A, et al. Antibiotic consumption as a driver for resistance in Staphylococcus aureus and Escherichia coli within a developing region[J]. American Journal of Infection Control, 2010, 38 (3): 212-216. |
| [5] | Shryock T R, Richwine A. The interface between veterinary and human antibiotic use[J]. Annals of the New York Academy of Sciences, 2010, 1213: 92-105. |
| [6] | Pruden A, Pei R T, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants: Studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40 (23): 7445-7450. |
| [7] | Kemper N. Veterinary antibiotics in the aquatic and terrestrial environment[J]. Ecological Indicators, 2008, 8 (1): 1-13. |
| [8] | Kü mmerer K. Antibiotics in the aquatic environment-A review-Part Ⅱ[J]. Chemosphere, 2009, 75 (4): 435-441. |
| [9] | Gonzalez J J, Andreu A, the Spanish Group for the Study of Perinatal Infection from the Spanish Society for Clinical Microbiology and Infectious Diseases. Multicenter study of the mechanisms of resistance and clonal relationships of Streptococcus agalactiae isolates resistant to macrolides, lincosamides, and ketolides in Spain[J]. Antimicrobial Agents and Chemotherapy, 2005, 49 (6): 2525-2527. |
| [10] | Brisson-Noël A, Arthur M, Courvalin P. Evidence for natural gene transfer from gram-positive cocci to Escherichia coli[J]. Journal of Bacteriology, 1988, 170 (4): 1739-1745. |
| [11] | Gupta A, Vlamakis H, Shoemaker N, et al. A new bacteroides conjugative transposon that carries an ermB gene[J]. Applied and Environmental Microbiology, 2003, 69 (11): 6455-6463. |
| [12] | Jiang H Y, Zhang D D, Xiao S C, et al. Occurrence and sources of antibiotics and their metabolites in river water, WWTPs, and swine wastewater in Jiulongjiang River basin, south China[J]. Environmental Science and Pollution Research, 2013, 20 (12): 9075-9083. |
| [13] | Koike S, Aminov R I, Yannarell A C, et al. Molecular ecology of macrolide-lincosamide-streptogramin B methylases in waste lagoons and subsurface waters associated with swine production[J]. Microbial Ecology, 2010, 59 (3): 487-498. |
| [14] | Hraoui M, Boubaker I B B, Rachdi M, et al. Macrolide and tetracycline resistance in clinical strains of Streptococcus agalactiae isolated in Tunisia[J]. Journal of Medical Microbiology, 2012, 61 (8): 1109-1113. |
| [15] | 国家环境保护总局. 水和废水监测分析方法[M]. (第四版).北京: 中国环境科学出版社, 2002. |
| [16] | Munir M, Wong K, Xagoraraki I. Release of antibiotic resistant bacteria and genes in the effluent and biosolids of five wastewater utilities in Michigan[J]. Water Research, 2011, 45 (2): 681-693. |
| [17] | da Silva M F, Tiago I, Veríssimo A, et al. Antibiotic resistance of enterococci and related bacteria in an urban wastewater treatment plant[J]. FEMS Microbiology Ecology, 2006, 55 (2): 322-329. |
| [18] | Clinical and laboratory standards institute (CLSI). Performance standards for antimicrobial susceptility testing; twenty-second informational supplement, M100-S22[Z]. Wayne, PA: Clinical and Laboratory Standards Institute, 2012. |
| [19] | Chen J, Yu Z T, Michel F C Jr, et al. Development and application of real-time PCR assays for quantification of erm genes conferring resistance to macrolides-lincosamides-streptogramin B in livestock manure and manure management systems[J]. Applied and Environmental Microbiology, 2007, 73 (14): 4407-4416. |
| [20] | Sutcliffe J, Grebe T, Tait-Kamradt A, et al. Detection of erythromycin-resistant determinants by PCR[J]. Antimicrobial Agents and Chemotherapy, 1996, 40 (11): 2562-2566. |
| [21] | Quiroga M P, Andres P, Petroni A, et al. Complex class 1 integrons with diverse variable regions, including aac(6')-Ib-cr, and a novel allele, qnrB10, associated with ISCR1 in clinical enterobacterial isolates from Argentina[J]. Antimicrobial Agents and Chemotherapy, 2007, 51 (12): 4466-4470. |
| [22] | Stokes H W, Nesbø C L, Holley M, et al. Class 1 integrons potentially predating the association with Tn402-like transposition genes are present in a sediment microbial community[J]. Journal of Bacteriology, 2006, 188 (16): 5722-5730. |
| [23] | Ma L P, Zhang X X, Cheng S P, et al. Occurrence, abundance and elimination of class 1 integrons in one municipal sewage treatment plant[J]. Ecotoxicology, 2011, 20 (5): 968-973. |
| [24] | Thumu S C R, Halami P M. Acquired resistance to macrolide-lincosamide-streptogramin antibiotics in lactic acid bacteria of food origin[J]. Indian Journal of Microbiology, 2012, 52 (4): 530-537. |
| [25] | Luna V A, Coates P, Eady E A, et al. A variety of gram-positive bacteria carry mobile mef genes[J]. Journal of Antimicrobial Chemotherapy, 1999, 44 (1): 19-25. |
| [26] | Portillo A, Ruiz-Larrea F, Zarazaga M, et al. Macrolide resistance genes in Enterococcus spp.[J]. Antimicrobial Agents and Chemotherapy, 2000, 44 (4): 967-971. |
| [27] | Higgins C F. Multiple molecular mechanisms for multidrug resistance transporters[J]. Nature, 2007, 446 (7137): 749-757. |
| [28] | Kim S, Jensen J N, Aga D S, et al. Fate of tetracycline resistant bacteria as a function of activated sludge process organic loading and growth rate[J]. Water Science & Technology, 2007, 55 (1-2): 291-297. |
 2015, Vol. 36
2015, Vol. 36


