石油主要由烷烃、芳香烃及环烷烃组成,此外还有少量的非烃类化合物. 在石油的开采、运输及使用过程中由于泄漏导致了各种环境污染[1, 2]. 石油黏着力强,进入土壤后一方面会影响土壤中的微生物组成和结构,另一方面,会阻碍植物根系呼吸及对营养的吸收,导致植物生长不良甚至死亡[3]. 石油中的有害物质可以通过食物链进入人体,直接危害人体健康,尤其是多环芳烃类物质具有致癌、致畸、致突变等作用[4]. 微生物修复法由于操作简便费用低、对周围环境污染小、基本不产生二次污染等优势,成为土壤石油污染修复的重要技术[5, 6, 7].
目前,很多研究者致力于筛选高效石油降解菌对油污土壤进行生物修复的研究,文献[8, 9, 10, 11, 12, 13, 14, 15]从不同石油污染土壤中筛选不同的高效石油降解菌、烷烃降解菌并对土壤进行修复研究; 也有学者对石油污染土壤的修复机制进行研究,王莉丽等[16]利用降解菌研究油污土壤中可溶性有机物的变化; 文献[17, 18, 19, 20]则对降解菌表面活性剂进行研究. 本文利用投菌法和生物刺激法对石油污染土壤进行修复,并对修复过程中催化基因、降解菌数目变化以及油污土壤菌群多样性与微生物修复效果之间的相关性进行了实验研究. 1 材料与方法 1.1 供试土壤及石油烃降解菌
取陕北子长县某油井周围石油污染土壤,过20目筛并混合搅拌均匀,作为石油污染降解修复供试土壤. 利用Bush-hass培养基从土壤中分离筛选出的石油烃降解菌 SZ-1(不动杆菌属) 为接种菌对土壤进行修复研究. 土壤理化性质测定、菌株分离及菌株理化性质测定见文献[21].
1.2 培养基及试剂牛肉膏蛋白胨培养基制备方法见文献[22]; Bush-hass培养基、1.2%琼脂糖凝胶、50×TAE缓冲液、PBS缓冲溶液的制备方法见文献[21]; 土壤DNA提取试剂盒: PowerSoil DNA Isolation Kit,美国MOBIO公司; 克隆试剂盒: PMD19T,TAKARA公司; 混合PAHs溶液: 菲10 g,蒽1 g,芘1 g,二苯并噻吩1 g,用二氯甲烷溶解并定容到1 L[23]; 碘代硝基四唑兰 (INT)(3 g ·L-1)溶液: 准确称取碘代硝基四唑兰粉末3 g,用灭菌纯水定容至1 L; 1.5%的琼脂糖凝胶: 量取1×TAE缓冲液50 mL置于250 mL锥形瓶中,取琼脂糖粉0.75 g加入到缓冲液中,锡纸封住瓶口,微波炉中火加热2 min使得琼脂糖粉完全溶解.
1.3 石油污染土壤微生物修复实验方案分别称取500 g供试土壤分装于圆形瓷盆,按表 1所示方案对土壤进行8周的生物修复处理,修复期间每天均匀翻动土壤保持透气性.
| 表 1 石油污染土壤微生物修复处理方案1)Table 1 Bioremediation treatments of petroleum contaminated soil |
取以上不同处理的土壤样品,风干研磨后,分别准确称取1 g各处理土样于称量瓶中,加入25 mL四氯化碳,利用超声波细胞粉碎机(JY92-Ⅱ,美国SONICS)超声萃取15 min,功率为170 W. 重复萃取两次,过滤后收集滤液于50 mL容量瓶中,定容,每个样品做3个平行. 利用OCMA-350红外测油仪测定土壤中石油烃. 1.5 土壤中催化基因的测定方法
利用土壤DNA提取试剂盒提取1、3、4号土壤样品DNA. 采用5倍系列稀释法将提取的土壤DNA稀释. 查找文献得到可降解链烷烃和可降解芳烃的催化基因引物[包括2种芳烃催化基因①(nahAcfor/nahAcrew)α-亚基萘双加氧酶基因、②(AJ025/AJ026)α-亚基环羟基双加氧酶基因和2种烷烃催化基因、③(alk-3F/alk-3R)链烷烃羟化酶基因、④(alkFa/aklRa) 链烷烃单加氧酶基因],其引物碱基序列、PCR扩增程序以及基因检测方法见文献[21, 25, 26, 27, 28]. 1.6 土壤中降解菌数目测定方法(MPN法)
分别称取1 g的1、3、4号土壤,加入9 mL PBS缓冲溶液,摇匀. 30℃,130 r ·min-1,振荡培养30 min,取上清液进行10倍系列稀释,依次获得101、102、…、1010土壤稀释液. 分别以石油烃(取自陕北某油田),正十六烷及混合PAHs溶液为MPN底物. 按文献所述的方法测定石油烃降解菌、烷烃降解菌和多环芳烃降解菌的数目[23]. 1.7 土壤微生物群落分析方法(PCR-DGGE)
利用土壤DNA提取试剂盒提取土壤样品的DNA.
利用细菌通用引物对338F/518R (5′-ACTCC TACGGGAGGCAGCAG-3′)/(5′-ATTACCGCGGCTG CTGG-3′)对土壤DNA进行PCR扩增,50 μL PCR反应体系配置: 22 μL ddH2O,25 μL 2×Go Taq酶,2 μL引物F,2 μL引物R. PCR反应升温程序: 94℃预变性5 min,94℃变性30 s,57℃退火30 s,72℃延伸30 s,重复 30个循环,最后72℃再延伸7 min. PCR扩增产物利用1.5%琼脂糖凝胶电泳进行检测[29].
检测条带清晰无拖尾表示扩增结果良好,将PCR扩增产物进行变性梯度凝胶电泳(DGGE)分析. DGGE程序为: 丙烯酰胺凝胶浓度10%,变性梯度范围40%~60%,PCR产物上样量为10 μL. 60℃,60V条件下运行15 h. 电泳结束用Gelred染色1 h,利用基因成像系统捕获图像并利用Quantity one软件(Quantity one-4.6.2)分析DGGE图像,切割DGGE条带,利用PMD19T载体试剂盒(PMD19T,TAKARA公司)进行克隆测序分析. 2 结果与分析 2.1 不同修复处理土壤中石油烃去除效果
不同生物处理对土壤石油烃的去除效果如图 1所示. 本次针对陕北子长某油井周围石油污染土壤修复实验中,生物刺激(添加氮磷营养的4、5号处理)对石油污染土壤的修复效果最好,修复8周时对石油烃去除率分别达到59.7%和57.5%; 其次为生物强化处理修复的土壤(投加外源菌的3、6号处理),修复8周时对石油烃去除率达到33%左右; 但是在自然条件下,保持土壤含水率为40%时(1号处理)以及灭菌处理(2号处理)对土壤的修复效果均不明显. 由以上结果可知,本次修复过程中土著菌对石油烃的降解效果较差,而在不添加氮磷营养时,外源菌比土著菌的环境适应性更强,石油烃去除效果更明显. 研究结果表明在修复土壤时,只有在碳氮磷比适宜条件下土著菌才能有效降解石油烃,而外加降解菌进行生物强化也可以对该土壤起到一定的修复作用.
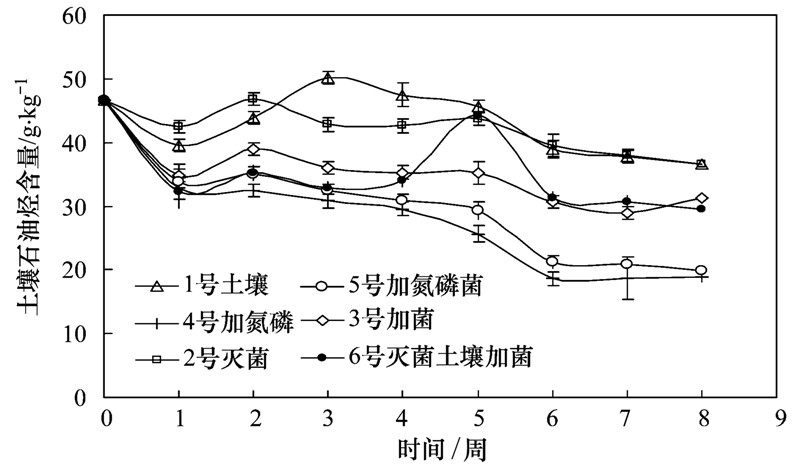 | 图 1 不同修复处理石油污染土壤中石油烃含量 Fig. 1 TPH of different treatment soils |
对1、3和4号土壤中可降解烷烃和多环芳烃的催化基因进行测定,结果如表 2所示.
修复前(第0周,即生物修复未开始时),油污土壤中只含有 AJ025/AJ026引物、 alkFa/aklRa引物控制的基因. 随修复时间增加,这两种引物控制基因呈现出先增加后稳定或者降低的趋势. 且3号处理中 AJ025/AJ026引物基因数量明显多于其他两种处理. 根据以前的研究工作可知菌株SZ-1含有AJ025/AJ026引物控制的基因[26],由此推断,3号处理土壤中增加的该引物基因是由于投加外源菌SZ-1引起的.
在微生物修复初期(第1周),AJ025/AJ026控制降解芳烃的催化基因含量增加较快,并且1、3号处理出现新的nahAcfor/nahAcrew可降解芳烃的催化基因; 在修复期(2~7周时),alkFa/aklRa控制降解烷烃的催化基因含量增加较快,同时检测到新 的alk-3F/alk-3R可降解烷烃催化基因. 研究表明,
| 表 2 生物修复土壤基因检测结果 1)Table 2 Catabolic genes in different treatment soils |
1、3、4号土壤中石油烃、烷烃、多环芳烃降解菌数的测定结果如图 2~4所示.
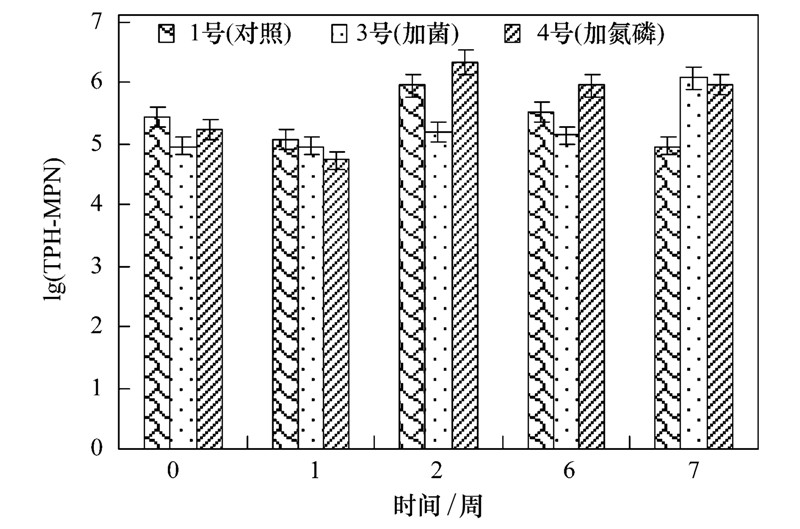 | 图 2 不同处理土壤中石油烃降解菌数 Fig. 2 Number of TPH degrading bacteriain different treatment soil |
由图 2、3、4可知,4号处理中石油烃、烷烃、多环芳烃降解菌数目增加幅度均最大,并且石油烃和烷烃降解菌数目可达105 CFU ·mL-1以上,多环芳烃降解菌数目最多可达到102 CFU ·mL-1. 在石油污染土壤修复过程中石油烃和烷烃降解菌个数明显多于多环芳烃降解菌.
根据图 5可知,修复后期4号处理中总降解菌数远远多于其他两种处理土壤中的总降解菌,并且不同处理中总降解菌数目呈现修复期(第0~6周)先减少后增加,修复后期(第7周)减少的变化规律. 土壤中总降解菌数目变化趋势与土壤中石油烃去除效果变化规律一致,由此可以看出,不同的生物修复处理可以促进油污土壤中总降解菌数的增多,土壤中降解菌数目越多,降解菌用于合成自身生长所需物质、加快生物新陈代谢所需要利用的唯一碳源石油烃量更多,从而使得土壤中剩余石油烃量减少,对石油烃去除效果越好.
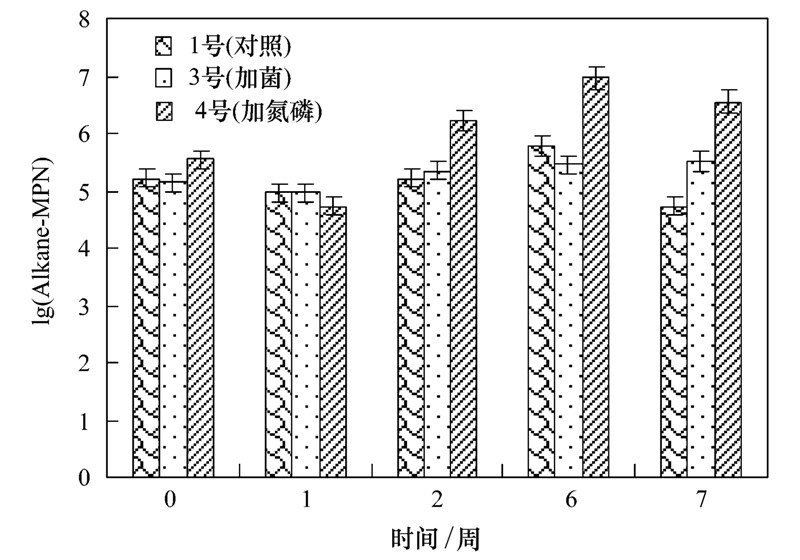 | 图 3 不同处理土壤中烷烃降解菌数 Fig. 3 Number of alkane degrading bacteria indifferent treatment soils |
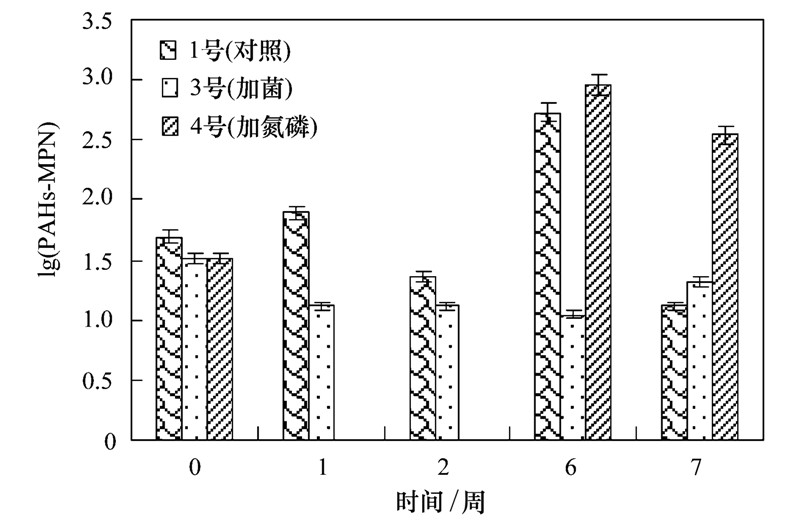 | 图 4 不同处理土壤中多环芳烃降解菌数 Fig. 4 PAHs degrading bacteria in different treatment soils |
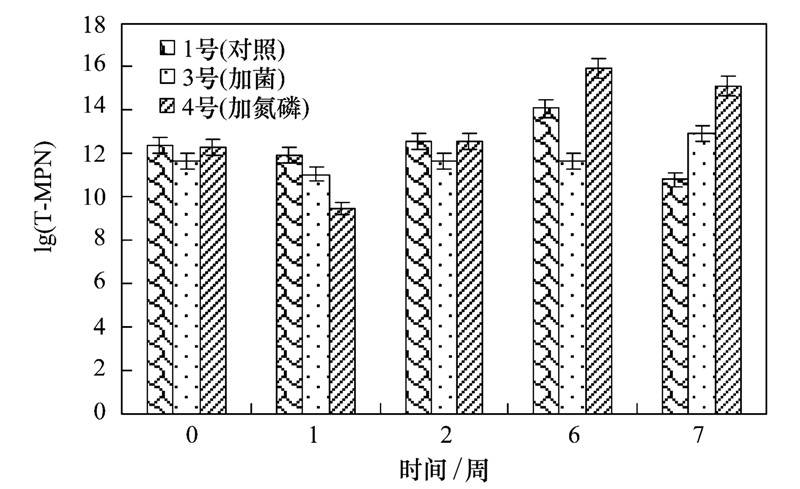 | 图 5 不同处理土壤中总降解菌数 Fig. 5 Total TPH degrading bacteria in different treatment soils |
提取1、3、4号处理土壤DNA,利用细菌通用引物对338F/518R进行PCR扩增,结果如图 6所示,扩增条带清晰明亮,未出现非特异性扩增,扩增片段约180 bp,扩增过程未污染. 继续对其进行PCR-DGGE分析,结果如图 7所示.
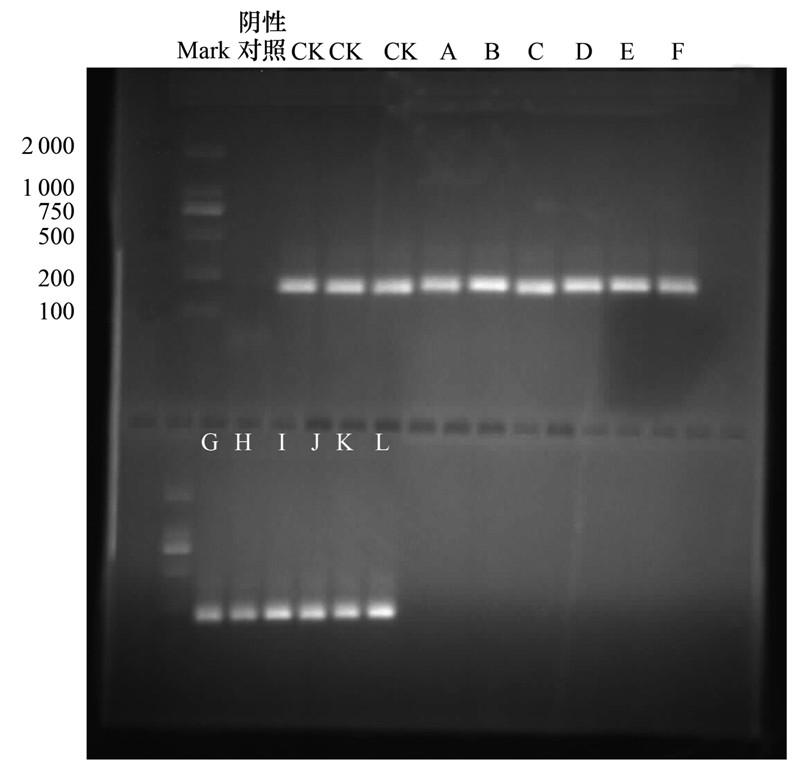 | 泳道CK: 空白对照,第0周不做处理的石油污染土壤,切割条带: 4、5、10; 泳道 A、D、G、J: 1号(土壤)处理第1、2、6、7周修复土壤,切胶条带: 7、9、12、15、18、22; 泳道 B、E、H、K: 3号(土壤加菌SZ-1)处理第1、2、6、7周修复土壤,切胶条带: 2、3、6; 泳道 C、F、I、L: 4号(土壤加氮磷)处理第1、2、6、7周修复土壤,切胶条带: 1、8、11、13、14、16、17、19、20、21、23 图 6 PCR扩增产物电泳 Fig. 6 Profiles of PCR |
 |
图注同图 6 图 7 PCR扩增产物变性梯度凝胶电泳图谱 Fig. 7 Profiles of DGGE |
由图 7(PCR-DGGE图谱)可以看出,空白对照(CK)中主要条带数目为7~9条; 与空白对照(CK)相比,在修复第1周时3种处理土壤中的条带数变化不大(泳道A、B、C),而修复2~7周,修复土壤中条带数目明显增多(泳道D~L),菌群结构变化较大.
对3号处理第1、2、6、7周 (对应B、E、H、K泳道) 时的条带2进行切割并克隆测序,结果发现该条带属于不动杆菌属,Blast比对结果表明条带2与降解菌SZ-1的相似度超过99%,因此可初步断定该条带有可能是外加降解菌SZ-1.
利用Quantity one软件对DGGE泳道主条带的DNA浓度进行定量分析(trace quantity),结果如图 8所示. 修复1周时,与空白对照相比(CK),不同处理的土壤样品中DNA浓度变化不大(泳道 A、B、 C),但是随修复时间的增加,不同处理的土壤中条带DNA总浓度基本上呈增加趋势.
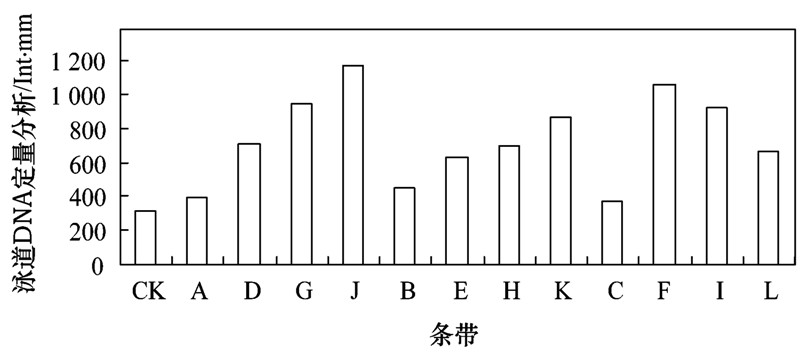 | 图 8 DGGE泳道条带定量分析结果 Fig. 8 Results for band trace quantity |
对DGGE图谱进行聚类分析 (cluster analysis)的结果如图 9所示. 修复1周时,土壤的菌群结构与修复前相比,变化不大. 当修复进行2周后,菌群结构变化较大,且相同处理的土壤在不同修复时期的菌群结构相似度较高.
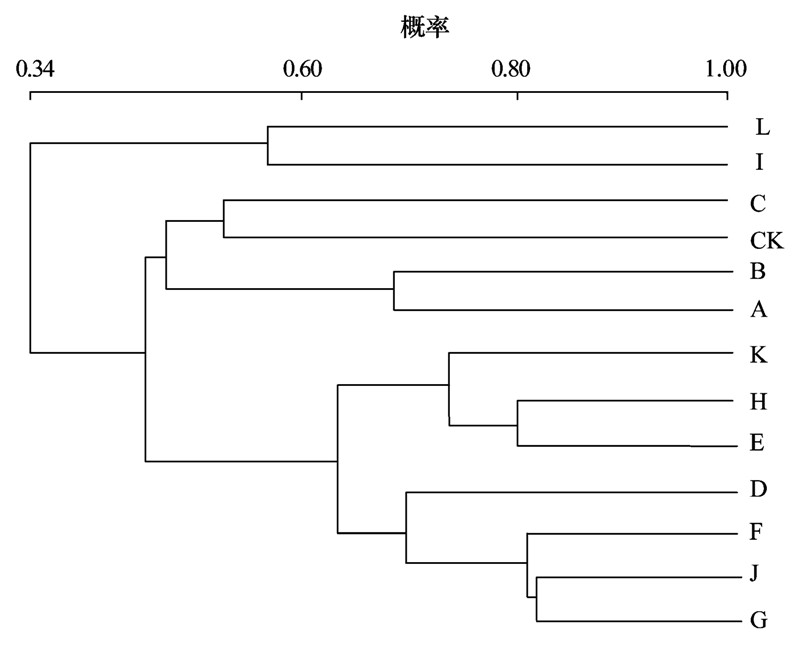 | 图 9 PCR-DGGE图谱的UPGAMA聚类分析结果 Fig. 9 Results of cluster analysis |
根据泳道条带定量分析(trace quantity)结果计算细菌群落结构多样性指标辛普森指数(Simpsons diversity index,D) [公式(1)]和香农指数(Shannon diversity index,H)[公式(2)],结果如表 3所示.
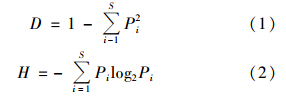
从表 3中可知,与原油污土壤相比,修复土壤中香农指数和辛普森指数都有增大的趋势,说明在修复的前6周对油污土壤进行不同修复处理均可以使细菌群落多样性增加; 而在第7周时土壤的群落多样性开始降低. 结合石油烃去除效果可知,土壤中细菌群落多样性结构越复杂,对石油烃的降解效果相对越好.
| 表 3 不同处理土壤的香农指数和辛普森指数 Table 3 Results of Simpsons and Shannon diversity index |
3号处理中的香农指数和辛普森指数最大. 说明向油污土壤投加降解菌SZ-1可以显著提高土壤细菌群落的多样性,但生物强化对该种土壤中石油烃的去除效果却低于生物刺激.
切割DGGE条带克隆培养测序,序列信息输入NCBI数据库进行BLAST比对,并申请菌种序列号,比对结果如表 4所示. 根据16SrRNA序列检测结果进行系统发育树分析,分析结果如图 10所示. 由表 4可知,在3号处理不同时期土壤样品中多次出现条带2、3、23,经检测其分别属于不动杆菌属(条带2、23)、产甲烷原核微生物(条带3),并且条带2仅在3号处理土壤中出现. 在4号处理中除了检测到的共有条带外,还多次出现条带21、23,序列分析发现二者属于不动杆菌属. 因此可知修复处理编号为3、4号的处理中不动杆菌活性较强,为优势菌群.
| 表 4 DGGE条带16S rRNA序列分析 Table 4 16S rRNA sequence analysis results of DGGE bands |
根据图 10可知,检测出的微生物总体分为八大类群: 不动杆菌属(Acinetobacter)、假单胞菌属(Pseudomonas)、副球菌属(Paracoccus)、变形菌门(Proteobacterium)、黄杆菌属(Flavobacteriaceae)、芽孢杆菌属(Bacillus)、芽单胞菌属(Gemmatimonas)、放线菌(Actinobacterium). 其中,条带2和条带12亲缘性较近(98%),条带4与 JX624259假单胞菌属(100%)、条带1与JQ400417芽单胞菌属(100%)、条带15与KF856725副球菌属(100%)几乎为同一种属,条带11与AB628746的假单胞菌的亲缘性较近(99%).
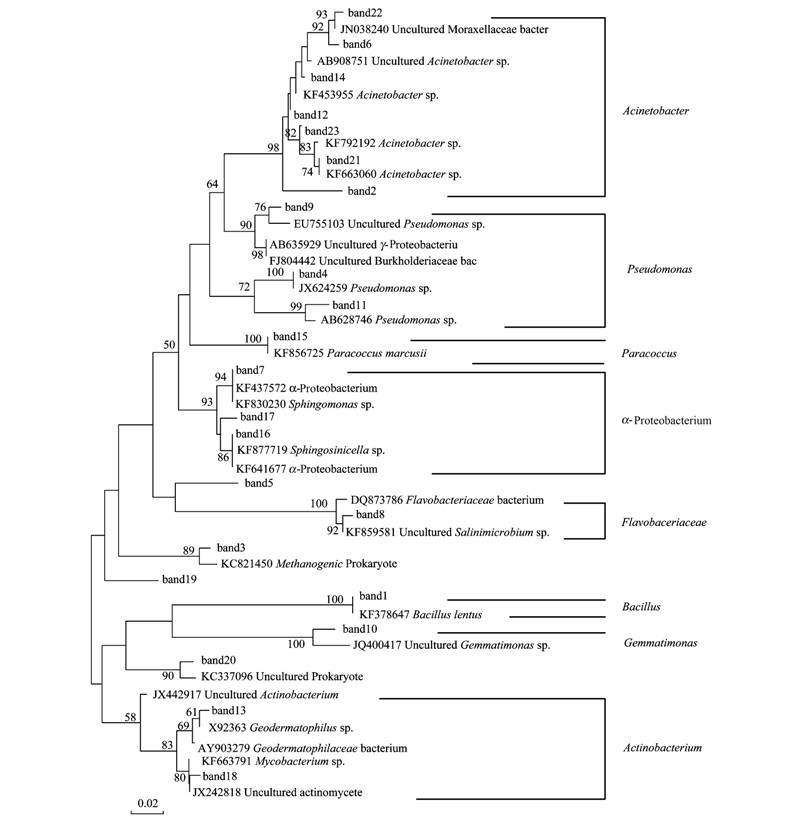 | 图 10 DGGE条带系统发育树 Fig. 10 Phylogenetic tree of DGGE |
(1)陕北子长石油污染土壤样品修复效果由好到差的顺序依次为: 向土壤中补充N、P营养物质 >向土壤中投加不动杆菌SZ-1 > 其他处理. 土著菌只有在碳氮磷比适宜条件下才能有效降解石油烃; 外源菌SZ-1比土著菌环境适应性强,能有效地降解土壤中的石油烃.
(2)土壤中可降解烷烃和多环芳烃的催化基因总量和总降解菌数量越多,土壤中石油烃的去除效果越好. 修复土壤中石油烃和烷烃降解菌数量明显多于多环芳烃降解菌数量.
(3)向油污土壤中投加SZ-1降解菌可以显著提高土壤细菌群落的多样性,该外加菌与土壤中的土著菌存在着协同修复作用. 细菌群落多样性结构越复杂,对石油烃的降解效果相对较好.
| [1] | 郝春博, 王广才, 董建楠, 等. 石油污染地下水中细菌多样性研究[J]. 环境科学, 2009, 30 (8): 2464-2472. |
| [2] | 山宝琴, 张永涛, 曹巧玲, 等. 6种陕北适生豆科植物生长对原油污染土壤的响应[J]. 环境科学, 2014, 35 (3): 1125-1130. |
| [3] | 王红旗, 刘新会, 李国学, 等. 土壤环境学[M]. 北京: 高等教育出版社, 2007. 88-89. |
| [4] | 廉景燕, 哈莹, 黄磊, 等. 石油污染土壤物化修复前后生物毒性效应[J]. 环境科学, 2011, 32 (3): 870-874. |
| [5] | 卢晓霞, 李秀利, 马杰, 等. 焦化厂多环芳烃污染土壤的强化微生物修复研究[J]. 环境科学, 2011, 32 (3): 864-869. |
| [6] | Chaineau C H, Rougeux G, Yéprémian C, et al. Effects of nutrient concentration on the biodegradation of crude oil and associated microbial populations in the soil[J]. Soil Biology and Biochemistry, 2005, 37 (8): 1490-1497. |
| [7] | 韩慧龙, 陈镇, 杨健民, 等. 真菌-细菌协同修复石油污染土壤的场地试验[J]. 环境科学, 2008, 29 (2): 454-461. |
| [8] | Wang Z Y, Gao D M, Li F M, et al. Petroleum hydrocarbon degradation potential of soil bacteria native to the Yellow River delta[J]. Pedosphere, 2008, 18 (6): 707-716. |
| [9] | Karamalidis A K, Evangelou A C, Karabika E, et al. Laboratory scale bioremediation of petroleum-contaminated soil by indigenous microorganisms and added Pseudomonas aeruginosa strain Spet[J]. Bioresource Technology, 2010, 101 (16): 6545-6552. |
| [10] | 吴涛, 谢文军, 依艳丽, 等. 耐盐石油烃降解菌的筛选鉴定及其特性研究[J]. 环境科学, 2012, 33 (11): 3949-3955. |
| [11] | Hassanshahian M, Ahmadinejad M, Tebyanian H, et al. Isolation and characterization of alkane degrading bacteria from petroleum reservoir waste water in Iran (Kerman and Tehran provenances)[J]. Marine Pollution Bulletin, 2013, 73 (1): 300-305. |
| [12] | 汪杰, 郑维爽, 礼晓, 等. 高效石油降解菌的筛选鉴定及修复能力研究[J]. 环境科学学报, 2010, 30 (6): 1228-1234. |
| [13] | Xu Y, Sun G D, Jin J H, et al. Successful bioremediation of an aged and heavily contaminated soil using a microbial/plant combination strategy[J]. Journal of Hazardous Materials, 2014, 264: 430-438. |
| [14] | Maria S K, Irena B I, Tatiana N K, et al. Survival of cryogel-immobilized Rhodococcus strains in crude oil-contaminated soil and their impact on biodegradation efficiency[J]. International Biodeterioration & Biodegradation, 2013, 84: 118-125. |
| [15] | 韩慧龙, 汤晶, 江皓, 等. 真菌-细菌修复石油污染土壤的协同作用机制研究[J]. 环境科学, 2008, 29 (1): 189-195. |
| [16] | 王莉丽, 吴蔓莉, 丁艺, 等. 油污土壤修复过程中水溶性有机物光谱特性研究[J]. 安全与环境学报, 2012, 12 (6): 27-30. |
| [17] | Zhang X S, Xu D J, Zhu C Y, et al. Isolation and identification of biosurfactant producing and crude oil degrading Pseudomonas aerugin[J]. Chemical Engineering Journal, 2012, 209: 138-146. |
| [18] | 刘魏魏, 尹睿, 林先贵, 等. 生物表面活性剂-微生物强化紫花苜蓿修复多环芳烃污染土壤[J]. 环境科学, 2010, 31 (4): 1076-1084. |
| [19] | 任春燕. 产微生物表面活性剂菌株的筛选、性能及其应用研究[D]. 西安: 西安建筑科技大学, 2009. |
| [20] | 高小朋, 姜钊, 高秀梅, 等. 石油降解菌产表面活性剂的条件优化[J]. 环境工程学报, 2013, 7 (8): 3244-3248. |
| [21] | 杨茜, 吴蔓莉, 曹碧霄, 等. 石油降解菌的筛选、降解特性及其与基因的相关性研究[J]. 安全与环境学报, 2014, 14 (1): 187-192. |
| [22] | 张宏波, 林爱军, 刘爽, 等. 芘高效降解菌的分离鉴定及其降解特性研究[J]. 环境科学, 2010, 31 (1): 243-248. |
| [23] | Wrenn B A, Venosa A D. Selective enumeration of aromatic and aliphatic hydrocarbon degrading bacteria by a most-probable-number procedure[J]. Canadian Journal of Microbiology, 1996, 42 (3): 252-258. |
| [24] | Mancera-López M E, Sparza-García F E, Chávez-Gómez B, et al. Bioremediation of an aged hydrocarbon-contaminated soil by a combined system of biostimulation-bioaugmentation with filamentous fungi[J]. International Biodeterioration & Biodegradation, 2008, 61 (2): 151-160. |
| [25] | 刘怡辰, 曹娟, 高国庆, 等. 萘降解菌N19-3的分离、鉴定和萘双加氧酶基因的检测[J]. 环境科学研究, 2008, 21 (5): 27-31. |
| [26] | Taylor P M, Medd J M, Schoenborn L, et al. Detection of known and novel genes encoding aromatic ring-hydroxylating dioxygenases in soils and in aromatic hydrocarbon-degrading bacteria[J]. FEMS Microbiology Letters, 2002, 216 (1): 61-66. |
| [27] | Widada J, Nojiri H, Kasugak K, et al. Molecular detection and diversity of polycyclic aromatic hydrocarbon-degrading bacteria isolated from geographically diverse sites[J]. Applied Microbiology and Biotechnology, 2002, 58 (2): 202-209. |
| [28] | Powell S M, Bowman J P, Ferguson S H, et al. The importance of soil characteristics to the structure of alkane-degrading bacterial communities on sub-Antarctic Macquarie Island[J]. Soil Biology and Biochemistry, 2010, 42 (11): 2012-2021. |
| [29] | Wu M L, Chen L M, Tian Y Q, et al. Degradation of polycyclic aromatic hydrocarbons by microbial consortia enriched from three soils using two different culture media[J]. Environmental Pollution, 2013, 178: 152-158. |
 2015, Vol. 36
2015, Vol. 36


