在污水厌氧处理、 污泥浓缩与消化以及垃圾填埋过程中有大量的甲烷生成. 甲烷属于温室气体,其温室效应是二氧化碳的26倍[1]. 而且甲烷还是危险气体,其在空气中的体积分数为5%~15%时极易发生爆炸. 甲烷的厌氧氧化是减少甲烷的主要途径之一. 每年有0.3 Gt (1 Gt=1015 g)的甲烷经厌氧反应被消除. 多位研究者相继发现在海底沉积层中,伴随甲烷的减少,有硫酸盐还原反应发生,并且检测到能够利用硫酸盐厌氧氧化甲烷的活性物质[2,3]. 在厌氧条件下,厌氧甲烷氧化菌利用硫酸盐将甲烷氧化为二氧化碳,硫酸盐还原为硫离子. 垃圾渗滤液、 食品医药生产废水、 工业废水以及矿山废水中含有大量的硫酸盐. 硫酸盐排入水体,会引起水体酸化、 土壤板结等问题[4]. 利用含硫酸盐的废水处理含甲烷的废气,是废气与废水协同处理的一个有效途径.
本研究采用硫酸盐作为电子受体氧化甲烷. 驯化培养硫酸盐还原-甲烷厌氧氧化菌群,采用聚合酶链反应-变性梯度胶电泳(PCR-DGGE)指纹图谱技术分析菌群多样性和群落结构特征,通过其甲烷氧化效果,以期为该菌群应用于甲烷与硫酸盐的协同处理提供科学依据.
1 材料与方法 1.1 甲烷厌氧氧化-硫酸盐还原菌群的驯化方法
甲烷厌氧氧化-硫酸盐还原菌群的驯化接种物取自北京某污水处理厂厌氧段污泥,污泥性质见表 1. 将接种物与营养液按一定比例混合,混合液的污泥浓度为2800~3500 mg ·L-1,K2SO4的浓度为5 g ·L-1. 营养液的成分为:NaCl 2.0 g ·L-1,CaCl20.1 g ·L-1,MgCl2 ·6H2O 3.4 g ·L-1,FeCl2 ·4H2O 0.5 g ·L-1,NH4Cl 1 g ·L-1,pH 7.2,所用试剂均为分析纯,国药集团化学试剂有限公司生产. 混合液加入100 mL血清瓶中再通入50 mL甲烷(60%,余气为氦气,北京华元气体化工有限公司),将血清瓶密封后放入恒温摇床(30℃,120 r ·min-1)厌氧培养. 每隔30 d,取出10 mL混合液转入相同营养液中,再通入50 mL浓度为60%甲烷,相同条件下继续培养、 驯化. 定期取样,测定反应瓶中甲烷、 硫酸钾及其产物的量,监测反应效果. 3个月后,反应瓶内的甲烷浓度持续减少,驯化完成. 为了对比不同条件下的驯化效果及菌群特征,设计了系列驯化试验,试验条件列于表 2. 用驯化好的混合菌群在相同条件下进行甲烷厌氧氧化研究.
|
|
表 1 污泥性质 Table 1 Characteristics of sludge |
|
|
表 2 试验条件 Table 2 Conditions of test |
甲烷,二氧化碳和硫化氢均采用气相色谱仪(安捷伦6890N型,美国)检测,外标法计算气相中相应气体浓度.
CH4:氢火焰离子检测器,N2为载气; HP-5毛细管柱(30 m×0.32 mm×0.25 μm,Agilent); 进样口温度、 柱温和检测器温度分别为:50、 100 和250℃.
CO2:热导检测器,He为载气; 不锈钢填充柱Tbx-01(2 m,2 mm,60~80目); 进样口温度、 柱温和检测器温度分别为:110、 100和250℃.
H2S:火焰光度检测器,N2为载气; DB-1701(30 m×0.32 mm×0.25 μm,Agilent); 进样口温度、 柱温和检测器温度分别为:100、 50和200℃.
SO42-浓度:DIONEX ICS1000离子色谱仪(美国)测定.
化学需氧量(COD):HACH COD测定仪(美国).
总有机碳(TOC):TOC-V CPH测定仪(日本)测定.
总悬浮固体(TSS)和挥发性悬浮固体(VSS):重量法测定(国家标准GB/T 11901-1989).
生物相分析:用MOBIO Power Soil基因组提取试剂盒(美国Mobio)提取基因组DNA. 选取引物357F-GC,517R和344F-GC,522R对提取的DNA分别进行细菌和古菌16S rDNA基因V3区扩增,其序列见表 3. 扩增反应体系(50 μL)包括:DNA模板5 μL,10×Buffer 5 μL,dNTP 4 μL,MgCl2 4 μL,引 物(10 μmol ·L-1)各1 μL,Ex Taq酶(Takara,中国大连)0.3 μL,灭菌超纯水29.7 μL. 细菌扩增程序为:94℃预变性 5 min,94℃变性 1 min,48℃退火 1 min,72℃延伸 1 min,30个循环,72℃延伸 10 min. 古菌扩增程序为:94℃预变性 5 min,94℃变性 30 s,68~58℃退火 30 s,72℃延伸 1 min,10个循环,94℃变性 30 s,58℃退火 30 s,72℃延伸 1 min,20个循环,72℃延伸10 min.
采用DCode 通用突变检测系统(美国Bio-Rad)对PCR反应产物进行电泳分离,聚丙烯酰胺凝胶浓度梯度:6%~12%(质量浓度),凝胶变性梯度:40%~70%,上样量:15 μL,60℃下65 V电泳16 h,SYBRE Green染色后用Gel Doc XR凝胶成像仪(美国Bio-Rad)捕获凝胶数字图像,并用图形分析软件Quantity One对DGGE指纹图谱进行分析.
对目的条带进行切胶、 扩增、 纯化和克隆[7],使用 T7引物(5′-TAATACGACTCACTATAGGG-3′)测序. 将获得的序列在NCBI(National Center for Biotechnology Information,http://blast.ncbi.nlm.nih.gov)数据库进行BLAST (Basic local alignment search tools)分析.
以Shannon-Weiner index(H)表示反应体系的微生物群落多样性,计算公式如式(1)[8]:
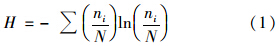
2 结果与讨论 2.1 硫酸盐还原型甲烷厌氧氧化菌群结构特征 2.1.1 细菌菌群结构特征
细菌的DGGE谱图如图 1所示. 接种物(MS2)与未加硫酸盐的驯化菌种(MS3)的DGGE谱图条带数量和迁移位置相似. MS2与MS3体系驯化时,都没有加入硫酸盐,MS3加入了60%的甲烷,驯化后这两个体系的菌群群落结构相近,丰度值差异不明显. 与MS2和MS3相比,加有硫酸盐的MS1体系的群落结构差异显著,其香农指数为3.79,明显高于MS2和MS3. 细菌群落聚类分析结果显示,MS2和MS3体系的菌群群落相似性为78%,MS1与前两者的相似性仅为63%. 细菌群落结构因硫酸盐的加入发生了明显变化,并且多样性增加. 在驯化过程中,细菌和古菌的群落结构和种群数量存在明显的演替过程,在演替过程中,既有原始种群的消亡,也有一些种群的增强,还有始终处于优势的种群,种群的功能地位处于动态变化中. 对比MS1、 MS2和MS3的DGGE谱图,MS1中增强的条带有11个,即B2、 B3、 B7、 B9~B11、 B14、 B16、 B17、 B19和B24. 条带B4、 B8、 B12、 B15、 B18、 B20、 B22、 B23和B25在MS1、 MS2和MS3中均有检出,表明这些在接种物中原有的菌种在驯化过程中一直存在. 同时条带B1、 B5、 B6、 B13、 B21和B26消失,这些种群可能因受到硫酸盐的抑制而被淘汰.
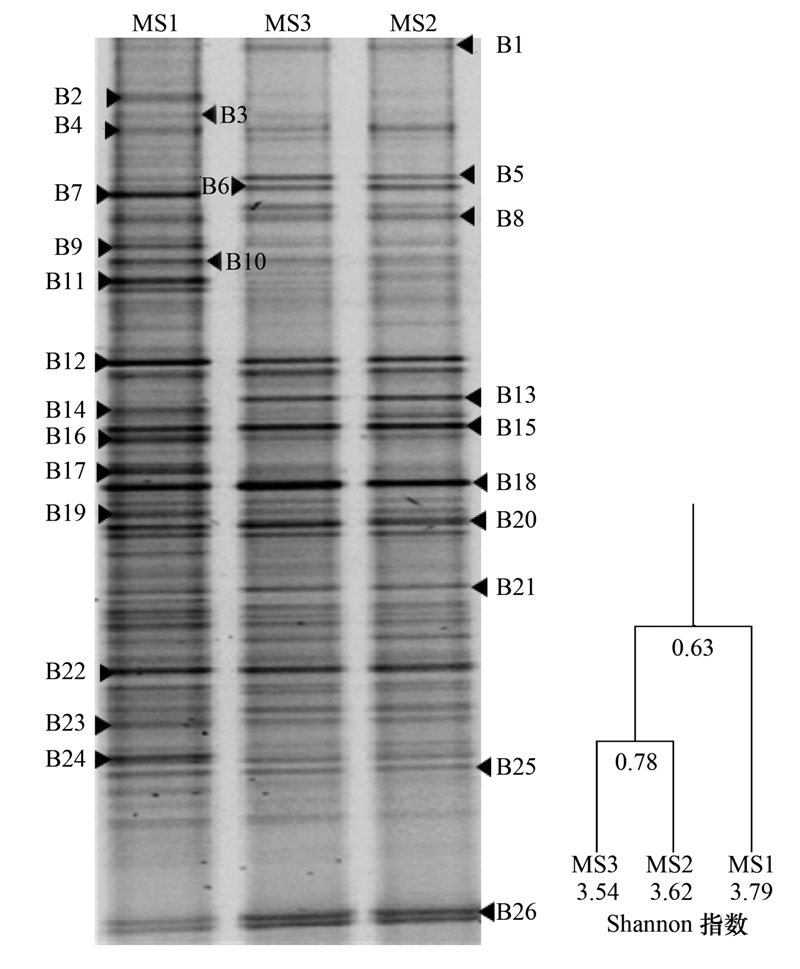 | 图 1 驯化过程中细菌的DGGE谱图和群落聚类分析 Fig. 1 DGGE fingerprint and community clustering analysis of bacteria during acclimation |
选择26个荧光强度较亮的DGGE条带(B1~B26)进行切胶、 纯化、 克隆和测序,所得序列在GenBank数据库中用BLAST进行同源性比较,确定各条带所代表的细菌菌群分类地位,分析MS1、 MS2和MS3中的优势菌群. 各体系中优势细菌及其相似菌列于表 4. 大部分相似菌(相似度大于98%)是从污水处理的厌氧消化污泥中分离得到的,它们分别属于螺旋菌门、 拟杆菌门、 厚壁菌门、 热袍菌门、 绿弯菌门、 变形菌门、 浮霉菌门、 放线菌门及未知菌. 与MS2和MS3相比,MS1中热袍菌门增强.
|
|
表 4 MS1、MS2、MS3体系中优势细菌的相似菌 Table 4 Similar bacteria of the dominant bacteria in MS1,MS2,MS3 |
条带B14和B2是MS1独有的种群,不存在于MS2和MS3中,属于驯化过程中丰度提高显著的2个种群. 其中,条带B14属于除硫单胞菌目(Desulfuromonadales),其相似菌分离自处理含硫酸盐废水的颗粒污泥,能以硫酸盐或硫磺为电子受体进行无氧呼吸,此种微生物能很好地以硫酸盐为底物进行生长[9,10]. B2与紫单胞菌科中的Proteiniphilum相似,可以将蛋白质水解为乙酸和丙酸[11]. 大部分增强的种群具有降解有机物的性质,B7相似于热袍菌门的Mesotoga,该类菌能在高温环境下分解碳水化合物,产物氢可被产甲烷菌利用[12,13]. 与B10相似的鞘脂单胞菌属(Sphingomonas)对芳香化合物有极广泛的代谢能力,具有特殊的代谢调控机制,可以适应营养物缺乏的环境[14]. B11、 B16、 B17、 B19和B23的相似菌均属于螺旋体门(Spirochaetes),它们经常出现在厌氧消化污泥中,厌氧发酵碳水化合物. 通过条带亮度可以看出,条带B12和B18是驯化过程一直存在的优势种群,从加乙酸盐的厌氧反应器分离的BH62与B18相似[15]. B12与在水稻田中发现的浮霉菌门(Planctomycetes)相似,浮霉菌门的某些种属具有与甲烷氧化作用相关的C转移基因[16,17].
经过驯化,MS1的细菌菌群中的优势功能种群包括硫酸盐还原菌、 有机物降解菌以及产甲烷菌. 2.1.2 古菌菌群结构特征
古菌的DGGE谱图显示接种物MS2与未加硫酸盐的驯化物MS3古菌菌群的群落结构相似(图 2),相似性达到96%,而与MS1有明显区别. MS1、 MS2和MS3的香农指数分别为2.51、 2.53和2.53,古菌的多样性差别不明显,加入硫酸盐的MS1体系古菌群落多样性略微降低.
11个条带(A1~A11)经切胶、 测序比对后,各体系中优势古菌及其相似菌列于表 5. 结果表明,这些菌分别属于广古菌门和泉古菌门. 大部分相似菌(相似度大于98%)
分离自厌氧生物反应器.
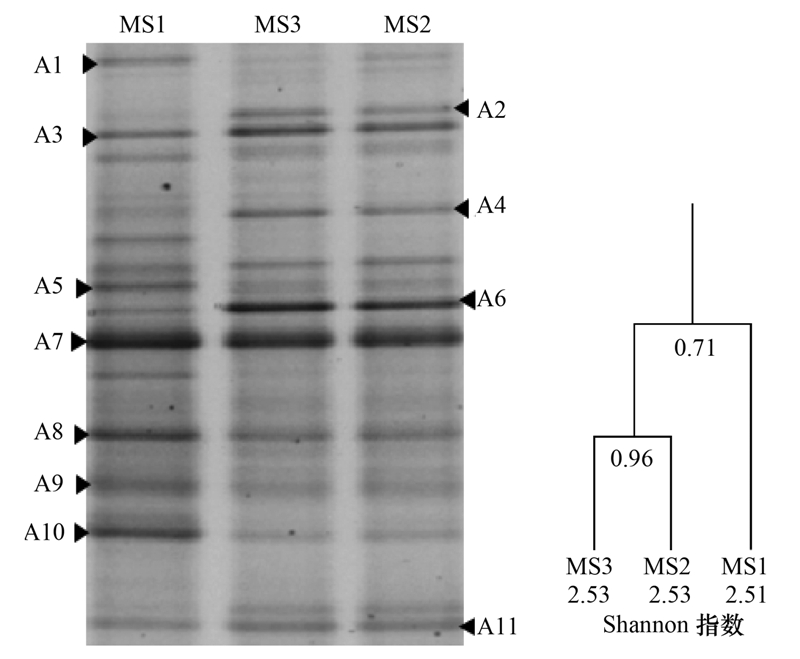 | 图 2 驯化过程中古菌的DGGE谱图和群落聚类分析 Fig. 2 DGGE fingerprint and community clustering analysis of archea during acclimation |
MS1体系中增强5个条带,即A1、 A5、 A8、 A9和A10. 其中,A1与甲烷丝状菌属的相似度为100%,Valentine等[18]在研究土壤中的甲烷厌氧氧化反应时发现该类菌具有甲烷氧化功能. 根据系统发育分析,通常将甲烷厌氧氧化古菌(ANME)分为3个类群,即ANME-1、 ANME-2 和ANME-3. 这3个类群都属于广古菌门(Euryarchaeota),具有催化甲烷的厌氧氧化的生理学特征. 其中ANME-1与甲烷八叠球菌目(Methanosarcinales)和甲烷微菌目(Methanomicrobiales)的亲缘关系较近,ANME-2属于甲烷八叠球菌目(Methanosarcinales),ANME-3与拟甲烷球菌属(Methanococcoides)亲缘关系较近[19]. A5的相似菌属于甲烷微菌目,与ANME-1亲缘关系较近. Hinrichs等[20]在研究某海底甲烷冷泉附近的沉积物中古菌群落发现有该类菌存在,并有甲烷水合物的分解现象. A9与甲烷球菌目的相似度为99%,该类菌属于能与硫酸盐还原菌共生的ANME-2[21,22].
MS1、 MS2和MS3有一些相同的微生物种群,如条带A3、 A7、 A8、 A9和A10,虽然亮度有所变化,但存在于整个驯化过程中,可能在微生物群落的物质和能量代谢中发挥着重要作用. 其中,A7与甲烷丝状菌属(Methanosaeta)相似度为99%. A3与甲烷囊菌属相似,该类菌是稻田土壤里发现的产甲烷菌中的一种[23]. A4号条带与甲烷八叠球菌属的相似度为99%,Bryant[24]发现其能将乙酸或甲醇代谢生成甲烷. A11条带与甲烷杆菌科相似,属产甲烷菌. 条带A2和A6驯化后明显减弱,A2的相似菌属于甲烷囊菌属,而A6的相似菌属于甲烷八叠球菌目(相似度为100%),该菌株(Methanosarcinales)分离自处理市政污水的厌氧消化污泥. 硫酸盐的抑制可能是此2种微生物数量减少的原因.
综上所述,驯化过程中,产甲烷古菌始终处于优势的种群. 硫酸盐的加入,极有可能增强了硫酸盐还原型甲烷厌氧氧化古菌的丰度,使得它们也成为优势菌群.
|
|
表 5 MS1、MS2、MS3体系中优势古菌的相似菌 Table 5 Similar archea of the dominant archea in MS1,MS2,MS3 |
定期取样,测定驯化体系中甲烷的量,监测甲烷的变化. 随驯化时间延长,MS1的甲烷浓度均逐渐降低(图 3). 在驯化过程中,体系内种群的功能地位发生变化,MS1中的甲烷氧化古菌和硫酸盐还原细菌逐渐成为优势菌群,导致甲烷浓度下降. 由于产甲烷菌存在于整个驯化过程中,甲烷氧化与甲烷产生同时存在并相互竞争,引起MS1中的甲烷浓度出现波动. 与硫酸根浓度较低的MS4相比,MS1的甲烷减少更多,由初始57.29%减少为35.45%. 在驯化初期,MS4的甲烷浓度由41.95%迅速升高至58.56%,10 d后缓慢下降,下降幅度低于MS1. 硫酸盐的量影响甲烷的氧化效果,在硫酸盐浓度较高的体系中甲烷氧化效果更好.
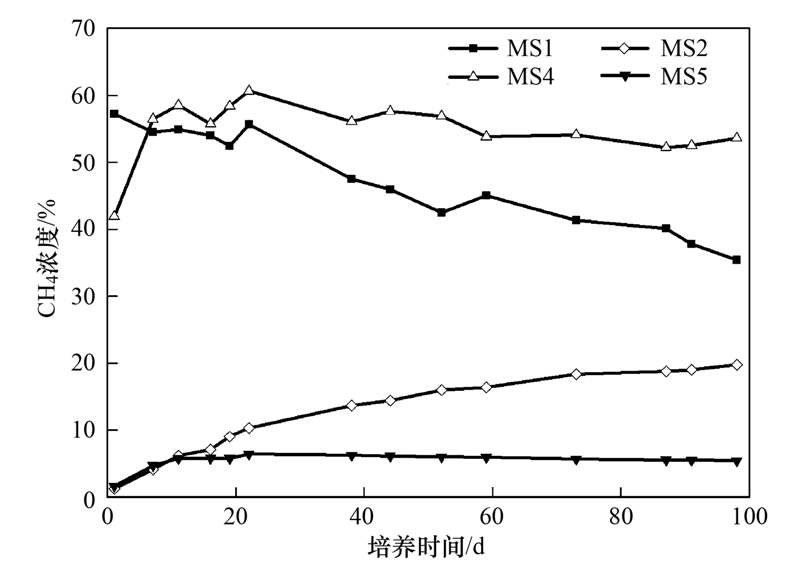 | 图 3 MS1、MS2、MS4和MS5驯化时甲烷浓度变化 Fig. 3 Concentration of methane in MS1,MS2,MS4and MS5 |
同样条件下,接种物MS2中的甲烷浓度持续升高. 接种物为污水处理厂厌氧消化池的污泥,其有机质和无机质比例为1.13(表 1),有机质含量较高. MS2中产甲烷细菌、 产甲烷古菌以及有机物降解菌是优势功能菌,在厌氧条件下,发生厌氧消化反应. 污泥中的碳水化合物、 蛋白质、 脂肪、 纤维素等非水溶性高分子有机物水解成溶解性的有机物质. 乙酸菌将水解产物,有机物、 乙醇等转变为乙酸. 污泥厌氧消化后期,乙酸在产甲烷菌作用下转化为CH4[(式(2)][25]. 因此,MS2中出现甲烷浓度持续升高的现象.
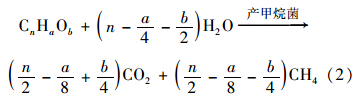
在加硫酸盐的MS5体系中,其驯化初期甲烷浓度升高显著,10 d后甲烷浓度不再升高,保持稳定,并随驯化时间延长有下降趋势. 这可能是由于有机物降解菌和产甲烷菌是驯化初始时期的优势菌群,因而体系以产甲烷为主; 随驯化时间的延长,产甲烷菌受到硫酸盐的抑制,而甲烷厌氧氧化古菌逐渐成为优势菌,因此甲烷氧化反应逐渐占优势. 2.3 甲烷厌氧氧化-硫酸盐还原反应产物形式和菌群结构
用驯化好的混合菌群进行甲烷厌氧氧化研究,定期取样分析反应物和产物,监测甲烷氧化效果,分析结果如图 4所示. 随甲烷减少二氧化碳逐渐增多,减少的甲烷被氧化为二氧化碳. 产生的二氧化碳一部分溶于水形成碳酸盐,使溶液中保持一定量的无机碳. 同时反应体系中溶解性总有机碳也增加(图 5). 以往的研究显示,硫酸盐还原型的甲烷厌氧氧化的途径有3种,每种途径的甲烷氧化过程中,甲烷经过一系列的中间反应最终被氧化为二氧化碳,其间反应步骤和中间产物较多,并且反应速率缓慢. 甲醇和甲酸[26]、 乙酸和甲基化合物[18]以及乙酰[27]为不同氧化途径的中间产物. 因此,除了微生物的自身分解和污泥的厌氧消化以外,甲烷氧化的中间产物的产生也会增加溶解性总有机碳的量.
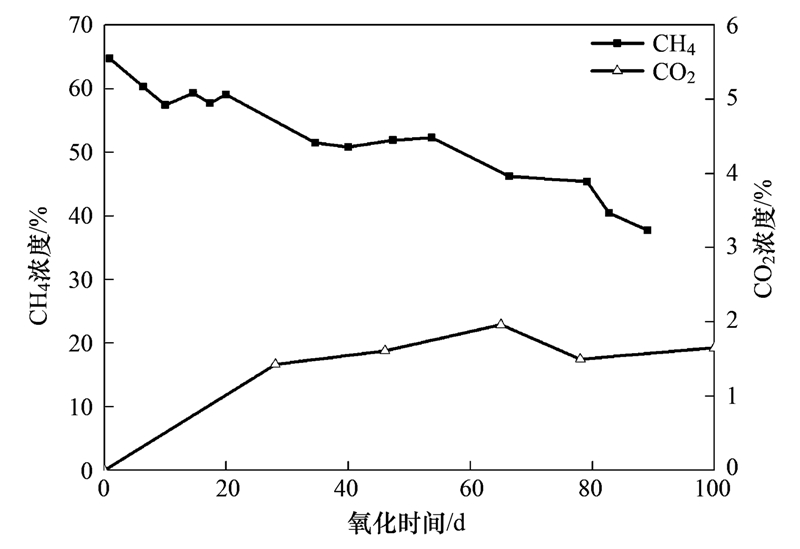 | 图 4 甲烷氧化过程的甲烷-二氧化碳浓度变化 Fig. 4 Concentration of methane and carbon dioxide during methane oxidation |
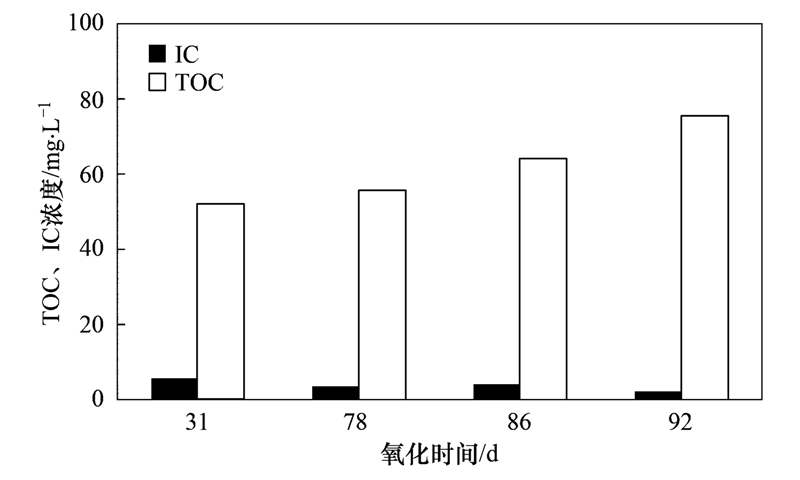 | 图 5 甲烷氧化过程的无机碳和总有机碳浓度变化 Fig. 5 Concentration of inorganic carbon and total organic carbon during methane oxidation |
硫酸盐还原型甲烷厌氧氧化的特点是甲烷厌氧氧化的同时伴随硫酸盐的还原,反应过程是以SO42-作为最终的电子受体,产物为HCO3-和HS-. 当体系中有H+时,HCO3-转化为二氧化碳,HS-转化为H2S. 体系中伴随硫酸盐的减少,可以检测到硫化氢的产生(表 6). 体系中发生了甲烷氧化-硫酸盐还原反应,反应式如式(3)[19].
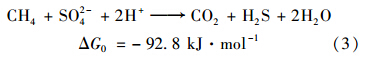
另外,硫酸盐的浓度还会影响产物的形式,当SO42-浓度高于6200 mg ·L-1时,检测到有硫单质生成,同时硫化氢的产生量减少.
| 表 6 甲烷氧化过程的硫酸盐和硫化氢浓度变化 Table 6 Concentration of sulfate and sulfide hydrogen during methane oxidation |
3 结论
(1)经过驯化,硫酸盐还原甲烷氧化体系中种群的功能地位发生了明显变化,既有原始种群的消亡和新种群的增强,也有始终处于优势的种群. 硫酸盐的加入,使参与甲烷厌氧氧化反应的甲烷氧化古菌和硫酸盐还原细菌逐渐成为优势菌群. 同时,有机物降解菌以及产甲烷菌也是体系中的优势种群. 可见,体系中同时存在甲烷的氧化与产生两个反应过程,它们在体系中同时发生并存在相互竞争.
(2)对驯化菌群甲烷厌氧氧化功能的研究结果表明,甲烷厌氧氧化的同时伴随硫酸盐的还原. 甲烷的氧化产物为二氧化碳. 二氧化碳一部分存在于气相中,一部分溶于水形成碳酸盐. 中间产物的形成增加了反应体系中溶解性总有机碳的量. 硫酸盐的转化产物为硫化氢和硫单质. 硫化氢是一种恶臭物质,极易从溶液中逸散到空气中,形成恶臭污染. 因此,可通过调节硫酸盐浓度,使硫酸盐的还原产物以硫单质为主,以实现氧化甲烷、 还原硫酸盐的同时,回收硫单质,避免恶臭.
| [1] | 吕镇梅, 闵航, 陈中云, 等. 水稻田土壤甲烷厌氧氧化在整个甲烷氧化中的贡献率[J]. 环境科学, 2005, 26 (4): 13-17. |
| [2] | Boetius A, Ravenschlag K, Schubert C J, et al. A marine microbial consortium apparently mediating anaerobic oxidation of methane[J]. Nature, 2000, 407 (6804): 623-626. |
| [3] | Joye S B, Boetius A, Orcutt B N, et al. The anaerobic oxidation of methane and sulfate reduction in sediments from Gulf of Mexico cold seeps[J]. Chemical Geology, 2004, 205 (3): 219-238. |
| [4] | 李宽峰, 吴鹏, 沈耀良, 等. 含硫酸盐废水处理研究现状与展望[J]. 水处理技术, 2013, 39 (11): 17-22. |
| [5] | Kimura M, Shibagaki T, Nakajima Y, et al. Community structure of the microbiota in the floodwater of a Japanese paddy field estimated by restriction fragment length polymorphism and denaturing gradient gel electrophoresis pattern analyses[J]. Biology and Fertility of Soils, 2002, 36 (4): 306-312. |
| [6] | Vetriani C, Jannasch H W, MacGregor B J, et al. Population structure and phylogenetic characterization of marine benthic archaea in deep-sea sediments[J]. Applied and Environmental Microbiology, 1999, 65 (10): 4375-4384. |
| [7] | Zhou X, Guo X, Han Y, et al. Enhancing nitrogen removal in an Orbal oxidation ditch by optimization of oxygen supply: practice in a full-scale municipal wastewater treatment plant[J]. Bioprocess and Biosystems Engineering, 2012, 35 (7): 1097-1105. |
| [8] | Li L, Gao M, Liu J. Distribution characterization of microbial aerosols emitted from a wastewater treatment plant using the Orbal oxidation ditch process[J]. Process Biochemistry, 2011, 46 (4): 910-915. |
| [9] | Borin S, Brusetti L, Daffonchio D, et al. Biodiversity of prokaryotic communities in sediments of different sub-basins of the Venice lagoon[J]. Research in Microbiology, 2009, 160 (5): 307-314. |
| [10] | Cheng T, Lin L, Wang P, et al. Methane-related microbial processes and metabolic stratification in a terrestrial mud volcano, southwestern Taiwan[C]. American Geophysical Union, Fall Meeting Abstracts, 2009. |
| [11] | Chen S, Dong X. Proteiniphilum acetatigenes gen. nov., sp. nov., from a UASB reactor treating brewery wastewater[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55 (6): 2257-2261. |
| [12] | Conners S B, Mongodin E F, Johnson M R, et al. Microbial biochemistry, physiology, and biotechnology of hyperthermophilic Thermotoga species[J]. FEMS Microbiology Reviews, 2006, 30 (6): 872-905. |
| [13] | Muralidharan V, Rinker K D, Hirsh I S, et al. Hydrogen transfer between methanogens and fermentative heterotrophs in hyperthermophilic cocultures[J]. Biotechnology and Bioengineering, 1997, 56 (3): 268-278. |
| [14] | 胡杰, 何晓红, 李大平, 等. 鞘氨醇单胞菌研究进展[J]. 应用与环境生物学报, 2007, 13 (3): 431-437. |
| [15] | Chien I C, Meschke J S, Gough H L, et al. Characterization of persistent virus-like particles in two acetate-fed methanogenic reactors[J]. PLoS One, 2013, 8 (11): e81040. |
| [16] | Cahyani V R, Murase J, Ikeda A, et al. Bacterial communities in iron mottles in the plow pan layer in a Japanese rice field: Estimation using PCR-DGGE and sequencing analyses[J]. Soil Science and Plant Nutrition, 2008, 54 (5): 711-717. |
| [17] | Chistoserdova L, Jenkins C, Kalyuzhnaya M G, et al. The enigmatic planctomycetes may hold a key to the origins of methanogenesis and methylotrophy[J]. Molecular Biology and Evolution, 2004, 21 (7): 1234-1241. |
| [18] | Valentine D L, Reeburgh W S. New perspectives on anaerobic methane oxidation[J]. Environmental Microbiology, 2000, 2 (5): 477-484. |
| [19] | Caldwell S L, Laidler J R, Brewer E A, et al. Anaerobic oxidation of methane: mechanisms, bioenergetics, and the ecology of associated microorganisms[J]. Environmental Science & Technology, 2008, 42 (18): 6791-6799. |
| [20] | Hinrichs K U, Hayes J M, Sylva S P, et al. Methane-consuming archaebacteria in marine sediments[J]. Nature, 1999, 398 (6730): 802-805. |
| [21] | Hallam S J, Girguis P R, Preston C M, et al. Identification of methyl coenzyme M reductase A (mcrA) genes associated with methane-oxidizing archaea[J]. Applied and Environmental Microbiology, 2003, 69 (9): 5483-5491. |
| [22] | Orphan V J, House C H, Hinrichs K U, et al. Methane-consuming archaea revealed by directly coupled isotopic and phylogenetic analysis[J]. Science, 2001, 293 (5529): 484-487. |
| [23] | Le Mer J, Roger P. Production, oxidation, emission and consumption of methane by soils: a review[J]. European Journal of Soil Biology, 2001, 37 (1): 25-50. |
| [24] | Bryant M. Microbial methane production-theoretical aspects[J]. Journal of Animal Science, 1979, 48 (1): 193-201. |
| [25] | Buswell A, Mueller H. Mechanism of methane fermentation[J]. Industrial & Engineering Chemistry, 1952, 44 (3): 550-552. |
| [26] | Zehnder A, Brock T. Methane formation and methane oxidation by methanogenic bacteria[J]. Journal of Bacteriology, 1979, 137 (1): 420-432. |
| [27] | Hinrichs K U, Boetius A. The anaerobic oxidation of methane: new insights in microbial ecology and biogeochemistry[A]. In: Ocean Margin Systems[M]. Berlin, Heidelberg: Springer, 2003. 457-477. |
 2014, Vol. 35
2014, Vol. 35


