2. 广东省微生物研究所, 广州 510070
2. Guangdong Institute of Microbiology, Guangzhou 510070, China
厌氧污泥是多种厌氧微生物形成的复杂聚集体,不同处理工艺内污泥中微生物群落的研究成为废水处理反应器的研究热点. 反应器内微生物的群落结构的变化及生态演替受生态因子如温度[1, 2]、 pH[2]、 氧化还原电位[3]、 反应器进水成分[4]、 进水碳硫比[5,6]等的影响; 同时,微生物种群的分布[10]、 不同微生物种群之间存在的协同[11]和竞争制约[12]生态学效应等,直接影响着反应器的处理效果.
厌氧污泥的驯化是UASB 等厌氧反应器运行的关键,对厌氧污泥驯化过程中微生物生态结构的变化及群落演替的研究具有重要的意义. 近年来不依赖纯培养的分子生物学的方法发展迅速,也得到广泛的应用[13],其中利用PCR-DGGE来研究未培养或不能培养的微生物,能够提供群落中优势种类信息和同时分析多个样品,适合于监测或调查种群的时空变化,自从1993年被Muyzer等[14]引入微生物生态学研究以来,已广泛应用于分析活性污泥[15]、 生物滤池[16]、 生物膜[17]、 土壤[18]、 底泥[19]、 水体[20]等各种环境中生物多样性、 种群演替等方面. 本研究采用变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)技术,通过不依赖分离培养的核酸信息对处理高浓度硫酸盐有机废水UASB反应器污泥驯化过程中微生物群落的变化进行了分析,以期为工艺优化和揭示厌氧污泥中硫酸盐生物处理机制提供科学依据.
1 材料与方法 1.1 硫酸盐还原反应器污泥的驯化采用UASB为硫酸盐还原反应器,容积为19.4 L. 接种污泥来源于某污水处理厂ABR的厌氧污泥和河涌底泥的混合污泥,以人工配水进行污泥驯化. 配水成分包含葡萄糖5 g ·L-1,酵母抽提物0.2 g ·L-1,Na2SO4 1.7 g ·L-1,MgSO4 0.03 g ·L-1,CaCl2 0.01 g ·L-1,KH2PO4 0.25 g ·L-1,NH4Cl 0.1 g ·L-1,NaHCO3 2 g ·L-1,(NH4)2CO3 0.1 g ·L-1,进水pH约为7.0.
反应器污泥驯化采用固定进水浓度,逐渐缩短水力停留时间的方法以提高反应器硫酸盐负荷. 整个驯化过程历时240 d,按水力停留时间的不同分为4个驯化阶段,其中第0~150 d为第1阶段,水力停留时间为80 h; 第150~180 d为第2阶段,水力停留时间为60 h; 第180~210 d为第3阶段,水力停留时间为48 h; 第210~240 d为第4阶段,水力停留时间为24 h.
1.2 采样本研究共采集污泥样品7个,自反应器污泥驯化的4个不同阶段,分别于驯化开始的第30、 60、 120、 150、 180、 210和240 d采用灭菌的离心管采集污泥样品,样品依次标记为A、 B、 C、 D、 E、 F和G. 所采集的样品直接用于基因组DNA的提取或冻存-80℃冰箱中待用,并测定MLVSS. 此外,定期采集反应器进出水水样,测定其中COD和硫酸盐含量的变化.
1.3 理化分析方法MLVSS、 COD、 硫酸盐采用文献[21]的方法进行测定.
1.4 变性梯度凝胶电泳(DGGE)分析微生物群落变化 1.4.1 细菌基因组总DNA的提取与纯化取5 g污泥样品,采用Zhou等[22]的方法进行总DNA提取. 所得DNA初提液采用天根公司的DNA纯化试剂盒进行纯化.
1.4.2 PCR扩增及变性梯度凝胶电泳(DGGE)分析以纯化后的细菌基因组总DNA为模板,采用细菌通用引物27F与1492R扩增以得到全长16S rDNA; 再以该扩增产物为模板,采用对大多数细菌和古生菌的16S rDNA基因V3区具有特异性的引物对GC-341F和534R[23]进行扩增.
PCR反应在MyCycler (Bio-Rad)上进行,50 μL反应体系为50 ng的模板、 20 pmol正反向引物、 200 μmol ·L-1 dNTP、 5 μL的10×PCR buffer (不含MgCl2)、 1.5 mmol ·L-1的MgCl2、 1 U的ExTaq DNA聚合酶和适量的双蒸水. 反应程序: 94℃ 4 min; 94℃ 45 s,55℃ 1 min,72℃ 45 s,30次循环; 72℃ 10 min.
采用D-code基因突变检测系统(Bio-Rad Laboratories Inc.USA)对 PCR 反应产物进行DGGE分离. 采用8%聚丙烯酰胺凝胶,变性剂范围为35%~55%(100%的变性剂定义为7 mol ·L-1尿素和40%去离子甲酰胺),在1×TAE(40 mmol ·L-1 Tris,20 mmol ·L-1乙酸,1 mmol ·L-1 EDTA,pH 7.4)中,60℃、150 V电泳6 h,用Goldenview染液染色,Bio-Rad凝胶成像系统观察并拍照.
采用Quantity One软件对DGGE图谱进行分析,对各泳道的条带数目、 位置及亮度,不同样品间细菌群落相似度用Dice系数计算,生成相似度矩阵.根据图谱中不同条带的光密度值,采用公式(1)和(2)计算微生物群落的Shannon-Wiener指数.

采用SPSS软件对驯化污泥化过程中反应器COD及硫酸盐去除效率、 微生物群落Shannon-Wiener指数等参数的变化进行相关性分析,选择Pearson相关分析选项,软件自动完成.
1.4.3 切胶测序及系统发育分析对DGGE图谱上的优势条带进行切胶回收,以V3区引物(不带GC夹)采用相同程序进行PCR扩增,扩增产物经纯化后采用PGM-T载体克隆至E.coli Top10后送交上海生物工程技术公司测序. 将测序结果提交NCBI数据库Blast搜索出相似性高的序列,利用Clustal X软件进行多序列比对,通过Mega 5构建系统发育树.
2 结果与讨论 2.1 微生物群落多样性及反应器处理效率变化为考察厌氧污泥驯化过程中群落多样性的变化情况,采集反应器驯化不同时期的污泥样品,对其进行微生物总DNA提取,并采用试剂盒对DNA初提液进行纯化,并以此为模板,采用巢式扩增,先用引物对27F/1492R 扩增得到污泥中微生物全长16S rDNA,再以此为模板,采用引物对341/534扩增得到微生物16S rDNA V3可变区基因,通过变性梯度凝胶电泳(DGGE)进行分析,不同泳道分别代表反应器驯化不同天数时所取反应器泥样. 污泥驯化不同时期微生物群落变性梯度凝胶电泳图谱分析如图 1所示. 结果表明,重复电泳的结果非常相似,重复间的相似性达99%,7个不同时期的污泥样品中微生物群落电泳图谱中条带的位置和数量不同,电泳条带呈现较明显的变化,显示反应器污泥驯化的不同时期,随着反应器的污泥驯化,污泥样品DGGE电泳图谱中条带数量增多,微生物群落结构和种群数量存在明显的演替过程. 既有一些相同的微生物种群,如条带1、 4、 7、 8、 9等,虽然亮度有所变化,但存在于整个驯化过程中; 也有一些种群(条带)逐渐消亡或者减弱的,而一些种群(条带)逐级出现或增强; 也有一些样品各自独有的种群,如第150 d样品的条带3; 另外还有相邻阶段共有的条带,如第180 d和第210 d样品的条带13.
 | 图 1 污泥驯化不同阶段的变性梯度凝胶电泳(DGGE)图谱 Fig. 1 DGGE patterns of different sludge samples |
采用凝胶分析软件Quantity One 对反应器驯化不同时期污泥样品电泳图谱相似度进行分析,分析结果见表 1. 结果显示,反应器驯化相邻阶段时期,污泥中微生物群落相似度均在30%以上,其中样品D与样品E,以及样品F与样品G之间相似度均为57%左右,表明反应器驯化的第150~180 d、 第210~240 d这2个阶段污泥中微生物群落变化较小. 而从不同时期反应器处理效率(图 2)可知,恰好这2个阶段是反应器硫酸盐处理效率高效和稳定的时期,去除率达90%~99%.
| 表 1 反应器驯化不同时期污泥样品DGGE图谱相似度 Table 1 Coefficients (Cs) comparing the similarities of the DGGE profiles at different sludge domestication stages |
为了解驯化过程中反应器处理效率与污泥中微生物群落多样性指数变化的关系,采用SPSS软件,对反应器驯化时间、 COD与硫酸盐负荷及其去除效果、 污泥浓度、 污泥中微生物群落多样性指数进行相关性分析,结果见表 2.
结合图 2和表 2来分析,可见反应器硫酸盐及COD去除率与驯化时间、污泥浓度、污泥中微生物群落多样性指数呈明显的正相关. 王爱杰等[24]在产酸脱硫反应器研究中发现,在不同的群落演替阶段,每个种群的代谢活性有一定差别,表现为COD 去除率、 SO2-4去除率的差异. 而本研究中随着污泥驯化的进行,微生物群落生物多样性得以提高,当微生物群落Shannon指数大于3.45、 污泥浓度大于40 g ·L-1时,硫酸盐去除率稳定在95%左右,这说明反应器污泥驯化成熟,处理系统中各菌群处于良好的协同状态.
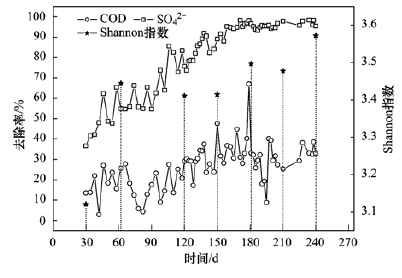 | 图 2 污泥驯化不同时期反应器处理效率及微生物群落多样性变化的关系 Fig. 2 Relation between removal efficiency and variation of microbial community at different sludge domestication stages |
| 表 2 各参数相关性分析 1) Table 2 Correlation analysis among parameters |
为了进一步分析微生物群落结构中的优势菌株和污泥中微生物的系统发育,对电泳图谱中较亮条带进行切割回收后,采用T载体克隆并测序进行系统发育分析见图 3.
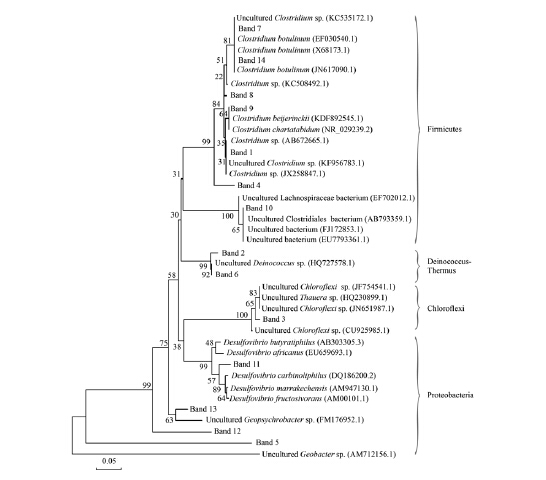 | 图 3 基于DGGE条带16S rDNA序列的不同泥样细菌系统发育树 Fig. 3 Phylogenetic tree of bacteria in different sludge samples based on the 16S rDNA sequences of DGGE bands |
结合图 1和图 3可知,反应器污泥中微生物群落中主要包含4大类群,其中Firmicutes占总数的50.0%,Proteobacteria占总数的28.6%,Deinococcus-Thermus占总数的14.3%,Chloroflexi占总数的7.1%.
污泥驯化过程中出现大量属于Firmicutes的发酵产酸菌,如条带1、4、7、8、9、10、14. 发酵产酸细菌能够将大分子的底物(如蔗糖、 葡萄糖和果糖等)转化为乙酸、乙醇、氢气、甲醇和甲酸等易被SRB利用的物质;在为SRB提供底物,缓冲碱度变化,维持系统适合的代谢环境等方面起着决定作用[24]. 本研究中的Firmicutes细菌均属于梭菌属Clostridium sp.,其中大部分菌群如条带1、4、7、8、9所代表的菌群在反应器驯化的4个阶段中均可监测到,为微生物群落中的优势菌属. Clostridium严格厌氧,能够产NH3H2S、H2,具有固氮、 发酵糖类产乙酸乳酸等功能[25]. Kaksonen等[26]在硫酸盐还原反应器中曾分离得到Clostridium 菌株. 此外,任南琪等[27]在硫酸盐还原反应器中也监测到Clostridium 的某些菌种随硫酸盐去除率的升高得以富集,在反应器中占优势. 有研究表明,随着不同种类的Clostridium属细菌的出现,其代谢产生大量发酵终产物作为硫酸盐还原过程的作用底物,从而影响硫酸盐还原菌群的丰度变化[28],由此造成硫酸盐还原菌群与Clostridia菌群呈平行的动态变化[29]. 本研究中Clostridium细菌在所监测的微生物群落中所占百分数为50.0%,并且随着反应器硫酸盐负荷的提高,属于该属的某些种类细菌相继出现或消亡,由此推测该属菌群在本反应器的硫酸盐还原过程中起着重要的作用.
Chloroflexi 、Geobacter、Geopsychrobacter是废水厌氧处理系统中的常见菌[30],在本研究的污泥中也存在. 条带3包含的序列属于Chloroflexi,在反应器污泥驯化过程中(第150 d)曾成为优势菌群,但后来随着反应器污泥的进一步驯化而消失. 颗粒污泥中经常监测到Chloroflexi 细菌,Roest 等[31]认为污泥中的Chloroflexi细菌可能直接或间接参与丁酸盐的降解. 条带5包含的序列属于变形菌门中的Geobacter sp.,自反应器驯化的第30 d后开始成为反应器中的优势菌群之一,驯化至第150 d后,条带亮度增强. Geobacter为厌氧菌,能够氧化有机物为CO2,以铁氧化物作为电子受体,被用于去除废水中有机及金属污染物[32]. 近年来在环境修复的研究中也发现该类菌的存在[33]. 条带12、 13包含的序列属于Geopsychrobacter sp.,其中,条带12在第60 d条亮度最强,在驯化的污泥过程中亮度减弱或消失,条带13在污泥驯化的第150~210 d内占优势,该属的某些细菌具有还原元素硫及Mn(Ⅳ),并氧化乙酸盐、 琥珀酸盐、 丙酸盐的功能[34].
本研究在污泥驯化的第1及第2阶段(第0~180 d)硫酸盐还原菌属均未占优势,但在驯化的最后2个阶段(第210~240 d)监测到Desulfovibrio sp. (条带11)占优势. Desulfovibrio sp.是革兰氏阴性菌,为快速生长的硫酸盐还原菌,能够部分氧化某些有机物为乙酸盐和CO2 [35],并利用氢为能源,以H+为电子受体,还原硫酸盐为硫化物[36]. 本研究中仅监测到这一类硫酸盐还原菌群,是由于人工配水驯化,底物单一的原因. 此外,Desulfovibrio占优势的2个阶段,反应器硫酸盐去除效率最高,为95%以上,可见Desulfovibrio 的数量与硫酸盐去除率间存在明显的正相关,因此该菌属的数量与活性是影响反应器硫酸盐去除效率的重要因素. 这些结果与任南琪等[27]的研究结果一致.
此外,条带2、 6所包含的序列属于异常球菌-栖热菌门(Deinococcus-Thermus),它们在微生物生态系统中所起的作用尚不明确,还有待进一步探索.
3 结论(1) 反应器污泥驯化过程中,微生物群落生物多样性与反应器硫酸盐及COD去除率呈明显的正相关关系. 当微生物群落Shannon指数大于3.45、 污泥浓度大于40 g ·L-1时,硫酸盐去除率稳定在95%左右,处理系统中各菌群处于良好的协同状态.
(2)反应器污泥中微生物群落主要包含Firmicutes、 Proteobacteria、Deinococcus-Thermus、Chloroflexi 4大类群,分别占总数的50.0%、28.6%、 14.3%和7.1%. 随着反应器污泥的驯化,污泥的微生物群落结构和种群数量存在明显的演替过程. 其中厌氧发酵细菌Clostridium sp.在驯化全过程中均占优势,但优势菌群的种类发生变化; 厌氧细菌Chloroflexi sp.在反应器驯化污泥第150 d曾成为优势菌群,但后来随着反应器污泥的进一步驯化而消失; 厌氧细菌Geobacter sp. 自反应器驯化的第30 d后开始成为反应器中的优势菌群之一,驯化至第150 d后,成为优势菌群; 厌氧细菌Geopsychrobacter sp.在污泥驯化过程中相继出现或消亡; Desulfovibrio sp.在污泥驯化的最后2个阶段占优势.
| [1] | 李津, 左剑恶, 邢薇. EGSB反应器在20℃下处理啤酒废水的工艺及微生物学研究[J]. 环境科学, 2008, 29 (4): 990-995. |
| [2] | Liu W T, Chan O C, Fang H H P. Microbial community dynamics during start-up of acidogenic anaerobic reactors[J]. Water Research, 2002, 36 (13): 3203-3210. |
| [3] | 赵丹, 任南琪, 王爱杰, 等. 产酸相稳定发酵类型微生物生态学研究[J]. 环境科学与技术, 2003, 26 (6): 37-38, 48. |
| [4] | 张斌, 孙宝盛, 刘慧娜, 等. 处理不同废水MBR系统中微生物群落结构的比较[J]. 环境科学, 2008, 29 (10): 2944-2949. |
| [5] | 任南琪, 王爱杰. 产酸脱硫反应器中碳硫比对群落生态特征的影响[J]. 环境科学, 2002, 23 (4): 52-56. |
| [6] | 王爱杰, 任南琪, 黄志, 等. 产酸脱硫反应器中COD/SO42-比制约的群落生态演替规律[J]. 环境科学, 2002, 23 (2): 34-38. |
| [7] | 苏俊峰, 马放, 王弘宇, 等. 利用PCR-DGGE技术分析生物陶粒硝化反应器中微生物群落动态[J]. 环境科学学报, 2007, 27 (3): 386-390. |
| [8] | Ding Y, Wu W X, Han Z Y, et al. Correlation of reactor performance and bacterial community composition during the removal of trimethylamine in three-stage biofilters[J]. Biochemical Engineering Journal, 2008, 38 (2): 248-258. |
| [9] | Miura Y, Hiraiwa M N, Ito T, et al. Bacterial community structures in MBRs treating municipal wastewater: Relationship between community stability and reactor performance[J]. Water Research, 2007, 41 (3): 627-637. |
| [10] | 任随周, 郭俊, 曾国驱, 等. 处理印染废水的厌氧折流板反应器中的微生物种群组成及分布规律[J]. 生态学报, 2005, 25 (9): 2297-2303. |
| [11] | 刘广民, 任南琪, 王爱杰, 等. 产酸-硫酸盐还原系统中产酸菌的发酵类型及其与SRB的协同作用[J]. 环境科学学报, 2004, 24 (5): 782-788. |
| [12] | Colleran E, Pender S. Mesophilic and thermophilic anaerobic digestion of sulphate-containing wastewaters[J]. Water Science and Technology, 2002, 45 (10): 231-235. |
| [13] | Sanz J L, Kochling T. Molecular biology techniques used in wastewater treatment: An overview[J]. Process Biochemistry, 2007, 42 (2): 119-133. |
| [14] | Muyzer G, de Waal E C, Uitterlinden A G. Profiling of complex microbial population by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes encoding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59 (3): 695-700. |
| [15] | 刘新春, 吴成强, 张昱, 等. PCR-DGGE法用于活性污泥系统中微生物群落结构变化的解析[J]. 生态学报, 2005, 25 (4): 842-847. |
| [16] | 李建军, 廖东奇, 许玫英, 等. 生物滴滤池对BTEX的去除及相应细菌群落分析[J]. 环境科学, 2013, 34 (7): 2552-2559. |
| [17] | 欧阳科, 刘俊新. 膜生物反应器与传统活性污泥反应器内生物群落特征[J]. 环境科学, 2009, 30 (2): 499-503. |
| [18] | 罗海峰, 齐鸿雁, 薛凯, 等. PCR-DGGE技术在农田土壤微生物多样性研究中的应用[J]. 生态学报, 2003, 23 (8): 1570-1575. |
| [19] | Aburto-Medina A, Adetutu E M, Aleer S, et al. Comparison of indigenous and exogenous microbial populations during slurry phase biodegradation of long-term hydrocarbon-contaminated soil[J]. Biodegradation, 2012, 23 (6): 813-822. |
| [20] | 刘敏, 朱开玲, 李洪波, 等. 应用PCR-DGGE技术分析黄海冷水团海域的细菌群落组成[J]. 环境科学, 2008, 29 (4): 1082-1091. |
| [21] | APHA. Standard methods for the examination of water and wastewater [M]. (20th ed.). USA Washington DC: American Public Health Association, 1998. |
| [22] | Zhou J Z, Bruns M A, Tiedje J M. DNA recovery from soils of diverse composition[J]. Applied and Environmental Microbiology, 1996, 62 (2): 316-322. |
| [23] | 郑小红,肖琳,任晶,等. 玄武湖微囊藻水华暴发及衰退期细菌群落变化分析[J]. 环境科学,2008, 29 (10): 2956-2962. |
| [24] | 王爱杰, 任南琪, 刘伟, 等. 产酸脱硫反应器中SRB种群的功能与地位[J]. 中国环境科学, 2001, 21 (2): 119-123. |
| [25] | Matthies C, Kuhner C H, Acker G, et al. Clostridium uliginosum sp. nov., a novel acid-tolerant, anaerobic bacterium with connecting filaments[J]. International Journal of Systematic and Evolutionary Microbiology, 2001, 51 (3): 1119-1125. |
| [26] | Kaksonen A H, Plumb J J, Robertson W J, et al. Culturable diversity and community fatty acid profiling of sulfate-reducing fluidized-bed reactors treating acidic, metal-containing wastewater[J]. Geomicrobiology Journal, 2004, 21 (7): 469-480. |
| [27] | 任南琪, 赵阳国, 王爱杰, 等. PCR-SSCP技术分析碱度影响下硫酸盐还原反应器中微生物群落动态[J]. 中国科学(C 辑): 生命科学, 2006, 36 (1): 51-58. |
| [28] | Vladár P, Rusznyák A, Márialigeti K, et al. Diversity of sulfate-reducing bacteria inhabiting the rhizosphere of phragmites australis in Lake Velencei (Hungary) revealed by a combined cultivation-based and molecular approach[J]. Microbial Ecology, 2008, 56 (1): 64-75. |
| [29] | Mallet C, Basset M, Fonty G, et al. Microbial population dynamics in the sediments of a eutrophic lake (Aydat, France) and characterization of some heterotrophic bacterial isolates[J]. Microbial Ecology, 2004, 48 (1): 66-77. |
| [30] | Bjornsson L, Hugenholtz P, Tyson G W, et al. Filamentous Chloroflexi (green non-sulfur bacteria) are abundant in wastewater treatment processes with biological nutrient removal[J]. Microbiology, 2002, 148 (8): 2309-2318. |
| [31] | Roest K, Heilig H G H J, Smidt H, et al. Community analysis of a full-scale anaerobic bioreactor treating paper mill wastewater[J]. Systematic and Applied Microbiology, 2005, 28 (2): 175-185. |
| [32] | Cord-Ruwisch R, Lovley D R, Schink B. Growth of Geobacter sulfurreducens with acetate in syntrophic cooperation with hydrogen-oxidizing anaerobic partners[J]. Applied and Environmental Microbiology, 1998, 64 (6): 2232-2236. |
| [33] | Ramos D T, da Silva M L B, Chiaranda H S, et al. Biostimulation of anaerobic BTEX biodegradation under fermentative methanogenic conditions at source-zone groundwater contaminated with a biodiesel blend (B20) [J]. Biodegradation, 2013, 24 (3): 333-341. |
| [34] | Vandieken V, Mussmann M, Niemann H et al. Desulfuromonas svalbardensis sp. nov. and Desulfuromusa ferrireducens sp. nov., psychrophilic, Fe(Ⅲ)-reducing bacteria isolated from Arctic sediments, Svalbard[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56 (5): 1133-1139. |
| [35] | Widdel F, Hansen T A. The dissimilatory sulfate-and sulfur-reducing bacteria[A]. In: The Procaryotes [M]. (2nd ed.). New York: Springer, 1992. 583-624. |
| [36] | Oude Elferink S J W H, Vorstman W J C, Sopjes A, et al. Characterization of the sulfate-reducing and syntrophic population in granular sludge from a full-scale anaerobic reactor treating papermill wastewater [J]. FEMS Microbiology Ecology, 1998, 27 (2): 185-194. |
 2014, Vol. 35
2014, Vol. 35


