2. 中国科学院大学, 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
抗生素抗性基因(antibiotic resistance genes,ARGs)作为一种新型环境污染物,自Pruden等[1]首次将其作为环境污染物被提出以来,引起了全球范围内广泛的关注. 因抗生素在医疗和养殖业的大量使用而造成的环境污染问题日趋严重[2]. 美国每年受到抗性病原体感染的人超过两百万,最终死亡14000人[1],且越来越多的研究表明,致病菌耐药性的扩散与环境微生物抗性和抗生素抗性基因有关[3].
由于抗生素具有预防疾病和刺激生长的作用,常以亚治疗的剂量长期添加于饲料中,在全球范围内广泛应用于养殖业中[4, 5, 6]. 猪饲料中的抗生素会显著改变猪肠道中的微生物群落结构,肠道中含有抗生素抗性基因的微生物群落显著增加. 同时研究证明大部分抗生素难以被动物吸收利用,约25%~75%的抗生素未经代谢而随粪尿排出体外进入周围环境[7]. 进一步的研究表明,动物肠道内的抗性细菌排出体外后,其携带的抗生素抗性遗传信息可以通过水平基因转移(horizontal gene transfer,HGT)机制转移到外部环境的微生物中[8,9]. 一旦抗生素抗性基因从环境微生物中进入致病菌,就可能对人类健康造成潜在的巨大风险.
目前,国内外关于抗生素抗性基因在城市污泥[3]污水[10]、 猪场土壤[11]、 养殖水体[12]和河流入海口[13]等环境中的污染分布情况及抗性基因多样性有较多相关研究,但是这些研究主要针对磺胺类抗生素抗性基因和四环素类抗性基因等有限的几种抗性基因,而且关于水稻土中的抗生素抗性基因的相关研究在国内外鲜见. 高通量定量研究可以从整体角度对环境抗生素抗性基因进行全面研究,并深入地寻找挖掘富集的基因. 本研究选用长期施用猪粪和未施用猪粪水稻土作为供试土壤,采用高通量荧光定量技术对水稻土的整体的抗生素抗性基因的丰度和多样性进行全面研究,并对各富集的抗生素抗性基因深入分析,以期对水稻土中抗生素抗性污染情况有较全面的认识并为可能的潜在生态环境风险评价提供理论支持. 1 材料与方法 1.1 样品采集及处理
水稻土是2013年10月采集自秋季水稻刚刚收割后的水稻田,位于福建省连城县. 连城县是福建省九龙江、 闽江和汀江的上游地区,是福建省水稻种植和生猪养殖大县. 长期施用猪粪的水稻土采样点15 m附近有一个建于2001年生猪养殖场,生猪粪尿和养殖废水常直接排入周围水稻土壤作为肥料,此外还采集了距离该养殖场约920 m外一处地势较高未施用猪粪的水稻土作为对照土样,这两块水稻田同属于一个农户,种植方式和农田管理方式相同. 两块水稻田面积分别约为0.0006 km2 (0.9亩)和0.008 km2(1.2亩),各都采集了3个重复水稻土样品约1 kg,土壤采集深度为20 cm,并且每个采样重复由5个小样组成,并现场均匀混合. 采集的水稻土去除水稻根和可见有机物残体后,存放于-20℃冰箱内用于DNA提取. 1.2 DNA提取
称取约0.5 g水稻土,采用FastDNA Spin Kit for Soil(MP Biomedicals)试剂盒,按照生产商提供的方法提取水稻土总DNA,随后用1%的琼脂糖凝胶进行电泳验证. 所提取的DNA样品用微量核酸蛋白质分析仪(Nanodrop ND1000,NanoDrop Technologies Inc.)测定浓度以及相应的纯度,所提取的DNA样品的A260/A280值在1.8~2.0之间,表明用试剂盒提取的DNA纯度较高. 1.3 抗生素抗性基因( ARGs)高通量荧光定量
采用SmartChip Real-time PCR Systems(WaferGen公司,美国)高通量荧光定量的反应平台. 荧光定量试剂是LightCycler 480 SYBR Green I Master (Roche公司,美国). 定量体系体积100 nL,其中各试剂终浓度为:LightCycler 480 SYBR Green I Master Mix 1×,Nuclease-free PCR-Grade water,BSA 1 μg ·μL-1,DNA浓度5 ng ·μL-1,引物1 μmol ·L-1. 定量PCR反应程序:95℃预变性10 min; 95℃变性30s,60℃退火延伸30s,40个循环; 程序自动升温进行熔解曲线分析. 抗性基因引物参照文献[11],该文献使用了334对引物,根据WaferGen公司的 SmartChip Real-Time PCR System仪器设置选择了295对抗性基因引物和一对细菌通用16S rRNA基因引物.
定量PCR数据处理根据文献[11,14]进行. 根据SmartChip Real-Time System的检测限和灵敏度,确定sT值为31时作为仪器的检测阈值. 每个样品进行3次技术重复试验,3次技术重复都扩增出来时认为是阳性; 同个采样点的3个采样重复至少有两个扩增出来,认为样品的目的基因被有效检出; 当sT值超出31时,对应的sT值被替换为31. 相对定量的统计方法用公式(1)、 (2)和(3)进行计算:
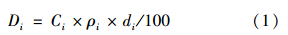
式中,ARGs是所要测定的295种抗生素抗性基因,16S是指待测样品的16S rRNA基因,sT是高通量荧光定量所测定的循环阈值,ΔsT某样品的目标基因sT值与16S rRNA基因sT值的差值,ΔΔsT是指待测(test)样品的目标基因ΔsT值与对照(control)样品的ΔsT差值,FC值是指待测(test)样品的目的基因是对照(control)样品的富集倍数. 1.4 数据分析
数据分析采用Excel 2010进行,计算FC下限值FC=2[-(ΔΔCT+s)], s是3个采样平行的功能基因sT标准方差同16S rRNA基因sT标准方差之间的平方和的根. s=(s12+s22)1/2. 式中,s1为3个采样平行的功能基因sT值的标准方差,s2为同一个采样位点的16S rRNA基因sT值的标准方差. 当FC=2[-(ΔΔCT+s)]>1,且student t检验具有显著性时认为待测样品相对于参照样品显著富集; Origin 9.0用于作图; 采用R3.0.0 (The R Foundation for Statistical Computing,Vienna,Austria) vegan 数据包(2.0~10)[15] 进行PCA分析,Pheatmap数据包 (0.7.7) 进行热图分析[16]. 2 结果与讨论 2.1 水稻土中抗生素抗性基因的多样性
施用猪粪的水稻土(PM)和未施用猪粪水稻土(PS)两种土壤中,总共检测到108种抗生素抗性基因. 其中未施用猪粪的水稻土(PS)中检测到了66种抗生素抗性基因[图 1(a)],施用猪粪水稻土(PM)则检测到了107种抗性基因[图 1(a)],两种水稻土中检测到的抗生素抗性基因主要包括抗生素失活(antibiotic deactivation)、 抗生素外排泵(efflux pump)、 细胞核糖体保护(cellular protection)和转座子移动元件(transposon)四类抗生素抗性机制[图 1(b)],这四类抗生素抗性机制在整体中比例分别为42.59%、 37.96%、 14.81%和2.78%. 水稻土中微生物对氨基糖苷类抗生素(aminoglycoside)、 β-内酰胺类抗生素(beta Lactam)、 喹诺酮类氯霉素类(FCA)、 大环内脂类林肯酰胺类链阳性菌素B(MLSB)、 四环素类抗生素(tetracycline)和万古霉素类抗生素(vancomycin)等表达出相应的抗生素抗性,分别占整体的13.89%、 23.15%、 12.96%、 10.19%、 10.19%和6.48%[图 1(c)]. 其它或者发挥外排泵机制的抗生素抗性基因则占到23.15%[图 1(c)]. 施用猪粪水稻土(PM)中检测到的抗性基因种类数目显著多于未施用猪粪水稻土(PS)中的抗性基因 (P<0.05). 检测到的抗生素抗性类型涵盖了目前已知的主要类型. 一般情况下,猪饲料中添加的抗生素主要包括氨基糖苷类的新霉素、 大观霉素,林肯酰胺类的林可霉素,大环内酯类的泰乐菌素、 北里霉素、 四环素类的土霉素、 金霉素等[17]. 但是施用猪粪的水稻土PM发现了β-内酰胺类抗生素抗性基因的cphA、 pbp等抗生素抗性基因,然而通常猪饲料中并未添加β-内酰胺类抗生素. Looft等[18]也发现未使用研究中涉及到的抗生素作为添加剂/催肥剂的情况下,生猪肠道微生物仍能发现一些新的抗生素抗性基因.
 | 图 1 水稻土中抗生素抗性基因种类Fig. 1 Variety of antibiotic resistance genes in rice paddy soil FCA:喹诺酮类氯霉素类抗性基因; MLSB:大环内脂类林肯酰胺类链阳性菌素B抗性基因 |
施用猪粪水稻土(PM)相较于未施用猪粪水稻土(PS),检测出了41种后者不具有的抗生素抗性基因,而未施用猪粪水稻土所检出的抗生素抗性基因中仅有aac(6′)-Ib(aka aacA4)基因在施用猪粪水稻土(PM)中未检出. 主成分分析表明,施用猪粪水稻土(PM)的长期猪粪施用显著改变了水稻土中抗生素抗性基因结构,PC1解释了抗性基因结构变化的89.7%,施用猪粪水稻土和对照水稻土沿第一轴分为两个簇,表明抗生素抗性基因在施用猪粪水稻土和未施用猪粪水稻土中有着不同的分布格局(图 2).
 | 图 2 水稻土中抗生素抗性基因多样性主成分分析Fig. 2 Principle component analysis of diversity of antibiotic resistance genes in rice paddy soil |
施用猪粪有机肥的水稻土(PM)相对于未施用猪粪水稻土(PS)的抗生素抗性基因的丰度和多样性均发生了明显变化,施用猪粪水稻土(PM)中抗生素抗性基因的种类和丰度显著增加,UPGMA cluster表明两类水稻土分别形成两个簇,施用猪粪水稻土中形成了新的抗生素抗性基因分布格局,表明施用猪粪有机肥的水稻土抗生素抗性基因污染加剧(图 3).
 | 图 3 水稻土中抗生素抗性基因污染分布格局Fig. 3 Antibiotic resistance genes profile of sampling sites of rice paddy soil |
施用猪粪水稻土(PM)除了检测到41种在未施用猪粪水稻土(PS)中未检出的抗生素抗性基因外,还有49种抗生素抗性基因丰度相对于未施用猪粪水稻土(PS)显著富集(P<0.05),这些显著富集的抗生素抗性基因类型涵盖了图 1(c)所描述的七大类抗生素抗性基因. 其中喹诺酮类/氯霉素类(FCA)抗性基因的mexF基因在施用猪粪水稻土(PM)中 的丰度是未施用猪粪水稻土(PS)的1792倍. β-内酰胺类抗生素(beta lactam)抗性基因中的blaSFO、 大环内脂类林肯酰胺类链阳性菌素B(MLSB)抗性基因中的oleC、 四环素类抗生素(tetracycline)抗性基因的tetD、 氨基糖苷类抗生素(aminoglycoside)抗性基因的aadE、 万古霉素类抗生素(vancomycin)抗性基因的vanHB 的丰度分别是未施用猪粪水稻土(PS)的101、 22、 12、 8、 2倍(图 4). 有研究表明,禽畜养殖场周围土壤抗生素抗性基因丰度较于正常土壤显著偏高,抗性基因污染严重[19]. Munir等[20]检测了猪粪和施用有机肥的土壤,结果表明,四环素类抗生素抗性基因丰度在猪粪中显著高于施用有机肥的土壤,但是土壤中抗生素抗性水平受到自然环境本底值和土壤性质的影响. 水稻土施用有机肥的土壤中(PM)中,检测到转座子基因tnpA和tp614的丰度分别是未施用猪粪水稻土的15.5倍和2.2倍,表明转座子类基因在施用猪粪水稻土中富集. 还有研究表明,作为饲料中抗生素添加剂的载体海泡石,也会增加抗生素抗性基因在细菌种类之间的水平基因转移[21]. Marti等[22]研究表明,施用猪粪的土壤以及土壤上种植的蔬菜都检测到了多种抗生素抗性基因,在施用新鲜猪粪的土壤中收获的蔬菜检测到一些独有的抗生素抗性基因. 一旦水稻土中的这些抗生素抗性基因进入致病菌,这种情况可能会对从事水稻种植的农民的健康造成潜在影响,并可能影响水稻这种农产品的生物食品安全. 此外,水稻土中的抗生素抗性基因可能会随着土壤微生物在农田雨水径流的冲刷下,基于水平基因转移机制进入不同的微生物物种并扩散到周围的池塘,河流水体和沉积物中,使得抗生素抗性基因在更大尺度的环境中出现和富集. 为减少抗生素抗性基因在土壤环境中传播,应当避免在农田土壤中施用含有抗生素残留的禽畜肥料[23]. 邰义萍等[24]的研究表明,长期施用粪肥菜地土壤中四环素类抗生素在土壤平面和土壤剖面造成明显的空间分布差异,由此造成的潜在的生态毒性和抗生素抗性应引起高度重视. 越来越多的研究表明,粪便是抗生素抗性和抗生素化合物的存储库(reservoir),禽畜粪便的施用造成土壤中抗生素抗性基因和抗性菌群的增加[25,26]. 因此,有必要加强对猪粪的合理使用研究,对水稻土中抗生素抗性基因污染状况做更深入的研究,重点关注抗生素抗性基因潜在的农业环境风险并作出科学评估.
 | 图 4 施用猪粪水稻土丰度增加的抗生素抗性基因富集倍数Fig. 4 Fold change of abundance of increased antibiotic genes in paddy soil amended with manure |
(1)施用猪粪改变了水稻土中抗生素抗性基因的多样性和丰度.
(2)高通量荧光定量基于多个样品和多种基因同时进行定量的高通量优势,从抗生素抗性基因集合的角度,较全面表征了水稻土中抗性基因总体分布情况,可以深入研究水稻中富集的抗性基因的污染状况.
| [1] | Pruden A, Pei R, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants: studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40 (23): 7445-7450. |
| [2] | 周启星,罗义,王美娥. 抗生素的环境残留、生态毒性及抗性基因污染[J]. 生态毒理学报, 2007, 2 (3): 243-251. |
| [3] | Zhang X X, Zhang T, Zhang M, et al. Characterization and quantification of class 1 integrons and associated gene cassettes in sewage treatment plants[J]. Applied Microbiology and Biotechnology, 2009, 82 (6): 1169-1177. |
| [4] | Chee-Sanford J C, Mackie R I, Koike S, et al. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste[J]. Journal of Environmental Quality, 2009, 38 (3): 1086-1108. |
| [5] | Pan X, Qiang Z, Ben W, et al. Residual veterinary antibiotics in swine manure from concentrated animal feeding operations in Shandong Province, China[J]. Chemosphere, 2011, 84 (5): 695-700. |
| [6] | Hvistendahl M. China takes aim at rampant antibiotic resistance[J]. Science, 2012, 336 (6083): 795-795. |
| [7] | Chee-Sanford J C, Aminov R I, Krapac I J, et al. Occurrence and diversity of tetracycline resistance genes in lagoons and groundwater underlying two swine production facilities[J]. Applied and Environmental Microbiology, 2001, 67 (4): 1494-1502. |
| [8] | El Salabi A, Walsh T R, Chouchani C. Extended spectrum β-lactamases, carbapenemases and mobile genetic elements responsible for antibiotics resistance in Gram-negative bacteria[J]. Critical Reviews in Microbiology, 2013, 39 (2): 113-122. |
| [9] | Akhtar M, Hirt H, Zurek L. Horizontal transfer of the tetracycline resistance gene tetM mediated by pCF10 among Enterococcus faecalis in the house fly (Musca domestica L.) alimentary canal[J]. Microbial Ecology, 2009, 58 (3): 509-518. |
| [10] | Pruden A. Balancing water sustainability and public health goals in the face of growing concerns about antibiotic resistance[J]. Environmental Science & Technology, 2014, 48 (1): 5-14. |
| [11] | Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110 (9): 3435-3440. |
| [12] | 高盼盼, 罗义, 周启星, 等. 水产养殖环境中抗生素抗性基因(ARGs)的研究及进展[J]. 生态毒理学报, 2009, 4 (6): 770-779. |
| [13] | 梁惜梅, 聂湘平, 施震. 珠江口典型水产养殖区抗生素抗性基因污染的初步研究[J]. 环境科学, 2013, 34 (10): 4073-4080. |
| [14] | Schmittgen T D, Livak K J. Analyzing real-time PCR data by the comparative CT method[J]. Nature Protocols, 2008, 3 (6): 1101-1108. |
| [15] | Oksanen J, Kindt R, Legendre P, et al. vegan: Community Ecology Package[EB/OL]. http://cran.r-project.org/web/packages/vegan/index.html,2013-12-12. |
| [16] | Kolde R. pheatmap: Pretty Heatmaps[EB/OL]. http://CRAN.R-project.org/package=pheatmap,2013-09-19. |
| [17] | 魏光伟, 魏文康, 余永鹏, 等. 饲料抗生素添加剂的应用技术[J]. 广东饲料, 2007, 16 (3): 15-16. |
| [18] | Looft T, Johnson T A, Allen H K, et al. In-feed antibiotic effects on the swine intestinal microbiome[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109 (5): 1691-1696. |
| [19] | 邹世春, 李青, 贺竹梅. 禽畜养殖场土壤抗生素抗性基因污染的初步研究[J]. 中山大学学报(自然科学版), 2012, 51 (6): 87-91. |
| [20] | Munir M, Xagoraraki I. Levels of antibiotic resistance genes in manure, biosolids, and fertilized soil[J]. Journal of Environmental Quality, 2011, 40 (1): 248-255. |
| [21] | Rodríguez-Beltrán J, Rodríguez-Rojas A, Yubero E, et al. The animal food supplement sepiolite promotes a direct horizontal transfer of antibiotic resistance plasmids between bacterial species[J]. Antimicrobial Agents And Chemotherapy, 2013, 57 (6): 2651-2653. |
| [22] | Marti R, Scott A, Tien Y C, et al. Impact of manure fertilization on the abundance of antibiotic-resistant bacteria and frequency of detection of antibiotic resistance genes in soil and on vegetables at harvest[J]. Applied and Environmental Microbiology, 2013, 79 (18): 5701-5709. |
| [23] | 吴楠, 乔敏. 土壤环境中四环素类抗生素残留及抗性基因污染的研究进展[J]. 生态毒理学报, 2010, 5 (5):618-627. |
| [24] | 邰义萍, 莫测辉, 李彦文, 等. 长期施用粪肥菜地土壤中四环素类抗生素的含量与分布特征[J]. 环境科学, 2011, 32 (4): 1182-1187. |
| [25] | Heuer H, Schmitt H, Smalla K. Antibiotic resistance gene spread due to manure application on agricultural fields[J]. Current Opinion in Microbiology, 2011, 14 (3): 236-243. |
| [26] | Jay-Russell M T, Madigan J E, Bengson Y, et al. Salmonella oranienburg isolated from horses, wild turkeys and an edible home garden fertilized with raw horse manure[J]. Zoonoses and Public Health, 2014, 61 (1): 64-71. |
 2014, Vol. 35
2014, Vol. 35


